文章信息
基金项目
- 广东省蔬菜新技术研究重点实验室开放课题基金(202001)
作者简介
- 赵添悦(2001—),女,在读硕士生,研究方向为食品加工与安全,E-mail:2956894331@qq.com.
通讯作者
- 高苏娟(1981—),女,博士,讲师,研究方向为食品生物技术,E-mail:gaoshj@126.com.
文章历史
- 收稿日期:2023-11-13
2. 广东省农业科学院蔬菜研究所/广东省蔬菜新技术研究重点实验室,广东 广州 510640
2. Vegetable Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory for New Technology Research on Vegetables, Guangzhou 510640, China
【研究意义】转录因子(Transcription factor,TF)是具有特殊结构域、能与基因启动子中的特定元件序列相结合从而行使调控基因表达的功能蛋白质[1]。植物基因组中存在大量转录因子,目前研究较多的转录因子有WRKY、NAC、bZIP、bHLH、MYB、HD、MADSbox、HSFs、AP2/EREBP等[2]。其中WRKY转录因子是最大的转录因子家族之一,在不同非生物胁迫(干旱、生理盐水、碱、温度、紫外线辐射等)的响应途径中发挥重要作用,其通过调控参与各种细胞过程的基因表达从而适应环境[3-5]。【前人研究进展】 WRKY基因最早从番薯(Ipomoea batatas)中鉴定,其编码一种名为SPF1(Sweet potato factor1)的蛋白质[3]。随后在野生燕麦(Avena fatua)和欧芹(Petroselinum crispum)等植物中发现WRKY转录因子的存在。目前已在多个物种中鉴定了WRKY基因家族,例如葡萄、高粱、油菜等[6-8]。前人研究指出,WRKY家族成员含有典型WRKY结构域(WRKY domain)和C2H2锌指基序(Motif zinc-finger),其中,WRKY结构域一般由60个氨基酸残基组成,含有DNA结合七肽WRKYGQK,而C2H2锌指基序位于WRKY结构域下游,具有不同的类型[9]。根据WRKY结构域的数量和锌指状基序的特征可将植物WRKY分为Ⅰ、Ⅱ和Ⅲ 3组,而第Ⅱ组中的WRKY根据其系统发育关系又可分为Ⅱa、Ⅱb、Ⅱc、Ⅱd和Ⅱe 5个亚组[9]。其中,Ⅰ组WRKY蛋白包含两个WRKY结构域,而Ⅱ组和Ⅲ组WRKY蛋白只包含1个WRKY结构域,且Ⅱ组的锌指基序与Ⅰ组相同,均为C-X4-5-C-X22-23-H-X1-H,而Ⅲ组的锌指基序为C-X7-C-X23-HX1-C[10]。功能研究表明,WRKY基因参与信号传导、胁迫响应、免疫反应等过程。例如将葡萄VvWRKY28转入拟南芥后,大大提高了拟南芥对低温和高盐的耐受性[6]。在高粱中,SbWRKY30通过与干旱胁迫响应基因SbRD19启动子的W-box/TTGAC(C/T)元件结合,促进SbRD19表达;并通过提高活性氧清除能力,保护植物细胞免受活性氧的伤害,增强其抗旱性[7]。油菜BcWRKY46基因在低温和ABA处理下被强烈诱导,从而激活ABA信号通路中的相关基因,提高植物的低温耐受性[8]。
【本研究切入点】综上,WRKY家族基因在许多植物的非生物胁迫响应过程中均起到重要作用。然而胁迫状态下冬瓜中WRKY家族基因表达情况研究存在空白。【拟解决的关键问题】本研究将基于冬瓜的基因组和转录组数据,通过基因鉴定、理化性质、系统进化、基因结构、保守基序、启动子顺式作用元件、qPCR等分析,探索WRKY基因在冬瓜胁迫响应过程中可能参与的潜在机制。
1 材料与方法 1.1 试验材料本研究所用材料为冬瓜高代自交系B227,由广东省农业科学院蔬菜研究所。
1.2 试验方法1.2.1 冬瓜WRKY家族成员的鉴定与染色体位置 分析根据已发表的拟南芥和水稻WRKY基因家族的相关研究,结合拟南芥WRKY基因,利用葫芦科基因组学数据库(CuGenDB)blastp功能,在冬瓜基因组中进行比对,定义阈值为e-5,获得候选冬瓜WRKY同源基因[11];使用HMMER软件(3.3版)的hmmsearch功能在冬瓜基因组中鉴定出候选WRKY基因[10];借助hmmbuild功能建立冬瓜特异性WRKY蛋白的HMM模型,E值阈值设置为e-5。利用新生成的HMM文件,再次从冬瓜基因组中鉴定出WRKY基因家族的成员[12]。采用美国国家生物技术信息中心(NCBI)的Batch CD-Search工具查找候选的WRKY蛋白保守结构域进行筛选[13]。整理冬瓜WRKY家族基因在染色体上的位置信息,并按其在染色体上的位置顺序进行重命名,利用TBtools软件绘制冬瓜WRKY基因的染色体位置图[14]。
1.2.2 冬瓜WRKY成员的系统发育分析与理化性质分析 通过MEGA 11.0软件构建冬瓜WRKY成员的系统发育树,将冬瓜、拟南芥蛋白进行多序列比对,方法为邻接法(Neighbor-joining, NJ),Bootstrap值设置为1 000次重复[15]。得到的进化树文件通过ITOL在线网站〔iTOL: Interactive Tree Of Life (embl.de)〕进行美化。利用ExPASy[16]网站(https://web.expasy.org/compute_pi/)和WoLF PSORT(https://wolfpsort.hgc.jp/)在线工具分析冬瓜WRKY蛋白质理化性质,获得碱基数、相对分子质量、不稳定性和等电点等信息。
1.2.3 冬瓜WRKY基因结构及保守基序分析 利用在线网站MEME(https://meme-suite.org/meme/tools/meme)[17]分析冬瓜WRKY蛋白序列的保守基序。利用葫芦科基因组数据库网站下载冬瓜WRKY蛋白对应的基因序列和cDNA序列,利用TBtools分析其内含子及外显子并进行可视化[11]。
1.2.4 冬瓜WRKY家族成员基因启动子顺式作用元件分析 利用TBtools软件提取冬瓜WRKY基因中起始密码子上游的2 000 bp启动子区域;使用在线工具PlantCARE预测基因启动子的顺式调控元件。挑选与激素和胁迫响应有关的顺式作用元件,剔除最为常见的启动子如ATAT-box、CAAT-box等,利用TBtools软件进行聚类分析[18]。
1.2.5 冬瓜WRKY基因组织表达分析 为研究冬瓜WRKY基因的组织表达特异性和不同发育时期表达特异性,使用先前发表的包含B227冬瓜花、果实、叶、根和茎等不同器官组织的RNA-seq数据作为数据源[19]。利用WRKY基因在不同组织中和不同发育时期的平均FPKM(Fragments per kilobase of transcript per million mapped reads)值进行表达谱分析。利用TBtools软件中的Heatmap绘制不同组织表达和不同发育时期表达热图,然后分别使用(FPKM+1)将FPKM值进行归一化处理[20]。
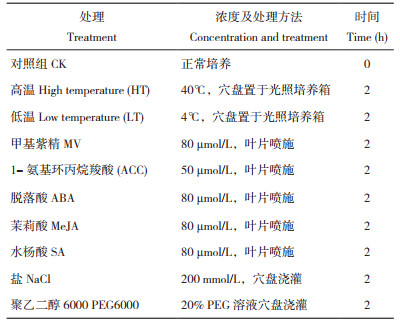
1.2.6 冬瓜植物材料处理 2022年4月,取饱满一致的B227冬瓜种子剥壳放入55 ℃水中浸泡8 h,于28~30 ℃恒温箱中催芽48 h,露白后播种在装有专用育苗基质的穴盘,放置在人工气候室(26 ℃,16/8 h的光/暗周期)培养。待长至两叶一心时将穴盘置于高温(40 ℃)、低温(4 ℃)、甲基紫精(MV)、1-氨基环丙烷羧酸(ACC)、脱落酸(ABA)、茉莉酸(JA)、水杨酸(SA)、NaCl、聚乙二醇6000(PEG6000)下进行处理,具体处理方式如表 1所示。2 d后每棵幼苗取第3片叶置于液氮中速冻,然后于-80 ℃保存。每种处理各设置3个生物学重复。
1.2.7 叶片RNA提取及荧光定量 利用北京全式金生物技术有限公司的高纯度RNA提取试剂盒提取冬瓜叶片的RNA。利用天根生化科技有限公司的FastKing gDNA Dispelling RT SuperMix试剂盒将提取的冬瓜总RNA反转录为cDNA。使用SuperReal PreMix Plus SYBR Green(天根试剂盒),在QuantStudio 5 Real-Time PCR仪(Thermo Scientific,Massachusetts,USA)上进行荧光定量分析,内参基因为BhUBQ。PCR结束后运用2-ΔΔCt方法计算基因的相对表达量[21]。
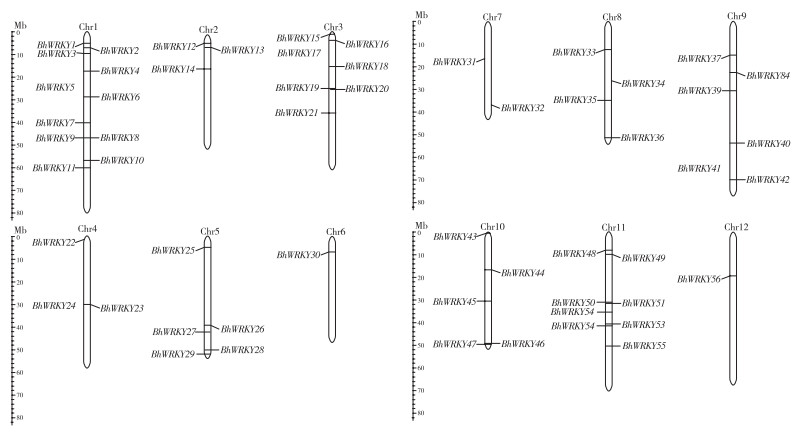
2 结果与分析 2.1 冬瓜WRKY基因家族成员的鉴定与理化性质分析通过生物信息学分析,去除冗余序列后共从冬瓜基因组鉴定到57个WRKY成员。根据冬瓜WRKY在染色体上的位置分别将其命名为BhWRKY1~BhWRKY57(图 1)。冬瓜WRKY家族基因集中分布在1号、11号染色体上,2号和4号染色体上有3个基因,7号染色体上有2个基因,6号和12号染色体上基因数最少,仅有1个基因;BhWRKY57基因被锚定在未组装的Contig上。此外,在1号染色体上鉴定出了1个WRKY串联重复簇,分别对应于2个基因(BhWRKY8和BhWRKY9)。这一现象表明,冬瓜WRKY基因在1号染色体上可能经历一次重复复制。
|
| 冬瓜WRKY基因是根据在染色体上的位置进行的重命名;Mb表示染色体的长度;染色体两侧是冬瓜WRKY基因在相应染色体上的物理位置 Wax gourd WRKY genes is renamed according to its position on the chromosome; Mb indicates the length of the chromosome; Both sides are the physical locations of the wax gourd WRKY genes on the corresponding chromosome 图 1 冬瓜WRKY基因染色体定位 Fig. 1 Mapping of wax gourd WRKY genes on chromosome |
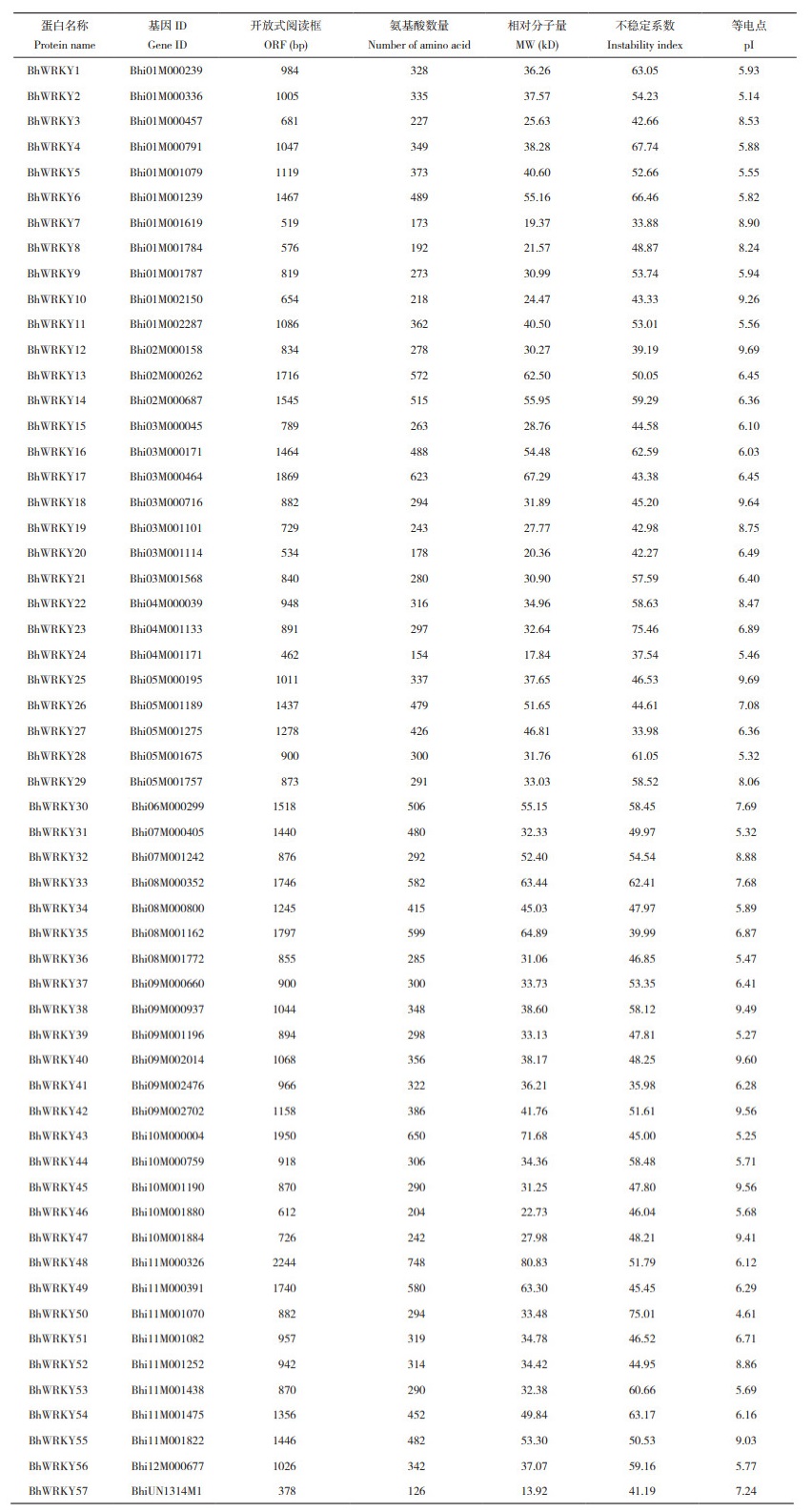
对冬瓜WRKY蛋白理化性质分析发现(表 3),BhWRKY蛋白氨基酸数目在126(BhWRKY43)~650 (BhWRKY57)之间,相对分子质量为13.92(BhWRKY43)~71.68(BhWRKY57)kD;蛋白等电点为4.61(BhWRKY50)~9.69(BhWRKY25),且BhWRKY3、BhWRKY7、BhWRKY8、BhWRKY10、BhWRKY12、BhWRKY18、BhWRKY19、BhWRKY22、BhWRKY25、BhWRKY26、BhWRKY29、BhWRKY30、BhWRKY32、BhWRKY33、BhWRKY38、BhWRKY40、BhWRKY42、BhWRKY45、BhWRKY47、BhWRKY52、BhWRKY55、BhWRKY57为碱性蛋白;不稳定系数为33.88(BhWRKY7)~75.46(BhWRKY23),其中只有BhWRKY7、BhWRKY24、BhWRKY27、BhWRKY35、BhWRKY41的不稳定系数低于40,蛋白质较稳定。
2.2 冬瓜WRKY家族的系统发育分析
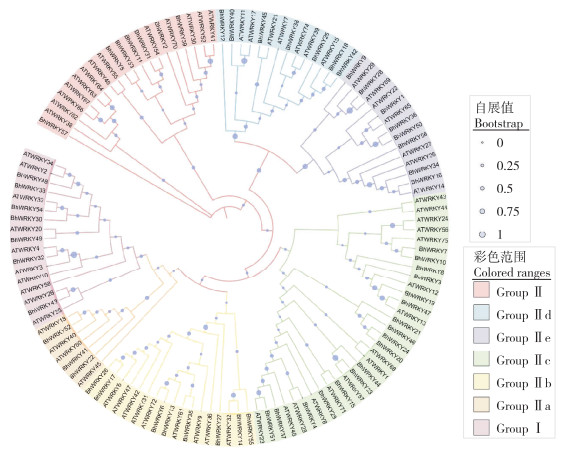
为了解冬瓜WRKY家族间亲缘关系与生物学功能,根据拟南芥WRKY家族转录因子的特征及分类方法,利用MEGA 11软件构建系统发育树,将57个BhWRKY蛋白分为Ⅰ组、Ⅱ组(Ⅱa、Ⅱb、Ⅱc、Ⅱd和Ⅱe)和Ⅲ组(图 2)。其中Ⅰ组、Ⅱd、Ⅲ组中都含有7个冬瓜WRKY成员。Ⅱa亚组中存在3个冬瓜WRKY成员;Ⅱb亚组和Ⅱe中的冬瓜成员较多,均为8个;Ⅱc亚组中冬瓜WRKY成员最多(17个)。各个组(或亚组)之间均与拟南芥WRKY家族成员有对应关系,暗示二者可能具有相似的生物学功能。
|
| 拟南芥(AT),冬瓜(Bh);类群(Ⅰ、Ⅱ、Ⅲ)和亚类群(Ⅱa、b、c、d、e)用不同的颜色进行区分;图中紫色的点代表同源的百分比 Arabidopsis thaliana (AT), Benincasa hispida (Bh); Groups (Ⅰ, Ⅱ and Ⅲ) and subgroups (Ⅱa, b, c, d and e) are distinguished in different colors; Purple dots in the figure represent the percentage of homologies 图 2 冬瓜WRKY成员的系统发育树 Fig. 2 Phylogenetic tree of WRKY members in wax gourd |
2.4 冬瓜WRKY基因结构和保守基序分析
基因结构是驱动多基因家族的进化重要原因之一,为探究冬瓜WRKY的进化关系,本研究对已鉴定出的57个BhWRKY基因的外显子-内含子结构进行分析。结果(图3)发现,Ⅱ c组含有2~4个外显子;Ⅱ a组含有3~5个外显子;Ⅱ b组含有4~6个外显子;Ⅲ组含有2~3个外显子;Ⅱ d组含有3个外显子;Ⅱ e组含有3个外显子;Ⅰ组除BhWRKY57含有1个外显子外,其他均含有4~5个,表明处于同一组的WRKY基因具有相似的基因结构。

|
使用MEME在线程序分析冬瓜WRKY蛋白保守基序,以探讨其基序组成的相似性和多样性。结果共检测到10个Motif(保守基序),其中,Motif 1和Motif 3包含1个WRKYGQK序列(图4),这是冬瓜WRKY的1个基本特征,Motif 1几乎在所有组中,Motif 3只存在于Ⅰ组中。且如图4所示,同一组或亚组内的冬瓜WRKY具有相似的Motif组成。
2.5 冬瓜WRKY基因的顺式作用元件分析启动子中的顺式作用元件对基因表达至关重要,是研究基因功能的重要组成部分。本研究利用PlantCARE在线软件检索冬瓜WRKY基因上游2 000 bp序列的顺式作用元件。为进一步研究WRKY转录因子参与应激反应的潜在调控机制,本研究选择了具有代表性的顺式元件进行定量分析。同时,根据其功能,将其分为环境应激、外源激素反应两类。
数量统计结果(图 5)表明,除(BhWRKY3、BhWRKY8、BhWRKY25、BhWRKY33、BhWRKY36、BhWRKY48、BhWRKY50)7个基因外,冬瓜其余WRKY成员均含有与外源激素处理相关的顺式作用元件,预测它们参与相应激素信号转导过程。在38个BhWRKY基因的启动子区域中发现了MeJA响应元件-CGTCA-motif,37个基因含有响应水杨酸的元件TCA,表明这些基因可能参与植物的非生物胁迫,这也与其他物种报道大量WRKY成员参与植物病虫害抗性的结果相符。此外,46个基因含有响应生长素元件TGA−element,7个基因含有响应赤霉素的元件GARE−motif。BhWRKY启动子上非生物胁迫相关的顺式作用元件统计显示,有40个BhWRKY含有W-box(TTGACC),发现(BhWRKY12、BhWRKY14、BhWRKY34、BhWRKY46、BhWRKY51)5个基因包含低温和脱水反应元件C-repeat/DRE,它们可能对低温和干旱胁迫产生反应(图 5)。在20个基因中发现参与干旱响应的特殊MYB结合位点MBS,表明部分冬瓜WRKY可能参与了干旱胁迫响应。多种顺式作用元件的存在说明这些基因可能受到复杂的调控,在冬瓜遭遇胁迫时协调发挥作用。
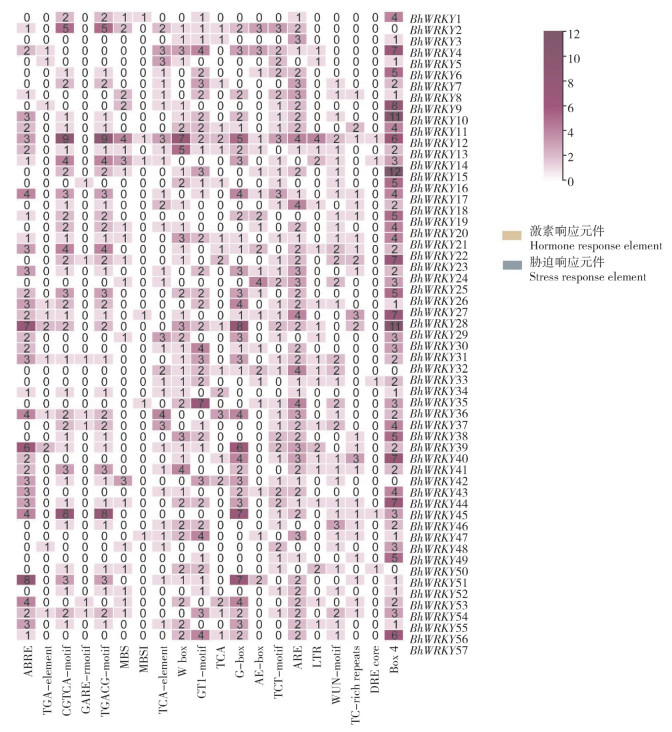
|
| 热图中不同颜色和数字表示不同顺式调节元件的数目 Different colors and numbers on the heat map indicate the number of different cis-regulating elements 图 5 冬瓜WRKY启动子区域的顺式调控元件分析 Fig. 5 Analysis of cis-acting elements in WRKY promoters of wax gourd |
2.6 冬瓜WRKY基因在不同组织与不同发育时期的表达分析
将冬瓜花、茎、叶、果实、根中WRKY基因的转录组数据分层聚类[22]。数据(图 6A)表明,WRKY基因在冬瓜各组织中有不同程度的表达。其中BhWRKY2、BhWRKY27、BhWRKY33、BhWRKY39、BhWRKY42、BhWRKY52、BhWRKY53在各组织中均有较高的表达量;BhWRKY8、BhWRKY19、BhWRKY37、BhWRKY10、BhWRKY56、BhWRKY57在各组织中的表达量较低;BhWRKY56、BhWRKY57在各组织的表达量均极低;有11个基因在根部的表达量高于其他组织;说明BhWRKY基因表达具有组织特异性,不同基因可能参与不同组织的生长发育调控。
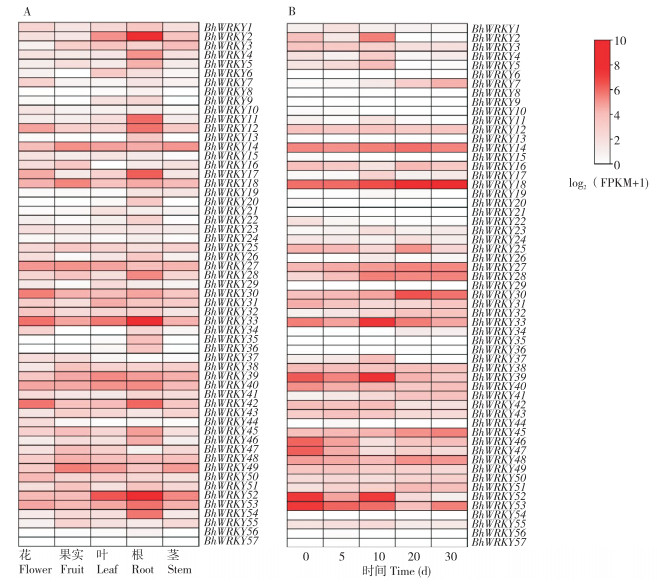
|
| A:冬瓜WRKY基因在花、果实、叶、根、茎中的表达量;B:冬瓜WRKY在授粉后0、5、10、20、30 d果实中的表达量;表达量值取对数,数值越大颜色越深,反之越浅 A: Expression levels of wax gourd WRKY in flowers, fruits, leaves, roots and stems; B: Expression levels of wax gourd WRKY in fruits 0, 5, 10, 20 and 30 days after pollination; The larger the value, the darker the color, conversely, the color is lighter 图 6 冬瓜WRKY基因在不同器官、不同发育阶段的表达谱 Fig. 6 Expression profiles of wax gourd WRKY genes in different tissues and development stages of fruits |
作为冬瓜的可食部分,果实发育是育种家关注的重点之一。利用授粉后不同天数冬瓜果实的转录组数据,将BhWRKY基因的表达数据分层聚类。由图 6B可知,大部分BhWRKY随着授粉时间呈现先升高后下降的趋势,并在授粉后10 d达到峰值。其中,BhWRKY12、BhWRKY14、BhWRKY18、BhWRKY27、BhWRKY38、BhWRKY40、BhWRKY49、BhWRKY55在授粉后的0~30 d内表达均较为稳定,BhWRKY2、BhWRKY3、BhWRKY39、BhWRKY46、BhWRKY47、BhWRKY52、BhWRKY53在授粉后30 d内表达逐渐下调;BhWRKY2、BhWRKY5、BhWRKY33、BhWRKY39、BhWRKY52、BhWRKY53在授粉后10 d时表达量最高。
2.7 冬瓜WRKY亚家族基因在不同处理表达分析由于WRKY在不同胁迫下起着重要的功能且冬瓜WRKY家族含有多种顺式作用元件,为进一步验证冬瓜WRKY在胁迫下可能发挥的生物学功能,本研究从57个冬瓜WRKY基因中选择了6个在不同器官中表达水平相对较高的WRKY成员,包括(BhWRKY11、BhWRKY5、BhWRKY53、BhWRKY2、BhWRKY39、BhWRKY31)在盐渍(NaCl)、高温(HT)、干旱(PEG)、低温(LT)、氧化胁迫(MV)及外源激素处理条件下,通过RT-PCR检测这些基因的表达模式,分析不同处理对冬瓜WRKY基因的表达影响。
结果(图 7)显示,冬瓜WRKY基因的表达至少对一种处理敏感。与对照组相比较,BhWRKY2表达在LT和MV处理下显著升高,其中尤以LT明显,表达量提高约80倍;与BhWRKY2表达相似,LT和MV亦能显著诱导BhWRKY5表达,在MV胁迫下该基因表达量提高约55倍;除LT和MV外,脱落酸(ABA)和水杨酸(SA)能显著诱导BhWRKY11的表达,而PEG、茉莉酸甲酯(JA)和NaCl处理则不影响其表达;BhWRKY31受SA诱导表达,而受PEG和JA抑制表达;BhWRKY39基因在LT下表达量提高约45倍,而在MV处理下表达量提高近10倍;BhWRKY53的表达水平在所有处理下的变化幅度较小,在SA和NaCl处理下分别提高约6.5倍和4.5倍。除BhWRKY2和BhWRKY53外,乙烯前体ACC处理能显著诱导其他BhWRKY基因的表达,而仅有BhWRKY31和BhWRKY53受高HT胁迫诱导。此外,BhWRKY31和BhWRKY53也同时响应JA处理,前者表达受JA抑制而后者受到诱导。
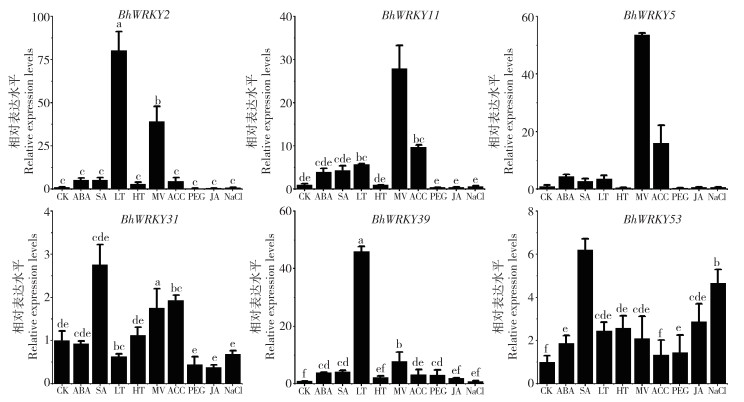
|
| 柱上小写英文字母不同者表示差异显著 Different lowercase letters above the columns represent significant differences 图 7 不同胁迫下部分冬瓜WRKY基因的表达分析 Fig. 7 Expression analysis of some WRKY genes in wax gourd under different stresses |
3 讨论
冬瓜在南菜北运及蔬菜市场周年供应中发挥重要作用。然而华南地区的高温高湿气候特点使得冬瓜生产面临病害发病率高、高温及干旱胁迫等不利条件。因此,从植物本身寻找抗性资源进行抗性育种是解决上述问题的关键手段之一。然而目前冬瓜的抗性资源及机理研究仍处于起步阶段,其核心基因组信息的解析可为深入研究相关机理提供条件。
WRKY转录因子是植物中调控网络成员之一,主要通过调控下游基因的表达参与植物的生长过程及响应不同胁迫,包括衰老、干旱、盐渍、低温、高温、激素处理等[23]。目前,拟南芥、水稻、蒲公英、茄子、马铃薯、辣椒、木薯和花生等植物基因组的WRKY基因均已被鉴定[24-29]。模式植物拟南芥中有74个WRKY基因;单子叶植物水稻中有100个WRKY基因[30]。黄瓜植物基因组中含有61个WRKY基因[31]。
本研究在冬瓜基因组中鉴定到57个WRKY基因,与拟南芥中WRKY的数目相近。然而,相较于黄瓜基因组,较大基因组的冬瓜(冬瓜、黄瓜基因组分别为950 Mb和360 Mb左右)具有较少的WRKY基因,这可能是由于相较于黄瓜,冬瓜基因组更为原始,其包含大量非编码序列。目前关于冬瓜WRKY基因还未有详细描述,本研究将预测到的冬瓜WRKY基因家族详细信息,包括基因名称、氨基酸长度、分子量、等电点与不稳定系数详细列于表 3。除BhWRKY57外的BhWRKY基因不均匀地分布于冬瓜12条染色体上,其中1号染色体上最多,有11个基因。BhWRKY57位于contig1314上,该片段由于测序原因并未定位到冬瓜12条染色体上。虽然BhWRKY57具有典型的WRKY结构域,但保守基序分析发现该基因与其他基因相比明显不同,BhWRKY57仅有1个Motif 2,因此BhWRKY57可能为假基因,这需要后续深入研究加以解析。同时本研究鉴定出1个WRKY串联重复簇,分别对应于2个基因(BhWRKY8/BhWRKY9)。类似现象也出现在其他物种中,例如StWRKY基因有15个重复:8个串联簇和7个片段[23];CaWRKY基因有11个串联簇[27];AdWRKY基因有4个串联簇和7个片段重复,AiWRKY基因有6个串联簇和10个片段重复。对拟南芥的研究表明,重复可能会影响基因家族的大小和分布,串联重复影响扩展,片段重复影响进化和功能预测[32]。
为了研究冬瓜WRKY蛋白与拟南芥已知WRKY之间的进化关系,本研究对冬瓜和拟南芥WRKY结构域的预测氨基酸序列进行多重比对,建立系统发育树。在拟南芥中,基于WRKY基因家族的基因组数据,建立了一个被广泛接受的WRKY基因分类系统[33]。根据结构域的数量和锌指基序的特征这一经典分类,将植物中的WRKY基因分为3组(Ⅰ、Ⅱ和Ⅲ)。另有报道指出,WRKY基因家族应纯粹根据系统发育数据分为Ⅰ、Ⅱa、Ⅱb、Ⅱc、Ⅱd、Ⅱe和Ⅲ组[34]。本研究中,57个WRKY蛋白被分为3个主要组群,第二组细分为5个亚组群。这与拟南芥、黄瓜、大豆、玉米中WRKY家族的分类相一致[9, 31, 35-36]。
在适应外界环境变化过程中,植物体内也发生信号转导,激发防御体系,从而提高自身抵抗胁迫能力,在此过程中各种转录因子与目标基因启动子上的顺式作用元件结合发挥了重要作用[37]。基因启动子中不同顺式作用元件的类型和数量与基因的表达差异有关,其中WRKY转录因子结合的核心基序W-box广泛存在于包括WRKY自身在内的基因的启动子中。如大豆中GmWRKY142能够结合ATCDT1,GmCDT1-1和GmCDT1-2启动子中的W-box元件[38];拟南芥中与防御应答相关的AtWRKY8和与衰老相关的AtWRKY22、AtWRKY28和AtWRKY70都可以结合W-box基序[39]。顺式作用元件分析显示冬瓜WRKY基因中也含有W-box基序,推测其可能也会与冬瓜WRKY基因结合从而调控自身及下游基因的表达。冬瓜WRKY基因启动子区也存在ABA、SA等响应激素顺式作用元件,多数WRKY基因启动子存在与低温及逆境响应元件。这些不同的BhWRKY所具有的不同顺式作用元件暗示冬瓜WRKY受不同环境信号调控,加之其本身构成的调控网络形成一个更加复杂的调控体系参与植物生长和胁迫反应的转录调控[31]。
基因表达结果显示,大部分BhWRKY在根中表达量较高,意味着BhWRKY可能通过根参与冬瓜对环境的响应。同时,多数BhWRKY在授粉后10 d达到最大值,以上结果表明BhWRKY可能也参与果实发育,尤其可能参与调节果实快速膨大过程。
BhWRKY39和BhWRKY2基因与拟南芥AtWRKY41基因同源性较高,拟南芥AtWRKY41可以通过直接调节ABI3的转录水平来控制拟南芥种子的休眠[40]。而BhWRKY39和BhWRKY2受低温诱导,暗示二者可能还具有参与植物低温胁迫响应;BhWRKY31和BhWRKY53基因与拟南芥的AtWRKY55、AtWRKY54基因同源性较高,AtWRKY55转录因子通过调节与拟南芥ROS和SA生物合成有关的基因的转录,正向调节叶片衰老和防御反应[37],而AtWRKY54则负调控SA生物合成[41-42]。BhWRKY31和BhWRKY53基因在SA处理下表达量较高,它们可能与AtWRKY54和AtWRKY55有类似的功能,可以参与SA生物合成过程以及调节植物的衰老。BhWRKY53基因与拟南芥AtWRKY53也有较高的同源性,AtWRKY53和CALMODULIN1(钙调蛋白1)通过负调控拟南芥JA生物合成途径在植物防御中发挥作用[43]。BhWRKY53基因受JA的诱导,说明BhWRKY53可能也具有与AtWRKY53相似的功能,通过参与JA生物合成途径从而参与植物防御;BhWRKY11和BhWRKY5基因受MV诱导,其与拟南芥AtWRKY70基因同源,AtWRKY70在拟南芥抗坏血酸过氧化物酶Apx1敲除(KO-APX1)植株受到光胁迫及氧化应激时表达上调[44]。同时AtWRKY70和AtWRKY54作为叶片衰老的负调控因子协同调控拟南芥叶片发育[45]。因此,推测BhWRKY11和BhWRKY5基因可能也参与了氧化胁迫与叶片衰老过程,然而这些基因的具体生物学功能仍需进一步研究。
4 结论本研究鉴定了57个冬瓜WRKY基因,并对其理化性质、系统进化、基因结构、蛋白保守基序、顺式作用元件等进行分析。通过RT-PCR分析,发现BhWRKY2、BhWRKY5、BhWRKY39参与了低温和甲基紫精胁迫过程中防御与生长的动态平衡。BhWRKY11、BhWRKY31、BhWRKY53参与了水杨酸胁迫响应。BhWRKY11也参与了甲基紫精胁迫响应,BhWRKY53也参与了盐渍胁迫响应。这些结果为解析胁迫响应机制提供了理论依据,可以进一步研究BhWRKY2、BhWRKY5、BhWRKY39、BhWRKY11、BhWRKY31、BhWRKY53的分子调控机制,有助于提高冬瓜的品质与产量。
| [1] |
SONG L, LI W T, CHEN X W. Transcription factor is not just a transcription factor[J]. Trends in Plant Science, 2022, 27(11): 1087-1089. DOI:10.1016/j.tplants.2022.08.001 |
| [2] |
KHAN S A, LI M Z, WANG S M, YIN H J. Revisiting the role of plant transcription factors in the battle against abiotic stress[J]. International Journal of Molecular Sciences, 2018, 19(6): 1634. DOI:10.3390/ijms19061634 |
| [3] |
CHEN F, HU Y, VANNOZZI A, WU K C, CAI H Y, QIN Y, MULLIS A, LIN Z G, ZHANG L S. The WRKY transcription factor family in model plants and crops[J]. Critical Reviews in Plant Sciences, 2018, 36(5-6): 311-335. DOI:10.1080/07352689.2018.1441103 |
| [4] |
张秀春, 吴坤鑫, 赵平娟, 刘志昕. 人工合成靶序列快速构建多靶标RNAi表达载体[J]. 广东农业科学, 2016, 43(7): 81-86. DOI:10.16768/j.issn.1004-874X.2016.07.013 ZHANG X C, WU K X, ZHAO P J, LIU Z X. Rapid construction of multiple target RNAi expression vector from synthesized fragment[J]. Guangdong Agricultural Sciences, 2016, 43(7): 81-86. DOI:10.16768/j.issn.1004-874X.2016.07.013 |
| [5] |
YAHYAOUI F E, KÜSTER H, AMOR B B, HOHNJEC N, PÜHLER A, BECKER A, GOUZY J, VERNIÉ T, GOUGH C, NIEBEL A, GODIARD L, GAMAS P. Expression profiling in Medicago truncatula identifies more than 750 genes differentially expressed during nodulation, including many potential regulators of the symbiotic program[J]. Plant Physiology, 2004, 136(2): 3159-3176. DOI:10.1104/pp.104.043612 |
| [6] |
LIU W, LIANG X Q, CAI W J, WANG H, LIU X, CHENG L F, SONG P H, LUO G J, HAN D G. Isolation and functional analysis of VvWRKY28, a Vitis vinifera WRKY transcription factor gene with functions in tolerance to cold and salt stress in transgenic Arabidopsis thaliana[J]. International Journal of Molecular Sciences, 2022, 23(21): 13418. DOI:10.3390/ijms232113418 |
| [7] |
YANG Z, CHI X Y, GUO F F, JIN X Y, LUO H L, HAWAR A, CHEN Y X, FENG K K, WANG B, QI J L, YANG Y H, SUN B. SbWRKY30 enhances the drought tolerance of plants and regulates a drought stress-responsive gene SbRD19 in sorghum[J]. Journal of Plant Physiology, 2020, 246-247: 153142. DOI:10.1016/j.jplph.2020.153142 |
| [8] |
WANG J Y, WANG L J, YAN Y, ZHANG S X, LI H, GAO Z, WANG C, GUO X Q. GhWRKY21 regulates ABA-mediated drought tolerance by fine-tuning the expression of GhHAB in cotton[J]. Plant Cell, 2021, 40(11): 2135-2150. DOI:10.1007/s00299-020-02590-4 |
| [9] |
EULGEM T, RUSHTON P J, ROBATZEK S, SOMSSICH I E. The WRKY superfamily of plant transcription factors[J]. Trends in Plant Science, 2000, 5(5): 199-206. DOI:10.1016/s1360-1385(00)01600-9 |
| [10] |
TIAN Y, LU X Y, PENG L S, FANG J. The structure and function of plant WRKY transcription factors[J]. Hereditas, 2000, 28(12): 1607-1612. DOI:10.1360/yc-006-1607 |
| [11] |
李鑫, 陈筱瑶, 尹丽娟, 侯尹婕, 马含月, 陈秀红, 于蓉, 岳贞, 张显, 魏春华. 甜瓜SWEET基因家族鉴定及表达分析[J/OL]. 分子植物育种, 1-12[2023-11-13]. http://kns.cnki.net/kcms/detail/46.1068.S.20230426.1122.006.html. LI X, CHEN X Y, YIN L J, HOU Y J, MA H Y, CHEN X H, YU R, YUE Z, ZHANG X, WEI C H. Identification and expression analysis of SWEET gene family in melon[J/OL]. Molecular Plant Breeding, 1-12[2023-11-13]. http://kns.cnki.net/kcms/detail/46.1068.S.20230426.1122.006.html. |
| [12] |
LOZANO R, HAMBLIN M T, PROCHNIK S, JANNINK J L. Identification and distribution of the NBS-LRR gene family in the Cassava genome[J]. BMC Genomics, 2015, 16(1): 360. |
| [13] |
BLU M M, CH A NG H Y, CH UGU R A NSK Y S, GR EG O T, KANDASAAMY S, MITCHELL S, NUKA G, PAYSAN-LAFOSSE T P, QURESHI M, RAJ S, RICHARDSON L, SALAZAR G, WILLIAMS L, BORK P, BRIDGE A, GOUGH J, HAFT D H, LETUNIC I, MARCHLER-BAUER A M, MI H, NATALE D A, NECCI M, ORENGO C A, PANDURANGAN A P, RIVOIRE C, SIGRIST C J, SILLITOE I, THANKI N, THOMAS P, TOSATTO S C, WU C H, BATEMAN A, FINN R D. The InterPro protein families and domains database: 20 years on[J]. Nucleic Acids Research, 2021, 49: 344-354. DOI:10.1093/nar/gkaa977 |
| [14] |
谢炳春, 黄俊霖, 温松森, 李涛, 李颖, 徐小万, 徐晓美, 吴智明, 衡周. 中国辣椒BCAT基因家族鉴定、表达分析及克隆[J]. 广东农业科学, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 XIE B C, HUANG J L, WEN S S, LI T, LI Y, XU X W, XU X M, WU Z M, HENG Z. Identification, expression analysis and cloning of capsicum chinense BCAT gene family[J]. Guangdong Agricultural Sciences, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 |
| [15] |
LI Y Q, AN S, CHENG Q Q, ZONG Y, CHEN W R, GUO W D, ZHANG L. Analysis of evolution, expression and genetic transformation of TCP transcription factors in blueberry reveal that VcTCP18 negatively regulates the release of flower bud dormancy[J]. Frontiers in Plant Science, 2021, 12: 697609. DOI:10.3389/fpls.2021.697609 |
| [16] |
赵建荣, 杨圆, 秦改花, 刘春燕, 于晴, 贾波涛, 苏颖, 曹榛, 黎积誉. 石榴HAK/KUP/KT家族基因鉴定及钾转运功能分析[J]. 园艺学报, 2022, 49(4): 758-768. |
| [17] |
李雨欣, 王鹏, 刘雯, 周扬. 海马齿NHX基因家族的鉴定及表达分析[J]. 广东农业科学, 2023, 50(10): 85-96. DOI:10.16768/j.issn.1004-874X.2023.10.010 LI Y X, WANG P, LIU W, ZHOU Y. Identification and expression analysis of NHX gene family of Sesuvium portulacastrum[J]. Guangdong Agricultural Sciences, 2023, 50(10): 85-96. DOI:10.16768/j.issn.1004-874X.2023.10.010 |
| [18] |
SZKLARCZYK D, GABLE A L, NASTOU K C, LYON D, KIRSCH R, PYYSALO S, DONCHEVA N T, LEGEAY M, FANG T, BORK P, JENSEN L, MERING C V. The STRING database in customizable protein-protein networks and functional characterization of user-uploaded gene measurement sets[J]. Nucleic Acids Research, 2021, 49(D1): 605-612. DOI:10.1093/nar/gkaa1074 |
| [19] |
XIE D S, XU Y C, WANG J P, LIU W R, ZHOU Q, LUO S B, HUANG W, HE X M, LI Q, PENG Q W, YANG X Y, YUAN J Q, YU J G, WANG X Y, LUCAS W J, HUANG S W, JIANG B, ZHANG Z H. The wax gourd genomes offer insights into the genetic diversity and ancestral cucurbit karyotype[J]. Nature Communications, 2019, 10(1): 5158. DOI:10.1038/s41467-019-13185-3 |
| [20] |
周荣, 刘嘉超, 杨凤玺. 墨兰CsAP1-A基因克隆及表达分析[J]. 广东农业科学, 2023, 50(9): 99-107. DOI:10.16768/j.issn.1004-874X.2023.09.010 ZHOU R, LIU J C, YANG F X. Analysis on cloning and expression of CsAP1-A gene in Cymbidium sinense[J]. Guangdong Agricultural Sciences, 2023, 50(9): 99-107. DOI:10.16768/j.issn.1004-874X.2023.09.010 |
| [21] |
CHENG Z K, LIU Z G, MA L L, CHEN J Y, GOU J Y, SU L W, WU W T, YU W J, WANG P, XU Y C, CHEN Y. Fine mapping and identification of the candidate gene BFS for fruit shape in wax gourd (Benincasa hispida)[J]. Theoretical and Applied Genetics, 2021, 134: 3983. DOI:10.1007/s00122-021-03942-8 |
| [22] |
CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [23] |
GOYAL P, DEVI1 R, VERMA1 R, HUSSAIN S, ARORA P, TABASSUM R, GUPTA R. WRKY transcription factors: Evolution, regulation, and functional diversity in plants[J]. Protoplasma, 2023, 260(2): 331-348. DOI:10.1007/s00709-022-01794-7 |
| [24] |
ZHAO N N, HE M J, LI L, CUI S L, HOU M Y, WANG L, MU G J, LIU L F, YANG X L. Identification and expression analysis of WRKY gene family under drought stress in peanut (Arachis hypogaea L.)[J]. PloS One, 2020, 15(4). DOI:10.1371/journal.pone.0231396 |
| [25] |
CHENG Y F, LUO J X, LI H, WEI F, ZHANG Y Q, JIANG H Y, PENG X J. Identification of the WRKY gene family and characterization of stress-responsive genes in taraxacum kok-saghyz rodin[J]. International Journal of Molecular Sciences, 2022, 23(18): 10270. DOI:10.3390/ijms231810270 |
| [26] |
YANG Y, LIU J, ZHOU X H, LIU S Y, ZHUANG Y. Identification of WRKY gene family and characterization of cold stress-responsive WRKY genes in eggplant[J]. PeerJ, 2020, 8: 8777. DOI:10.7717/peerj.8777 |
| [27] |
ZHANG C, WANG D D, YANG C H, KONG N, SHI Z, ZHAO P, NAN Y, NIE T K, WANG R Q, MA H L, CHEN Q. Genome-wide identification of the potato WRKY transcription factor family[J]. PloS One, 2017, 12(7): 0181573. DOI:10.1371/journal.pone.0181573 |
| [28] |
DIAO W P, SNYDER J C, WANG S B, LIU J B, PAN B G, GUO G J, WEI G. Genome-wide identification and expression analysis of WRKY gene family in Capsicum annuum L.[J]. Frontiers in Plant Science, 2016, 7: 211. DOI:10.3389/fpls.2016.00211 |
| [29] |
W EI, Y X, SHI H T, XIA Z Q, TIE W W, DING Z H, YAN Y, WANG W Q, HU W, LI K. Genome-wide identification and expression analysis of the WRKY gene family in cassava[J]. Frontiers in Plant Science, 2016, 7: 25. DOI:10.3389/fpls.2016.00025 |
| [30] |
ROSS C A, LIU Y, SHEN Q X J. The WRKY gene family in rice (Oryza sativa)[J]. Journal of Integrative Plant Biology, 2007, 49(6): 827-842. DOI:10.1111/j.1744-7909.2007.00504.x |
| [31] |
CHEN C H, CHEN X Q, HAN J Q, LU W L, REN Z H. Genome-wide analysis of the WRKY gene family in the cucumber genome and transcriptome-wide identification of WRKY transcription factors that respond to biotic and abiotic stresses[J]. BMC Plant Biology, 2020, 20(1): 443. DOI:10.1186/s12870-020-02625-8 |
| [32] |
CANNON S B, MITRA A, BAUMGARTEN A, YOUNG N D, MAY G. The roles of segmental and tandem gene duplication in the evolution of large gene families in Arabidopsis thaliana[J]. BMC Plant Biology, 2004, 4(1): 10. DOI:10.1186/1471-2229-4-10 |
| [33] |
WU K L, GUO Z J, WANG H H, LI J. The WRKY family of transcription factors in rice and Arabidopsis and their origins[J]. DNA Research, 2004, 12(1): 9. DOI:10.1093/dnares/12.1.9 |
| [34] |
GOEL R, PANDEY A, TRIVEDI P K, ASIF M H. Genome-wide analysis of the musa WRKY gene family evolution and differential expression during development and stress[J]. Frontiers in Plant Science, 2016, 7: 299. DOI:10.3389/fpls.2016.00299 |
| [35] |
BENCKE-MALATO M, CABREIRA C, WIEBKE-STROHM B, BÜCKER-NETO1 L, MANCINI E, OSORIO M B, HOMRICH M S, TURCHETTO-ZOLET A C, CARVALHO M C D, STOLF R, WEBER R L, WESTERGAARD G, CASTAGNARO A, ABDELNOOR R V, MARCELINO-GUIMARÃES F C, MARGIS-PINHEIRO M, BODANESE-ZANETTINI M H. Genome-wide annotation of the soybean WRKY family and functional characterization of genes involved in response to Phakopsora pachyrhizi infection[J]. BMC Plant Biology, 2014, 14(1): 236. DOI:10.1186/s12870-014-0236-0 |
| [36] |
WEI K F, CHEN J, CHEN Y F, WU L J, XIE D X. Molecular phylogenetic and expression analysis of the complete WRKY transcription factor family in maize[J]. DNA Research, 2012, 19(2): 153-164. DOI:10.1093/dnares/dsr048 |
| [37] |
WALLEY J W, DEHESH K. Molecular mechanisms regulating rapid stress signaling networks in Arabidopsis[J]. Journal of Integrative Plant Biology, 2010, 52(4): 354-359. DOI:10.1111/j.1744-7909.2010.00940.x |
| [38] |
CAI Z D, XIAN P Q, WANG H, LIN R B, LIAN T X, CHENG Y B, MA Q B, NIAN H. Transcription factor GmWRKY142 confers cadmium resistance by up-regulating the cadmium tolerance 1-like genes[J]. Frontiers in Plant Science, 2020, 11: 724. DOI:10.3389/fpls.2020.00724 |
| [39] |
DHATTERWAL P, BASU S, MEHROTRA S, MEHROTRA R. Genome wide analysis of W-box element in Arabidopsis thaliana reveals TGAC motif with genes down regulated by heat and salinity[J]. Scientific Reports, 2019, 9: 1681. DOI:10.1038/s41598-019-38757-7 |
| [40] |
DING Z J, YAN J Y, LI G X, WU Z C, ZHANG S Q, ZHENG S J. WRKY41 controls Arabidopsis seed dormancy via direct regulation of ABI3 transcript levels not downstream of ABA[J]. The Plant Journal, 2014, 79(5): 810-823. DOI:10.1111/tpj.12597 |
| [41] |
WANG Y Q, CUI X, YANG B, XU S, WEI S Y, ZHAO P Y, NIU F F, SUN M T, WANG C, CHENG H, JIANG Y Q. WRKY55 transcription factor positively regulates leaf senescence and defense response through modulating the transcription of genes implicated in ROS and SA biosynthesis in Arabidopsis[J]. Development, 2020, 147: 189647. DOI:10.1242/dev.189647 |
| [42] |
LI J, ZHONG R S, PALVA T. WRKY70 and its homolog WRKY54 negatively modulate the cell wall-associated defenses to necrotrophic pathogens in Arabidopsis[J]. PloS One, 2017, 12(8): 0183731. DOI:10.1371/journal.pone.0183731 |
| [43] |
JIAO C Y, LI K X, ZUO Y X, GONG J Q, GUO Z Q, SHEN Y B. CALMODULIN1 and WRKY53 function in plant defense by negatively regulating the jasmonic acid biosynthesis pathway in Arabidopsis[J]. International Journal of Molecular Sciences, 2022, 23: 7718. DOI:10.3390/ijms23147718 |
| [44] |
DAVLETOVA S, RIZHSKY L, LIANG H J, ZHONG S Q, OLIVER D J, COUTU J, SHULAEV V, SCHLAUCH K, MITTLER R. Cytosolic ascorbate peroxidase 1 is a central component of the reactive oxygen gene network of Arabidopsis[J]. The Plant Cell, 2005, 17(1): 268-81. DOI:10.1105/tpc.104.026971 |
| [45] |
BESSEAU B, LI J, PALVA E T. WRKY54 and WRKY70 co-operate as negative regulators of leaf senescence in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2012, 63(7): 2667-2679. DOI:10.1093/jxb/err450 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51