文章信息
基金项目
- 岭南现代农业科学与技术广东省实验室河源分中心项目(DT20220021);广东省现代农业产业技术体系创新团队项目(2023KJ114);广州市农村科技特派员项目(20212100032);广东省现代畜牧业示范推广(畜牧业新技术示范推广)项目
作者简介
- 唐雪颖(2000—),女,在读硕士生,研究方向为反刍动物遗传育种,E-mail:tangxueying0502@126.com.
通讯作者
- 李大刚(1974—),男,博士,研究员,研究方向为反刍动物健康养殖,E-mail:lidagang@gdaas.cn.
文章历史
- 收稿日期:2023-11-08
2. 广东省农业科学院动物科学研究所,广东 广州 510641;
3. 岭南现代农业科学与技术广东省实验室河源分中心,广东 河源 517000
2. Institute of Animal Science, Guangdong Academy of Agricultural Sciences, Guangzhou 510641, China;
3. Heyuan Sub-center, Guangdong Laboratory for Lingnan Modern Agricultural Science and Technology, Heyuan 517000, China
【研究意义】荷斯坦牛是世界上最受欢迎的奶牛品种之一。奶牛养殖业中,泌乳及繁殖性能是重点关注问题,泌乳能力差与繁殖力低是母牛淘汰的主要因素。尽管目前我国的奶牛淘汰率低于欧美国家,但奶牛平均胎次与之相比仍有较大差距。骨骼肌作为哺乳动物最大的组织,参与动物的生长、发育、繁殖及免疫等众多生命过程[1]。挖掘荷斯坦牛肌肉组织中与妊娠末期、泌乳期及繁殖力性状相关的关键候选基因,能够为荷斯坦牛核心群选育和分子育种提供参考。
【前人研究进展】研究发现,奶牛肌肉组织中关键基因(如MSTN、GBRH、PPARG)的表达对繁殖系统的生长发育、器官成熟及泌乳性能维持具有重要作用[2-4]。繁殖性状是奶牛重要的经济性状,主要包括产犊难易、产犊间隔、胚胎发育等指标,受特定基因表达严格调控。研究表明,荷斯坦牛FoxO1基因的CC、TT、CT基因型,对奶牛的产犊间隔、初产日龄及空怀等繁殖性状有显著影响[5]。PIK3CB基因多态性被证实与荷斯坦牛繁殖性状和产奶性状存在关联,其上的g.130433743 A>G等SNP位点可作为潜在的分子标记[6]。繁殖性状相关关键基因及其功能的深入挖掘对奶牛的遗传改良、性能提升至关重要。
加权基因共表达网络分析(Weighted gene co-expression network analysis, WGCNA)是一种基于相关系数的共表达网络分析方法,通过将高通量基因表达数据转化为基因共表达网络,使复杂的高通量基因表达数据转化为简洁的模块[7]。蛋白互作(Protein-protein interaction,PPI)网络分析是研究蛋白质之间相互作用的网络分析方法,通过构建蛋白质相互作用网络可以揭示蛋白质之间的功能联系、信号传导通路和调控机制[8]。近年来,WGCNA技术逐步被应用于畜禽关键性状调控标记基因的挖掘研究工作中[9]。【本研究切入点】目前,对荷斯坦牛肌肉组织的研究多与肉品质相关,而肌肉组织与妊娠末期、泌乳期和繁殖力的相关研究未见报道。因此,开展对其肌肉组织与不同生理时期及繁殖力的研究很有必要,可探明妊娠末期、泌乳期肌肉中基因的差异表达,以及肌肉中基因表达对繁殖力的影响。【拟解决的关键问题】本研究通过WGCNA、PPI等生物信息学联合分析技术构建荷斯坦牛肌肉中与妊娠末期、泌乳期及繁殖力相关的基因共表达网络,为深入挖掘不同生理时期荷斯坦牛肌肉组织中繁殖力调控关键基因及其应答网络奠定基础,进而为高繁殖力奶牛的遗传调控解析提供理论依据。
1 材料与方法 1.1 试验材料本研究数据来源于GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)中有关不同生理时期及不同繁殖力荷斯坦牛的肌肉组织基因表达数据集GSE62159。研究数据包含48头荷斯坦牛的胎次(1~2胎次)、产奶量(200~900 kg)、不同生理时期肌肉组织的基因表达数据,其中妊娠末期(分娩前48 d)、泌乳早期(分娩后1 d)、泌乳中期(分娩后147 d)荷斯坦牛各16头。产犊间隔长短按照爱尔兰荷斯坦牛国家数据库中产犊间隔最短20% 与产犊间隔最长5% 的荷斯坦牛平均产犊间隔进行划分。低繁殖力奶牛、高繁殖力奶牛各24头,从分娩到再次配种成功的平均时间间隔分别为113.8、85.6 d[10-11]。
1.2 试验方法1.2.1 基因共表达网络构建 使用R语言中的WGCNA包[12]对样本数据进行分析。首先将基因表达模式相似的样本进行聚类,并根据聚类高度30 000去除离群样本;根据pickSoftThreshold函数,以无尺度网络拟合指数R2>0.9为标准,选择出最佳软阈值。使用adjacency函数构建拓扑重叠矩阵(TOM),根据相异度(dissTOM=1-TOM)和模块合并阈值0.15对基因进行聚类,得到共表达模块。
将共表达模块与表型信息进行关联分析,计算出各模块与表型的相关系数r值与P值。依据|r|>0.35、P<0.05选择出与不同生理时期和繁殖力相关的模块作为目标模块,以各目标模块内连接度排名前30的基因作为各模块的枢纽基因(Hub基因),构建基因共表达网络。
1.2.2 模块基因富集分析 使用DAVID网站(https://david.ncifcrf.gov)对所有目标模块内的基因进行GO与KEGG富集分析,并使用R语言中的ggplot2包将富集结果可视化。
1.2.3 蛋白互作网络构建及目标基因确定 使用String网站(https://cn.string-db.org)进行蛋白互作网络(PPI)构建。使用Cytoscape软件以PPI网络中节点的度值为基础,筛选出排名前30的基因作为核心基因。将每个目标模块得到的Hub基因与核心基因取交集,得到与性状相对应的目标基因。
2 结果与分析 2.1 荷斯坦牛肌肉组织基因共表达网络构建对数据集数据进行样本聚类分析,剔除离群样本后得到33个样本,其中妊娠末期样本15个、泌乳早期样本2个、泌乳中期样本16个;高繁殖力样本18个,低繁殖力样本15个。对这33个样本进行分析,得到样本的树状图与性状热图(图 1A)。根据样本自由度和连通度分析,确定最佳软阈值为12,并进行无尺度网络构建。根据模块内的特征基因进行聚类,将红线(高度0.15)以下的模块进行合并(图 1B),共得到13个模块(图 1C)。将表型信息与模块特征值进行相关性分析,选择相关性最高的模块作为目标模块,妊娠末期选择Green模块(r=0.45,P=0.009)、泌乳中期选择Blue模块(r=0.52,P=0.002)、高繁殖力选择Magenta模块(r=0.38,P=0.04)为目标模块(图 1D)。
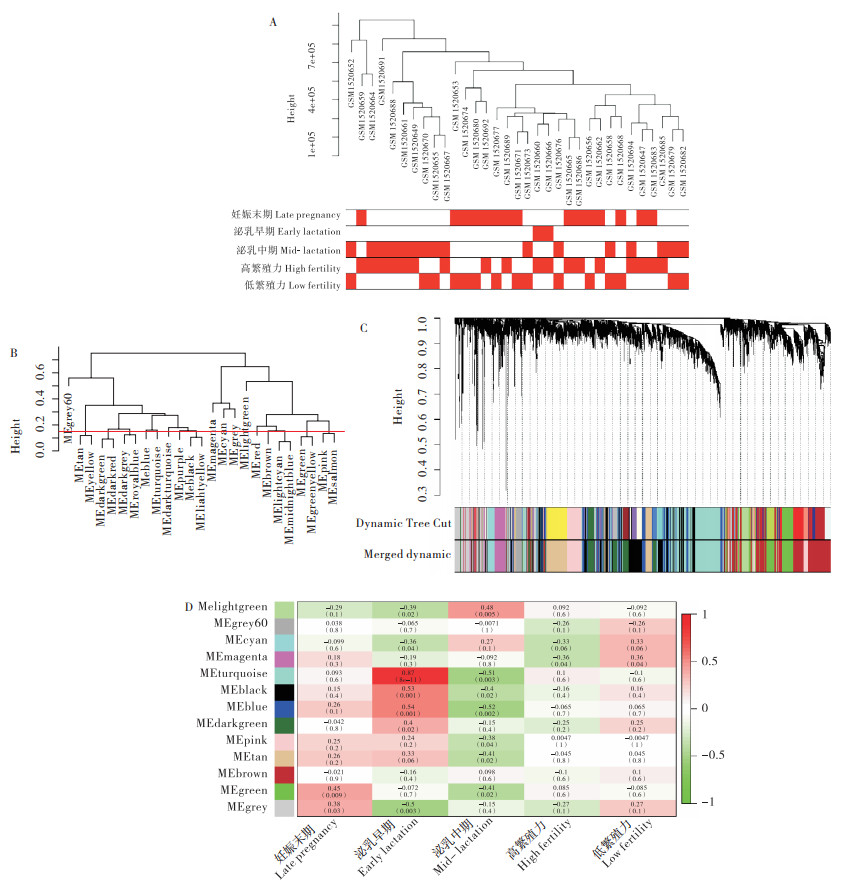
|
| A: 样本聚类树和性状热图;B: 模块特征基因聚类树;C: 模块聚类树;D: 表型信息与模块特征值相关性 A: Sample clustering tree and trait heatmap; B: Modular characteristic gene clustering tree; C: Module clustering tree; D: Correlation between phenotypic information and module eigenvalues 图 1 荷斯坦牛肌肉组织基因共表达网络构建 Fig. 1 Construction of gene co-expression network in genes of muscle tissue of Holstein cattle |
2.2 妊娠末期荷斯坦牛肌肉组织基因共表达网络及功能分析
各目标模块内连接度排名前30的Hub基因如表 1所示。对Green模块的30个Hub基因进行GO富集分析,结果(图 2A)显示,与Green模块相关联的主要生物学过程有三羧酸循环、尾部锚定膜蛋白插入内质网膜、线粒体自噬调节、肌动蛋白丝聚合调节、蛋白质运输、靶向线粒体的蛋白质、蛋白质K48连接泛素化、通过终止 - 转移膜 - 锚定序列将蛋白质插入内质网膜、蛋白酶体介导的泛素依赖性蛋白质分解代谢过程、蛋白质定位到细胞核正向调控、线粒体裂变正向调节、pH值降低、泛素特异性蛋白酶活性负调节、蛋白质K48相关去泛素化负调控、β- 淀粉样蛋白形成负调节、线粒体翻译、线粒体呼吸链复合物Ⅰ组装、氢离子跨膜转运、细胞对氧化应激的反应和对细胞内氧化剂的解毒;与Green模块相关联的主要细胞成分包括突触小泡、蛋白酶体核心复合物、β- 亚基复合物、细胞质的核周区、核浆、线粒体、线粒体小核糖体亚基、线粒体呼吸链复合物Ⅰ、线粒体基质、线粒体大核糖体亚基、线粒体内膜前序列易位酶复合物、线粒体内膜、线粒体内膜组成部分、内质网膜组成部分、内质网膜蛋白复合物、核内体、内质网膜、内膜系统、胞液、细胞质和网格蛋白包被的囊泡膜;与Green模块相关联的主要分子功能包括泛素结合、翻译延伸因子活性、转录辅阻遏物活性、苏氨酸型内肽酶活性、核糖体结构组成、细胞骨架结构组成、质子传输ATP酶活性,旋转机制、蛋白质同二聚化活性、蛋白质结合、桥接参与泛素化的底物识别、蛋白结合、NEDD8- 特异性蛋白酶活性、NADH结合、金属肽酶活性、金属离子结合、膜插入酶活性、大分子复合物结合、异肽酶活性、相同蛋白质结合、伴侣蛋白结合和1-酰基甘油 -3- 磷酸O- 酰基转移酶(AGPAT)活性。KEGG富集分析结果(图 2B)表明,与Green模块相关联的主要信号通路有2- 氧化羧酸代谢、三羧酸循环、蛋白酶体、FcγR介导的吞噬作用、非酒精性脂肪肝、cGMP-PKG信号通路、内质网中的蛋白质加工、脊髓小脑性共济失调、内吞作用、氧化磷酸化、生热作用,以及糖尿病、心肌病、致癌作用—活性氧、朊病毒病、帕金森综合症、亨廷顿病、肌萎缩侧索硬化症、阿尔茨海默病、神经退行性变的途径等多种疾病和代谢途径。Green模块中连接度排名前30的Hub基因与PPI网络分析的前30个核心基因取交集,得到3个目标基因:EIF5A、ACO2和EEF1G(图 2C、D)。
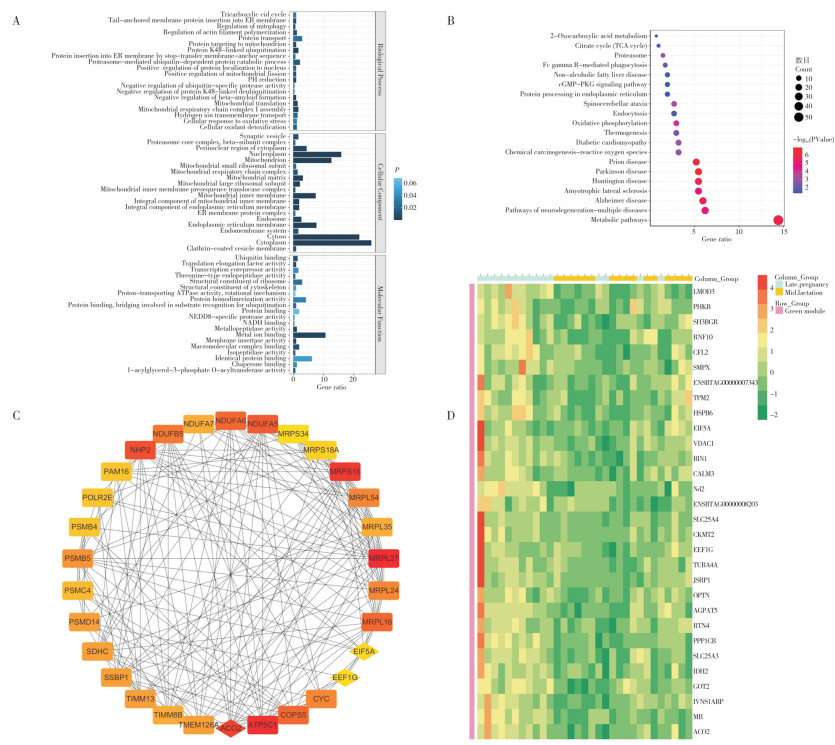
|
| A: GO富集分析;B: KEGG富集分析;C: PPI网络核心基因互作图;D: Hub基因在不同生理时期的表达水平热图 A: GO enrichment analysis; B: KEGG enrichment analysis; C: Core gene interaction map of PPI Network; D: Heat map of expression levels of Hub gene at different physiological stages 图 2 Green模块富集分析、PPI及Hub基因表达验证结果 Fig. 2 Green module enrichment analysis, PPI and Hub gene expression verification results |
2.3 泌乳中期荷斯坦牛肌肉组织基因共表达网络及功能分析
对Blue模块中连接度排名前30的Hub基因(表 1)进行GO富集分析,结果(图 3A)显示,与Blue模块相关联的主要生物学过程有翻译起始、干细胞群维持、RNA拼接、蛋白质运输、蛋白质稳定化、蛋白质输入细胞核、蛋白质折叠、蛋白酶体介导的泛素依赖性蛋白质分解代谢过程、蛋白靶向膜易位的翻译后蛋白、翻译负调节、mRNA剪接等,与Blue模块相关联的主要细胞成分包括泛素连接酶复合物、P小体、核质、核仁、核斑点、核膜、中体、高尔基体、纤维中心、内质网 - 高尔基体中间室、内质网、细胞质、中心体和催化步骤2剪接体;与Blue模块相关联的主要分子功能包括未折叠蛋白结合、泛素蛋白连接酶结合、tRNA结合、翻译起始因子活性、翻译因子活性、RNA结合、RNA解旋酶活性、蛋白磷酸酶调节活性、蛋白磷酸酶2A结合、核输出信号受体活性、mRNA结合、大分子复合物结合、酶结合、ATP酶活性、ATP结合和7SK snRNA结合。KEGG富集分析结果(图 3B)表明,与Blue模块相关联的主要信号通路有长寿调节途径、抗原加工和呈递、GnRH信号通路、癌症中PD-L1表达和PD-1检查点通路、卵母细胞减数分裂、弓形体病、恰加斯病、泛素介导的蛋白水解、细胞衰老、流体剪切应力和动脉粥样硬化、真核生物中的核糖体生物发生、mRNA监测途径、核质转运、内质网中的蛋白质加工、沙门氏菌感染、脂质与动脉粥样硬化、MAPK信号通路、肌萎缩侧索硬化症和剪接体。Blue模块中连接度排名前30的Hub基因与PPI网络分析的前30个核心基因取交集,得到1个核心基因:EIF4A2(图 3C、D)。
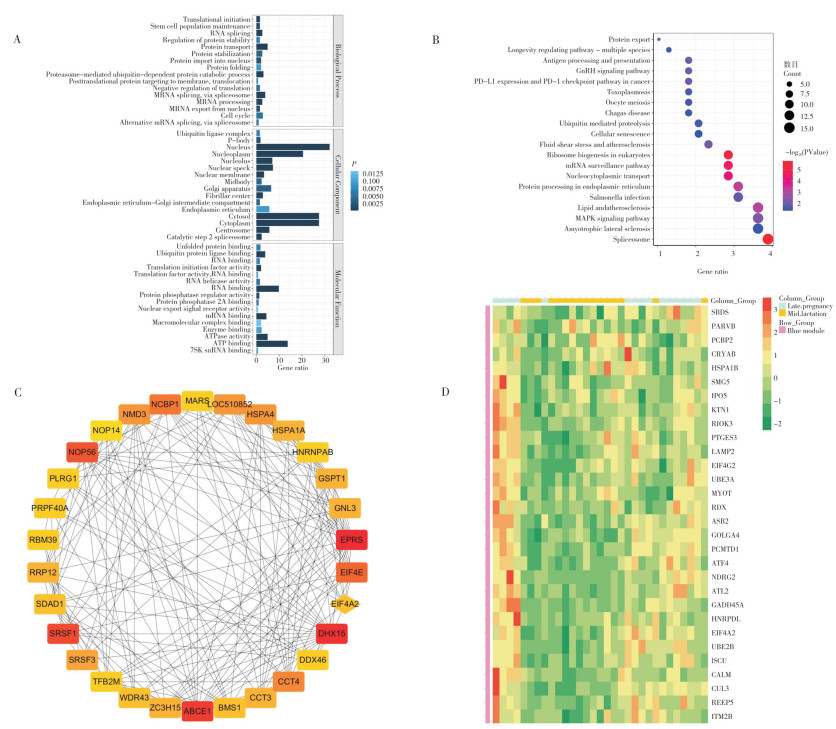
|
| A: GO富集分析;B: KEGG富集分析;C: PPI网络核心基因互作图;D: Hub基因在不同生理时期组的表达水平热图 A: GO enrichment analysis; B: KEGG enrichment analysis; C: Core gene interaction map of PPI Network; D: Heat map of expression levels of Hub gene at different physiological stages 图 3 Blue模块富集分析、PPI及Hub基因表达验证结果 Fig. 3 Blue module enrichment analysis, PPI and Hub gene expression verification results |
2.4 高繁殖力荷斯坦牛肌肉组织基因共表达网络及功能分析
对Magenta模块进行GS-MM相关性验证,结果表明,Magenta模块内的基因与泌乳中期显著相关(P=1.5×10-7),模块内连接度排名前30的Hub基因见表 1。GO富集分析结果(图 4A)显示,与Magenta模块相关联的主要生物学过程有血管形成、受体内化、蛋白质定位于质膜、平滑肌细胞增殖正向调节、粘着斑组装正向调控、细胞迁移正向调节、血管生成正向调节、内皮细胞凋亡负调控、子宫内胚胎发育、细胞对转化生长因子β刺激的反应、细胞粘附、细胞间粘附、ATP合成耦合电子传递、血管新生和肌动蛋白丝组织;与Magenta模块相关联的主要细胞成分包括褶皱膜、受体复合物、细胞质的核周区、膜筏、膜、粘着斑、细胞外空间、质膜外侧、细胞骨架、细胞质、细胞表面、细胞皮层、基底膜、顶质膜和粘连节点;与Magenta模块相关联的主要分子功能包括转化生长因子β结合、受体结合、蛋白激酶结合、蛋白质同源二聚体活性、蛋白质结合、NADH脱氢酶(泛醌)活性、离子通道结合、整合素结合、相同蛋白质结合、细胞外基质结构成分、酶抑制剂活性、钙调蛋白结合、钙离子结合、肌动蛋白丝结合和肌动蛋白结合。KEGG富集分析结果(图 4B)表明,与Magenta模块相关联的主要信号通路有粘连结点、小细胞肺癌、Th1和Th2细胞分化、氧化磷酸化、细胞外基质 - 受体相互作用、细胞粘附分子、Rap1信号通路、弓形体病、白细胞跨内皮细胞迁移、生热作用、肌动蛋白细胞骨架调节、人乳头瘤病毒感染、附着斑、PI3K-Akt信号通路和癌症途径。Magenta模块中连接度排名前30的Hub基因与PPI网络分析的前30个核心基因取交集,得到8个核心基因:ITGB1、MYH9、TLN1、CAV1、COL4A1、COL4A2、FLNA和HSPG2(图 4C、D)。
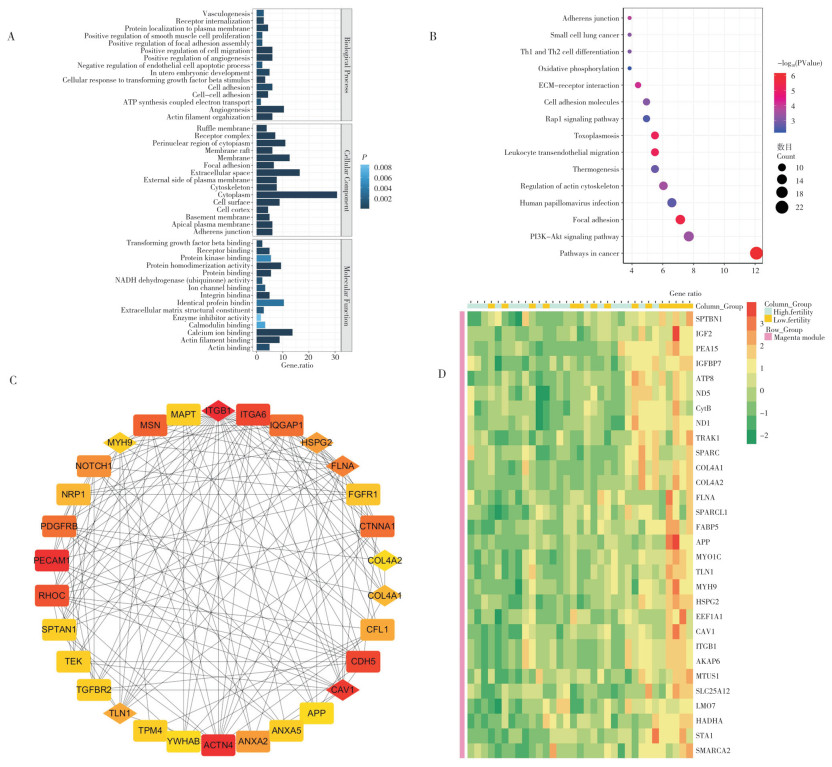
|
| A: GO富集分析;B: KEGG富集分析;C: PPI网络核心基因互作图;D: Hub基因在不同繁殖力组的表达水平热图 A: GO enrichment analysis; B: KEGG enrichment analysis; C: Core gene interaction map of PPI Network; D: Heat map of expression levels of Hub gene at different physiological stages 图 4 Magenta模块富集分析、PPI及Hub基因表达验证结果 Fig. 4 Magenta module enrichment analysis, PPI and Hub gene expression verification results |
3 讨论
肌肉组织的基因参与机体代谢的各个环节,因而与奶牛生理时期及繁殖力密切相关[13]。本研究通过对荷斯坦牛肌肉组织中与泌乳期及繁殖力相关基因的共表达网络构建,得到与妊娠末期、泌乳中期相关的Green模块、Blue模块以及与繁殖力相关的Magenta模块,并在各模块中筛选出1~8个目标性状相关的关键基因。尽管原数据集中包含泌乳早期样品数据,但由于在样品聚类分析时该组多数样品不符合分析要求而被剔除(仅剩余2个样品可用),因此本研究没有对泌乳早期相关信息进行后续分析。
妊娠末期是奶牛泌乳周期中特殊的生理时期,期间奶牛要经历分娩和泌乳等生理过程,由于这一时期机体营养和内分泌功能改变,奶牛面临特殊生理过程产生的应激。本研究在Green模块中最终锚定与妊娠末期相关的关键基因为EIF5A、ACO2和EEF1G。尽管目前未有EIF5A基因在反刍动物中的功能研究,但有报道指出EIF5A在真核生物中高度保守,其能够调控增殖、凋亡和细胞分化等细胞过程相关基因mRNA的转录,同时也与炎症相关蛋白的翻译有关[14]。Forde等[15]通过蛋白质组学与转录组学分析发现,ACO2等基因在妊娠和非妊娠母牛体内表达具有显著差异,推测ACO2等基因具有作为妊娠鉴定标志物的潜在价值。此外,本研究对筛选到的关键基因进行功能富集分析发现,Green模块中基因功能包括蛋白合成代谢、三羧酸循环及分泌相关的生物学过程。这与妊娠末期(分娩前1 d)荷斯坦牛由非泌乳状态向大量合成和分泌乳汁的状态进行转变,物质与能量代谢加强、蛋白质合成与分泌增加、机体对能量需求急剧增加[16-17]、机体免疫应答功能响应[18]等生理现象相符合。
泌乳中期奶牛在较长时间内需要稳定供应乳腺组织合成蛋白以满足泌乳需要[19]。本研究在Blue模块中的富集分析结果大部分与蛋白质的合成及分泌有关。在Blue模块寻找到1个目标基因EIF4A2,此基因作为真核翻译起始因子之一,不仅参与蛋白质合成,还在肌肉发育中起重要作用[20],此外,EIF4A2基因通过控制编码多能性因子的mRNA从而调控干细胞维持及机体发育[21]。Docquier等[22]研究表明,同为真核反义起始因子基因的EIF3f会影响胚胎发育,并依赖mTOR调控肌肉的生长发育,表明EIF家族可能通过调控蛋白合成调控奶牛的生产及繁殖性能。
品种、遗传、个体因素、饲养管理水平、繁殖技术与疾病等均会影响奶牛的繁殖性能[23]。以往开展繁殖性状候选基因相关研究,通常以繁殖系统相关组织器官如子宫、卵巢、胎盘等为研究对象,挖掘到如FSH[24]、LH[25]、GnRH[26]、PRLR[27]等繁殖相关关键基因。本研究选取荷斯坦牛的肌肉组织为研究对象,是认为奶牛繁殖性能的体现是动物体功能系统的综合体现,肌肉组织作为机体最大的组织器官,存在对繁殖性能的贡献性与复杂性,因而本研究在繁殖力相关的Magenta模块中最终挖掘到的关键基因多达8个。其中ITGB1基因调控整合素的生成,可以促进精子在生殖道内的存活和运动能力,同时也能增强卵子和胚胎的黏附能力,提高奶牛受孕率[28-29]。良好的卵子质量、卵泡成熟度以及胚胎发育与MYH9和TLN1基因的高表达水平有直接关系[29-30]。CHSPG2基因可以影响泌乳激素的分泌和作用,进而对卵巢功能和雌激素水平产生调节作用[31]。HSPG2参与胎盘和子宫内膜的形成和发育,对胚胎植入和胎儿发育具有重要影响[32]。OL4A1和COL4A2在鸡体内编码Ⅳ胶原蛋白,该蛋白是组成基底膜的关键成分,这两个基因可影响基底膜的稳定性[33],但对于牛的作用尚不明确。
PPI网络应用有助于揭示蛋白质功能和调控的复杂性。蛋白质相互作用是细胞内许多生物过程的基础,包括信号传导、代谢调控和细胞周期等。通过构建和分析PPI网络,可以识别关键的蛋白质节点和模块,进而了解它们在细胞功能中的作用和相互关系,这对于揭示生物体内复杂的生物学过程至关重要[34-36]。本研究将WGCNA与PPI网络相结合来挖掘荷斯坦牛肌肉组织中与泌乳期及繁殖力相关的基因,将各性状模块内的Hub基因与核心基因取交集,得到各性状相关的目标关键基因,有助于后续深入了解荷斯坦牛肌肉组织对生产和繁殖性能的调控机制。目前本研究尚未对筛选到的目标关键基因及其特异性功能与作用进行深入验证,后续研究将围绕筛选到的基因开展分子遗传学研究,以期为高产及高繁殖力的奶牛选育积累更加丰富的理论资料。
4 结论本研究运用WGCNA方法分析得到与荷斯坦牛妊娠末期相关的Green模块、与泌乳中期相关的Blue模块以及与繁殖力相关的Magenta模块,结合PPI网络分析结果鉴定到荷斯坦牛肌肉中与妊娠末期、泌乳中期相关的关键基因,分别有3个(EIF5A、ACO2和EEF1G)和1个(EIF4A2),与繁殖力相关的关键基因有8个(ITGB1、MYH9、TLN1、CAV1、COL4A1、COL4A2、FLNA和HSPG2)。本研究构建了荷斯坦牛肌肉中与妊娠末期、泌乳期和繁殖力性状相关的基因共表达网络,基因功能富集结果显示,共表达网络中的基因主要富集在氧化磷酸化、细胞外基质 - 受体相互作用、细胞粘附分子、Rap1信号通路、白细胞跨内皮细胞迁移、生热作用、肌动蛋白细胞骨架调节、PI3K-Akt信号通路等分子途径,与奶牛繁殖力性状具有关联性。
| [1] |
SCIORATI C, RIGAMONTI E, MANFREDI A A, ROVERE-QUERINI P. Cell death, clearance and immunity in the skeletal muscle[J]. Cell Death and Differentiation, 2016, 23(6): 927-37. DOI:10.1038/cdd.2015.171 |
| [2] |
贺鹏迦, 马正才, 敏妍滢, 董艳娇, 朱静, 马永生, 马彦男. PPARG基因Q448H突变位点对中国荷斯坦牛繁殖性状的影响[J]. 农业生物技术学报, 2018, 26(2): 253-262. DOI:10.3969/j.issn.1674-7968.2018.02.008 HE P J, MA Z C, MIN Y Y, DONG Y J, ZHU J, MA Y S, MA Y N. The Effects of a Q448H mutation of the PPARG gene on reproductive traits in Chinese Holstein cattle (Bos taurus)[J]. Journal of Agricultural Biotechnology, 2018, 26(2): 253-262. DOI:10.3969/j.issn.1674-7968.2018.02.008 |
| [3] |
高清, 张海亮, 胡丽蓉, 张帆, 许静漪, 徐昊祺, 罗汉鹏, 刘林, 郭刚, 王雅春. GnRH1基因多态性与奶牛繁殖、产奶和热应激反应性状的关联分析[J]. 中国畜牧杂志, 2023, 59(4): 90-95. DOI:10.19556/j.0258-7033.20220413-04 GAO Q, ZHANG H L, HU L R, ZHANG F, XU J L, XU H Q, LUO H P, LIU L, GUO G, WANG Y C. Association analysis of GnRH1 gene polymorphisms and breeding, milk production and heat stress response traits in dairy cows[J]. Chinese Journal of Animal Science, 2023, 59(4): 90-95. DOI:10.19556/j.0258-7033.20220413-04 |
| [4] |
韩圣忠. MSTN基因缺失对猪繁殖性能的影响研究[D]. 延边: 延边大学, 2021. DOI: 10.27439/d.cnki.gybdu.2021.000905. HAN S Z. Study on the effect of MSTN gene deletion on pig reproductive performance[D]. Yanbian : Yanbian University, 2021. DOI: 10.27439/d.cnki.gybdu.2021.000905. |
| [5] |
赵佳强, 周妍, 于倩楠, 陶林林, 张淑君, 易建明, 滕茂平, 李丛远, 张军伟. 中国荷斯坦奶牛FoxO1基因多态性及与繁殖性状关系研究[J]. 东北农业大学学报, 2013, 44(6): 54-57. DOI:10.19720/j.cnki.issn.1005-9369.2013.06.010 ZHAO J Q, ZHOU Y, YU Q N, TAO L L, ZHANG S J, YI J M, TENG M P, LI C Y, ZHANG J W. Polymorphism of FoxO1 gene and associations with reproduction traits in Holstein cattle of China[J]. Journal of Northeast Agricultural University, 2013, 44(6): 54-57. DOI:10.19720/j.cnki.issn.1005-9369.2013.06.010 |
| [6] |
徐昊祺, 许静漪, 胡丽蓉, 张帆, 罗汉鹏, 张海亮, 师睿, 李想, 刘林, 刘巧香, 郭刚, 王雅春. 中国荷斯坦牛PIK3CB基因多态性及其与繁殖和产奶性状的关联分析[J]. 中国畜牧兽医, 2022, 49(9): 3438-3452. DOI:10.16431/j.cnki.1671-7236.2022.09.018 XU H Q, XU J Y, HU L R, ZHANG F, LUO H P, ZHANG H L, SHI R, LI X, LIU L, LIU Q X, GUO G, WANG Y C. Polymorphism of PIK3CB gene and its association analysis with reproductive and milk production traits in Chinese Holstein cattle[J]. China Animal Husbandry & Veterinary Medicine, 2022, 49(9): 3438-3452. DOI:10.16431/j.cnki.1671-7236.2022.09.018 |
| [7] |
宋长新, 雷萍, 王婷. 基于WGCNA算法的基因共表达网络构建理论及其R软件实现[J]. 基因组学与应用生物学, 2013, 32(1): 135-141. SONG C X, LEI P, WANG T. Gene co-expression network analysis based on WGCNA algorithm-theory and implementation in R software[J]. Genomics and Applied Biology, 2013, 32(1): 135-141. |
| [8] |
李明阳, 马春霞, 吴翰欣, 吴小海, 俞建昆. 鼻咽癌组织中基因差异表达及蛋白互作网络分析[J]. 生物技术通讯, 2018, 29(3): 345-350. LI M Y, MA C X, WU H X, WU X H, YU J K. Gene differential expression and protein interaction networks analysis in nasopharyngeal cancer[J]. Letters in Biotechnology, 2018, 29(3): 345-350. |
| [9] |
王京, 米芳, 高慧杰, 郑惠玲. 加权基因共表达网络分析方法及其应用[J]. 家畜生态学报, 2023, 44(3): 92-96. WANG J, MI F, GAO H J, ZHENG H L. Analytical method and application of weighted gene co-expression network[J]. Journal of Domestic Animal Ecology, 2023, 44(3): 92-96. |
| [10] |
MORAN B, CUMMINS S B, CREEVEY C J, BUTLER S T. Transcriptomics of liver and muscle in Holstein cows genetically divergent for fertility highlight differences in nutrient partitioning and inflammation processes[J]. BMC Genomics, 2016, 17(1): 603. DOI:10.1186/s12864-016-2938-1 |
| [11] |
CUMMINS S B, LONERGAN P, EVANS A C, BERRY D P, EVANS R D, BUTLER S T. Genetic merit for fertility traits in Holstein cows: I. Production characteristics and reproductive efficiency in a pasture-based system[J]. Journal of Dairy Science, 2012, 95(3): 1310-1322. DOI:10.3168/jds.2011-4742 |
| [12] |
BOTIA J A, VANDROVCOVA J, FORABOSCO P, GUELFI S, D'SA K, HARDY J, LEWIS C M, RYTEN M, WEALE M E. An additional k-means clustering step improves the biological features of WGCNA gene co-expression networks[J]. BMC Systems Biology, 2017, 11(1): 47. DOI:10.1186/s12918-017-0420-6 |
| [13] |
GESSNER D K, GRONE B, ROSENBAUM S, MOST E, HILLEN S, BECKER S, ERHARDT G, REINER G, EDER K. Effect of dietary fish oil on the expression of genes involved in lipid metabolism in liver and skeletal muscle of lactating sows[J]. Journal of Animal Physiology and Animal Nutrition, 2016, 100(2): 337-347. DOI:10.1111/jpn.12324 |
| [14] |
ANDERSON-BAUCUM E, PINEROS A R, KULKARNI A, WEBB-ROBERTSON B J, MAIER B, ANDERSON R M, WU W, TERSEY S A, MASTRACCI T L, CASIMIRO I, SCHEUNER D, METZ T O, NAKAYASU E S, EVANS-MOLINA C, MIRMIRA R G. Deoxyhypusine synthase promotes a pro-inf lammatory macrophage phenotype[J]. Cell Metabolism, 2021, 33(9): 1883-1893. DOI:10.1016/j.cmet.2021.08.003 |
| [15] |
FORDE N, BAZER F W, SPENCER T E, LONERGAN P. Conceptualizing the endometrium: identification of conceptus-derived proteins during early pregnancy in cattle[J]. Biology of Reproduction, 2015, 92(6): 156. DOI:10.1095/biolreprod.115.129296 |
| [16] |
NORVEZH F, RAZI J M, TABANDEH M R, HAJIKOLAEI M, GOORANINEJAD S. Serum Apelin-36 alteration in late pregnancy and early lactation of dairy cows and its association with negative energy balance markers[J]. Research in Veterinary Science, 2019, 125: 285-289. DOI:10.1016/j.rvsc.2019.07.012 |
| [17] |
淡新刚, 韩元, 史远刚. 添加不同浓度过瘤胃脂肪对奶牛产后MUN、NEFA、BHBA及繁殖的影响[J]. 广东农业科学, 2014, 41(19): 118-121. DOI:10.16768/j.issn.1004-874X.2014.19.042 DAN X G, HAN Y, SHI Y G. Effects of adding different concentrations of undegradable fatty on MUN, NEFA, BHBA and reproduction in postpartum cows[J]. Guangdong Agricultural Sciences, 2014, 41(19): 118-121. DOI:10.16768/j.issn.1004-874X.2014.19.042 |
| [18] |
孙光野, 夏成, 徐闯, 张洪友. 围生期奶牛能量负平衡与免疫抑制的关系[J]. 黑龙江畜牧兽医, 2019(6): 36-40. DOI:10.13881/j.cnki.hljxmsy.2018.03.0147 SUN G Y, XIA C, XU C, ZHANG H Y. The relationship between negative energy balance and immunosuppression in perinatal dairy cow[J]. Heilongjiang Animal Science and Veterinary Medicine, 2019(6): 36-40. DOI:10.13881/j.cnki.hljxmsy.2018.03.0147 |
| [19] |
曹志勇, 黄伟, 邓斌, 陈民, 李晓君, 李贵元, 杜子居, 杨秀娟. 不同胎次荷斯坦牛泌乳量的变化规律研究[J]. 中国奶牛, 2021(5): 14-18. DOI:10.19305/j.cnki.11-3009/s.2021.05.004 CHAO Z Y, HUANG W, DENG B, CHEN M, LI X J, LI G Y, DU Z J, YANG X J. Study on the effect of parity on the milk production of Holstein cows[J]. China Dairy Cattle, 2021(5): 14-18. DOI:10.19305/j.cnki.11-3009/s.2021.05.004 |
| [20] |
ZHANG Z, PENG M, WEN Y, CHAI Y, LIANG J, YANG P, LIU X, LI J, HUANG Y, LI L, HUANG W, QI Z, YANG G, CHEN F, SHI Q, LI Z, RU B, LEI C, WANG E, HUANG Y. Copy number variation of EIF4A2 loci related to phenotypic traits in Chinese cattle[J]. Veterinary Medicine and Science, 2022, 8(5): 2147-2156. DOI:10.1002/vms3.875 |
| [21] |
LI D, YANG J, HUANG X, ZHOU H, WANG J. eIF4A2 targets developmental potency and histone H3.3 transcripts for translational control of stem cell pluripotency[J]. Science Advances, 2022, 8(13): eabm0478. DOI:10.1126/sciadv.abm0478 |
| [22] |
DOCQUIER A, PAVLIN L, RAIBON A, BERTRAND-GADAY C, SAR C, LEIBOVITCH S, CANDAU R, BERNARDI H. eIF3f depletion impedes mouse embryonic development, reduces adult skeletal muscle mass and amplifies muscle loss during disuse[J]. Journal of Physiology-London, 2019, 597(12): 3107-3131. DOI:10.1113/JP277841 |
| [23] |
张运杰. 影响奶牛繁殖力的因素及提高繁殖力技术研究[D]. 泰安: 山东农业大学, 2021. ZHANG Y J. Study on the factors affecting the fecundity of dairy cows and the techniques for improving fecundity[D]. Tai'an: Shandong Agricultural University, 2021. DOI: 10.27277/d.cnki.gsdnu.2021.000526. |
| [24] |
GROMOLL J, PEKEL E, NIESCHLAG E. The structure and organization of the human follicle-stimulating hormone receptor (FSHR) gene[J]. Genomics, 1996, 35(2): 308-311. DOI:10.1006/geno.1996.0361 |
| [25] |
刘继丰, 赵志辉, 戴立胜, 岳续朋, 赵锐锋, 张嘉保. 促黄体素LHβ基因的单核苷酸多态性与牛精液品质的相关分析[J]. 畜牧与兽医, 2009, 41(10): 8-12. LIU J F, ZHAO Z H, DAI L S, YUE X P, ZHAO R F, ZHANG J B. Single nucleotide polymorphism of the LHβ gene and its association with the semen quality in the cattle[J]. Animal Husbandry & Veterinary Medicine, 2009, 41(10): 8-12. |
| [26] |
黎金秀, 凌泽继, 叶世光, 秦津, 潘堂峰, 潘琼, 韩涛, 李瑞明, 谢莹雪, 周辰瑜, 宋小白. 哺乳动物促性腺激素释放激素(GnRH) 研究进展[J]. 现代农业科技, 2009(11): 220-221. LI J X, LIN Z J, YE S G, QIN J, PAN Q, HAN T, LI R M, XIE Y X, ZHOU C Y, SONG X B. Progress in gonadotropin-releasing hormone (GnRH)[J]. Modern Agricultural Science and Technology, 2009(11): 220-221. |
| [27] |
HAYES H, LE CHALONY C, GOUBIN G, MERCIER D, PAYEN E, BIGNON C, KOHNO K. Localization of ZNF164, ZNF146, GGTA1, SOX2, PRLR and EEF2 on homoeologous cattle, sheep and goat chromosomes by fluorescent in situ hybridization and comparison with the human gene map[J]. Cytogenet Cell Genet, 1996, 72(4): 342-346. DOI:10.1159/000134218 |
| [28] |
邱梅玉, 黄涛, 胡九英, 汪德明, 公红斌, 王斌虎, 孙敬礼, 来红霞. ESR与ITGB1基因多态性对大白猪繁殖性能的影响[J]. 中国兽医学报, 2018, 38(9): 1798-1803. QIU M Y, HUANG T, HU J Y, WANG D M, GONG H B, WANG B H, SUN J L, LAI H X. Effects of polymorphisms of ESR and ITGB1 on reproductive performance of Yorkshire sows[J]. Chinese Journal of Veterinary Science, 2018, 38(9): 1798-1803. |
| [29] |
BRIDGER P S, HAUPT S, LEISER R, JOHNSON G A, BURGHARDT R C, TINNEBERG H R, PFARRER C. Integrin activation in bovine placentomes and in caruncular epithelial cells isolated from pregnant cows[J]. Biology of Reproduction, 2008, 79(2): 274-282. DOI:10.1095/biolreprod.108.067637 |
| [30] |
LAMY J, NOGUES P, COMBES-SOIA L, TSIKIS G, LABAS V, MERMILLOD P, DRUART X, SAINT-DIZIER M. Identification by proteomics of oviductal sperm-interacting proteins[J]. Reproduction, 2018, 155(5): 457-466. DOI:10.1530/REP-17-0712 |
| [31] |
ZACHUT M, SOOD P, LEVIN Y, MOALLEM U. Proteomic analysis of preovulatory follicular fluid reveals differentially abundant proteins in less fertile dairy cows[J]. Journal of Proteomics, 2016, 139: 122-129. DOI:10.1016/j.jprot.2016.03.027 |
| [32] |
VASCONCELOS G L, SARAIVA M V, COSTA J J, PASSOS M J, SILVA A W, ROSSI R O, PORTELA A M, DUARTE A B, MAGALHAES-PADILHA D M, CAMPELO C C, FIGUEIREDO J R, VAN DEN HURK R, SILVA J R. Effects of growth differentiation factor-9 and FSH on in vitro development, viability and mRNA expression in bovine preantral follicles[J]. Reproduction Fertility and Development, 2013, 25(8): 1194-1203. DOI:10.1071/RD12173 |
| [33] |
杜宏婷. FOXL2对鸡颗粒细胞胶原蛋白基因COL4A1的表达调控研究[D]. 武汉: 华中农业大学, 2020. DU H T. EFfect of FOXL2 on the expression of COL4A1 collagen gene in chicken granulosa cells[D]. Wuhan: Huazhong Agricultural University, 2020. DOI: 10.27158/d.cnki.ghznu.2020.000211. |
| [34] |
江学斌, 陈嘉蔚, 杨军, 胡位荣, 肖安吉, 卢志鹏, 楚品品, 张玲华. 猪PR39真核表达载体构建[J]. 广东农业科学, 2016, 43(3): 148-151. DOI:10.16768/j.issn.1004-874X.2016.03.029 JIANG X B, CHEN J W, YANG J, HU W R, XIAO A J, LU Z P, CHU P P, ZHANG L H. Construction of eukaryotic expression vector of porcine PR39[J]. Guangdong Agricultural Sciences, 2016, 43(3): 148-151. DOI:10.16768/j.issn.1004-874X.2016.03.029 |
| [35] |
张爱玲, 孙显月, 尹琪, 李加琪, 张豪. 猪羰基还原酶1(CBR1) 基因克隆及组织表达谱分析[J]. 广东农业科学, 2016, 43(8): 151-157. DOI:10.16768/j.issn.1004-874X.2016.08.025 ZHANG A L, SUN X Y, YIN Q, LI J Q, ZHANG H. Cloning and expression of porcine carbonyl reductase1 (CBR1) gene[J]. Guangdong Agricultural Sciences, 2016, 43(8): 151-157. DOI:10.16768/j.issn.1004-874X.2016.08.025 |
| [36] |
ATHANASIOS A, CHARALAMPOS V, VASILEIOS T, ASHRAF G M. Protein-protein interaction (PPI) network: Recent advances in drug discovery[J]. Current Drug Metabolism, 2017, 18(1): 5-10. DOI:10.2174/138920021801170119204832 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51