文章信息
基金项目
- 清远市2023年省科技创新战略专项(大专项+ 任务清单)项目(2023DZX012);科技创新战略专项资金(高水平农科院建设)(R2020PY-JC001);广东省农业科学院驻镇帮扶村农村科技特派员项目(KTP20210010);广东省畜禽疫病防治研究重点实验室项目(2023B1212060040)
作者简介
- 符德佳(1998—),男,在读硕士生,研究方向为中药和动物肠道菌群,E-mail:2635322868@qq.com.
通讯作者
- 彭新宇(1967—),男,硕士,研究员,研究方向为中药和动物肠道菌群,E-mail:996202250@qq.com.
文章历史
- 收稿日期:2023-11-30
2. 广东省农业科学院动物卫生研究所/广东省畜禽疫病防治研究重点实验室/农业农村部兽用药物与诊断技术广东科学观测实验站/广东省中兽药工程技术研究中心,广东 广州 510640;
3. 广州市江丰实业股份有限公司,广东 广州 510510;
4. 广州市江丰实业翁源有限公司,广东 韶关 512600;
5. 广东容大生物股份有限公司,广东 清远 511517
2. Institute of Animal Health, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory of Animal Disease Control/Guangdong Scientifi c Observation and Experiment Station of Veterinary Medicine and Diagnostic Technology, Ministry of Agriculture and Rural Affairs/Guangdong Engineering and Technology Research Center of Chinese Veterinary Medicine, Guangzhou 510640, China;
3. Guangzhou Jiangfeng Industrial Co., Ltd., Guangzhou 510510, China;
4. Guangzhou Jiangfeng Industrial Wengyuan Co., Ltd., Shaoguang 512600, China;
5. Guangdong Rongda Biological Co., Ltd., Qingyuan 511517, China
【研究意义】肺炎克雷伯氏菌(Klebsiella pneumoniae)是一种常见的革兰氏阴性杆菌,广泛存在于自然环境中,是感染人和动物的重要病原菌之一,可寄生于动物的鼻咽、肠胃等粘膜表面上,可引起尿路感染、菌血症和肺炎等感染性疾病,受到全球性关注 [1-2]。该细菌的耐药性问题严重危害公共卫生安全,不仅影响畜禽健康和生产性能,还可能通过食品链传播给人类,危害公共卫生 [3-4]。因此,研究肺炎克雷伯氏菌的耐药表型和耐药基因的携带情况具有重要意义。
【前人研究进展】肺炎克雷伯氏菌原本是一种常见的医院获得性感染源病原体,而近年来大量研究发现,肺炎克雷伯氏菌也存在于鸡、猪、牛、兔等动物中 [5-8]。目前,关于肺炎克雷伯氏菌的耐药性研究已取得一定成果。早期的研究主要集中在耐药基因的检测和分析上,已鉴定到多种与耐药性相关的基因,如β-内酰胺酶类、耐碳青霉烯类、氨基糖苷类、四环素类等耐药基因 [9-12]。董瑞等 [13]发现,猪源肺炎克雷伯氏菌株对阿莫西林和氟苯尼考等抗生素耐药尤为严重,且氨基糖苷类耐药基因检出率较高;黄攀等 [14]研究发现,鸡源肺炎克雷伯氏菌对氟苯尼考、四环素和氯霉素等药物耐药,通过基因型PCR检测确定该菌株携带CTX-M-27、SHV-26等β-内酰胺酶类耐药基因。菌株耐药性的出现使得临床上治疗肺炎克雷伯氏菌感染的可选药物明显减少,给临床治疗带来挑战。肺炎克雷伯氏菌的多重耐药不仅影响着畜禽业的发展,同时也威胁着人类的健康。因此,明确畜禽源肺炎克雷伯氏菌的耐药性和耐药基因,有利于有效治疗和预防相关疾病及进一步探讨肺炎克雷伯氏菌的耐药机制。
【本研究切入点】近年来,肺炎克雷伯氏菌耐药性研究大多集中在人源菌株上,对动物源菌株的研究相对较少。研究表明,鸡养殖场饮水系统易受到细菌污染,但关于养殖场鸡饮水中肺炎克雷伯氏菌的报道较少 [15-16]。【拟解决的关键问题】本研究拟从养殖场鸡饮水中分离、鉴定肺炎克雷伯氏菌,开展药敏试验检测其耐药性表型,并通过PCR检测常见重要耐药基因,及借助基因组测序分析预测分离菌株所携带的耐药基因。
1 材料与方法 1.1 试验材料1.1.1 样品来源 于2020—2022年,从广州市某公司花都肉鸡养殖场鸡饮用水供水系统的前端、中端和末端提取水样,每个样品2 000 mL,用无菌聚丙烯容器装样,4 ℃保存,运送到实验室。
1.1.2 药敏纸片 25种抗生素药敏纸片:阿莫西林、氨苄西林、利福平、恩诺沙星、环丙沙星、替米考星、磺胺甲噁唑、杆菌肽、阿米卡星、四环素、多西环素、氟苯尼考、甲硝唑、多粘菌素、链霉素、卡那霉素、克林霉素、新霉素、大观霉素、庆大霉素、红霉素、阿奇霉素、克拉霉素、氯霉素、林可霉素,购自杭州微生物试剂有限公司和英国Oxoid公司。
1.1.3 试验用试剂 LB固体培养基、麦康凯培养基和革兰氏染色试剂盒,购自环凯生物有限公司;DEPCH2O和2×EsTaqMasterMix,购自生工生物工程(上海)股份有限公司;微量微生物生化鉴定条和KOVACS氏试剂,购自海博生物公司;甲基红试剂,购自天津市天新精细有限公司;氯化铁(分析纯),购自天津市百世化工有限公司;细菌基因组DNA提取试剂盒,购自天根生化科技有限公司。
1.2 单菌株分离摇匀水样,取100 μL样品均匀涂布接种于麦康凯琼脂平板培养基,37 ℃恒温培养16~18 h;从麦康凯琼脂平板培养基上挑选生长状态良好的单菌落,再划线接种于麦康凯琼脂平板培养基,37 ℃培养箱内恒温培养16~18 h,无菌操作刮取单菌落培养物备用。
1.3 菌株鉴定1.3.1 16S rDNA序列鉴定 采用煮沸法提取细菌DNA模板。取少许单菌落培养物加入100 μL无核酸水中,水浴煮沸加热10 min,4 ℃、12 000 r/min离心10 min,取上清液为模板。
对细菌基因组16S rDNA片段进行PCR扩增,引物序列为F:5'-AGAGTTTGATCMTGGCTC AG-3',R:5'-GGTTACCTTGTTACGACTT-3',PCR反应体系(20 μL)包含1 μL模板,上、下游引物各1 μL、4.5 μL DEPC H2O和12.5 μL 2×Es Taq MasterMix。PCR反应程序:94 ℃预热2 min;94 ℃变性1 min、50 ℃退火1 min、72 ℃延伸1 min,共30个循环;72 ℃延伸10 min,最后保持在10 ℃。
琼脂糖凝胶电泳检测PCR产物,目的条带大小为1 415 bp。将阳性PCR扩增产物送往生工生物工程(上海)股份有限公司进行序列检测,在NCBI官网对各产物序列进行基因型序列比对及同源性分析,初步确定菌株物种。
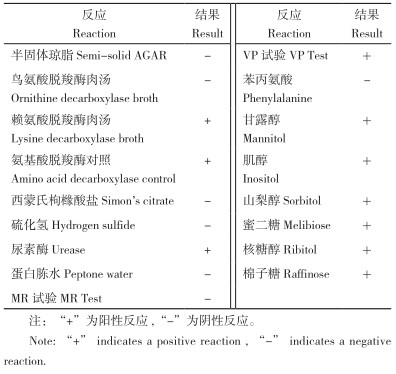
1.3.2 生化试验 按革兰氏染色试剂盒使用说明对分离纯化的细菌进行革兰氏染色并镜检。根据肠菌科细菌生化鉴定条使用说明书比对序列,对初步鉴定为肺炎克雷伯氏菌的分离菌株进行生化鉴定。
生化鉴定项目包括半固体琼脂、鸟氨酸脱羧酶肉汤、赖氨酸脱羧酶肉汤、氨基酸脱羧酶对照、西蒙氏枸橼酸盐、硫化氢、尿素酶、蛋白胨水、MR实验、VP试验、苯丙氨酸、甘露醇、肌醇、山梨醇、蜜二糖、核糖醇、棉子糖。
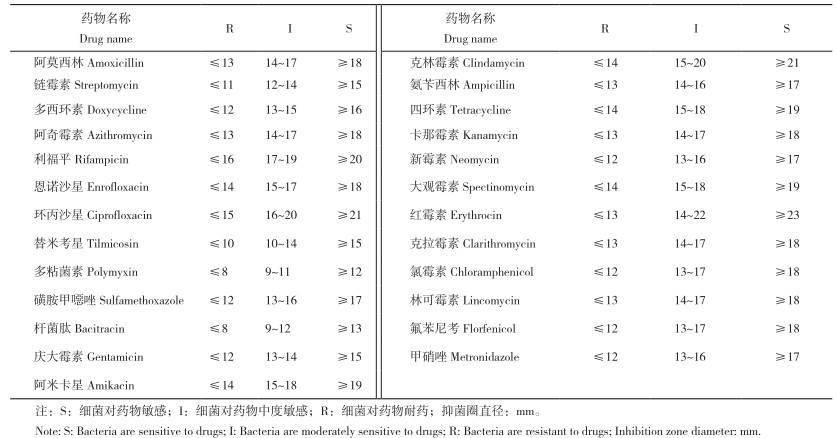
1.4 药敏试验采用纸片扩散法(K-B法),对分离的肺炎克雷伯氏菌菌株进行药敏试验,评定其耐药性。先将菌液浓度调整到接近0.5麦氏比浊度,再将其均匀涂布接种到LB平板上,接着在LB平板上均匀放置各抗菌药敏纸片,每种药敏纸片重复3次,37 ℃恒温培养24 h,测量各抑菌圈大小并记录。耐药性评价标准参考美国临床和实验室标准化协会CLSI 2022修订的《抗菌药物敏感性试验标准》。判断标准见表 1。
1.5 细菌耐药基因PCR检测
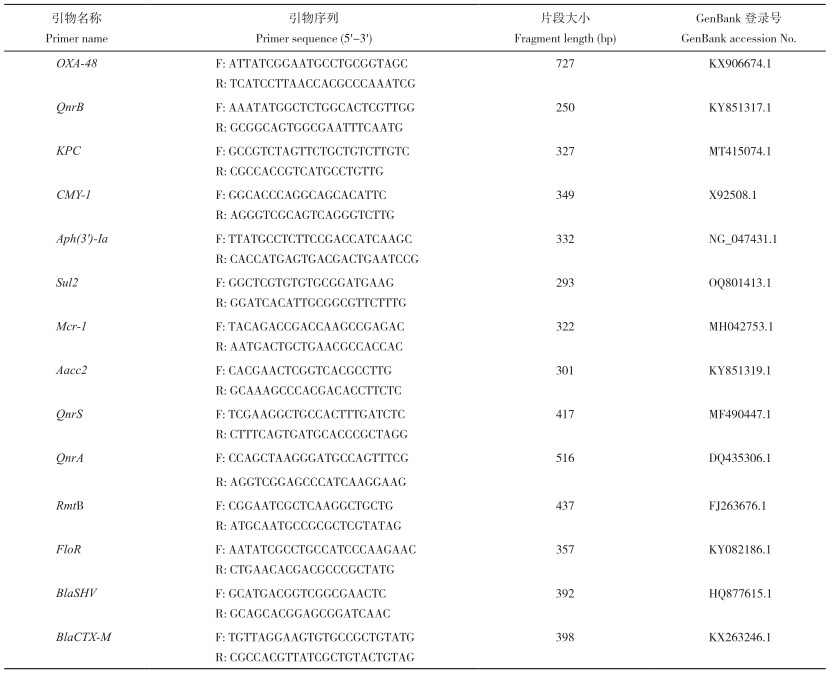
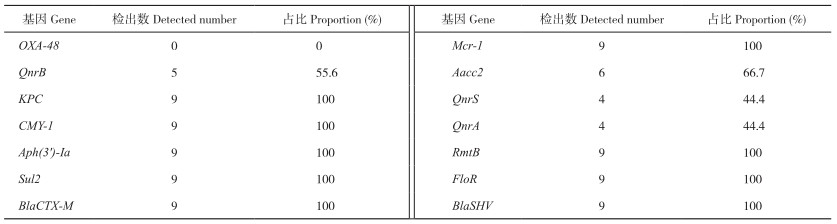
1.5.1 引物合成 选择广泛报道、高发生率的14种重要耐药基因,包括β-内酰胺酶类耐药基因BlaSHV、BlaCTX-M、CMY-1,碳青霉烯酶类耐药基因OXA-48、KPC,喹诺酮类耐药基因QnrB、QnrA、QnrS,氟苯尼考类耐药基因FloR,磺胺类耐药基因Sul2,氨基糖苷类耐药基因Aacc2、Aph(3')-Ia、RmtB和多粘菌素耐药基因Mcr-1 [17-20],在NCBI网站查找到相应耐药基因序列,并通过生工生物工程(上海)股份有限公司合成相应引物。各引物序列及预期目标片段大小见表 2。
1.5.2 PCR检测 采用煮沸法提取细菌DNA,方法见1.3.1。PCR反应体系(20 μL),包括1 μL模板,上、下游引物各1 μL,4.5 μL DEPC H2O和12.5 μL 2×Es Taq MasterMix。PCR反应程序:94 ℃预热2 min;94 ℃变性1 min、50 ℃退火1 min、72 ℃延伸1 min,共30个循环;72 ℃延伸10 min,最后保持在10 ℃。
用1% 琼脂糖凝胶电泳检测PCR产物,并送生工生物工程(上海)股份有限公司测序,比较测序结果与预期序列。
1.6 细菌的全基因组测序取4株菌株的单克隆培养液按细菌基因组DNA提取试剂盒进行基因组DNA提取。检测合格后,委托上海美吉生物医药科技股份有限公司进行测序,并在该公司云平台上分析测序数据。
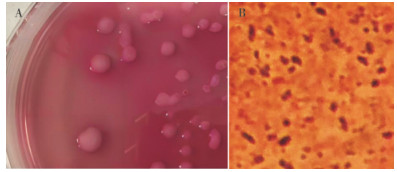
2 结果与分析 2.1 细菌分离鉴定2.1.1 菌株生长形态 从广州市某公司花都肉鸡养殖场鸡饮用水供水系统采集的水样中共分离到9株疑似肺炎克雷伯氏菌。分离菌株在麦康凯琼脂平板培养基表面上形成的菌落呈中间圆突起状、浅白色、半透明,边缘整齐光滑带有粉红色(图 1A)。革兰氏染色结果为红色(图 1B),可以判断该菌株为革兰氏阴性菌。
|
| 图 1 分离菌株的菌落形态(A)及革兰氏染色结果(B) Fig. 1 Colony morphology (A) and gram staining (B) of isolated strains |
2.1.2 16S rDNA鉴定 分离菌株16S rDNA PCR扩增产物的琼脂糖凝胶电泳结果见图 2,所有分离菌株均可扩增出1 445 bp左右的明亮条带。将PCR产物测序结果与肺炎克雷伯氏菌比对后发现,同源性达98.0%~99.9%,初步判断分离到的9株菌株均为肺炎克雷伯氏菌。

|
| M:2 000 bp DNA Marker; 1~9:分离菌株; 0:空白对照 M: 2 000 bp DNA Marker; 1-9: Isolated strains; 0 : Blank control 图 2 分离菌株16S rDNA基因扩增电泳结果 Fig. 2 Electrophonesis results of 16S rDNA gene amplification of isolated strains |
2.2 生化试验
分离菌株生化试验结果(表 3)表明,赖氨酸脱羧酶肉汤、氨基酸脱羧酶对照、尿素酶、VP试验、甘露醇、肌醇、山梨醇、蜜二糖、核糖醇、棉子糖表现为阳性;而半固体琼脂、鸟氨酸脱羧酶肉汤、西蒙氏枸橼酸盐、硫化氢、蛋白胨水、MR实验、苯丙氨酸表现为阴性。将该结果对比《伯杰细菌鉴定手册》 [21],可以判定菌株为肺炎克雷伯氏菌。
2.3 药敏试验结果
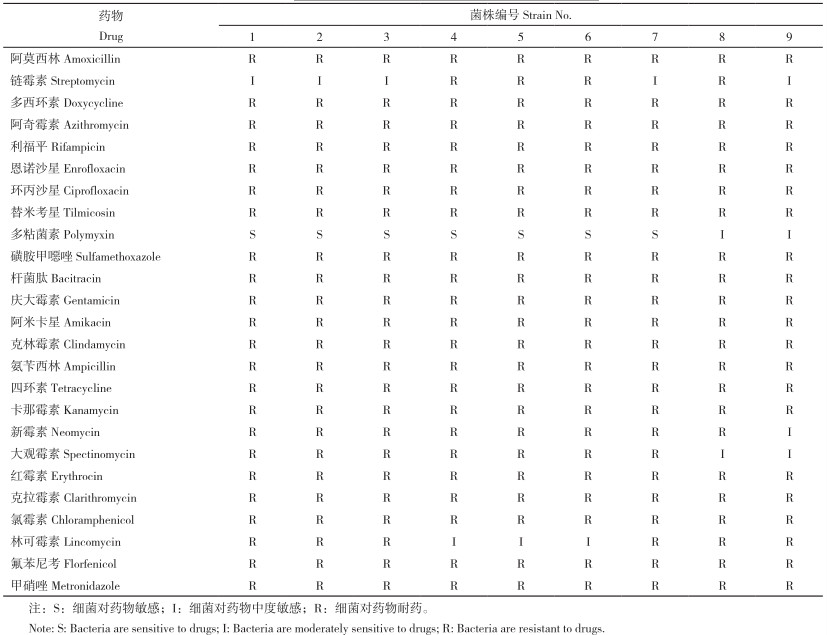
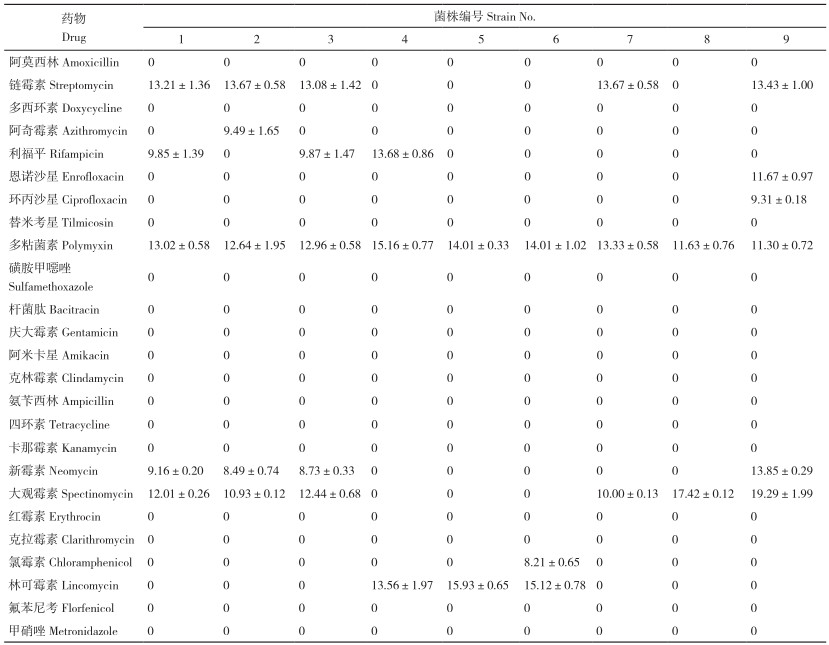
细菌药敏试验结果如表 4、表 5所示。由表 5可知,9株肺炎克雷伯氏菌分离菌株均对阿莫西林、多西环素、阿奇霉素、利福平、恩诺沙星、环丙沙星等20种药物表现为耐药;其中部分菌株对阿奇霉素、利福平、恩诺沙星、环丙沙星和氯霉素等5种药物表现出抑菌圈,但结果判定为耐药。
|
而部分菌株对链霉素、新霉素、大观霉素、林可霉素等4种药物表现出抑菌圈的结果判定为中度敏感;9株分离菌株均对多粘菌素表现为敏感。
2.4 耐药基因PCR检测结果由图 3可知,PCR共检测出13种耐药基因,分别是QnrB、KPC、CMY-1、Aph(3')-Ia、Sul2、Mcr-1、Aacc2、QnrS、QnrA、RmtB、FloR、BlaSHV和BlaCTX-M,总检出率为92.86%。各耐药基因的PCR产物序列与在NCBI网站上的肺炎克雷伯氏菌参考序列同源性在95.0%~99.9% 之间。
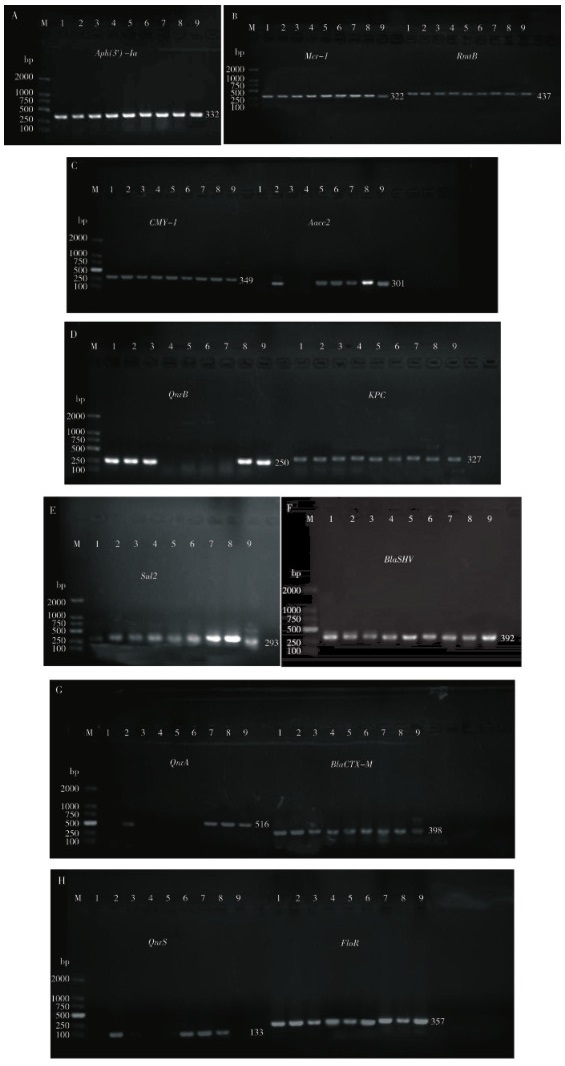
|
| M: 2000 bp DNA Marker 图 3 耐药基因PCR扩增结果 Fig. 3 Results of PCR amplification of drug resistance genes |
各耐药基因检出结果如表 6所示,KPC、CMY-1、Aph(3')-Ia、Sul2、Mcr-1、RmtB、FloR、BlaSHV和BlaCTX-M在分离菌株中的检出率为100%(9/9);Aacc2的检出率为66.7%(6/9);QnrB的检出率为55.6%(5/9);QnrS和QnrA的检出率为44.4%(4/9);所有菌株均未检测到OXA-48耐药基因。由此可知,这9株肺炎克雷伯氏菌分离菌株携带多种耐药基因,检测到的耐药基因数量在10~13个之间。
2.5 全基因组测序结果
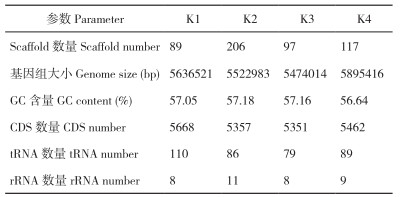
肺炎克雷伯氏菌K1菌株测序得到Scaffolds为89条,总长度5 636 521 bp,鸟嘌呤-胞嘧啶(G+C)含量为57.05%,编码区基因总数量为5 668个(表 7)。K2菌株测序得到Scaffolds为206条,总长度5 522 983 bp,鸟嘌呤-胞嘧啶(G+C)含量为57.18%,编码区基因总数量为5 357个。K3菌株测序得到Scaffolds为97条,总长度5 474 014 bp,鸟嘌呤-胞嘧啶(G+C)含量为57.16%,编码区基因总数量为5 351个。K4菌株测序得到Scaffolds为117条,总长度5 895 416 bp,鸟嘌呤-胞嘧啶(G+C)含量为56.64%,编码区基因总数量为5 462个。
GC含量是每个物种基因组的重要指标,不同的基因组具有不同的GC含量。4株肺炎克雷伯氏菌的GC_depth分布图(图 4)中多数的点集中分布在一个比较窄的范围内,表明样本不存在污染。
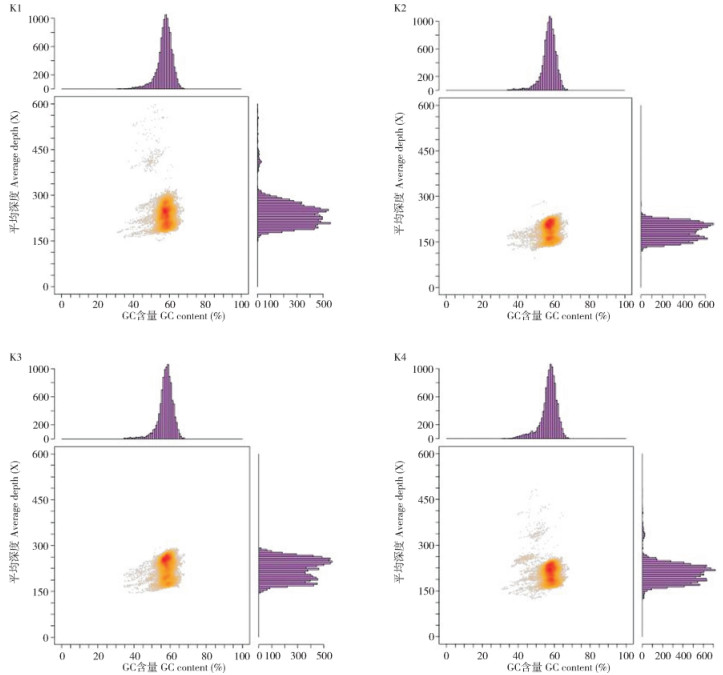
|
| 图 4 4株肺炎克雷伯氏菌GC_depth分布 Fig. 4 GC_depth distribution of 4 strains of Klebsiella pneumoniae |
对4株肺炎克雷伯氏菌进行全基因组测序和比较基因组学分析。通过CARD抗性基因数据库(The Comprehensive Antibiotic Research Database)对耐药基因预测,结果(图 5)显示,4株肺炎克雷伯氏菌耐药基因种类及数量相似,共检测到59种耐药基因,其中31种为针对抗生素的耐药基因,分别为四环素类抗生素、大环内酯类抗生素、氟喹诺酮类抗生素、青霉烷、头孢菌素、酚类抗生素、头霉素、碳青霉烯类、氨基糖苷类抗生素、氨基香豆素抗生素、肽类抗生素、利福霉素抗生素、培南、林可酰胺抗生素、单环内酰胺、甘氨酰环素、截短侧耳素、二氨基嘧啶类抗生素、链霉素抗生素、硝基咪唑类抗生素、糖肽类抗生素、噁唑烷酮类抗生素、核苷类抗生素、磷霉素、磺胺类抗生素、硝基呋喃类抗生素、莫匹罗星、双环霉素、苯扎氯铵、砜类抗生素、阿尔法霉素抗生素。此外,预测针对四环素类抗生素的耐药基因数量最多,其次是大环内酯类抗生素和氟喹诺酮类抗生素。
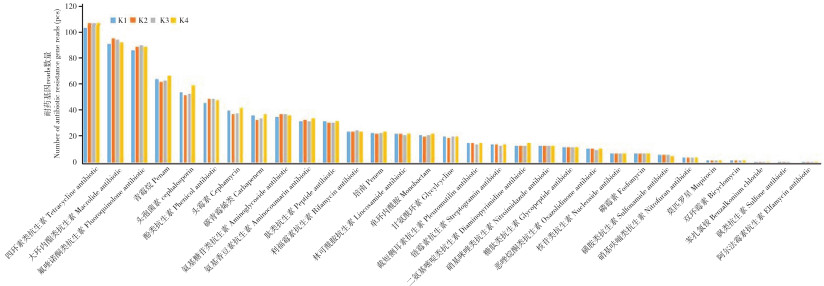
|
| 图 5 4株肺炎克雷伯氏菌针对不同抗生素耐药基因的reads数量统计 Fig. 5 Statistics of the number of antibiotic resistance gene reads of 4 strains of Klebsiella pneumoniae |
预测4个菌株携带耐药基因334~344个。将覆盖度和同源性均设定在98% 以上时,预测得到的耐药基因有23个,分别为FosA6、CRP、KpnF、KpnFG、KpnE、OqxA、OqsB、SHV-11、Tet(D)、CTX-M-27、QnrB65、DHA-1、Aph(4')-Ia、MsrE、QnrB2、Aph(3')-Ia、Sul1、CmlA6、FloR、AadA12、Ant(3'')-IIa、ArmA、Sul2(表 8)。
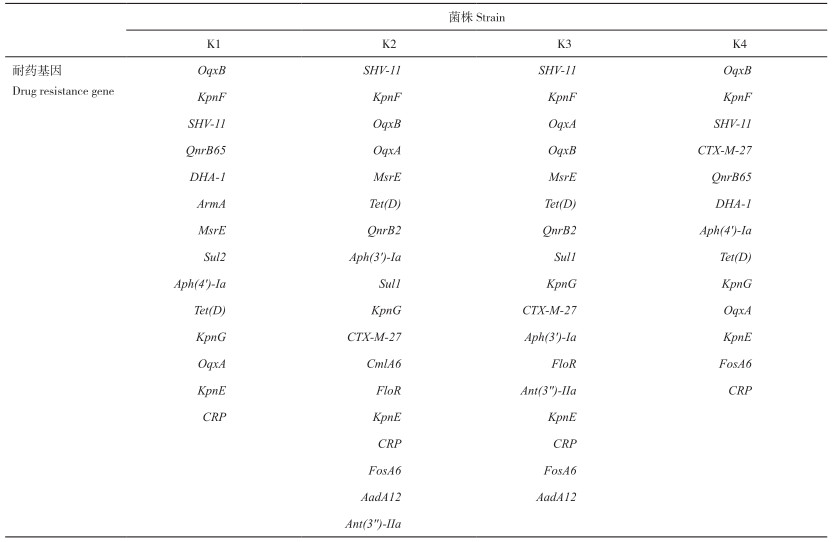
|
3 讨论
肺炎克雷伯氏菌是一种重要的条件致病菌,其高度耐药性引起了全球广泛关注。近年来,肺炎克雷伯氏菌感染在全球范围内广泛流行,对由其引发疾病的治疗也日益困难,对于如何预防及有效治疗肺炎克雷伯氏菌的感染已成为迫在眉睫的问题 [22-23]。
本研究结果显示,从鸡养殖场饮水系统中分离所得9株肺炎克雷伯氏菌均对阿莫西林、多西环素、四环素、氟苯尼考等20种药物耐药,对大观霉素、林可霉素等4种药物中度敏感,对多粘菌素表现为敏感。具体来看,所分离的9株肺炎克雷伯氏菌菌株均为多重耐药,对β-内酰胺酶类、碳青霉烯类、氨基糖苷类、四环素类等抗生素具有很强的耐药性。究其原因,其一可能是抗生素的滥用导致肺炎克雷伯氏菌的耐药机制发生了改变,即其关键靶位发生了改变;其二可能通过基因传递获得耐药基因,例如希瓦氏菌的碳青霉烯类耐药基因KPC和喹诺酮类耐药基因QnrB存在于质粒之中,通过质粒可将这些基因转移到肺炎克雷伯氏菌,使得肺炎克雷伯氏菌对该抗生素耐药 [24]。在实际养殖生产中,动物发生细菌性感染,人们常会根据经验使用抗菌药,如阿莫西林和庆大霉素等。而本试验中,肺炎克雷伯氏菌对阿莫西林和庆大霉素等常用抗生素表现出较强的耐药性,这可能导致疾病治疗效果不佳,同时增加药物残留风险。本试验所测试的25种抗菌药物中,多粘菌素和甲硝唑已经被禁止用于动物促生长方面,氯霉素已经被禁用于所有食品动物;阿莫西林、多西环素、恩诺沙星、环丙沙星、氟苯尼考、氨苄西林、磺胺甲噁唑、卡那霉素和替米考星已被禁止在鸡产蛋期使用 [25-27]。本研究中,从鸡养殖场饮水系统中分离得到的肺炎克雷伯氏菌对这些药物(尤其是已被禁用的药物)仍然表现出较强的耐药性。前人研究发现,鸡源中的沙门氏菌和大肠杆菌同样耐药严重,主要对四环素、阿莫西林、多西环素等药物表现出耐药,部分菌株对大观霉素、多粘菌素敏感 [28-32],这与该文肺炎克雷伯氏菌的药敏试验结果一致。综上,在养殖场发生细菌感染时,应进行药敏试验,选择敏感性高的药物,以提高治疗效果、降低养殖成本、减少药物残留风险。
一般来说,细菌的耐药基因与耐药表型是一致的,不过也经常有二者不一致的情形。本研究发现,9株肺炎克雷伯氏菌分离菌株均携带CMY-1、BlaSHV和BlaCTX-M等β-内酰胺酶类耐药基因和碳青霉烯类耐药基因KPC。具有这些基因的细菌可以产生水解抗生素β-内酰胺环的β-内酰胺酶 [33],从而使得β-内酰胺酶类抗生素失效。药敏试验结果显示,这9株菌对阿莫西林等β-内酰胺酶类药物表现出很强的耐药性,耐药表型和耐药基因一致。KPC基因于2001年首次从美国北卡罗来纳州分离的肺炎克雷伯氏菌中发现 [34],KPC耐药基因可以在细菌的质粒间进行移动传播。尽管本试验未测试肺炎克雷伯氏菌对亚胺培南、美罗培南、帕尼培南等碳青霉烯类药物的敏感性,但热那古·艾山等 [35]研究表明,试验菌株对上述药物的耐药率均为100%。本研究中,喹诺酮类耐药基因QnrB的检出率为55.6%,QnrS和QnrA耐药基因的检出率均为44.4%,且9种肺炎克雷伯氏菌分离株均对恩诺沙星、环丙沙星等喹诺酮类药物表现为耐药性,说明其耐药表型和耐药基因型密切相关,这与张召兴等 [36]的研究结果一致。已有研究指出,肺炎克雷伯氏菌对喹诺酮药物耐药的原因,一是拥有相应的喹诺酮类耐药基因,可以将喹诺酮药物排出细菌细胞之外;二是由于β-内酰胺酶类耐药基因BlaSHV的基因编码位点与喹诺酮类药物耐药基因的位点相似,故对β-内酰胺酶类耐药的细菌也对喹诺酮类药物耐药;再者由于抗生素的过度使用或者是接收了其他细菌的耐药质粒导致肺炎克雷伯氏菌对喹诺酮药物耐药 [36-38]。氨基糖苷类耐药基因Aph(3')-Ia、氟苯尼考类耐药基因FloR和磺胺类耐药基因Sul2在9株肺炎克雷伯氏菌的检出率均为100%,药敏试验结果显示,这些菌株对氨基糖苷类药物如庆大霉素、大观霉素和卡那霉素,以及氟苯尼考和磺胺类药物如磺胺甲噁唑等表现出耐药性,耐药表型和耐药基因高度一致。细菌的耐药性与耐药基因一致,表明细菌可能通过基因突变或水平基因转移等方式获得了耐药性,即细菌可以通过自身的遗传变异来对抗抗生素的作用,从而导致抗生素治疗失效。同时,说明细菌在面对抗生素压力时可以迅速适应和发展耐药性,这对医疗和公共卫生构成严重挑战。
此外,Mcr-1基因是对多粘菌素类药物耐药的基因,位于细菌质粒上,能够在多种细菌间传播 [39]。有学者认为,Mcr-1耐药基因的出现并不意味着它对该药物表现出耐药性,只是对低浓度的多粘菌素耐药,而对高浓度的多粘菌素敏感 [39]。郑常委 [28]研究发现,大肠杆菌Mcr-1阳性菌对多粘菌素敏感的比例为35%,阴性菌则为70%;吴植等 [29]研究发现,鸡源沙门氏菌对多粘菌素药物敏感,但同时也检测出Mcr-1基因。本试验中,9株肺炎克雷伯氏菌分离菌株均对多粘菌素敏感,而它们的Mcr-1耐药基因检出率为100%,表明携带Mcr-1耐药基因的菌株不一定对多粘菌素表现出耐药性,这与前述研究结果一致。同时,体现出防止耐药性传播工作的复杂性,检测耐药基因有利于防止多粘菌素的耐药质粒传播和及时阻止耐药菌的发展,暗示有必要严格控制和规范多粘菌素在畜禽养殖中的应用。
苯唑西林酶OXA-48是一种D类碳青霉烯类酶,可水解碳青霉烯类药物,从而对治疗重度细菌感染的抗生素(如亚胺培南,美罗培兰等)产生耐药性,其基因可在多种肠杆菌中快速传播,形成所谓的“超级细菌” [40]。OXA-48对碳青霉烯类药物的水解能力较弱,产OXA-48菌株常表现为对这些药物呈一定程度的敏感性 [41]。因此,即便此类菌株已广泛流行,但仍然难以被监测体系发现 [42]。据报道,携带OXA-48基因的肺炎克雷伯氏菌在欧美广泛流行,主要存在于土耳其、中东、北非和欧洲国家 [43]。近年来,在中国医院临床及动物上也发现产OXA-48肠杆菌,依然以肺炎克雷伯菌为主,不过主要呈散发流行 [41, 44]。姚佳彤 [45]研究发现,医院分离耐碳青霉烯类肺炎克雷伯氏菌和大肠埃希菌通过PCR检测,均未检测到OXA-48基因。在本研究中,9株肺炎克雷伯氏菌分离菌株PCR检测结果均未检测到OXA-48基因,与前述研究结果一致,表明OXA-48基因尚未在我国广泛流行。
4 结论本研究从鸡场水样中共分离到9株肺炎克雷伯氏菌。这些菌株具有多重耐药性,对20种抗生素耐药,包括阿莫西林、多西环素、环丙沙星等。部分分离菌株对链霉素、新霉素、大观霉素、林可霉素等4种药物表现出中度敏感,而所有分离菌株均对多粘菌素敏感。所有菌株均携带了多种耐药基因,如多粘菌素类耐药基因Mcr-1,β-内酰胺酶类耐药基因CMY-1、BlaSHV和BlaCTX-M,碳青霉烯类耐药基因KPC,喹诺酮类耐药基因QnrB、QnrS和QnrA,氨基糖苷类的耐药基因Aph(3')-Ia,磺胺类耐药基因Sul2和氟苯尼考类耐药基因FloR等。其中,多粘菌素类耐药基因与耐药表型不一致,其他耐药基因与其耐药表型为一致。此外,分离菌株均未检测到OXA-48基因。这些结果有助于家禽用药和公共卫生参考。未来需要进一步探索肺炎克雷伯氏菌耐药性的传播途径和机制,以采取有效的控制措施。
| [1] |
WU H, WANG M Y, LIU Y Q, WANG X H, WANG Y K, LU J X, XU H. Characterization of antimicrobial resistance in Klebsiella species isolated from chicken broilers[J]. International Journal of Food Microbiology, 2016, 232: 95-102. DOI:10.1016/j.ijfoodmicro.2016.06.001 |
| [2] |
WANG G Y, ZHAO G, CHAO X Y, XIE L X, WANG H J. The characteristic of virulence, biofilm and antibiotic resistance of Klebsiella pneumoniae[J]. International Journal of Environmental Research and Public Health, 2020, 17(17): 6278. DOI:10.3390/ijerph17176278 |
| [3] |
SMET A, MARTEL A, PERSOONS D, DEWULF J, HEYNDRICKX M, HERMAN L, HAESEBROUCK F, BUTAYE P. Broad-spectrum beta-lactamases among enterobacteriaceae of animal origin: Molecular aspects, mobility and impact on public health[J]. FEMS Microbiol Reviews, 2010, 34(3): 295-316. DOI:10.1111/j.1574-6976.2009.00198.x |
| [4] |
LI P, LIU D, ZHANG X Z, TUO H M, LEI C W, XIE X J, GU J, ZHANG A Y. Characterization of plasmid-mediated quinolone resistance in gram-negative bacterial strains from animals and humans in China[J]. Microbial Drug Resistance, 2019, 25(7): 1050-1056. DOI:10.1089/mdr.2018.0405 |
| [5] |
LI Y, WANG Q, XIAO X, LI R C, WANG Z Q. Emergence of bla(NDM-9)-bearing tigecycline-resistant Klebsiella aerogenes of chicken origin[J]. Journal of Global Antimicrobial Resistance, 2021, 26: 66-68. DOI:10.1016/j.jgar.2021.04.028 |
| [6] |
欧冠标, 周雨晴, 彭昊, 陆泽宁, 廖玉英, 马东鑫, 袁敬知, 葛强, 李珣, 王晓晔. 猪源肺炎克雷伯氏菌耐药性及毒力基因分析[J]. 中国动物检疫, 2021, 38(6): 102-111. DOI:10.3969/j.issn.1005-944X.2021.06.021 OU G B, ZHOU Y Q, PENG H, LU Z N, LIAO Y Y, MA D X, YUAN J Z, GE Q, LI X, WANG X Y. Analysis on drug resistance and virulence genes of porcine Klebsiella pneumoniae[J]. China Animal Heathl Inspection, 2021, 38(6): 102-111. DOI:10.3969/j.issn.1005-944X.2021.06.021 |
| [7] |
张保海, 罗正中, 罗梓丹, 骆巧, 沈留红, 姚学萍, 曹随忠. 致奶牛乳房炎肺炎克雷伯氏菌的分离鉴定及防治建议[J]. 中国奶牛, 2020(3): 33-36. DOI:10.19305/j.cnki.11-3009/s.2020.03.010 ZHANG B H, LUO Z Z, LUO Z D, LUO Q, SHEN L H, YAO X P, CAO S Z. Isolation, identification and prevention and control of Klebsiella pneumoniae in dairy cattle[J]. China Dairy Cattle, 2020(3): 33-36. DOI:10.19305/j.cnki.11-3009/s.2020.03.010 |
| [8] |
孙雪岩, 刘玉梅, 张天琪, 孟冰冰, 张自强. 兔源性肺炎克雷伯氏菌的研究现状及进展[J]. 中国养兔杂志, 2021(3): 24-29. SUN X Y, LIU Y M, ZHANG T Q, MENG B B, ZHANG Z Q. Research satus and progress of rabbit-derived Klebsiella pneumoniae[J]. Chinese Journal of Rabbit Farming, 2021(3): 24-29. |
| [9] |
SHEN X H, YIN L, MA H T, PAN X C, ZHANG D J, ZHAO R H, DAI Y, HOU H Y, HU X M. Comprehensive genomic analysis and characterization of a new ST 174 type Klebsiella variicola strain isolated from chicken embryos[J]. Infection, Genetics and Evolution, 2021, 90: 104768. DOI:10.1016/j.meegid.2021.104768 |
| [10] |
JIN X, CHEN Q, SHEN F, JIANG Y, WU X Q, HUA X T, FU Y, YU Y S. Resistance evolution of hypervirulent carbapenem-resistant Klebsiella pneumoniae ST11 during treatment with tigecycline and polymyxin[J]. Emerging Microbes & Infections, 2021, 10(1): 1129-1136. DOI:10.1080/22221751.2021.1937327 |
| [11] |
KARAISKOS I, GALANI I, PAPOUTSAKI V, GALANI L, GIAMARELLOU H. Carbapenemase producing Klebsiella pneumoniae: Implication on future therapeutic strategies[J]. Expert Review of Anti-infective Therapy, 2022, 20(1): 53-69. DOI:10.1080/14787210.2021.1935237 |
| [12] |
LYU L C, WAN M, WANG C Z, GAO X, YANG Q W, PARTRIDGE S R, WANG Y, ZONG Z Y, DOI Y H, SHEN J Z, JIA P Y, SONG Q H, ZHANG Q H, YANG J, HUANG X H, WANG M G, LIU J H. Emergence of a plasmid-encoded resistance-nodulation-division efflux pump conferring resistance to multiple drugs, including tigecycline, in Klebsiella pneumoniae[J]. mBio, 2020, 11(2): e2919-e2930. DOI:10.1128/mBio.02930-19 |
| [13] |
董瑞, 孙晓. 沧州地区猪源肺炎克雷伯菌分离菌株、耐药性及耐药基因检测[J]. 中国兽医杂志, 2020, 56(4): 76-79. DONG R, SUN X. Isolation, drug resistance and drug resistance gene detection of Klebsiella pneumoniae from pigs in Cangzhou area[J]. Chinese Journal of Veterinary Medicine, 2020, 56(4): 76-79. |
| [14] |
黄攀, 于惠迪, 金文杰, 秦爱建. 鸡源产ESBLs肺炎克雷伯菌的分离鉴定及其耐药性分析[J]. 中国家禽, 2018, 40(18): 57-59. DOI:10.16372/j.issn.1004-6364.2018.18.012 HUANG P, YU H D, JIN W J, QIN A J. Isolation, identification and drug resistance analysis of Klebsiella pneumoniae producing ESBLs from chickens[J]. China Poultry, 2018, 40(18): 57-59. DOI:10.16372/j.issn.1004-6364.2018.18.012 |
| [15] |
万意, 季志远, 马瑞钰, 杜强, 杨荣斌, 詹凯. 冬春季层叠式蛋鸡舍乳头式饮水系统细菌含量检测及其对生产性能的影响[J]. 中国农业大学学报, 2021, 26(5): 72-82. DOI:10.11841/j.issn.1007-4333.2021.05.08 WAN Y, JI Z Y, MA R Y, DU Q, YANG R B, ZHAN K. Detection of bacterial content in the nipple watering system of layer house with cascading cages in winter and spring and its influence on the production performance[J]. Journal of China Agricultural University, 2021, 26(5): 72-82. DOI:10.11841/j.issn.1007-4333.2021.05.08 |
| [16] |
朱志梅. 养鸡饮水系统生物膜形成过程及预防措施[J]. 北方牧业, 2023(1): 26. ZHU Z M. Biofilm formation process and preventive measures of chicken drinking water system[J]. Northern animal husbandry, 2023(1): 26. |
| [17] |
张聪, 肖亦辰, 陈怀君, 袁敬知, 韦德源, 王晓晔. 伴侣动物源肺炎克雷伯氏菌的分离鉴定及毒力和耐药性分析[J]. 中国畜牧兽医, 2020, 47(5): 1583-1592. DOI:10.16431/j.cnki.1671-7236.2020.05.033 ZHANG C, XIAO Y C, CHEN H J, YUAN J Z, WEI D Y, WANG X Y. Isolation identification and analysis of virulence and drug resistance of Klebsiella pneumoniae from companion animals[J]. China Animal Husbanry & Veterinry Medicine, 2020, 47(5): 1583-1592. DOI:10.16431/j.cnki.1671-7236.2020.05.033 |
| [18] |
隋明, 王静霞, 唐贤华, 张彩, 舒学香, 叶绍勇. 四川地区牦牛源肺炎克雷伯氏菌的分离鉴定及其耐药性分析[J]. 中国畜牧兽医, 2019, 46(6): 1816-1824. DOI:10.16431/j.cnki.1671-7236.2019.06.030 SUI M, WANG J X, TANG X H, ZHANG C, SHU X X, YE S Y. Isolation identification and drug resistance analysis of Klebsiella pneumoniae from yak in Sichuan[J]. China Animal Husbanry & Veterinry Medicine, 2019, 46(6): 1816-1824. DOI:10.16431/j.cnki.1671-7236.2019.06.030 |
| [19] |
ROBATJAZI S, NIKKHAHI F, NIAZADEH M, MARASHI S M A, PEYMANI A, JAVADI A, KASHANI A H. Phenotypic identification and genotypic characterization of plasmid-mediated AmpC beta-Lactamase-producing Escherichia coli and Klebsiella pneumoniae isolates in Iran[J]. Current Microbiology, 2021, 78(6): 2317-2323. DOI:10.1007/s00284-021-02479-9 |
| [20] |
RODRIGUEZ O L, SOUSA A, PEREZ-RODRIGUEZ M T, MARTINEZ-LAMAS L, SUAREZ R L, MARTINEZ C T, PINO C P, VIDAL F V, PERZ-LANDEIRO A, CASAL M C. Mortality-related factors in patients with OXA-48 carbapenemase-producing Klebsiella pneumoniae bacteremia[J]. Medicine (Baltimore), 2021, 100(14): e24880. DOI:10.1097/MD.0000000000024880 |
| [21] |
RUCHANAN R E, G IBBES N E. Bergey's manual of systematic bacteriology: Volume eight: Gram-negative facultative anaerobic bacillus[M]. Beijing: Science Press, 1984.
|
| [22] |
刘卫国. 肺炎克雷伯菌临床分布及耐药性分析[J]. 实用中医药杂志, 2011, 27(3): 192-193. LIU W G. Clinical distribution and drug resistance analysis of Klebsiella pneumoniae[J]. Journal of Practical Traditional Chinese Medicine, 2011, 27(3): 192-193. |
| [23] |
HOLT K E, WERTHEIM H, ZADOKS R N, BAKER S, WHITEHOUSE C A, DANCE D, JENNEY A, CONNOR T R, HSU L Y, SEVERIN J, BRISSE S, CAO H, WILKSCH J, GORRIE C, SCHULTZ M B, EDWARDS D J, NGUYEN K V, NGUYEN T V, DAO T T, MENSINK M, MINH V L, NHU N T, SCHULTSZ C, KUNTAMAN K, NEWTON P N, MOORE C E, STRUGNELL R A, THOMSON N R. Genomic analysis of diversity, population structure, virulence, and antimicrobial resistance in Klebsiella pneumoniae, an urgent threat to public health[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(27): E3574-E3581. DOI:10.1073/pnas.1501049112 |
| [24] |
MARTINEZ-MARTINEZ L, PASCUAL A, JACOBY G A. Quinolone resistance from a transferable plasmid[J]. The Lancet, 1998, 351(9105): 797-799. DOI:10.1016/S0140-6736(97)07322-4 |
| [25] |
中华人民共和国农业部公告第2428号[EB/OL]. http://www.moa.gov.cn/govpublic/SYJ/201608/t20160801_5224428.html. Announcement No. 2428, Ministry of Agriculture of the People's Republic of China[EB/OL]. http://www.moa.gov.cn/govpublic/SYJ/201608/t20160801_5224428.html. |
| [26] |
农业农村部权威公布的禁用兽药目录汇总[EB/OL]. http://www.scyanbian.gov.cn/snfw/scxx/4214266.shtml. The list of prohibited veterinary drugs published by the authority of the Ministry of Agriculture and Rural Affairs[EB/OL]. http://www.scyanbian.gov.cn/snfw/scxx/4214266.shtml. |
| [27] |
食品安全国家标准食品中兽药最大残留限量(GB 31650-2019). 农业农村部, 国家卫生健康委员会, 国家市场监管理总局[S]. 北京: 中国农业出版社. 2019. National food safety standard-maximum residue limits for veterinary drugs in foods. Ministry of Agriculture and Rural Affairs, National Health Commission, State Administration for Market Regulation (GB 31650-2019)[S]. Beijing: China Agriculture Publishing House. 2019. |
| [28] |
郑常委. 鲁西南地区畜禽大肠杆菌mcr-1耐药基因携带情况调查[D]. 泰安: 山东农业大学, 2019. ZHENG C W. Prevalence of mcr-1 resistance genes in Escherichia coli from livestock and poultry in southwest of Shandong Province[D]. Taian: Shandong Agricultural University, 2019. |
| [29] |
吴植, 张聪, 袁慧莎, 董洪燕, 戴利霞, 贾鲁起, 吴双, 朱善元. 黄鸡源沙门菌分离鉴定及毒力和耐药性分析[J]. 中国畜牧兽医, 2023, 50(4): 1695-1706. DOI:10.16431/j.cnki.1671-7236.2023.04.040 WU Z, ZHANG C, YUAN H S, DONG H Y, DAI L X, JIA L Q, WU S, ZHU S Y. Isolation, identification, virulence and drug resistance of Salmonella from yellow chicken[J]. China Animal Husbanry & Veterinry Medicine, 2023, 50(4): 1695-1706. DOI:10.16431/j.cnki.1671-7236.2023.04.040 |
| [30] |
杨永亚, 刘志欢, 宋雪艳, 谢翠萍, 刘洋, 邢家宝, 陈环环, 刘建华. 携带MCR-1鸡源大肠杆菌耐药性及传播特性的初步分析[J]. 中国预防兽医学报, 2021, 43(7): 706-710. DOI:10.3969/j.issn.1008-0589.202009021 YANG Y Y, LIU Z H, SONG X Y, XIE C P, LIU Y, XING J B, CHEN H H, LIU J H. Preliminary study on the drug resistance and transmission characteristics of chicken-derived Escherichia coli carrying MCR-1[J]. Chinese Journal of Preventive Veterinary Medicine, 2021, 43(7): 706-710. DOI:10.3969/j.issn.1008-0589.202009021 |
| [31] |
王迪轩, 高东阳, 张天姿, 刘维, 周祖涛, 徐晓娟, 蔡旭旺. 华中地区鸡源沙门菌分离鉴定及耐药性分析[J]. 中国畜牧兽医, 2019, 46(3): 931-939. DOI:10.16431/j.cnki.1671-7236.2019.03.035 WANG D X, GAO D Y, ZHANG T Z, LIU W, ZHOU Z T, XU X J, CAI X W. Isolation identification and drug resistance analysis of Avian Salmoella in central China[J]. China Animal Husbanry & Veterinry Medicine, 2019, 46(3): 931-939. DOI:10.16431/j.cnki.1671-7236.2019.03.035 |
| [32] |
林居纯, 岳飞利, 李杰, 魏峰, 金山, 曹光秀. 鸡源大肠杆菌对氨基糖苷类抗生素耐药性检测[J]. 广东农业科学, 2012, 39(4): 90-91. LIN J C, YUE F L, LI J, WEI F, JIN S, CAO G X. Detection of aminoglycoside antibiotic resistance in chicken E. coli[J]. Guangdong Agricultural Sciences, 2012, 39(4): 90-91. |
| [33] |
翟俊斌, 曹小利, 程莉, 周万青, 张之烽, 沈瀚. 肺炎克雷伯菌的敏感性及其质粒介导的耐药基因的分布特点分析[J]. 临床输血与检验, 2020, 22(4): 425-429. DOI:10.3969/j.issn.1671-2587.2020.04.021 ZHAI J B, CAO X L, CHENG L, ZHOU W Q, ZHANG Z F, SHEN H. Sensitivity of Klebsiella pneumoniae to antibiotics and its genetic distribution of plasmid mediated resistance to quinilone[J]. Journal of Clinical Transfusion and Laboratory Medicine, 2020, 22(4): 425-429. DOI:10.3969/j.issn.1671-2587.2020.04.021 |
| [34] |
YIGIT H, QUEENAN A M, ANDERSON G J, DOMENECH- SANCHEZ A, BIDDLE J W, STEWARD C D, ALBERTI S, BUSH K, TENOVER F C. Novel carbapenem-hydrolyzing beta-lactamase, KPC-1, from a carbapenem-resistant strain of Klebsiella pneumoniae[J]. Antimicrob Agents Chemother, 2001, 45(4): 1151-1161. DOI:10.1128/AAC.45.4.1151-1161.2001 |
| [35] |
热那古·艾山, 帕丽扎提·阿布都依木, 李娟, 季萍. 产KPC型碳青霉烯酶细菌的耐药基因研究[J]. 中华医院感染学杂志, 2016, 26(10): 2169-2171. DOI:10.11816/CN.NI.2016-160605 AISHAN R N G, ABUDUYIMU P L Z T, LI J, JI P. Study on drug resistance genes in KPC carbapenemase-producing bacteria[J]. Chinese Journal of Nosocomiology, 2016, 26(10): 2169-2171. DOI:10.11816/CN.NI.2016-160605 |
| [36] |
张召兴, 刘少杰, 苏硕青, 周琦, 贾青辉, 张艳英, 史秋梅. 貉源致病性肺炎克雷伯氏菌对喹诺酮类药物耐药表型及耐药基因检测[J]. 野生动物学报, 2019, 40(4): 906-911. DOI:10.19711/j.cnki.issn2310-1490.2019.04.013 ZHANG Z X, LIU S J, SU S Q, ZHOU Q, JIA Q H, ZHANG Y Y, SHI Q M. Detection of quinolone resistance phenotype and the gene resistant to Klebsiella pneumoniae from raccoon dog[J]. Chinese Journal of Wildlife, 2019, 40(4): 906-911. DOI:10.19711/j.cnki.issn2310-1490.2019.04.013 |
| [37] |
蒙正群, 冷依伊, 任梅渗, 刘亚东, 王印, 姚学萍, 杨泽晓. 一株牛源肺炎克雷伯氏菌的分离鉴定与耐药基因型检测[J]. 浙江农业学报, 2017, 29(4): 534-541. DOI:10.3969/j.issn.1004-1524.2017.04.03 MENG Z Q, LENG Y Y, REN M S, LIU Y D, WANG Y, YAO X P, YANG Z X. Isolation, identification and drug resistance detection of a calf Klebsiella pneumoniae strain[J]. Acta Agriculturae Zhejiangensis, 2017, 29(4): 534-541. DOI:10.3969/j.issn.1004-1524.2017.04.03 |
| [38] |
李淑梅, 郝海玲, 宁红梅, 齐永华. 猪源沙门氏菌氟喹诺酮耐药基因突变的SSCP分析[J]. 广东农业科学, 2012, 39(20): 139-141. DOI:10.16768/j.issn.1004-874X.2012.20.003 LI S M, HAO H L, NING H M, QI Y H. SSCP analysis of fluoroquinolone resistance gene mutation in porcine Salmonella[J]. Guangdong Agricultural Sciences, 2012, 39(20): 139-141. DOI:10.16768/j.issn.1004-874X.2012.20.003 |
| [39] |
LIU Y Y, WANG Y, WALSH T R, YI L X, ZHANG R, SPENCER J, DOI Y H, TIAN G B, DONG B L, HUANG X H, YU L F, GU D X, REN H W, CHEN X J, LYU L C, HE D D, ZHOU H W, LIANG Z S, LIU J H, SHEN J Z. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study[J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168. DOI:10.1016/S1473-3099(15)00424-7 |
| [40] |
PITOUT J D D, PEIRANO G, KOCK M M, STRYDOM K A, MATSUMURA Y. The global ascendency of OXA-48-type carbapenemases[J]. Clinical Microbiology Reviews, 2019, 33(1): e00102-19. DOI:10.1128/CMR.00102-19 |
| [41] |
GUO L, AN J N, MA Y N, YE L Y, LUO Y P, TAO C M, YANG J Y. Nosocomial outbreak of OXA-48-producing Klebsiella pneumoniae in a chinese hospital: Clonal transmission of ST147 and ST383[J]. PloS One, 2016, 11(8): e0160754. DOI:10.1371/journal.pone.0160754 |
| [42] |
BOYD S E, HOLMES A, PECK R, LIVERMORE D M, HOPE W. OXA-48-like β-lactamases: Global epidemiology, treatment options, and development pipeline[J]. Antimicrobial Agents and Chemotherapy, 2022, 66(8): e0021622-e0021622. DOI:10.1128/AAC.00216-22 |
| [43] |
PITOUT J D D, NORDMANN P, POIREL L. Carbapenemase-producing Klebsiella pneumoniae, a key pathogen set for global nosocomial dominance[J]. Antimicrobial Agents and Chemotherapy, 2015, 59(10): 5873-5884. DOI:10.1128/AAC.01019-15 |
| [44] |
ZHUANG Z L, LYU L C, LU J X, LIN J H, LIU J H. Emergence of Klebsiella pneumoniae and Enterobacter cloacae producing OXA-48 carbapenemases from retail meats in China, 2018[J]. Journal of Antimicrobial Chemotherapy, 2019, 74(12): 3632-3634. DOI:10.1093/jac/dkz394 |
| [45] |
姚佳彤. 吉林省某医院肺炎克雷伯菌和大肠埃希菌对碳青霉烯类药物的耐药情况分析[D]. 长春: 吉林大学, 2022. DOI: 10.27162/d.cnki.gjlin.2022.006296. YAO J T. Analysis of carbapenem resistance of Klebsiella pneumoniae and Escherichia coli in a hospital in Jilin province[D]. Changchun: Jilin University, 2022. DOI: 10.27162/d.cnki.gjlin.2022.006296. |
 2024, Vol. 51
2024, Vol. 51