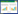文章信息
基金项目
- 国家重点研发计划项目(2023YFD2300802-1);广东省乡村振兴战略专项种业振兴行动项目(2023-NPY-00-001);国家香蕉产业技术体系专项(CARS-31-01)
作者简介
-
盛鸥,博士,广东省农业科学院果树研究所研究员,从事香蕉种质资源评价与利用研究,负责农业农村部广州香蕉种质资源圃的管理工作,是国家香蕉产业技术体系种质资源收集和评价岗位科学家团队核心骨干、广东省香蕉菠萝产业创新团队香蕉种质资源评价和品种培育岗位专家。主持国家自然科学基金、国家重点研发计划子课题、Bioverisity International等科研项目10余项。荣获广东省农业技术推广奖二等奖(排名第一)、广东省科技进步二等奖(排名第三)。作为主要执笔人之一制定国家农业行业标准3项;作为主要选育者之一育成中蕉8号、美食蕉1号等香蕉新品种12个;以第一作者或通信作者在《Plant Communications》《Food Chemistry》《Plant Disease》等期刊发表学术论文20多篇;担任《Diversity》《Frontiers in Sustainable Food Systems》等期刊编辑,是Frontiers、BMC等期刊社多个杂志的审稿人。获评国际园艺学会“ Miklos Faust International Travel Award for Young Pomologists”、广州市珠江科技新星、广东省农业科学院“金颖之星”和青年研究员,入选广东省农业项目专家库和广州市科技专家库。担任本期“香蕉遗传改良与栽培生理”专栏的客座主编。
盛鸥(1982—),男,博士,研究员,研究方向为香蕉种质资源评价与遗传改良,E-mail:shengou6@126.com.
通讯作者
- 易干军(1966—),男,博士,研究员,研究方向为香蕉遗传改良,E-mail:yiganjun@vip.163.com.
文章历史
- 收稿日期:2024-01-22
2. 中国科学院华南植物园/广东省应用植物学重点实验室,广东 广州 510650
2. South China Botanical Garden, Chinese Academy of Sciences/Guangdong Key Laboratory of Applied Botany, Guangzhou 510650, China
香蕉(Musa spp.)是重要的热带水果之一,全世界有130多个国家和地区种植香蕉,主要分布在南北回归线之间(南纬30°至北纬30°)[1]。印度、中国、菲律宾、哥伦比亚、印度尼西亚、厄瓜多尔和巴西是全球主要的香蕉生产大国,其中菲律宾和厄瓜多尔为全球主要的香蕉出口国[2-3]。FAO数据显示,2021年世界香蕉的总收获面积达1 212.92万hm2,总产量达17 030.02万t[4]。香蕉的栽培品种可简单分为鲜食蕉和主食蕉,其中鲜食蕉约占总产量的70%(0.97亿t),最常食用的品种为香牙蕉(Cavendish),其占鲜食蕉产量的82.5%,占全世界香蕉总产量的57%(约0.80亿t)[3]。主食蕉(如Plantain、东非高原蕉)的产量占世界香蕉总产量的25%,其需要烹煮或加工才能食用,是热带贫困地区4亿多人口的主要食物来源、营养来源和经济来源,被认为是仅次于水稻、小麦和玉米的第四大粮食作物[2-3]。因此,香蕉又是关系到世界粮食安全的重要作物之一[2-3]。
香蕉种质资源的鉴定和评价是香蕉遗传改良的基础。研究栽培香蕉品种的起源和演化,对加快香蕉育种进程具有重要的理论指导意义。近年来香蕉种质资源的分子系统学研究取得了重要进展,栽培品种的起源和演化过程不断被揭示,虽然还未完全清晰,但已经为香蕉育种工作提供了重要信息。本文综述了近年来香蕉品种资源的分类、栽培品种的起源和演化等方面的研究进展,以期为挖掘对香蕉育种有重要价值的优异种质和基因资源、指导香蕉育种提供借鉴。
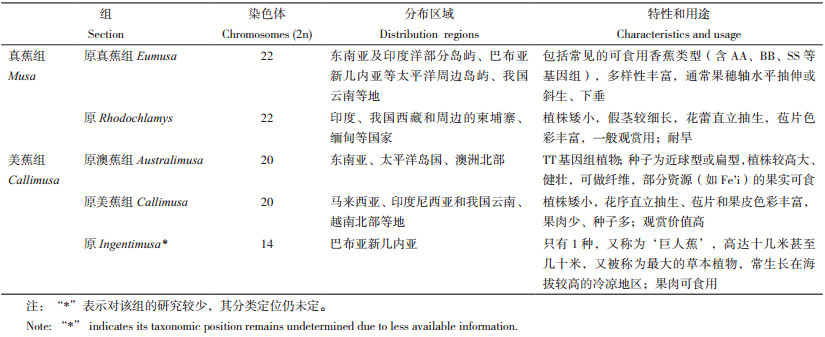
1 香蕉品种资源的分类 1.1 野生种质分类香蕉是芭蕉科(Musaceae)芭蕉属(Musa)的单子叶、多年生、大型草本植物[5]。芭蕉科除芭蕉属外还有象腿蕉属(Ensete)和地涌金莲属(Musella)[5]。芭蕉属内植物有60~70个种,至今还在通过分类评价和野外发现等进行修订和补充新物种[6-7]。基于形态学特征和染色体数目,芭蕉属内植物通常被分为5组,分别为Eumusa(真蕉组,2n = 22)、Rhodochlamys(2n = 22)、Australimusa(澳蕉组,又被称为南蕉组,2n = 20)、Callimusa(美蕉组,2n = 20)和Ingentimusa(2n = 14)[8-9]。基于分子标记的结果,香蕉分类学家Häkkinen[10]于2013年将以上5组植物又进行了归类,芭蕉属只分为真蕉组(Musa)和美蕉组(Callimusa),新的真蕉组涵盖原真蕉组Eumusa和Rhodochlamys的全部种,新的美蕉组则包括剩余3组的全部种。芭蕉属内分组和组内资源的特征和区域分布详见表 1和图 1。
象腿蕉属又称为衣蕉属,属内有7~10种植物,大部分用于园林景观用途[9, 12]。我国云南地区分布有象腿蕉属内野生种E. glaucum,具有耐旱、耐贫瘠等性状,假茎粗壮,果肉可食,内有大粒种子[12-13]。象腿蕉属中最重要的经济种为E. ventricosum,是非洲埃塞俄比亚2 000万人口的主粮作物之一[14-15],其假茎和球茎部位提取的淀粉可制成食物(当地称为Koch、Bulla和Amicho等)[16]。E. ventricosum在埃塞俄比亚有多个地方品种,被广泛栽培[17-18]。
地涌金莲属内植物仅有1个种Musella lasiocarpa,为我国特有芭蕉科种质,在国外又被称为Golden lotus banana或Chinese yellow banana[19]。它是佛教中的“五树六花”之一,民间俗称为千瓣莲花、千叶佛莲、宝兰花等[12]。现主要分布在我国云南、贵州、四川及越南邻近云南的部分地区,一般生长在海拔1 450~2 500 m,丛生,花期长,具有较高的观赏价值[20-21]。我国云南的科研单位从中选育出观赏价值较高的品种[21],也有学者对其进行化学成分及药用价值研究[22]。
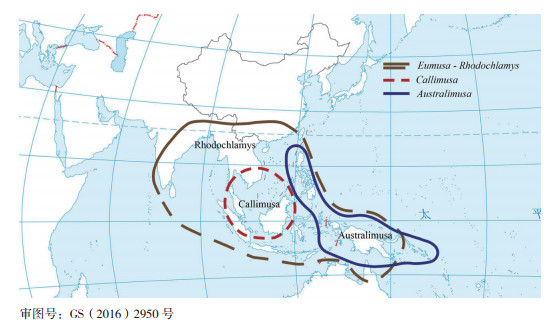
1.2 主要栽培类型全世界常见的香蕉栽培品种有200~300个,一般只含有A或B基因组,如二倍体(AA、AB)、三倍体(AAA、AAB、ABB)和四倍体(AAAA、AAAB),其中三倍体AAA和AAB品种数量较多[1, 12, 23]。除A和B染色体基因组外,少数品种中还存在S基因组(来源于Musa schizocarpa)和T基因组(来源于美蕉组Callimusa),因此还存在AS、AAT等基因组类型[24-25]。然而,栽培香蕉品种的分类比较混乱,同物异名和同名异物现象比较突出。例如,同样是Lady finger,在澳大利亚是AAB类型[26],而在我国被直译为“夫人指”、认为是AA类型[27]。Plantain在我国有多个译名,如“大蕉”“饭蕉”“菜蕉”等,不仅不能准确反映Plantain的特征特性,还会与基因组类型为ABB的我国特有地方品种群“大蕉”混淆[12]。
因此,近年来各个国家逐渐统一了分类方法,以“栽培类型+ 基因组”的双名法进行命名。栽培香蕉品种可根据不同的基因组类型和特征性状分为不同的栽培类型。根据近年来出版的英文书籍和涉及分类的文献[1, 9, 12, 23, 28-31],我们将全球常见的栽培类型及其特点汇总整理如表 2所示。
2 香蕉栽培品种的起源及迁徙路径
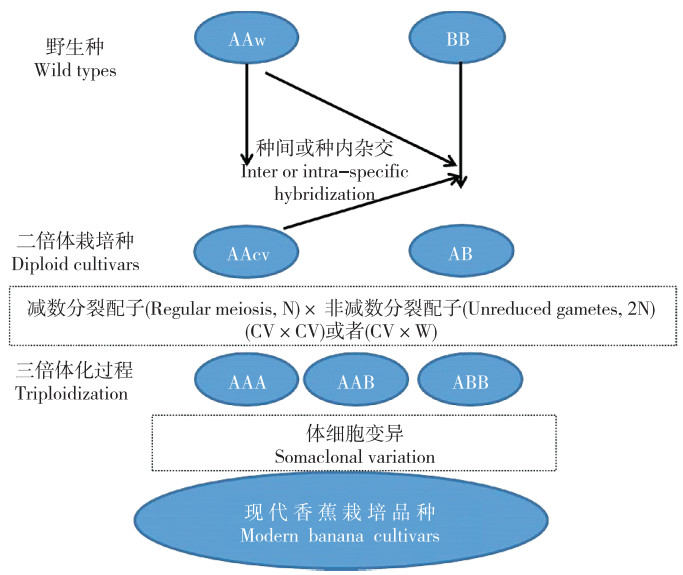
现代栽培香蕉品种大多源于两个野生种M. acuminata(尖苞野蕉,AA)和M. balbisiana(长梗野蕉,BB)的种内和种间杂交及体细胞变异而来[24]。M. acuminata的遗传多样性较丰富,目前鉴定的亚种有9个,分别是banksii、zebrina、malaccensis、burmannicoides、burmannica、siamea、truncata、microcarpa、errans[32];其中,burmannica与burmannicoides、siamea相近,可以统称为burmannica组[32-34]。原始的野生种多籽、果肉少,基本没有食用价值;野生种经过自然界的相互杂交、突变和人工选择,逐渐演化成具有单性结实(无籽)、果肉饱满等性状的栽培二倍体种[25, 32]。二倍体非减数分裂的配子(2n)与减数分裂的配子(n)进行杂交,产生三倍体种质;三倍体种质经过长期的体细胞变异和人工驯化,演化成AAA、AAB、ABB等基因组类型的栽培品种(图 2)[25, 30]。
|
| 图 2 三倍体栽培种香蕉演化示意图 Fig. 2 Evolution diagram of triploid cultivated banana |
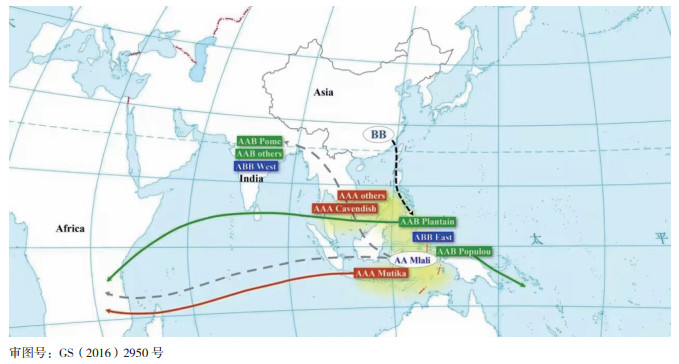
比较重要的栽培品种类型有3个,分别是香牙蕉、Plantain和东非高原蕉(East African Highland bananas,又称为Mutika)[25]。非洲中西部低洼湿地是Plantain的多样性分布中心,同时非洲东部高原地带是Mutika的多样性中心[32, 35-36]。Plantain曾是与传统意义上的栽培香蕉(Banana)齐名的独立物种,西班牙人第一次接触这个香蕉品种时感觉其与悬铃木(Plane tree)比较像,而取名为“Platano” [37]。基于Plantain的重要性,联合国粮农组织(FAO)将Plantain的统计数据单列[2-4],因此FAO的Banana数据并未包含Plantain的信息。
目前还不清楚栽培香蕉品种是如何从起源地扩散到世界各地。考古学家在巴布亚新几内亚境内的库克早期农业遗址(Kuk Early Agricultural Site)发现了香蕉的植物化石[38-39],并认为大约在公元前8 000年人类就开始驯化香蕉;而且香蕉的进化中心可能不仅限于巴布亚新几内亚,还包括周边的马来半岛、印度尼西亚、菲律宾等区域[25, 32]。据《巴利经》(Pali canon)记载,做贸易的印度人在马来西亚地区尝到了香蕉,就将香蕉带回国内种植;公元前327年,亚历山大大帝入侵印度,又把香蕉带至欧洲和非洲;香蕉最初的拉丁名Musa sapientum,意思为“智者的果实”来源于西印度[40]。但有学者认为,是阿拉伯人将香蕉从小亚细亚(亚洲西部半岛,现为土耳其国土)带至非洲,因为香蕉的英文单词“Banana”源自阿拉伯词语“Banan”(意为手指)[37, 41]。在进化后最初的2 000年内(约公元前6 000年),香蕉从进化中心扩散至整个热带地区,包括印度尼西亚、马来西亚、澳大利亚和印度等国家。大约在公元前3 000年,东部非洲地区已经有Plantain类型香蕉的种植,在公元前1 000年到达马达加斯加。有确切的考古学证据表明,非洲大陆在公元前500年至公元后500年间存在Plantain类型香蕉[31-32]。然而,Plantain如何扩散到非洲大陆仍是个谜,香蕉迁徙路径的假想见图 3。
2.1 香蕉栽培品种中A基因组的起源和演化
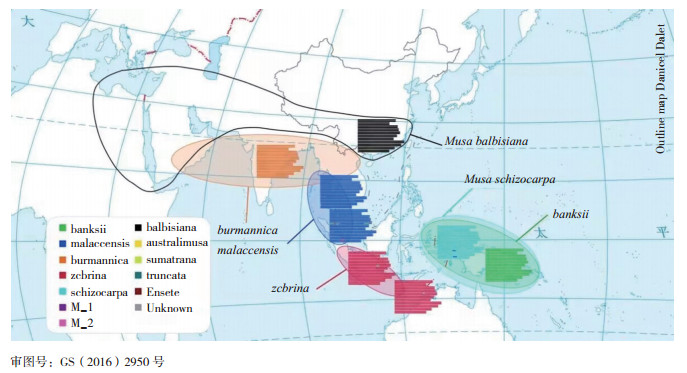
A基因组是栽培香蕉最重要的遗传资源,其对现代栽培种的品质、单性结实等性状有重要贡献[42]。目前AA野生种质的分布较广,横跨印度、斯里兰卡到巴布亚新几内亚、澳大利亚的北缘[25]。最新的系统进化和系统地理学的研究结果认为,在1 000万年前M. acuminata首先诞生在全球生物多样性热点之一的印- 缅地区(Indo-Burma)北部[39, 41]。burmannica亚种首先分化出来,然后通过马来半岛扩散至爪哇岛,接着扩散至新几内亚岛,最后又回到印度- 马来地区(Indo-Malayan region)的南部[43-45]。如图 4所示,在M. acuminata的亚种演化过程中,banksii出现在新几内亚,zebrina和microcarpa诞生于爪哇和婆罗洲,malaccensis和truncata出现在马来半岛,errans出现在菲律宾[25, 46]。亚种间的基因渗入现象较频繁,如泰国分布的malaccensis种质中存在burmannica/siamea的渗入[47]。
AA的亚种中最重要的群体是banksii,被认为对现代香蕉栽培种贡献最大,当前在巴布亚新几内亚还存在大量banksii的野生种质或衍生的栽培种[35, 45]。不仅如此,巴布亚新几内亚内也是SS的起源中心,且研究发现banksii与schizocarpa存在频繁的基因渗入现象[35, 45]。另外4个被认为对品种演化具有不同程度贡献的AA亚种(或种群)为burmannica/siamea、malaccensis、zebrina和errans[32, 48-49]。之前研究推测,香蕉栽培品种源于种间(Interspecific hybrids)或种内(Intraspecific hybrids)的杂交演化而来(如图 2)。然而这种观点现在被认为过于简单化,主要原因有2个:(1)种间的杂交演化过程中伴随着回交(Back-crossing)事件[50-51];(2)M. acuminata的亚种之间的频繁杂交,导致亚基因组中形成类似马赛克的染色体镶嵌结构(Sub-genomes mosaics)[47]。因此,AA的野生种- 栽培种的基因渗入也可能是现代栽培种演化出生态适应性各异的重要原因[35]。
基于基因组序列的研究结果显示,现代AA栽培种中还存在未知的AA始祖亚种。Martin等[35, 47]鉴定到2个未知亚种的信息,命名为M_1和M_2,并认为M_1可能与新发现的1个野生亚种M. acuminata ssp. halabanensis比较接近,在小部分AA栽培种(如Pisang Jari Buaya)中发现存在M_1的序列信息,而M_2则可能已经渗入到多个AA栽培种中,在东南亚及巴布亚新几内亚的很多AA栽培种中存在M_1的序列信息。Jeensae等[52]研究结果也证实M_1的存在。Sardos等[45]进一步发现有3个基因库(Genepools)的信息与已知的AA野生亚种基因组信息匹配不上,这其中就包含Martin等发现的M_1和M_2序列信息,因此他们认为可能存在3个未知的祖先亚种。图 4绘制了栽培香蕉的祖先种的起源和分布[35]。
2.2 AAA和AAB栽培类群的演化基于简化基因组测序(RADSeq)的研究结果表明,对现代香蕉栽培品种贡献最大的亚种为banksii、zebrina、malaccensis[35, 45]。大部分AAA中还存在祖先种M_2及S基因组渗入,部分AAA品种的9号染色体存在祖先种M_1[35]。对于AAB品种,Maia Maoli/Popoulu(太平洋岛国地区特有Cooking类型品种)中banksii占比较高,伴随着zebrina及S基因组的渗入;Plantain、Iholena和Laknao的结果与Maia Maoli/Popoulu类似,但这3个栽培类型中还存在一定比例的malaccensis信息[35];Silk中则是malaccensis占主导,同时存在banksii、zebrina和S祖先种的基因组信息[35]。
最近,有学者对几个代表性的AAA和AAB品种进行了全基因组测序和精确组装[53-55]。结果显示,亚基因组A经历了极其复杂的杂交过程,而亚基因组B是几乎同质的;对于Plantain品种类型,banksii对A基因组贡献度高达85.54%,malaccensis和zebrina分别为5.07% 和3.11%;对于Silk品种类型,malaccensis对A基因组贡献最多、达到59.86%,banksii和zebrina分别贡献29.22% 和9.55%[53]。AAA品种类型(香牙蕉和大蜜舍)的基因组组装结果明确了3个亚基因组Ban、Dh和Ze主要来源于banksii、malaccensis和zebrina[52-53]。这与前述基于简化基因组测序的研究结果高度吻合。此外,还发现AAA栽培种在香蕉果实成熟调控和抗病基因分布等方面呈现一定程度的亚基因组优势,品质等性状与Ban关联性较大,Ze亚基因组分布更多抗病基因,可能贡献了较多的抗性[54-55]。
2.3 ABB和AB栽培类群的演化与Maia Maoli/Popoulu品种祖先种A的溯源结果类似的是,ABB的Bluggoe、Monthan、Ney Mannan、Saba和Pelipita中banksii占比较高,同时有zebrina及S基因组的基因渗入现象[35, 56];粉蕉(Pisang Awak)的结果与Silk类似,其A基因组中malaccensis占主导地位[35, 51]。
异源多倍体(如小麦、油菜)的演化过程中经常发生同源交换事件(Homeologous exchanges,HEs)原因是具有高序列相似度的部分同源染色体之间比较容易通过配对交叉而导致大规模染色体片段交换,从而影响基因组的结构变化和基因表达[57-59]。Cenci等[51]通过分析36个ABB品种的简化基因组序列,发现至少存在9次不同的A与B亚基因组的HEs,同其他科研人员一致认为这可能是造成ABB多样性的重要原因[56, 60];Bluggoe、Monthan、Peyan和Ney Mannan的HEs区域较接近,表明它们具有类似的祖先种[51];此外,部分ABB品种类型(如Pelipita和Klue Teparod)中B与A的比例超过2︰1,也表明ABB品种演化过程中可能先形成AB,然后再与BB进行杂交形成ABB[51]。
AB类型的品种较少,印度主栽品种Kunnan属于AB类型。与AAA和AAB类似,AB类型中的A主要来源于malaccensis、banksii和zebrina,其中malaccensis占比最高,此外也存在S基因的渗入[51]。
3 展望近年来大多数研究集中在AA或A基因组的遗传信息解析上,BB基因组虽然已经组装[61],但对BB种质的研究仍旧较少。M. balbisiana虽然广泛分布于中国、印度、东南亚和巴布亚新几内亚等国,其带有一些优良的农艺性状,如抗病虫、耐旱和抗寒,但至今未有学者对其进行亚种的鉴定或分类,这可能是因为BB的多样性较缺乏所致[12, 25]。AAB的基因组解析结果显示,Plantain和Silk中B的亚基因组同源性很高[53]。这是否间接说明在进化过程中BB的分化原本就较少,导致B参与栽培香蕉的品种演化程度较低,或者含B的香蕉种质在自然进化或人工选择过程中大部分被淘汰,原因尚待揭示。
具有经济价值的香蕉二倍体栽培种还有TT基因组的Abaca(Musa textilis)和Fe’i(Musa troglodytarum)[9, 23]。Abaca中可提取蕉麻纤维,其被认为具有超强的纤维性能和耐腐蚀性,可被用作汽车车身部件和航空航天材料所用纤维复合材料的替代原料[62];Fe’i香蕉在太平洋地区被独立驯化,其果实富含类胡萝卜素[23]。笔者所在课题组以这两个材料组装了染色体水平的TT基因组,并初步解析蕉麻纤维性能形成及类胡萝卜素积累的分子机制[63]。后续将继续挖掘或验证纤维增强的基因资源,用于高性能生物纤维的利用,或改良香蕉或其他物种的抗倒伏性等研究。
从现代香蕉品种的演化过程来看,香蕉的遗传背景十分复杂。整体而言,Cooking类型品种(如Plantain和东非高原蕉)中banksii的贡献度较高,而鲜食蕉类型中malaccensis的贡献度较高[35, 45, 48]。几乎所有含A基因组的品种都含有banksii、zebrina、malaccensis等祖先种以及S基因组的渗入[35]。因此,AA野生种群的亚种间或种内杂交和基因渗入等复杂的进化事件,是栽培香蕉起源问题一直存在诸多争议的重要原因[35, 45]。此外,多样性丰富的各类香蕉二倍体种质资源,不仅是现代香蕉品种的演化基础,更是蕴含抗性、品质、产量等农艺和经济性状的基因库[64]。AA野生种经过漫长的演化形成多样性丰富的AA栽培种(AAcv),其中部分二倍体栽培种质(如Pisang Jari Buaya、Mlali)被认为对香牙蕉、大蜜舍、Plantain等品种的形成具有重要贡献[45, 47]。因此,进一步了解这些二倍体种群的遗传信息,对解析三倍体香蕉品种的起源和演化历程、挖掘对育种有重要价值的优异种质和基因资源、加快香蕉的选育种工作等方面,具有重要的应用价值和理论意义[65]。
| [1] |
ROBINSON J C, SAUCO V G. Bananas and plantains[M]. 2nd ed.. Wallingford, UK: CABI Publishing, 2010.
|
| [2] |
FAO. Banana market review-preliminary results[EB/OL]. [2023-05-16]. http://www.fao.org/3/cc3421en/cc3421en.pdf.
|
| [3] |
FAO. Banana factsand figures[EB/OL]. [2024-02-06]. https://www.fao.org/economic/est/est-commodities/oilcrops/bananas/bananafacts/en/.
|
| [4] |
FAOSTAT. [2024-01-08]. https://www.fao.org/faostat/en/#data.
|
| [5] |
SIMMONDS N W, SHEPHERD K. The taxonomy and origins of the cultivated bananas[J]. The Journal of the Linnean Society of London, 1955, 55(359): 302-312. DOI:10.1111/j.1095-8339.1955.tb00015.x |
| [6] |
SACHTER-SMITH G, SARDOS J. Bananas of cook islands: A catalog of banana diversity seen on the islands of Rarotonga and Aitutaki [EB/OL]. [2024-01-08] https://cgspace.cgiar.org/bitstream/handle/10568/113375/Bananas_Sachter_2021.pdf.
|
| [7] |
SACHTER-SMITH G, SARDOS J. Bananas of samoa: A catalogue of banana diversity seen on the island of Upolu[EB/OL]. [2024-01-08]. https://cgspace.cgiar.org/bitstream/handle/10568/113376/Samoa_catalog_2021.pdf.
|
| [8] |
SIMMONDS N W. The evolution of the bananas[M]. London: Longmans Green, 1962.
|
| [9] |
PILLAY M, TENKOUANO A. Banana breeding: Progress and challenges[M]. Boca Raton, USA: CRC Press, 2011.
|
| [10] |
HÄKKINEN M. Reappraisal of sectional taxonomy in Musa (Musaceae)[J]. Taxon, 2013, 62(4): 809-813. DOI:10.12705/624.3 |
| [11] |
DE LANGHE E, VRYDAGHS L, DE MARET P, PERRIER X, DENHAM T. Why bananas matter: An introduction to the history of banana domestication[J]. Ethnobotany Research and Applications, 2009, 7: 165-177. DOI:10.17348/era.7.0.165-177 |
| [12] |
盛鸥, 易干军. 香蕉种质资源图鉴[M]. 广州: 广东科技出版社, 2022. SHENG O, YI G J. Catalog of selected Musa accessions in China[M]. Guangzhou: Guangdong Science & Technology Press, 2022. |
| [13] |
WANG Z, ROUARD M, BISWAS M K, DROC G, CUI D, ROUX N, BAURENS F C, GE X J, SCHWARZACHER T, HESLOPHARRISON P, LIU Q. A chromosome-level reference genome of Enseteglaucum gives insight into diversity, chromosomal and repetitive sequence evolution in the Musaceae[J]. GigaScience, 2022, 11: 1-21. DOI:10.1093/gigascience/giac027 |
| [14] |
BORRELL J S, BISWAS M K, GOODWIN M, BLOMME G, SCHWARZACHER T, HESLOP-HARRISON J S, WENDAWEK A M, BERHANU A, KALLOW S, JANSSENS S, MOLLA E L, DAVIS A P, WOLDEYES F, WILLIS K, DEMISSEW S, WILKIN P. Enset in Ethiopia: A poorly characterized but resilient starch staple[J]. Annals of Botany, 2019, 123(5): 747-766. DOI:10.1093/aob/mcy214 |
| [15] |
MORROW N, BORRELL J S, MOCK N B, BÜCHI L, GATTO A, LULEKAL E. Measure of indigenous perennial staple crop, Enseteventricosum, associated with positive food security outcomes in southern Ethiopian highlands[J]. Food Policy, 2023, 117: 102451. DOI:10.1016/j.foodpol.2023.102451 |
| [16] |
PIJLS L T J, TIMMER A A M, WOLDE-GEBRIEL Z, WEST C E. Cultivation, prepa ration and consumption of ensete (Enseteventricosum) in Ethiopia[J]. Journal of the Science of Food and Agriculture, 1995, 67(1): 1-11. DOI:10.1002/jsfa.2740670102 |
| [17] |
NURAGA G W, EYISSA T, TESFAYE K, BISWAS M K, SCHWARZACHER T, BORRELL J S, WILKIN P, DEMISSEW S, TADELE Z, HESLOP-HARRISON J S. The genetic diversity of Enset (Enseteventricosum) landraces used in traditional medicine is similar to the diversity found in non-medicinal landraces[J]. Frontiers in Plant Science, 2022, 12: 756182. DOI:10.3389/fpls.2021.756182 |
| [18] |
KIDANE S A, HAUKELAND S, MERESSA B H, HVOSLEF-EIDE A K, COYNE D L. Planting material of Enset (Enseteventricosum), a key food security crop in Southwest Ethiopia, is a key element in the dissemination of plant-parasitic nematode infection[J]. Frontiers in Plant Science, 2021, 12: 664155. DOI:10.3389/fpls.2021.664155 |
| [19] |
傅本重, 刘丽, 伍建榕. 地涌金莲研究进展[J]. 中国农学通报, 2010, 26(15): 164-167. FU B Z, LIU L, WU J R. Research progress on Musellalasiocarpaplant[J]. Chinese Agricultural Science Bulletin, 2010, 26(15): 164-167. |
| [20] |
LIU A Z, KRESS W J, LONG C L. The ethnobotany of Musellalasiocarpa (Musaceae), an endemic plant of southwest China[J]. Economic Botany, 2003, 57(2): 279-281. DOI:10.1663/0013-0001 |
| [21] |
MA H, PAN Q J, WANG L, LI Z H, WAN Y M, LIU X X. Musellalasiocarpa var. rubribracteata (Musaceae), a new variety from Sichuan, China[J]. Novon, 2011, 21: 349-353. DOI:10.3417/2010125 |
| [22] |
唐靓, 李丕睿, 冯博雅, 赵万里, 陈雨. 中国特有植物地涌金莲的化学成分及应用价值研究进展[J]. 安徽农业科学, 2023, 51(3): 15-22, 36. DOI:10.3969/j.issn.0517-6611.2023.03.004 TANG L, LI P R, FENG B Y, ZHAO W L, CHEN Y. Research progress on chemical constituents and application values of Chinese endemic plant Musellalasiocarpa[J]. Journal of Anhui Agricultural Sciences, 2023, 51(3): 15-22, 36. DOI:10.3969/j.issn.0517-6611.2023.03.004 |
| [23] |
KANG MS, PRIYADASHAN PM. Breeding major food staples[M]. Ames, IA, USA: Blackwell Publishing, 2007.
|
| [24] |
ORTIZ R, SWENNENR. From crossbreeding to biotechnology-facilitated improvement of banana and plantain[J]. Biotechnology Advances, 2014, 32(1): 158-169. DOI:10.1016/j.biotechadv.2013.09.010 |
| [25] |
PERRIER X, DE LANGHE E, DONOHUE M, LENTFER C, VRYDAGHS L, BAKRY F, CARREEL F, HIPPOLYTE I, HORRY J P, JENNY C, LEBOT V, RISTERUCCI A M, TOMEKPE K, DOUTRELEPONT H, BALL T, MANWARING J, DE MARET P, DENHAM T. Multidisciplinary perspectives on banana (Musa spp.) domestication[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108: 11311-11318. DOI:10.1073/pnas.1102001108 |
| [26] |
HAMILL S D, DOOGAN V J, SMITH M K, DANIELLS J W. Characterisation and early detection of an offtype from micropropagated Lady Finger bananas[J]. Australian Journal of Experimental Agriculture, 1999, 39(8): 1017-1023. DOI:10.1071/EA99098 |
| [27] |
胡春华, 魏岳荣, 盛鸥, 邝瑞彬, 杨乔松, 李春雨, 易干军. 香蕉ACO反义基因转化夫人指蕉的研究[J]. 分子植物育种, 2015, 13(7): 1553-1558. DOI:10.13271/j.mpb.013.001553 HU C H, WEI Y R, SHENG O, KUANG R B, YANG Q S, LI CY, YI G J. Study on transformation of Furenzhi Banana (Musa spp. AA group) with an antisense ACO gene[J]. Molecular Plant Breeding, 2015, 13(7): 1553-1558. DOI:10.13271/j.mpb.013.001553 |
| [28] |
黄秉智. 香蕉种质资源描述规范和数据标准[M]. 北京: 中国农业科学出版社, 1998. HUANG B Z. Descriptors and data standard for banana (Musa spp.)[M]. Beijing: China Agriculture Press, 1998. |
| [29] |
李丰年, 李载忠, 郭富华. 广东香蕉菠萝图谱[M]. 广州: 广东科技出版社, 1998. LI F N, LI Z Z, GUO F H. The altas of banana and pineapple in Guangdong[M]. Guangzhou: Guangdong Science & Technology Press, 1998. |
| [30] |
KEMA G H J, DRENTH A. Achieving sustainable cultivation of bananas. Volume 2: Germplasm and genetic improvement[M]. Cambridge: Burleigh Dodds Science Publishing, 2020.
|
| [31] |
NAYAR N M. The bananas: Botany, origin, dispersal[J]. Horticultural Reviews, 2010, 36(3): 117-16. DOI:10.1002/9780470527238.ch2 |
| [32] |
PERRIER X, BAKRY F, CARREEL F, JENNY C, HORRY JP, LEBOT V, HIPPOLYTE I. Combining biological approaches to shed light on the evolution of edible bananas[J]. Ethnobotany Research and Applications, 2009, 7: 199-216. DOI:10.17348/era.7.0.199-216 |
| [33] |
HÄKKINEN M, VÄRE H. Typification and check-list of Musa L. names (Musaceae) with nomenclatural notes[J]. Adansonia, 2008, 30: 63-112. |
| [34] |
SHEPHERD K. Observations on Musa taxonomy[C] // JARRET R L. Identification of genetic diversity in the genus Musa[M]. Montpellier, France: INIBAP, 1990.
|
| [35] |
MARTIN G, COTTIN A, BAURENS FC, LABADIE K, HERVOUET C, SALMON F, PAULO-DE-LA-REBERDIERE N, DEN HOUWE I V, SARDOS J, AURY JM, D'HONT A, YAHIAOUI N. Interspecific introgression patterns reveal the origins of worldwide cultivated bananas in New Guinea[J]. The Plant Journal, 2023, 113(4): 802-818. DOI:10.1111/tpj.16086 |
| [36] |
PERRIER X, JENNY C, BAKRY F, KARAMURA D, KITAVI M, DUBOIS C, HERVOUET C, PHILIPPSON G, DE LANGHE E. East African diploid and triploid bananas: A genetic complex transported from South-East Asia[J]. Annals of Botany, 2019, 123(1): 19-36. DOI:10.1093/aob/mcy156 |
| [37] |
STOVER R H, SIMMONDS N W. Bananas[M]. Essex, London: Longman Scientific and Technical Publishers, Inc., 1987.
|
| [38] |
DENHAM T P, HABERLE S G, LENTFER C, FULLAGAR R, FIELD J, THERIN M, PORCH N, WINSBOROUGH B. Origins of agriculture at Kuk Swamp in the highlands of New Guinea[J]. Science, 2003, 301(5630): 189-193. DOI:10.1126/science.1085255 |
| [39] |
DENHAM T, HABERLE S, LENTFER C. New evidence and revised interpretations of early agriculture in Highland New Guinea[J]. Antiquity, 2004, 78(302): 839-857. DOI:10.1017/S0003598X00113481 |
| [40] |
FULLER D Q, MADELLA M. Banana cultivation in South Asia and East Asia: A review of the evidence from archaeology and linguistics[J]. Ethnobotany Research and Applications, 2009, 7: 333-351. DOI:10.17348/era.7.0.333-351 |
| [41] |
JANSSENS S B, VANDELOOK F, DE LANGHE E, VERSTRAETE B, SMETS E, VANDENHOUWE I, SWENNEN R. Evolutionary dynamics and biogeography of Musaceae reveal a correlation between the diversification of the banana family and the geological and climatic history of Southeast Asia[J]. The New Phytologist, 2016, 210: 1453-1465. DOI:10.1111/nph.13856 |
| [42] |
HESLOP-HARRISON J S, SCHWARZACHER T. Domestication, genomics and the future for banana[J]. Annals of Botany, 2007, 100: 1073-1084. DOI:10.1093/aob/mcm191 |
| [43] |
FU N, JI M, ROUARD M, YAN H F, GE X J. Comparative plastome analysis of Musaceae and new insights into phylogenetic relationships[J]. BMC Genomics, 2022, 23: 223. DOI:10.1186/s12864-022-08454-3 |
| [44] |
ROUARD M, DROC G, MARTIN G, SARDOS J, HUEBER Y, GUIGNON V, CENCI A, GEIGLE B, HIBBINS M S, YAHIAOUI N, BAURENS F, BERRY V, HAHN M W, D'HONT A, ROUX N. Three new genome assemblies support a rapid radiation in Musa acuminate (Wild banana)[J]. Genome Biology and Evolution, 2018, 10: 3129-3140. DOI:10.1093/gbe/evy227 |
| [45] |
SARDOS J, BRETON C, PERRIER X, VAN DEN HOUWE I, CARPENTIER S, PAOFA J, ROUARD M, ROUX N. Hybridization, missing wild ancestors and the domestication of cultivated diploid bananas[J]. Frontiers in Plant Science, 2022, 13: 969220. DOI:10.3389/fpls.2022.969220 |
| [46] |
CARREEL F, GONZALEZ DE LEON D, LAGODA P, LANAUD C, JENNY C, HORRY J P, DU MONTCEL H T. Ascertaining maternal and paternal lineage within Musa by chloroplast and mitochondrial DNA RFLP analyses[J]. Genome, 2002, 45: 679-692. DOI:10.1139/g02-033 |
| [47] |
MARTIN G, CARDI C, SARAH G, RICCI S, JENNY C, FONDI E, PERRIER X, GLASZMANN J C, D'HONT A, YAHIAOUI N. Genome ancestry mosaics reveal multiple and cryptic contributors to cultivated banana[J]. The Plant Journal, 2020, 102: 1008-1025. DOI:10.1111/tpj.14683 |
| [48] |
SARDOS J, ROUARD M, HUEBER Y, CENCI A, HYMA K E, VAN DEN HOUWE I, HRIBOVA E, COURTOIS B, ROUX N. A genomewide association study on the seedless phenotype in banana (Musa spp.) reveals the potential of a selected panel to detect candidate genes in a vegetatively propagated crop[J]. PLoS One, 2016, 11: e0154448. DOI:10.1371/journal.pone.0154448 |
| [49] |
CHRISTELOVÁ P, DE LANGHE E, HŘIBOVÁ E, ČÍŽKOVÁ J, SARDOS J, HUŠÁKOVÁ M, VAN DEN HOUWE I, SUTANTO A, KEPLER A K, SWENNEN R, ROUX N, DOLEŽEL J. Molecular and cytological characterization of the global Musa germplasm collection provides insights into the treasure of banana diversity[J]. Biodiversity and Conservation, 2017, 26: 801-824. DOI:10.1007/s10531-016-1273-9 |
| [50] |
BAURENS F C, MARTIN G, HERVOUET C, SALMON F, YOHOMÉ D, RICCI S, ROUARD M, HABAS R, LEMAINQUE A, YAHIAOUI N, D'HONT A. Recombination and large structural variations shape inter-specific edible bananas genomes[J]. Molecular Biology and Evolution, 2019, 36: 97-111. DOI:10.1093/molbev/msy199 |
| [51] |
CENCI A, SARDOS J, HUEBER Y, MARTIN G, BRETON C, ROUX N, SWENNEN R, CARPENTIER S C, ROUARD M. Unravelling the complex story of intergenomic recombination in ABB allotriploid bananas[J]. Annals of Botany, 2021, 127: 7-20. DOI:10.1093/aob/mcaa032 |
| [52] |
JEENSAE R, KONGSIRI N, FLUCH S, BURG K, BOONRUANGROD R. Cultivar specific gene pool may play an important role in Musa acuminate Colla evolution[J]. Genetic Resources and Crop Evolution, 2021, 68: 1589-1601. DOI:10.1007/s10722-020-01088-y |
| [53] |
XIE W Z, ZHENG YY, HE W, BI F, LI Y, DOU T, ZHOU R, GUO YX, DENG G, ZHANG W, YUAN M H, SANZ-JIMENEZ P, ZHU X T, XU X D, ZHOU Z W, ZHOU Z W, FENG J W, LIU S, LI C, YANG Q, HU C, GAO H, DONG T, DANG J, GUO Q, CAI W, ZHANG J, YI G, SONG J M, SHENG O, CHEN L L. Two haplotype-resolved genome assemblies for AAB allotriploid bananas provide insights into banana subgenome asymmetric evolution and Fusarium wilt control[J]. Plant Communications, 2024, 5: 100766. DOI:10.1016/j.xplc.2023.100766 |
| [54] |
LI X, YU S, CHENG Z, CHANG X, YUN Y, JIANG M, CHEN X, WEN X, LI H, ZHU W, XU S, WANG X, ZHANG C, WU J, LIN Z, AURY JM, PEER Y V, WANG Z, ZHOU X, WANG J, LYU P, ZHANG L. Origin and evolution of the triploid cultivated banana genome[J]. Nature Genetics, 2024, 56: 136-142. DOI:10.1038/s41588-023-01589-3 |
| [55] |
HUANG H R, LIU X, ARSHAD R, WANG X, LI W M, ZHOU Y, GE X J. Telomere-to-telomere haplotype-resolved reference genome reveals subgenome divergence and disease resistance in triploid Cavendish banana[J]. Horticulture Research, 2023, 10(9): uhad153. DOI:10.1093/hr/uhad260 |
| [56] |
MARTIN G, BAURENS F C, LABADIE K, HERVOUET C, SALMON F, MARIUS F, PAULO-DE-LA-REBERDIERE N, VAN DEN HOUWE I, AURY J-M, D'HONT A, YAHIAOUI N. Shared pedigree relationships and transmission of unreduced gametes in cultivated banana[J]. Annals of Botany, 2023, 131: 1149-1161065. DOI:10.1093/aob/mcad065 |
| [57] |
SALMON A, FLAGEL L, YING B, UDALL JA, WENDEL J F. Homoeologous nonreciprocal recombination in polyploid cotton[J]. New Phytologist, 2010, 186: 123-134. DOI:10.1111/j.1469-8137.2009.03093.x |
| [58] |
HE Z, WANG L, HARPER A L, HAVLICKOVA L, PRADHAN A K, PARKIN I A. Bancroft I. Extensive homoeologous genome exchanges in allopolyploid crops revealed by mRNA seq-based visualization[J]. Plant Biotechnology Journal, 2017, 15: 594-604. DOI:10.1111/pbi.12657 |
| [59] |
LLOYD A, BLARY A, CHARIF D, CHARPENTIER C, TRAN J, BALZERGUE S, DELANNOY E, RIGAILL G, JENCZEWSKI E. Homoeologous exchanges cause extensive dosage-dependent gene expression changes in an allopolyploid crop[J]. New Phytologist, 2018, 217(1): 367-377. DOI:10.1111/nph.14836 |
| [60] |
HIGGINS J, OSORIO-GUARÍN J A, OLAVE-ACHURY C, TOLOZA-MORENO D L, ENRIQUEZ A, DI PALMA F, YOCKTENG R, DE VEGA J J. Characterizing subgenome recombination and chromosomal imbalances in banana varietal lineages[J]. Annals of Botany, 2023, mcad192. DOI:10.1093/aob/mcad192 |
| [61] |
WANG Z, MIAO H, LIU J, XU B, YAO X, XU C, ZHAO S, FANG X, JIA C, WANG J, ZHANG J, LI J, XU Y, WANG J, MA W, WU Z, YU L, YANG Y, LIU C, GUO Y, SUN S, BAURENS FC, MARTIN G, SALMON F, GARSMEUR O, YAHIAOUI N, HERVOUET C, ROUARD M, LABOUREAU N, HABAS R, RICCI S, PENG M, GUO A, XIE J, LI Y, DING Z, YAN Y, TIE W, D'HONT A, HU W, JIN Z. Musa balbisiana genome reveals subgenome evolution and functional divergence[J]. Nature Plants, 2019, 5: 810-821. DOI:10.1038/s41477-019-0452-6 |
| [62] |
DELICANO J A. A review on abaca fiber reinforced composites[J]. Composite Interfaces, 2018, 25(12): 1039-1066. DOI:10.1080/09276440.2018.1464856 |
| [63] |
ZHOU R, WANG S, ZHAN N, HE W, DENG G, DOU T, ZHU X T, XIE W Z, ZHENG Y Y, HU C, BI F, GAO H, DONG T, LIU S, LI C, YANG Q, WANG L, SONG J M, DANG J, GUO Q, YI G, CHEN L L, SHENG O. High quality reference genome assemblies for two Australimusa bananas provide insights into genetic diversity of the Musaceae family and regulatory mechanisms of superior fiber properties[J]. Plant Communications, 2024, 5: 100681. DOI:10.1016/j.xplc.2023.100681 |
| [64] |
ROUARD M, SARDOS J, SEMPÉRÉ G, BRETON C, GUIGNON V, VAN DEN HOUWE I, CARPENTIER S C, ROUX N. A digital catalog of high density markers for banana germplasm collections[J]. Plants, People, Planet, 2022, 4(1): 61-67. DOI:10.1002/ppp3.10187 |
| [65] |
DESTA ZA, ORTIZ R. Genomic selection: Genome-wide prediction in plant improvement[J]. Trends in Plant Science, 2014, 19(9): 592-601. DOI:10.1016/j.tplants.2014.05.006 |
(责任编辑 张辉玲)
 2024, Vol. 51
2024, Vol. 51