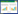文章信息
基金项目
- 广东省乡村振兴战略专项种业振兴行动项目(2022-NPY-03-001);广东省自然科学基金(2019A1515011587);广州市科技计划项目(202102010414)
作者简介
- 许玉富(1998—),女,在读硕士生,研究方向为蔬菜遗传育种,E-mail:1228056643@qq.com.
通讯作者
- 夏岩石(1976—),男,博士,副教授,研究方向为分子遗传育种,E-mail:xiayanshi922@gzhu.edu.cn.
文章历史
- 收稿日期:2023-11-25
2. 广东省良种引进服务公司,广东 广州 510091;
3. 广州市农业科学研究院,广东 广州 510308
2. Guangdong Provincial Improved Variety Introduce Service Corporation, Guangzhou 510091, China;
3. Guangzhou Academy of Agricultural Sciences, Guangzhou 510308, China
【研究意义】菜心(Brassica campestris L. ssp. Chinensis var. utilis Tsen et Lee)是十字花科芸薹属芸薹种白菜亚种中以花薹为产品的变种,是华南地区的特色蔬菜。随着市场需求的扩大和人们消费水平的提高,高产、优质和高抗菜心品种的选育已成为当前主要的研究方向[1-2],而开发多态性丰富的分子标记应用于菜心遗传育种,可有效拓宽菜心的遗传基础,加快菜心优异种质资源的改良创新,促进菜心新品种的选育。【前人研究进展】目前,RAPD(Random amplification polymorphic DNA)、ISSR(Inter-simple sequence repeat)、MFLP(Microsatellite-anchored fragment length polymorphism)、AFLP(Amplified fragment length polymorphism)和SSR(Simple sequence repeat)等分子标记均广泛地应用于菜心种质资源遗传多样性评价、遗传图谱构建和关联分析等研究[3-7],但这些分子标记的筛选成本大、稳定性差、效率低,制约了其在菜心遗传育种研究中的进一步应用。插入/ 缺失(Insertion/Deletion,InDel)标记是等位基因位点的核苷酸序列在个体间因插入、缺失引起序列长度存在多态性变异,具有基因组内分布广、密度高、变异稳定、多态性丰富和开化成本低等优点[8]。随着作物基因组序列的不断完善,基于重测序数据和生物信息学等手段进行基因组水平上InDel标记的开发变得简便快捷[9]。Liu等[10]以Chiifu-401-42的基因组序列为参考序列,通过对2份白菜材料的全基因组重测序来鉴定InDel位点,设计并验证了387个多态性InDel标记,可应用于白菜群体的遗传多样性分析。Guo等[11]通过全基因组重测序的方法,以C. annuum cv. CM334的基因组序列为参考序列,在2份辣椒材料中鉴定了InDel位点,筛选出194个InDel标记在5份辣椒材料中表现出多态性。RAD-seq(Restriction-site associated DNA sequencing)是Miller等[12]基于高通量测序方法发展起来的简化基因组测序技术,由于使用限制性内切酶消化技术降低了基因组的复杂性,在鉴定InDel位点方面具有低成本和高通量的优势。近年来,利用该技术在西红柿[13]、芝麻[14]和烟草[15]等作物中开发了大量的InDel标记。【本研究切入点】至今,关于菜心InDel标记开发研究的报道较少。江定等[8]基于转录组数据进行了InDel位点的挖掘,但仅停留在统计和分析,尚未对位点进行InDel标记开发和验证。而孟霖等[16]通过白菜型油菜R-O-18和菜薹L58的基因组重测序数据进行InDel标记开发,500个InDel位点中仅有106对引物在2个亲本间表现出多态性。【拟解决的关键问题】为获得大量多态性丰富的InDel标记应用于菜心的遗传育种,本研究选用低成本高通量的RAD技术,对熟性和耐热程度差异明显的4份菜心种质材料进行重测序,以Chiifu-401-42的基因组序列为参考序列进行比对,在全基因范围内鉴定4份菜心材料间具有多态性的InDel位点。运用生物信息学方法,筛选4份菜心种质材料间具有潜在多态性的InDel位点,并根据InDel位点两端的DNA序列设计PCR引物开发易于凝胶电泳检测的InDel标记,利用部分InDel标记分析菜心群体的遗传多样性,为菜心遗传育种提供研究基础。
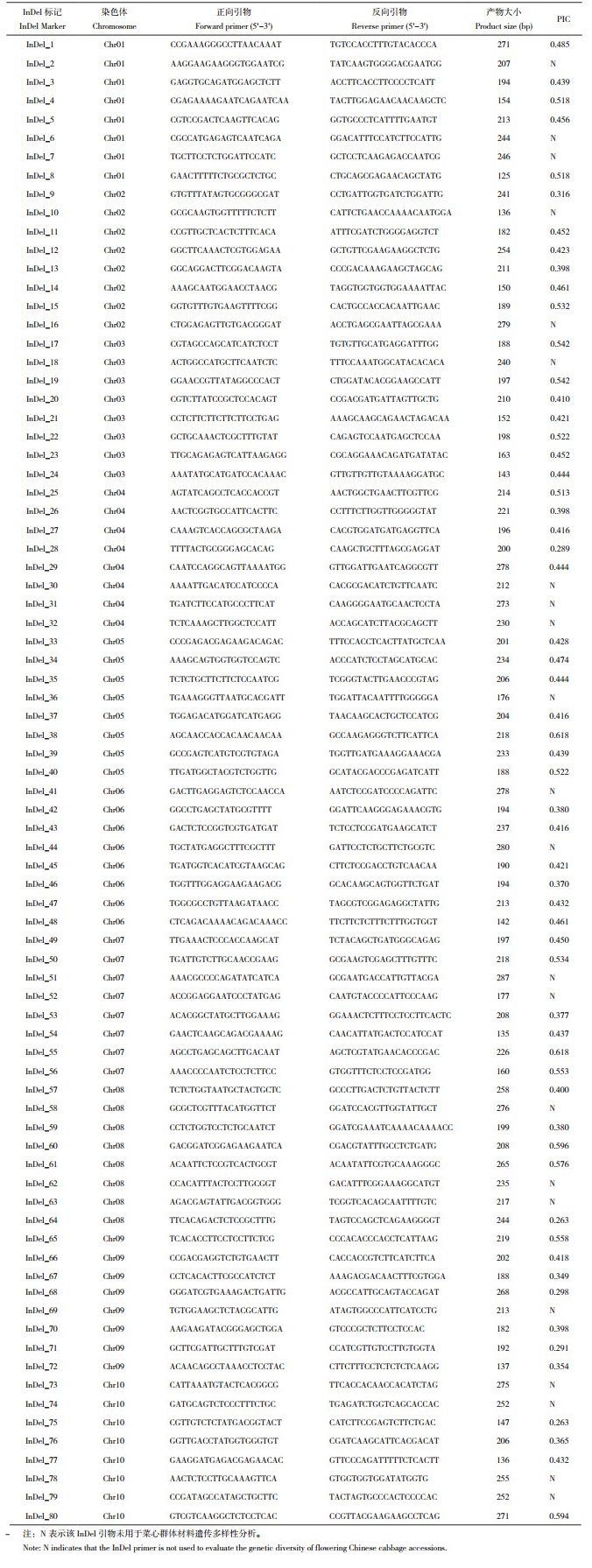
1 材料与方法 1.1 试验材料本试验选取四九-19号菜心、油绿501菜心、3T6和柳叶50等4份菜心种质材料进行RAD重测序筛选InDel标记,其中四九-19号菜心和油绿501菜心为早熟耐热菜心种质,3T6和柳叶50为中熟不耐热菜心种质[17-19]。另选取55份菜心种质材料用于InDel标记应用性验证及遗传多样性分析,以上菜心种质材料均由广州市农业科学研究院蔬菜研究所提供。55份菜心种质材料根据其耐热程度不同可分为耐热性差、耐热性中等和耐热性强等3种类型[20],其叶色及耐热性的情况如表 1所示。
|
1.2 试验方法
1.2.1 RAD重测序及InDel位点的挖掘 利用改良的CTAB法提取4份菜心种质材料的基因组DNA,然后采用紫外分光光度法和1.2% 琼脂糖凝胶电泳检测DNA的浓度和质量。检测合格的DNA样品送至广州基迪奥科技股份有限公司完成RAD文库构建和二代测序。重测序原始数据(Raw reads)经过质控,过滤低质量的reads,得到高质量的clean reads。使用BWA软件[21]将高质量的测序数据与参考基因组Brara_Chiifu_V3.0(http://brassicadb.cn/#/Download/)进行比对,随后利用Samtools软件[22]和GATK(https://github.com/broadinstitute/gatk)进行去重复和InDel位点的挖掘。以参考基因组Brara_Chiifu_V3.0的注释基因信息(gff3数据)为参考位置,利用软件ANNOVAR[23]对InDel位点进行基因注释。
1.2.2 InDel位点的筛选及标记有效性验证 从上述鉴定到的InDel位点中筛选出InDel长度大于5 bp、测序深度大于10×、且在4份重测序菜心材料间具有多态性的位点。从中挑选80个分布于10条染色体上的InDel位点进行引物设计,提取该位点上下游各250 bp碱基序列,利用Primer 3.0软件设计引物,设置引物长度为18~25 bp,GC含量为40%~60%,Tm值为52~62 ℃,产物大小为100~300 bp。以4份重测序菜心种质材料的基因组DNA为模版进行PCR扩增,然后采用Liu等[24]建立的非变性聚丙烯酰胺凝胶电泳方法检测PCR扩增产物,验证80个InDel引物的有效性。
1.2.3 InDel标记分析菜心种质群体的遗传多样性 选取扩增条带清晰且多态性明显的InDel标记,以55份菜心种质材料的基因组DNA为模板,进行PCR扩增和6% 的非变性聚丙烯酰胺凝胶电泳检测,根据条带的有无,确定等位变异位点,并构建1、0二元数据矩阵,完成菜心种质群体的基因分型。使用Power Marker(PM)3.2[25]计算每个InDel标记的PIC值,同时利用软件NTSYS-pc2.10进行1、0二元数据矩阵分析,计算菜心材料间的遗传相似系数,然后使用SAHN程序和UPGMA算法进行聚类分析,参数为默认,生成聚类树状图。
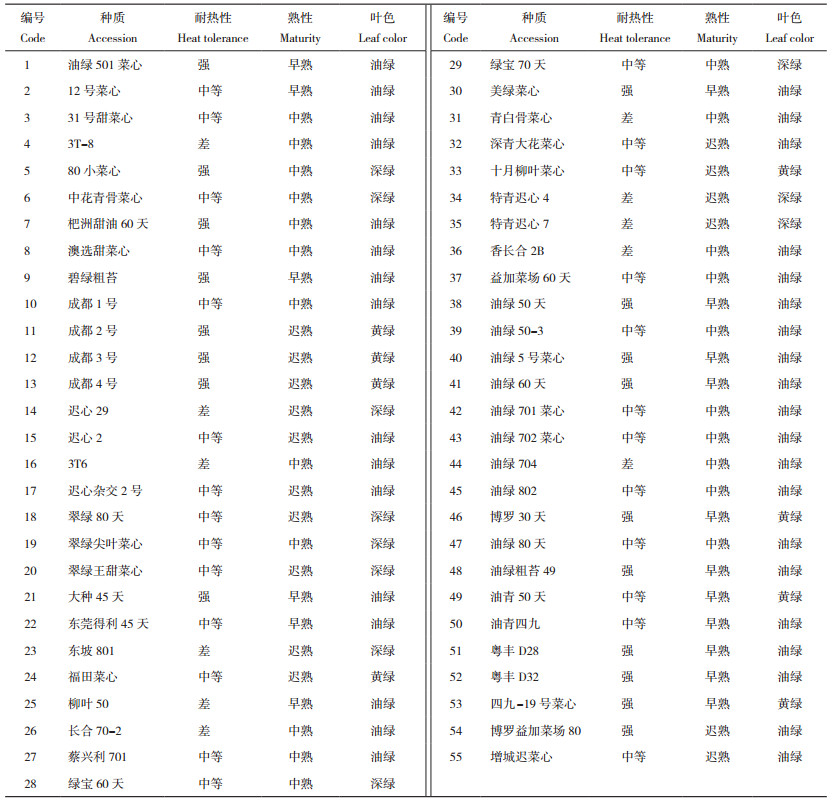
2 结果与分析 2.1 菜心RAD重测序数据的统计分析通过Illumina测序,4份菜心种质材料经过滤后共得到10.38 Gb clean data,各样本的clean data在2.50~2.66 Gb之间,GC含量在38.37%~39.90% 之间,Q30均在93% 以上。与参考基因组Brara_Chiifu_V3.0比对,4份菜心材料的比对率在80.21%~90.35% 之间,平均测序深度为10.89×(表 2)。由表 2可知,4份菜心材料获得的测序数据充足、质量可靠,可进一步用于InDel标记的开发。
|
2.2 菜心InDel位点的鉴定
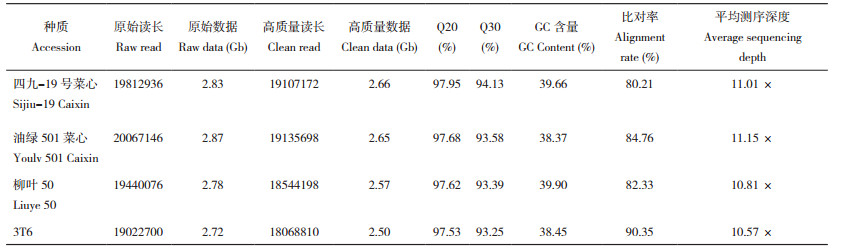
通过与参考基因组Brara_Chiifu_V3.0比对,四九-19号菜心、油绿501菜心、柳叶50和3T6等4份菜心种质材料分别检测到22 944、23 425、34 425和51 157个InDel位点,总计84 510个,其中43 437个(51.39%)为缺失突变,41 073个(48.60%)为插入突变。Indel位点的差异长度在1~61 bp之间,其中1 bp为主要差异长度,其次为2~4 bp,长度大于5 bp的InDel数量较少(图 1A)。基因注释结果(图 1B)显示,位于基因内含子区域的InDel位点最多,占比32.77%;其次为位于间隔区,占比24.47%;位于基因上游和下游的数量接近,分别为16.01% 和17.14%;位于外显子区域和剪接点的InDel个数较少,仅有3.94% 和0.43%。
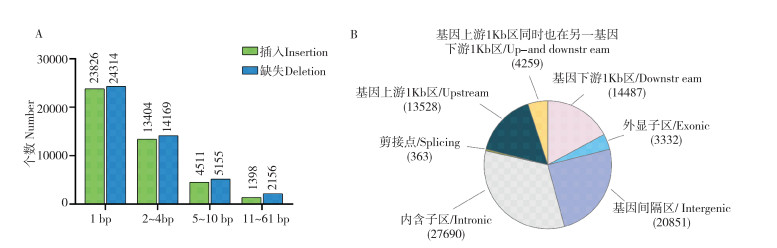
|
| A: 不同长度InDel位点的数量;B: InDel位点的注释信息 A: Number of InDel loci with different length; B: Annotated information of InDel loci 图 1 84 510个InDel位点的统计信息 Fig. 1 Statistical information of 84 510 InDel loci |
2.3 菜心多态性InDel位点的筛选及有效性验证
基于生物信息学分析,筛选4份重测序菜心材料间具有多态性的InDel位点,且InDel长度大于5 bp、测序深度大于10×,共获得3 609个。在每条染色体上随机挑选8个InDel位点,共80个,设计PCR扩增引物,以4份菜心材料的基因组DNA为模板进行PCR扩增,非变性聚丙烯酰胺凝胶电泳检测扩增产物的大小及多态性。结果(图 2)显示,80对InDel引物中,73对引物均能扩增出与预测产物大小基本相同的条带、占比91.25%,其中58对引物在4份菜心材料间检测出多态性、占比72.5%,这些具有多态性的InDel标记可用于检测菜心群体材料的遗传多样性。
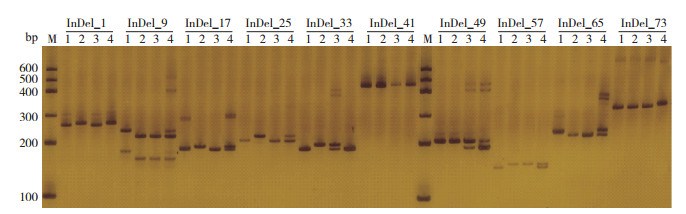
|
| M:DNA分子量标准;1~4:分别代表四九-19号菜心、柳叶50、油绿501菜心和3T6 M: DNA molecular weight marker; 1-4: Refer to Sijiu-19 Caixin, Liuye 50, Youlv 501 Caixin and 3T6, respectively 图 2 10对InDel引物在4份菜心种质中的PCR扩增结果 Fig. 2 PCR results of 10 pairs of InDel primers in four flowering Chinese cabbage accessions |
2.4 基于InDel标记菜心群体的遗传多样性分析
利用具有多态性的58个InDel标记对55份菜心种质材料进行基因型分析,图 3是InDel_43标记对55份菜心种质材料的基因分型结果。58个InDel标记在55份菜心种质材料中共检测出133个等位基因,变幅为2~3个,平均每个标记2.26个,多态信息量(PIC)的分布范围在0.263~0.618之间、平均值为0.443(表 3)。通过NTSYS-pc2.1软件分析,55份菜心种质材料间的遗传相似系数(Genetic similarity coefficient,GS)变异范围为0.366~0.756、平均值为0.570。其中,绿宝60天与澳选甜菜心的GS最小、为0.366,其次是绿宝60天与迟心杂交2号、油青四九与博罗益加菜场80,GS分别为0.397和0.398,表明这3组菜心材料的亲缘关系较远,遗传差异比较大。油绿501菜心与油绿50-3、迟心杂交2号与油绿50天的GS最大、均为0.756,表明两组菜心材料的亲缘关系较近。可见,本研究开发的InDel标记在菜心种质群体中具有良好的多态性和特异性,能有效区分种质材料间的亲缘关系,可用于菜心品种的鉴别和育种。
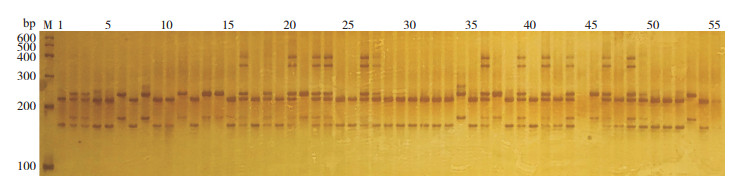
|
| M:DNA分子量标准;1~55:55份菜心种质材料 M: DNA molecular weight marker; 1-55: 55 flowering Chinese cabbage accessions 图 3 InDel_43标记在55份菜心种质中的PCR扩增结果 Fig. 3 PCR amplification result of InDel_43 marker in 55 flowering Chinese cabbage accessions |
|
根据种质材料间的GS值,使用非加权平均法(UPGMA)将55份菜心材料进行聚类,在GS值为0.564处可分为Ⅰ、Ⅱ、Ⅲ和Ⅳ等4个类群(图 4)。类群Ⅰ包含8份菜心种质,其中耐热性强和耐热性差的种质材料各2份,耐热性中等的种质材料4份;类群Ⅱ包含37份菜心种质材料,其中耐热性强的种质材料15份,耐热性中等的种质材料16份,耐热性差的种质材料6份;类群Ⅲ仅包含4份菜心种质材料,耐热性中等和耐热性差的种质材料分别为3份和1份;类群Ⅳ包含6份菜心种质材料,其中耐热性中等和耐热性差的种质材料均为3份,耐热性强的种质材料为1份。研究中还发现,4个类群中菜心种质材料的叶色和熟性也没有明显的聚类。
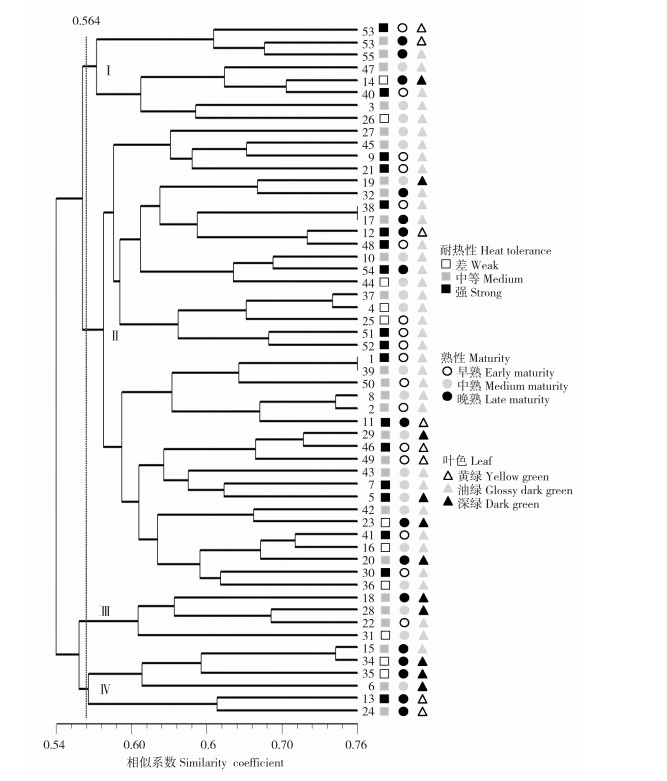
|
| 图 4 基于InDel标记55份菜心种质的遗传聚类图 Fig. 4 Genetic cluster graph of 55 flowering Chinese cabbage accessions based on InDel markers |
3 讨论
InDel标记具有变异稳定、准确性高、检测方便的特点,随着高通量测序技术的快速发展,基于高通量测序数据来鉴定和开发InDel标记已广泛应用于作物的遗传育种。本研究通过4份菜心种质材料的基因组RAD重测序,检测到84 510个InDel位点,其中1 bp为主要差异长度、其次为2~4 bp,长度大于5 bp的InDel数量较少。基因注释结果显示,位于基因内含子区域的InDel位点最多,占比32.77%;其次为位于间隔区,占比24.47%;位于外显子区域和剪接点的InDel个数少,仅有3.94% 和0.43%。这一结果与芥蓝[26]、油菜[16]、白菜[27]、大豆[28]等作物基因组中InDel类型的分布特征一致。江定等[8]基于菜心转录组测序数据检测的InDel位点结果也显示基因内含子区域内InDel位点最多,但基因间隔区分布很少,这可能是由于转录组数据中缺少基因间隔区域的序列所导致的差异。
根据检测到的InDel位点,本研究筛选了均匀分布于全基因组80个InDel位点,并设计引物,结果发现73对引物均能扩增出与预测产物大小基本相同的条带、占比91.25%,其中58对引物在4份菜心材料间检测出多态性、占比72.5%。Liu等[10]利用2份白菜材料的重测序数据开发InDel标记,验证了639个InDel位点,其中491个(76.8%)具有多态性;洪晓如等[26]基于2份芥蓝材料的重测序数据开发InDel标记367个,验证后发现284个(77.4%)具有多态性。在白菜和芥蓝的研究中,InDel位点的选择标准都是基于插入或缺失碱基数小于5 bp,而本研究为便于凝胶电泳分析,InDel标记的选择标准为插入或缺失碱基数大于5 bp。由于所选择的插入/ 缺失碱基数较大,其假阳性也相应增加,从而导致InDel标记的多态率降低。高通量测序的深度对InDel位点鉴定的准确性也有影响,测序深度越高,鉴定的InDel位点越准确,一般测序深度为10×时,可满足InDel标记的开发[28]。例如,孟霖等[16]利用测序深度为3×的重测序数据检测了白菜型油菜与菜薹间的InDel位点,设计了500个InDel标记,仅有106个(21.2%)在二者间呈多态性;张志刚等[29]利用测序深度为8×的大白菜自交系材料的重测序数据进行InDel标记的开发,验证了933个InDel位点,阳性率为63.6%,验证的InDel位点多态率均低于本研究结果。
种质资源是作物遗传改良的基础,分析菜心种质资源材料的遗传多样性和亲缘关系对拓宽其种质资源及遗传育种具有重要作用。利用58对InDel标记检测了55份菜心种质材料的基因型,共检测出133个等位基因,55份菜心种质材料间的遗传相似系数(GS)在0.366~0.756之间,平均值为0.570,这一结果比利用SSR标记[30]、SCoT标记[31]和SRAP标记[32]分析菜心群体的遗传相似系数低,其可能的原因是本次选用的菜心群体材料遗传差异较大,也可能是InDel标记具有良好的多态性和特异性,更能区分菜心种质材料间较小的差异,进而达到良好的分离效果[33]。基于遗传相似系数的聚类分析显示,在GS值为0.564处,55份菜心种质分为4个类群,各类群包含的菜心材料数量差异较大,不同耐热型、不同熟性和不同叶色类型的菜心材料分散在各类群之间,这与夏岩石等[7]、史卫东等[31]和卢宇鹏等[34]的研究结果基本一致,其原因可能为菜心的耐热性和叶色受多基因调控[35-36],较少的分子标记不能有效地区分这两个性状。
4 结论本研究通过RAD重测序和比对分析,在4份菜心种质材料中共鉴定出84 510个InDel位点,筛选出3 609个InDel位点在4份材料间具有潜在多态性,挑选80个InDel标记进行PCR扩增验证,其中58个(72.5%)在4份测序样品中具有多态性。利用58个InDel标记对55份菜心种质材料的遗传多样性分析显示,筛选的InDel标记多态性良好,PIC值在0.263~ 0.618之间,能较好地区分55份菜心种质材料间的遗传差异,在GS值为0.564处可将55份菜心种质分为4个类群。本研究利用重测序数据开发的InDel标记,将在菜心种质资源遗传多样性分析、遗传图谱的构建和分子标记辅助育种等工作中发挥重要作用。
| [1] |
王先琳, 王翠叶, 陈利丹, 苏运诗, 周成良, 谢月华. 我国薹用不结球白菜主栽品种现状及育种趋势[J]. 蔬菜, 2022(3): 52-55. WANG X L, WANG C Y, CHEN L D, SU Y S, ZHOU C L, XIE Y H. Current situation and breeding trend of non-heading Chinese cabbage in China[J]. Vegetables, 2022(3): 52-55. |
| [2] |
黄依琳, 许玉富, 李荣华, 李光光, 张华, 郭培国, 夏岩石. 菜心群体中AUF2基因的遗传变异及其与农艺性状的关联分析[J]. 广东农业科学, 2023, 50(4): 33-41. DOI:10.16768/j.issn.1004-874X.2023.04.004 HUANG Y L, XU Y F, LI R H, LI G G, ZHANG H, GUO P G, XIA Y S. Genetic variations of AUF2 gene and their association with agronomic traits in flowering Chinese cabbage[J]. Guangdong Agricultural Sciences, 2023, 50(4): 33-41. DOI:10.16768/j.issn.1004-874X.2023.04.004 |
| [3] |
谭雪, 郭爽, 林锦英, 张华, 郑岩松, 谢伟平. 菜薹航天诱变后代变异及其RAPD标记多态性分析[J]. 中国蔬菜, 2011(10): 54-57. DOI:10.19928/j.cnki.1000-6346.2011.10.010 TAN X, GUO S, LIN J Y, ZHANG H, ZHENG Y S, XIE W P. Cabbage offspring variation of space flight mutation and analysis of genetic diversity by RAPD[J]. China Vegetables, 2011(10): 54-57. DOI:10.19928/j.cnki.1000-6346.2011.10.010 |
| [4] |
陈兆贵, 王愈. 菜心ISSR分子标记技术初步研究[J]. 种子, 2010, 29(3): 29-32. DOI:10.16590/j.cnki.1001-4705.2010.03.078 CHEN Z G, WANG Y. Preliminary study on identification flowering Chinese cabbage varieties by ISSR molecular markers[J]. Seed, 2010, 29(3): 29-32. DOI:10.16590/j.cnki.1001-4705.2010.03.078 |
| [5] |
郭培国, 王直亮, 陈静芳, 夏岩石, 李荣华, 张华, 李光光, 郑岩松, KADAMBOT S. 具通用性芸薹属SSR标记在菜心RIL群体中的偏分离分析[J]. 分子植物育种, 2016, 14(8): 2139-2146. DOI:10.13271/j.mpb.014.002139 GUO P G, WANG Z L, CHEN J F, XIA Y S, LI R H, ZHANG H, LI G G, ZHENG Y S, SIDDIQUE K. Segregation distortion analysis of transferable Brassica SSR marker in RIL population of flowering Chinese cabbage[J]. Molecular Plant Breeding, 2016, 14(8): 2139-2146. DOI:10.13271/j.mpb.014.002139 |
| [6] |
康红卫, 史卫东, 罗海玲, 周建辉, 踞茜茜, 张力, 农贵雄. 菜心抗虫性的cDNA-AFLP分析[J]. 西南农业学报, 2018, 31(6): 1135-1142. DOI:10.16213/j.cnki.scjas.2018.6.007 KANG H W, SHI W D, LUO H L, ZHOU J H, JU X X, ZHANG L, NONG G X. cDNA-AFLP analysis of insect resistance in flowering Chinese cabbage (Brassica rapa var. parachinensis)[J]. Southwest China Journal of Agricultural Sciences, 2018, 31(6): 1135-1142. DOI:10.16213/j.cnki.scjas.2018.6.007 |
| [7] |
夏岩石, 张润霖, 卢宇鹏, 李荣华, 李光光, 张华, 郭培国. 菜薹品质性状与SSR标记的关联分析[J]. 园艺学报, 2019, 46(10): 1960-1972. DOI:10.16420/j.issn.0513-353x.2019-0036 XIA Y S, ZHANG R L, LU Y P, LI R H, LI G G, ZHANG H, GUO P G. Association analysis of quality traits with SSR markers in flowering Chinese cabbage[J]. Acta Horticulturae Sinica, 2019, 46(10): 1960-1972. DOI:10.16420/j.issn.0513-353x.2019-0036 |
| [8] |
江定, 李光光, 张华, 戴修纯, 任海龙, 郑岩松. 基于转录组测序技术分析菜心SNP/InDel标记[J/OL]. 分子植物育种, 1-16[2023-11-26]. http://kns.cnki.net/kcms/detail/46.1068.S.20220303.1133.006.html. JIANG D, LI G G, ZHANG H, DAI X C, REN H L, ZHENG Y S. Analysis on SNP/InDel markers based on RNA-seq technology in flowering Chinese cabbage[J/OL]. Molecular Plant Breeding, 1-16[2023-11-26]. http://kns.cnki.net/kcms/detail/46.1068.S.20220303.1133.006.html. |
| [9] |
李欢, 张文洋, 田志强, 张震, 叶文超, 周子键, 陈甲法, 吴建宇. 高通量分子标记检测方法的研究进展[J]. 玉米科学, 2022, 30(3): 1-9. DOI:10.13597/j.cnki.maize.science.20220301 LI H, ZHANG W Y, TIAN Z Q, ZHANG Z, YE W C, ZHOU Z J, CHEN J F, WU J Y. Research progress of high-throughput molecular marker detection methods[J]. Journal of Maize Sciences, 2022, 30(3): 1-9. DOI:10.13597/j.cnki.maize.science.20220301 |
| [10] |
LIU B, WANG Y, ZHAI W, DENG J, WU J. Development of InDel markers for Brassica rapa based on whole-genome resequencing[J]. Theoretical and Applied Genetics, 2013, 126(1): 231-239. DOI:10.1007/s00122-012-1976-6 |
| [11] |
GUO G J, ZHANG G L, PAN B G, DIAO W P, LIU J B, GE W, GAO C Z, ZHANG Y, JIANG C, WANG S B. Development and application of InDel markers for Capsicum spp. based on whole-genome re-sequencing[J]. Scientific Reports, 2019, 9(1): 1-14. DOI:10.1038/s41598-019-40244-y |
| [12] |
MILLER M R, DUNHAM J P, AMORE A, CRESKO W A, JOHNSON E A. Rapid and costeffective polymorphism identification and genotyping using restriction site associated DNA (RAD) markers[J]. Genome Research, 2007, 17(2): 240-248. DOI:10.1101/gr.5681207 |
| [13] |
JIN L, ZHAO L P, WANG Y L, ZHOU R, SONG L X, XU L P, CUI X, LI R, YU W G, ZHAO T M. Genetic diversity of 324 cultivated tomato germplasm resources using agronomic traits and InDel markers[J]. Euphytica, 2019, 215: 69. DOI:10.1007/s10681-019-2391-8 |
| [14] |
KIZIL S, BASAK M, GUDEN B, TOSUN HS, UZUN B, YOL E. Genome-wide discovery of InDel markers in sesame (Sesamum indicum L.) using ddRADSeq[J]. Plants, 2020, 9(10): 1262. DOI:10.3390/plants9101262 |
| [15] |
LI H Y, IKRAM M, XIA Y S, LI R H, YUAN Q H, ZHAO W C, SIDDIQUE K H M, GUO P G. Genome-wide identification and development of InDel markers in tobacco (Nicotiana tabacum L.) using RAD-seq[J]. Physiology and Molecular Biology of Plants, 2022, 28(5): 1077-1089. DOI:10.1007/s12298-022-01187-3 |
| [16] |
孟霖, 刘博, 林良斌, 程峰, 王晓武, 武剑. 白菜型油菜和菜薹的InDel标记开发及其RILs群体遗传连锁图谱的构建[J]. 园艺学报, 2012, 39(8): 1491-1500. DOI:10.16420/j.issn.0513-353x.2012.08.009 MENG L, LIU B, LIN L B, CHENG F, WANG X W, WU J. Development of InDel markers for Brassica campestris and genetic linkage map construction of the RILs population[J]. Acta Horticulturae Sinica, 2012, 39(8): 1491-1500. DOI:10.16420/j.issn.0513-353x.2012.08.009 |
| [17] |
李光光, 黄红弟, 张华, 郑岩松. 菜心种子的活力及其品种耐热性鉴定[J]. 湖南农业大学学报(自然科学版), 2013, 39(1): 31-35. DOI:10.3724/SP.J.1238.2013.00031 LI G G, HUANG H D, ZHANG H, ZHENG Y S. Seed vigor and assessment for heat tolerance in different Chinese flowering cabbage cultivars[J]. Journal of Hunan Agricultural University (Natural Sciences), 2013, 39(1): 31-35. DOI:10.3724/SP.J.1238.2013.00031 |
| [18] |
李荣华, 郭培国, 张华, 黄红弟, 郑岩松, 夏岩石. 高温胁迫对菜心农艺性状的影响[J]. 长江蔬菜, 2011(22): 39-43. DOI:10.3865/j.issn.1001-3547.2011.22.015 LI R H, GUO P G, ZHANG H, HUANG H D, ZHENG Y S, XIA Y S. Effects of heat stress on agronomic traits of flowering Chinese cabbages[J]. Journal of Changjiang Vegetables, 2011(22): 39-43. DOI:10.3865/j.issn.1001-3547.2011.22.015 |
| [19] |
AHMED W, LI R H, XIA Y S, BAI G H, SIDDIQUE K, ZHANG H, ZHENG Y S, GUO P G. Comparative analysis of miRNA expression profiles between heat-tolerant and heat-sensitive genotypes of flowering Chinese cabbage under heat stress using high-throughput sequencing[J]. Genes, 2020, 11(3): 264. DOI:10.3390/genes11030264 |
| [20] |
许兰桂. 菜薹耐热性状的关联分析[D]. 广州: 广州大学, 2015. XU L G. Association analysis of heat tolerance in flowering Chinese cabbage[D]. Guangzhou: Guangzhou University, 2015. |
| [21] |
LI H, DURBIN R. Fast and accurate long-read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2010, 26(5): 589-595. DOI:10.1093/bioinformatics/btp698 |
| [22] |
LI H, HANDSAKER B, WYSOKER A, FENNELL T, RUAN J, HOMER N, MARTH G, ABECASIS G, DURBIN R. The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 |
| [23] |
WANG K, LI M Y, HAKONARSON H. ANNOVAR: Functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Research, 2010, 38(16): e164. DOI:10.1093/nar/gkq603 |
| [24] |
LIU W J, LI R H, AYALEW H, XIA Y S, BAI G H, YAN G J, SIDDIQUE K, GUO P G. Development of a simple and effective silver staining protocol for detection of DNA fragments[J]. Electrophoresis, 2017, 38(8): 1175-1178. DOI:10.1002/elps.201700009 |
| [25] |
LIU K J, MUSE S V. PowerMarker: An integrated analysis environment for genetic marker analysis[J]. Bioinformatics, 2005, 21(9): 2128-2129. DOI:10.1093/bioinformatics/bti282 |
| [26] |
洪晓如, 吴智明, 陈汉才, 黎庭耀, 沈卓, 张艳. 基于重测序的芥蓝(Brassica alboglabra) 全基因组InDel标记开发[J]. 分子植物育种, 2021, 19(4): 1190-1201. DOI:10.13271/j.mpb.019.001190 HONG X R, WU Z M, CHEN H C, LI T Y, SHEN Z, ZHANG Y. Development of InDel markers for Brassica alboglabra based on whole-genome re-sequencing data[J]. Molecular Plant Breeding, 2021, 19(4): 1190-1201. DOI:10.13271/j.mpb.019.001190 |
| [27] |
仪泽会, 卢有飞, 郭晓芹, 惠麦侠, 张鲁刚, 张明科. 大白菜简单序列重复(SSR) 和插入/缺失(InDel) 标记的开发及通用性分析[J]. 农业生物技术学报, 2012, 20(12): 1398-1406. DOI:10.3969/j.issn.1674-7968.2012.12.006 YI Z H, LU Y F, GUO X Q, HUI M X, ZHANG L G, ZHANG M K. Development of simple sequence repeat (SSR) and Insertion/Deletion (InDel) markers in Chinese cabbage (Brassica rapa ssp. pekinesis) and analysis of their transferability[J]. Journal of Agricultural Biotechnolog y, 2012, 20(12): 1398-1406. DOI:10.3969/j.issn.1674-7968.2012.12.006 |
| [28] |
陈正杰, 宛永璐, 钟文娟, 陈四维, 周永航, 石盛佳, 蒋理, 戢沛城, 杨泽湖, 毛正轩, 牟方生. 基于大豆基因组重测序的InDel标记开发及应用[J]. 植物遗传资源学报, 2021, 22(3): 815-833. DOI:10.13430/j.cnki.jpgr.20201028003 CHEN Z J, WAN Y L, ZHONG W J, CHEN S W, ZHOU Y H, SHI S J, JANG L, JI P C, YANG Z H, MAO Z X, MOU F S. Development and application of soybean InDel markers based on whole-genome resequencing datasets[J]. Journal of Plant Genetic Resources, 2021, 22(3): 815-833. DOI:10.13430/j.cnki.jpgr.20201028003 |
| [29] |
张志刚, 赵智中, 李巧云, 王晓, 刘栓桃, 王淑芬, 徐文玲, 刘贤娴, 刘辰. 大白菜InDels标记开发及其在剩余杂合体鉴定中的应用[J]. 农业生物技术学报, 2016, 24(4): 510-518. DOI:10.3969/j.issn.1674-7968.2016.04.005 ZHANG Z G, ZHAO Z Z, LI Q Y, WANG X, LIU S T, WANG S F, XU W L, LIU X X, LIU C. Development of InDels markers and their usage in detection of residual heterozygous lines in Chinese cabbage (Brassica rapa ssp. pekinensis)[J]. Journal of Agricultural Biotechnology, 2016, 24(4): 510-518. DOI:10.3969/j.issn.1674-7968.2016.04.005 |
| [30] |
李光光, 张华, 郑岩松, 李荣华. 利用SSR标记研究菜心资源的遗传多样性分析[J]. 基因组学与应用生物学, 2018, 37(3): 1257-1264. DOI:10.13417/j.gab.037.001257 LI G G, ZHANG H, ZHENG Y S, LI R H. The genetic diversity analysis of Chinese flowering cabbage resources based on SSR marker[J]. Genomics and Applied Biology, 2018, 37(3): 1257-1264. DOI:10.13417/j.gab.037.001257 |
| [31] |
史卫东, 琚茜茜, 张力, 罗海玲, 熊发前, 康红卫. 菜心种质资源遗传多样性的SCoT分析[J]. 南方农业学报, 2015, 46(8): 1350-1355. DOI:10.3969/j:issn.2095-1191.2015.08.1350 SHI W D, JU X X, ZHANG L, LUO H L, XIONG F Q, KANG H W. Analysis on genetic diversity of Chinese flowering cabbage (Brassica campestris L. ssp. chinensis Var. utilis Tsen et Le) germplasms based on SCoT markers[J]. Journal of Southern Agriculture, 2015, 46(8): 1350-1355. DOI:10.3969/j:issn.2095-1191.2015.08.1350 |
| [32] |
李桂花, 陈汉才, 张艳, 温艺敏, 陈琼贤, 张桂权. 菜心种质资源遗传多样性的SRAP分析[J]. 中国农学通报, 2012, 28(4): 110-114. DOI:10.3969/j.issn.1000-6850.2012.04.020 LI G H, CHEN H C, ZHANG Y, WEN Y M, CHEN Q X, ZHANG G Q. Genetic diversity of Brassica parachinensis germplasm revealed by SRAP analysis[J]. Chinese Agricultural Science Bulletin, 2012, 28(4): 110-114. DOI:10.3969/j.issn.1000-6850.2012.04.020 |
| [33] |
杨易, 黎庭耀, 李国景, 陈汉才, 沈卓, 周轩, 吴增祥, 吴新义, 张艳. 基于重测序的长豇豆基因组InDel标记开发及应用[J]. 园艺学报, 2022, 49(4): 778-790. DOI:10.16420/j.issn.0513-353x.2021-0347 YANG Y, LI T Y, LI G J, CHEN H C, SHEN Z, ZHOU X, WU Z X, WU X Y, ZHANG Y. Development and application of insertion-deletion (InDel) markers in Asparagus bean based on whole genome re-sequencing data[J]. Acta Horticulturae Sinica, 2022, 49(4): 778-790. DOI:10.16420/j.issn.0513-353x.2021-0347 |
| [34] |
卢宇鹏, 夏岩石, 温少波, 李荣华, 李光光, 张华, 郭培国. 不同熟性菜心品质性状的多样性分析[J]. 广东农业科学, 2020, 47(5): 29-36. DOI:10.16768/j.issn.1004-874X.2020.05.004 LU Y P, XIA Y S, WEN S B, LI R H, LI G G, ZHANG H, GUO P G. Diversity of quality traits of Chinese flowering cabbage varieties with different maturity[J]. Guangdong Agricultural Sciences, 2020, 47(5): 29-36. DOI:10.16768/j.issn.1004-874X.2020.05.004 |
| [35] |
IKRAM M, CHEN J F, XIA Y S, LI R H, SIDDIQUE K, GUO P G. Comprehensive transcriptome analysis reveals heat-responsive genes in flowering Chinese cabbage (Brassica campestris L. ssp. chinensis) using RNA sequencing[J]. Frontiers in Plant Science, 2022, 13: 1077920. DOI:10.3389/fpls.2022.1077920 |
| [36] |
林婷婷, 王立, 张琳, 王建军, 侯喜林, 李英. 不结球白菜叶绿素含量的主基因+多基因混合遗传分析[J]. 南京农业大学学报, 2014, 37(5): 34-40. DOI:10.7685/j.issn.1000-2030.2014.05.006 LIN T T, WANG L, ZHANG L, WANG J J, HOU X L, LI Y. Genetic analysis of chlorophyll content using mixed major gene plus polygene inheritance model in non-heading Chinese cabbage[J]. Journal of Nanjing Agricultural University, 2014, 37(5): 34-40. DOI:10.7685/j.issn.1000-2030.2014.05.006 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51