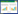文章信息
基金项目
- 广州市重点研发计划项目(202206010192);广东省科技计划项目(2021B1212050021);广东省畜禽疫病防治研究重点实验室项目(2023B1212060040)
作者简介
- 常鑫(1997—),女,在读本科生,研究方向为猪传染病防控,E-mail:2965392542@qq.com.
通讯作者
- 翟少伦(1983—),男,博士,研究员,研究方向为猪传染病防控,E-mail:zhaishaolun@163.com.
文章历史
- 收稿日期:2023-11-10
2. 广东省农业科学院动物卫生研究所/广东省畜禽疫病防治研究重点实验室/农业农村部兽用药物与诊断技术广东科学观测实验站,广东 广州 510640;
3. 广东永顺生物制药股份有限公司,广东 广州 511356
2. Institute of Animal Health, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory of Animal Disease Control and Research/Guangdong Scientific Observation and Experiment Station of Veterinary Drugs and Diagnostic Technology, Ministry of Agriculture and Rural Affairs, Guangzhou 510640, China;
3. Guangdong Winsun Biopharmaceutical Co., Ltd, Guangzhou 511356, China
【研究意义】猪圆环病毒(Porcine circovirus,PCV)属于圆环病毒科圆环病毒属,病毒粒子约17 nm,呈正20面体对称结构,是目前已知最小的DNA病毒[1]。PCV病毒粒子无囊膜,仅由蛋白质外壳和内部芯髓构成,对氯仿、乙醚等脂溶性消毒剂不敏感[2]。该病毒对环境的适应力极强,在56 ℃下可以存活,70 ℃条件下30 min才能被杀灭[3]。PCV2全基因组长度仅1 770 bp左右,但基因组极易发生组间突变和重组,导致PCV2具有遗传多样性,使得PCV2的流行呈现复杂化和多样化[4]。为了解广东省PCV2流行毒株的遗传变异情况,丰富PCV2基因数据库,有必要对广东省猪群开展系统流行病学调查及序列分析。【前人研究进展】PCV最早于1974年在PK-15细胞传代中被发现,当时被认为是细胞系污染源的一种,并无致病性[3]。1990年欧洲暴发仔猪断奶多系统衰竭综合征,后被证明其主要病原体为PCV,该致病性的PCV单克隆抗体无法对之前无致病性的PCV产生反应,因此把先前无致病性的PCV命名为PCV1,该致病性的PCV命名为PCV2[5]。2016年美国对疑似PCV2症状的临床样品通过宏基因组测序分析发现了PCV3[6-7]。2019年湖南大学研究团队在患有呼吸道症状的猪群中检测到PCV4[8]。目前共发现4个PCV血清型(PCV1~PCV4),致病性最强的是PCV2[9]。PCV2可以感染各年龄段的猪,是仔猪断奶多系统衰竭综合征、皮炎肾病综合征、母猪繁殖障碍综合征、仔猪先天性震颤等疾病的主要病原[10]。PCV2主要侵害猪群的免疫系统,使猪群抵抗外来病原微生物的能力降低,增加了猪群混合感染的风险。猪群感染PCV2后整体表现为饲料利用率下降,药物疗效降低[10-11],给养猪业造成巨大经济损失。PCV2共有11个开放阅读框,最大的2个阅读框是ORF1和ORF2,分别编码Rep蛋白(与病毒复制相关)、Cap蛋白(与病毒毒力相关)[5]。国内根据PCV2全基因组长度和Cap蛋白遗传进化距离将其分为5个基因亚型PCV2a~e[12-13],目前国内以PCV2d基因型流行为主[14]。【本研究切入点】课题组前期对广东省2022年临床样品进行PCV2检测时,获得1份PCV2病毒载量较高的阳性样品,荧光PCR结果显示该样品Ct值明显低于同期样品。为了探究该样品具有高病毒载量的原因,开展全基因组测序分析,从核酸和氨基酸水平分析该毒株与其他毒株的差异[15]。【拟解决的关键问题】通过全基因组PCR扩增、克隆后测序获得该毒株序列,并与GenBank中已经公布的PCV2毒株进行核苷酸相似性比对和遗传进化分析。深层次比对该毒株与同基因亚型毒株之间编码的Rep蛋白和Cap蛋白的氨基酸序列,探究其具有高病毒载量的原因。将该毒株序列与现有的PCV2疫苗株进行抗原指数比对,筛选适合本地区的疫苗株,以期为PCV2疫苗候选株的选用和研发提供参考。
1 材料与方法 1.1 试验材料1.1.1 病料来源 病料样品来自2022年广东省某猪场有PCV2临床症状的猪肺脏组织,编号为GD222858,样品保存在广东省农业科学院动物卫生研究所猪病研究室。
1.1.2 主要试剂 组织研磨用生理盐水购自辰欣药业股份有限公司(批号:H37022337),组织研磨陶瓷珠购自广州露卡测序仪器有限公司,核酸提取试剂盒购自康宁生命科学(吴江)有限公司,2×FastFire qPCR PreMix(Probe)、TOP10感受态细胞、DNase/RNase-Free Delonized Waster购自天根生化科技(北京)有限公司,PCRMIX购自南京诺唯赞生物科技有限公司,琼脂糖干粉购自上海贝晶生物技术有限公司,胶回收试剂盒购自苏州优逸兰迪生物科技有限公司,TSV-007VS-T载体连接试剂盒购自擎科生物科技有限公司,LB琼脂、LB肉汤购自广州环凯微生物科技有限公司,氨苄青霉素钠购自北京博奥拓达科技有限公司,质粒提取试剂盒购自Omega BioTek公司。
1.2 试验方法1.2.1 引物合成 荧光PCR(Probe)引物采用PCV2荧光PCR(qPCR)检测引物(GB/T 35901-2018)[16]。PCR全长扩增引物使用前期已建立的PCV2全基因组扩增引物(F:5'-TCCGCGGGCTGGCTGAACTTTTGA-3',R:5'-CCCGCGGAAATTTCTGACAAACGT-3')。引物均由生工生物(上海)工程股份有限公司合成。
1.2.2 样品DNA提取 将肺脏组织按照1 g样品加入1 mL生理盐水进行处理,混入研磨珠进行研磨,将研磨好的组织匀浆于-80 ℃下冻融3次,12 000 r/min离心2 min,取上清液按照病毒DNA提取试剂盒说明书提取核酸。
1.2.3 样品检测 将提取到的DNA进行PCV2 qPCR检测,反应体系(20 µL):2×FastFire qPCR PreMix(Probe)10 µL,上游引物、下游引物、探针各1 µL(稀释到标准使用浓度),DNA模板2 µL,ddH2O补足20 µL。反应程序:预反应50 ℃、2 min;预变性95 ℃、2 min;变性95 ℃、15 s;退火、延伸、采集荧光60 ℃、30 s,40个循环;冷却25 ℃、10 s。检测结束后根据扩增曲线和Ct值判定结果,筛选阳性样品。
1.2.4 PCV2全基因组扩增 PCR反应体系(25 µL):2×Taq plus Master Mix Ⅱ 12.5 µL,上、下游引物各1 µL,DNA模板2 µL,ddH2O补足25 µL。PCR反应程序:94 ℃预变性5 min;94℃变性20 s、63 ℃退火20 s、68 ℃延伸5 min,35个循环;68 ℃终延伸5 min。
1.2.5 目的片段克隆、测序 PCR产物经1.0%琼脂糖凝胶电泳检测,并于凝胶成像仪下切胶,使用胶回收试剂盒回收目的片段。将目的片段与TSV-007VS-T载体进行连接,连接体系10 µL:TSV-007VS-T载体2 µL,目的片段5 µL,DNase/RNase-Free Delonized Waster 3 µL。25 ℃金属浴30 min。将100 µL TOP10感受态细胞置于冰上融化,加入10 µL连接产物冰浴30 min,42 ℃热激90 s再冰浴2 min。加入900 µL不含抗生素的LB培养基震荡培养1 h,取200 µL培养后的菌液均匀涂布在含有氨苄青霉素抗性的LB琼脂平板上,37 ℃培养过夜。挑取单克隆菌落于氨苄青霉素抗性的LB肉汤培养基中震荡培养12 h。提取质粒并做鉴定,将阳性质粒送生工生物(上海)工程股份有限公司测序。
1.2.6 PCV2全基因组核苷酸序列比对与遗传进化分析 从GenBank数据库下载不同国家、地区、亚型的PCV2全基因组序列(表 1)。利用DNAStar中的MegAlign软件对本研究的PCV2克隆株与其他PCV2毒株序列进行核苷酸相似性比对,利用Mega7.0软件进行遗传进化分析。

|
1.2.7 PCV2 ORF1氨基酸序列比对 使用DNAStar中的MegAlign软件将样品ORF1基因编码的氨基酸序列与PCV2同亚型参考毒株进行比对,分析氨基酸序列的相似性。
1.2.8 PCV2 ORF2氨基酸序列比对及Cap蛋白分析 使用DNAStar中的MegAlign软件将样品ORF2基因编码的氨基酸序列与PCV2同亚型参考毒株进行比对,分析氨基酸序列的相似性;采用DNAStar中的Protean程序预测毒株的Cap蛋白二级结构及B细胞表位,并与4株疫苗株的Cap蛋白抗原指数进行比对分析。
2 结果与分析 2.1 PCV2荧光定量PCR检测结果使用PCV2荧光定量PCR检测方法(GB/T 35901-2018),检测出1株高病毒载量的毒株(GD222858),Ct值为13(图 1)。

|
| 1:GD222858扩增曲线 1: Amplification curve of GD222858 图 1 荧光定量PCR检测结果 Fig. 1 Fluorescence quantitative PCR test results |
2.2 PCV2全基因组扩增及测序
提取GD222858毒株核酸后进行全基因组扩增,PCR扩增产物凝胶电泳图上显示有1条约1 800 bp的条带,与预期大小一致(图 2)。对克隆后的阳性质粒进行测序,获得全基因组长度为1 767 bp,其中ORF1长945 bp、ORF2长705 bp,全基因组GC含量为48.73%。

|
| 1~5:全基因组扩增产物;M:DL2000 DNA maker;6:阴性对照 1-5: PCR amplification products of whole genome; M: DL2000 DNA maker; 6: Negative control 图 2 PCV2全基因组PCR扩增结果 Fig. 2 PCR amplification results of PCV2 whole genome |
2.3 PCV2全基因核苷酸序列比对与遗传进化分析
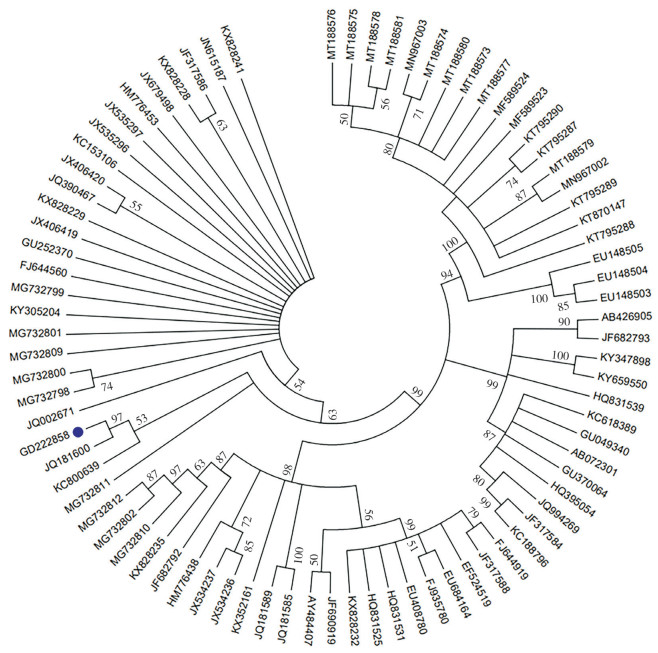
运用DNAStar、Mega7.0等生物信息学软件将获得的GD222858毒株序列与国内外82株PCV2毒株参考毒株(表 1)序列进行核苷酸序列比对与遗传进化分析。结果(图 3)显示,GD222858毒株与国内外82株参考毒株的核苷酸相似性为91.4%~99.6%,与越南毒株Han8(登录号:JQ181600)的亲缘关系最近,属于PCV2d亚型。
|
| 图 3 PCV2核苷酸序列系统进化树 Fig. 3 Phylogenetic tree of nucleic acid sequences of PCV2 |
2.4 PCV2 ORF1氨基酸序列比对分析
GD222858毒株的ORF1共945 bp,编码314个氨基酸。使用MegAlign软件将其ORF1基因核苷酸序列和编码的Rep蛋白氨基酸序列与PCV2同亚型的参考毒株进行比对,结果(图 4)显示,该毒株与参考毒株ORF1的核苷酸序列相似性为96.9%~100.0%,氨基酸序列的相似性为97.5%~100.0%。对GD222858毒株ORF1氨基酸位点进行分析,发现多个特异性突变位点F70Y、F77L、W202R、N256S。
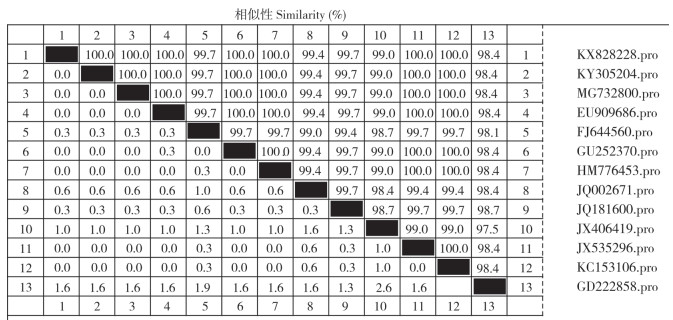
|
| 图 4 PCV2 Rep蛋白氨基酸序列相似性比对 Fig. 4 Similarity comparison of amino acid sequences of PCV2 Rep protein |
2.5 PCV2 ORF2氨基酸序列比对分析
GD222858毒株的ORF2共705 bp,编码234个氨基酸。使用MegAlign软件将GD222858毒株ORF2基因核苷酸序列和编码的Cap蛋白氨基酸序列与PCV2同亚型的参考毒株进行比对,结果(图 5)显示,该毒株与参考毒株ORF2的核苷酸序列相似性为97.3%~100.0%,氨基酸序列相似性为97.0%~100.0%。对GD222858毒株ORF2氨基酸位点进行分析,无发现特异性突变。

|
| 图 5 PCV2 Cap蛋白氨基酸序列相似性比对 Fig. 5 Similarity comparison of amino acid sequences of PCV2 Cap protein |
2.6 PCV2 Cap蛋白分析
GD222858毒株的Cap蛋白相对分子质量为27 957.93,等电点10.83,含有234个氨基酸,其中疏水氨基酸64个,占氨基酸总量的27.35%,偏亲水性。应用DNAStar中的Protean程序预测Cap蛋白二级结构,经Gamier-Robson方法预测显示,该Cap蛋白无α螺旋,β折叠占67.95%,转角区域占25.21%,卷曲区域占7.26%;经Chou-Fasman方法预测显示,该Cap蛋白α螺旋占9.83%,β折叠占39.32%,转角区域占41.03%。两种预测方法均表明该Cap蛋白整体偏向β折叠化。使用SWISS-MODEL在线网站建模的Cap蛋白三级结构与上述预测的Cap蛋白β折叠化相符(图 6)。使用Protean程序中Kyte-Doolittle、karplus-Schulz、JamesonWolf、Emini等方法对Cap蛋白进行亲水性、柔韧性、抗原性及表面可及性分析,结果(图 7)显示,在氨基酸第5~18、24~25、39~41、48~49、57~65、99、101、112~114、139~140、145~150、162~165、175~181、188~189、205~211、227~232位置处,均可能存在潜在的B细胞表位[17]。使用Jameson-Wolf方法对GD222858毒株与4株疫苗株进行抗原指数比对分析[18],结果(图 8)显示,该毒株Cap蛋白抗原指数与4株疫苗株均有差异,在氨基酸45~57、124~132、223~233位置处的抗原指数明显高于其他4株疫苗株,且与疫苗株HM038034差异最大。
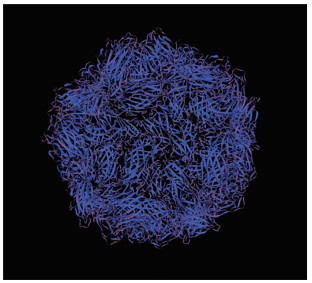
|
| 图 6 PCV2 Cap蛋白三级结构预测模型 Fig. 6 Prediction model of PCV2 Cap protein tertiary structure |
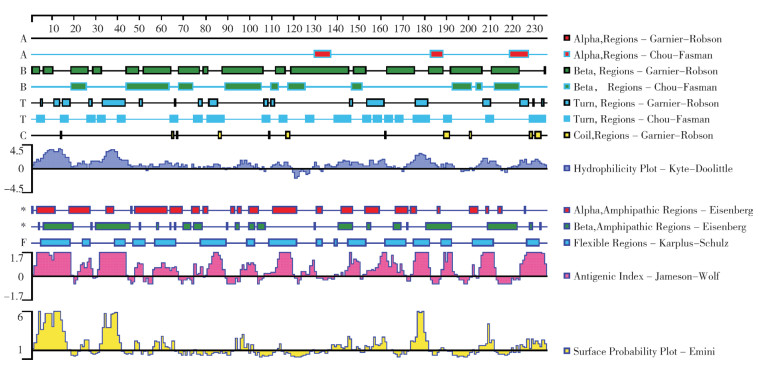
|
| 图 7 PCV2 Cap蛋白B细胞表位预测 Fig. 7 Prediction of B-cell epitope in PCV2 Cap protein |
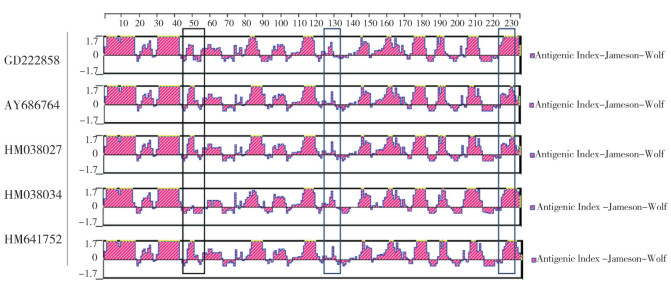
|
| 图 8 PCV2 Cap蛋白抗原指数预测 Fig. 8 Prediction of PCV2 Cap protein antigen index |
3 讨论
PCV2于2001年开始在我国南方地区流行,2003年在全国范围内流行和发生;2001—2012年间优势毒株逐渐由PCV2a演变为PCV2b;2012年至今,优势毒株又由PCV2b演变为PCV2d[19]。近年PCV2流行病学调查结果显示,我国流行的优势毒株仍然以PCV2d为主[20-21]。PCV2是一种高突变性、高传染性的病原,可以多途径传播,并且极易发生组间重组[4]。疫苗免疫是防制PCV2感染的最有效途径,选择安全有效的疫苗对PCV2的防控极为重要[10]。
PCV2 ORF1编码的Rep蛋白与病毒复制相关,Rep蛋白及其剪切体Rep′是PCV2复制所需的重要蛋白。研究表明,定点突变PCV2 Rep蛋白的部分氨基酸序列将严重影响PCV2的复制,或失去复制能力或增强复制能力且病毒载量远高于原感染毒株[22]。对GD222858毒株ORF1氨基酸位点进行多序列比对分析发现多个特异性突变位点(F70Y、F77L、W202R、N256S)。ORF2编码的Cap蛋白是PCV2的主要结构蛋白,与致病性有关。对GD222858毒株ORF2氨基酸位点进行多序列比对分析,并未发现特异性突变。推测该毒株具有高病毒载量的原因与多个Rep蛋白突变位点相关[23]。研究发现,如果抗原表位中有1个氨基酸突变,则突变后的病毒将不能被表位抗体识别[24]。使用Protean程序中Kyte-Doolittle、karplus-Schulz、Jameson-Wolf、Emini等方法对GD222858毒株的Cap蛋白进行亲水性、柔韧性、抗原性及表面可及性分析,结果显示,在氨基酸第5~18、24~25、39~41、48~49、57~65、99、101、112~114、139~140、145~150、162~165、175~181、188~189、205~211、227~232位置处均可能存在潜在B细胞表位,MegAlign软件对GD222858毒株的ORF2氨基酸位点分析发现,潜在的B细胞抗原表位上没有发生突变。综上,该毒株具有高病毒载量的原因与Cap蛋白无关。将GD222858毒株的Cap蛋白与4株疫苗株进行抗原指数比对分析,结果显示,GD222858毒株的Cap蛋白抗原指数与4株疫苗株均有差异,在氨基酸45~57、124~132、223~233位置处抗原指数明显高于其他4株疫苗株,且与疫苗株HM038034差异最大。病毒在机体中的增殖较为复杂,参考前人研究的试验数据[22],推测GD222858毒株感染猪群的原因可能是Rep蛋白多个位点发生特异性突变及疫苗株选用不当所致[25-26]。GD222858毒株的感染性和致病性有待进一步研究。
4 结论本研究对广东省PCV2毒株GD222858进行测序和遗传进化分析,以探究其具有高病毒载量的原因。结果表明,GD222858毒株为PCV2d亚型,全基因组长度为1 767 bp,其中ORF1长945 bp,编码的Rep蛋白处发现多个特异性突变位点(F70Y、F77L、W202R、N256S);ORF2长705 bp,编码的Cap蛋白相对保守,预测Cap蛋白氨基酸第5~18、24~25、39~41、48~49、57~65、99、101、112~114、139~140、145~150、162~165、175~181、188~189、205~211、227~232位置处均可能存在潜在的B细胞表位;对ORF2氨基酸位点分析表明,潜在的B细胞抗原表位上没有发生突变。研究发现,GD222858毒株的Cap蛋白抗原指数与4株疫苗株均有差异,在氨基酸45~57、124~132、223~233位置处抗原指数明显高于其他4株疫苗株,且与疫苗株HM038034差异最大。推测GD222858毒株感染猪群的原因可能是Rep蛋白多个位点发生特异性突变及疫苗株选用不当所致。
| [1] |
王卓. 猪圆环病毒跨物种传播研究进展[J]. 兽医导刊, 2021(13): 136-137. WANG Z. Research progress on cross-species transmission of porcine circovirus[J]. Veterinary Orientation, 2021(13): 136-137. |
| [2] |
李天芝, 于新友, 李峰. 我国猪圆环病毒的分类、流行现状及分子生物学检测方法研究进展[J]. 养猪, 2022(2): 124-128. DOI:10.13257/j.cnki.21-1104/s.2022.02.025 LI T Z, YU X Y, LI F. Research progress on classification, epidemic status and molecular biology detection methods of porcine circovirus in China[J]. Swine Production, 2022(2): 124-128. DOI:10.13257/j.cnki.21-1104/s.2022.02.025 |
| [3] |
何治富, 郑慧慧, 王万, 曹永斌. 猪圆环病毒病的研究进展[J]. 兽医导刊, 2021(7): 123. HE Z F, ZHENG H H, WANG W, CAO Y B. Research progress of porcine circovirus disease[J]. Veterinary Orientation, 2021(7): 123. |
| [4] |
池雪林, 程洁, 毛文潇, 杨新炜, 吴星星, 张莹, 曾显成. 福建地区猪圆环病毒2型全基因序列遗传演化分析及病理学观察[J]. 动物医学进展, 2020, 41(10): 6-14. DOI:10.16437/j.cnki.1007-5038.2020.10.002 CHI X L, CHENG J, MAO W X, YANG X W, WU X X, ZHANG Y, ZENG X C. Phylogenetic analysis of complete genome equences of porcine circovirus type 2 strains and histopatho logical observations in Fujian Province[J]. Progress in Veterinary Medicine, 2020, 41(10): 6-14. DOI:10.16437/j.cnki.1007-5038.2020.10.002 |
| [5] |
乔翠, 高凤山, 许崇波. 猪圆环病毒2型分子致病机理及相关疾病研究进展[J]. 中国畜牧兽医, 2012, 39(2): 170-173. QIAO C, GAO F S, XU C B. Studying on the development of molecula ar pathogenesis and related infectious disease in porcine e circle virus 2[J]. China Animal Husbandry & Veterinary Medicine, 2012, 39(2): 170-173. |
| [6] |
吕其壮, 覃婷, 龚紫凤, 农可懿, 梁小妹, 覃新云, 陈旭健. 猪圆环病毒3型流行病学研究进展[J]. 中国预防兽医学报, 2021, 43(5): 567-571. DOI:10.3969/j.issn.1008-0589.202008050 LYU Q Z, QIN T, GONG Z F, NONG K Y, LIANG X M, TAN X Y, CHEN X J. Epidemiological research progress on porcine circovirus type 3[J]. Chinese Journal of Preventive Veterinary, 2021, 43(5): 567-571. DOI:10.3969/j.issn.1008-0589.202008050 |
| [7] |
刘道泉. 猪圆环病毒3型感染流行病学研究进展[J]. 中国动物检疫, 2022, 39(8): 92-96. LIU D Q. Advances in the epidemiological researches on porcine circovirus 3 infection[J]. China Animal Health Inspection, 2022, 39(8): 92-96. |
| [8] |
祝羊, 梁海英, 曾智勇, 陈娟, 黄二素, 徐松平, 徐玉. 猪圆环病毒4型研究进展[J]. 中国动物传染病学报, 2023, 31(2): 197-202. DOI:10.19958/j.cnki.cn31-2031/s.20210611.001 ZHU Y, LIANG H Y, ZENG Z Y, CHEN J, HUANG E S, XU S P, XU Y. Research progress of porcine circovirus 4[J]. Chinese Journal of Animal Infectious Diseases, 2023, 31(2): 197-202. DOI:10.19958/j.cnki.cn31-2031/s.20210611.001 |
| [9] |
王圣伟, 李相钊, 李富金. 猪圆环病毒的研究进展及防控措施[J]. 今日养猪业, 2022(3): 17-20. WANG S W, LI X Z, LI F J. Research progress and prevention and control measures of porcine circovirus[J]. Pigs Today, 2022(3): 17-20. |
| [10] |
朱志刚. 猪圆环病毒的流行现状与防控进展[J]. 山东畜牧兽医, 2023, 44(5): 73-75. ZHU Z G. Epidemic status and prevention and control progress of porcine circovirus[J]. Shandong Journal of Animal Science and Veterinary Medicine, 2023, 44(5): 73-75. |
| [11] |
邓同炜, 薛亚南, 卢建洲, 夏艳勋, 陈露露, 郭懿文, 彭丽明, 蒋增海, 乔宏兴, 徐耀辉, 张晓战. 猪繁殖与呼吸综合征病毒、猪圆环病毒2型与副猪嗜血杆菌混合感染的诊断及病原分析[J]. 中国畜牧兽医, 2020, 47(10): 3401-3409. DOI:10.16431/j.cnki.1671-7236.2020.10.040 DENG T W, XUE Y N, LU J Z, XIA Y X, CHEN L L, GUO Y W, PENG L M, JIANG Z H, QIAO H X, XU Y H, ZHANG X Z. Diagnosis and pathogen analysis of mixed infection of porcine reproductive and respiratory syndrome virus, porcine circovirus type 2 and haemophilus parasuis[J]. China Animal Husbandry & Veterinary Medicine, 2020, 47(10): 3401-3409. DOI:10.16431/j.cnki.1671-7236.2020.10.040 |
| [12] |
CORTEY M, OLVERA A, GRAU-ROMA L, JOAQUIM S. Further comments on porcine circovirus type 2 (PCV2) genotype definition and nomenclature[J]. Veterinary Microbiology, 2011, 149(34): 522-523. DOI:10.1016/j.vetmic.2010.11.009 |
| [13] |
SEGALES J, OLVERA A, GRAU-ROMA L, CHARREYRE C, NAUWYNCK H, LARSEN L, DUPONT K, MCCULLOUGH K, ELLIS J, KRAKOWKA S, MANKERTZ A, FREDHOLM M, FOSSUM C, TIMMUSK S, STOCKHOFE-ZURWIEDEN N, BEATTIE V, ARMSTRONG D, GRASSLAND B, BAEKBO P, ALLAN G. PCV2 genotype definition and nomenclature[J]. Veterinary Record, 2008, 162(26): 867-868. DOI:10.1136/vr.162.26.867 |
| [14] |
BAO F, MI S, LUO Q, GUO H, T U C, ZHU G, GONG W. Retrospective study of porcine circovirus type 2 infection reveals a novel genotype PCV2f[J]. Transbound and Emerging Diseases, 2018, 65(2): 432-440. DOI:10.3389/fvets.2022.932612 |
| [15] |
黄冠雄, 许丹宁, 杨舒展, 郭斯璇, 曾依翎, 黄运茂, 田允波, 曹楠. 广东省鹅细小病毒的分离及序列分析[J]. 广东农业科学, 2018, 45(5): 98-10. DOI:10.16768/j.issn.1004-874X.2018.05.017 HUANG G X, XU D N, YANG S Z, GUO S X, ZENG Y L, HUANG Y M, TIAN Y B, CAO N. Isolation and phylogenetic analysis of goose parvovirus isolated in Guangdong Province[J]. Guangdong Agricultural Sciences, 2018, 45(5): 98-10. DOI:10.16768/j.issn.1004-874X.2018.05.017 |
| [16] |
赵凯, 杨丹, 张晓霞, 张琪, 张皖静, 浦心祎, 赵斌安, 赵悦琪, 钱忠辉, 张俊屏, 丁卫星, 张春玲, 赵本进, 王瑞阳, 张华弟. 一种猪圆环病毒2型的荧光定量PCR检测方法及试剂盒[P]. 中国: CN114015808A, 2022-02-08. ZHAO K, YANG D, ZHANG X X, ZHANG Q, ZHANG W J, PU X Y, ZHAO B A, ZHAO Y Q, QIAN Z H, ZHANG J P, DING W X, ZHANG C L, ZHAO B J, WANG R Y, ZHANG H D. Fluorescence PCR detection method and kit for porcine circovirus type 2[P]. China: CN114015808A, 2022-02-08. |
| [17] |
王丽娜, 贺显晶, 蒋剑成, 汪锋锋, 肖佳薇, 蒋凯, 赵鹏宇, 王天硕, 于思雯, 毕栏, 郭东华. 牛坏死杆菌43K OMP的生物信息学分析及B细胞表位预测[J]. 黑龙江八一农垦大学学报, 2022, 34(3): 41-49. DOI:10.3969/j.issn.1002-2090.2022.03.007 WANG L N, HE X J, JIANG J C, WANG F F, XIAO J W, JIANG K, ZHAO P Y, WANG T S, YU S W, BI L, GUO D H. Bioinformatics analysis and B Cell epitope prediction of 43K OMP from bovine fusobacterium necr rophrum[J]. Journal of Heilong jiang Bayi Agricultural University, 2022, 34(3): 41-49. DOI:10.3969/j.issn.1002-2090.2022.03.007 |
| [18] |
羊露露, 李安琪, 袁生, 黄淑坚, 温峰, 郭锦玥. 广东省4株PCV2型分离株全基因组进化分析[J]. 中国畜牧兽医, 2022, 49(3): 1015-1023. DOI:10.16431/j.cnki.1671-7236.2022.03.023 YANG L L, LI A Q, YUAN S, HUANG S J, WEN F, GUO J Y. Complete genome evolution analysis of four porcine circovirus type 2 isolates from Guangdong Province[J]. China Animal Husbandry & Veterinary Medicine, 2022, 49(3): 1015-1023. DOI:10.16431/j.cnki.1671-7236.2022.03.023 |
| [19] |
徐丽华, 蓝胜芝, 余斌, 李军星, 张鹏超, 李宝臣, 苏菲, 袁秀芳. 浙江地区猪圆环病毒2型检测及遗传变异分析[J]. 浙江农业学报, 2020, 32(11): 1970-1977. DOI:10.3969/j.issn.1004-1524.2020.11.06 XU L H, LAN S Z, YU B, LI J X, ZHANG P C, LI B C, SU F, YUAN X F. Detection and genetic variation analysis of prevalent porcine circovirus type 2 strains in Zhejiang Province[J]. Acta Agriculturae Zhejiangensis, 2020, 32(11): 1970-1977. DOI:10.3969/j.issn.1004-1524.2020.11.06 |
| [20] |
NAN W, WU J, HU H, PENG G, TAN S, DENG Z. Prevalence and genetic diversity of porcine circovirus type 2 in northern Guangdong Province during 2016-2021[J]. Frontiers in Veterinary Science, 2022(9): 932612. DOI:10.3389/fvets.2022.932612 |
| [21] |
YANG S, YIN S, SHANG Y, LIU B, YUAN L, ZAFAR KHAN M, LIU X, CAI J. Phylogenetic and genetic variation analyses of porcine circovirus type 2 isolated from China[J]. Transboundary and Emerging Diseases, 2018, 65(2): e383-e392. DOI:10.1111/tbed.12768 |
| [22] |
李荷然, 肖琦, 温立斌, 朱雪蛟, 芮荣, 何孔望. 猪圆环病毒2型ORF1部分位点突变对病毒复制能力的影响[J]. 畜牧与兽医, 2024, 56(1): 71-76. LI H R, XIAO Q, WEN L B, ZHU X J, RUI R, HE K W. Effect of partial site mutations of porcine circovirus type 2 ORF1 on its replication abillity[J]. Animal Husbandry & Veterinary Medicine, 2024, 56(1): 71-76. |
| [23] |
孟张丽, 梁鹏帅, 夏平安, 刘燕玲, 李艳, 蒋智勇, 蔡汝健, 宋长绪. 广东省猪圆环病毒2型毒株分离及全基因序列分析[J]. 广东农业科学, 2014, 41(7): 149-154. DOI:10.16768/j.issn.1004-874X.2014.07.006 MENG Z L, LIANG P S, XIA P A, LIU Y L, LI Y, JIANG Z Y, CAI R J, SONG C X. lsolation and sequence analysis of PCV2 strains from Guangdong Province[J]. Guangdong Agricultural Sciences, 2014, 41(7): 149-154. DOI:10.16768/j.issn.1004-874X.2014.07.006 |
| [24] |
LI D, DU Q, WU B, LI J, CHANG L, ZHAO X, HUANG Y, TONG D. Immunogenicity of adenovirus vaccines expressing the PCV2 capsid protein in pigs[J]. Vaccine, 2017, 35(36): 4722-4729. DOI:10.1016/j.vaccine.2017.07.031 |
| [25] |
薛瑞雪, 田野, 杨婉婷, 王贵升, 马慧玲, 李玉杰, 王苗利, 蔺晓月, 陈峰, 孙圣福, 李云岗. 山东部分地区猪圆环病毒2型遗传变异分析[J]. 中国预防兽医学报, 2021, 43(2): 198-201, 218. DOI:10.3969/j.issn.1008-0589.202005021 XUE R X, TIAN Y, YANG W T, WANG G S, MA H L, LI Y J, WANG M L, LIN X Y, CHEN F, SUN S F, LI Y G. Genetic variation analysis of porcine circovir rus type 2 strains in Shandong Province[J]. Chinese Journal of Preventive Veterinary, 2021, 43(2): 198-201, 218. DOI:10.3969/j.issn.1008-0589.202005021 |
| [26] |
曹宗喜, 郑心力, 张艳, 叶保国, 林哲敏, 王峰. PCV2海南株全基因组的克隆与序列分析[J]. 广东农业科学, 2015, 42(8): 118-123. DOI:10.16768/j.issn.1004-874X.2015.08.001 CAO Z X, ZHENG L X, ZHANG Y, YE B G, LIN Z M, WANG F. Cloning and sequence analysis of complete genome of porcine circovirusisolated from Hainan Province[J]. Guangdong Agricultural Sciences, 2015, 42(8): 118-123. DOI:10.16768/j.issn.1004-874X.2015.08.001 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51