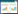文章信息
基金项目
- 上海市自然科学基金(20ZR1423600);上海扬帆人才计划项目(19YF1419400);国家自然科学基金(31902376)
作者简介
- 张纬宇(1997—),男,在读硕士生,研究方向为观赏鱼遗传育种,E-mail:zwyZWY971114@163.com.
通讯作者
- 陈再忠(1972—),男,博士,教授,研究方向为水产动物繁殖生物学、观赏鱼养殖,E-mail:chenzz@shou.edu.cn.
文章历史
- 收稿日期:2023-05-09
【研究意义】我国是观赏鱼产业大国,观赏鱼种类繁多[1]。具有透明性状的鱼类不仅有极高的观赏价值,而且因其内部脏器结构显而易见而成为重要的科研材料之一,目前已被广泛应用于器官发育和病原感染等相关领域的研究[2-3]。在生命初期阶段,大多数鱼类的身体都是透明的,随着组织中色素细胞的累积(尤其皮肤、腹膜组织),透明表型便在成年后逐渐褪去。成熟的色素细胞主要经由鳞片和皮肤中的神经嵴细胞迁移至特定部位后发育分化而成[4-5],根据其形态结构、色素色和呈色物质可分为黄色素细胞、黑色素细胞、白色素细胞、红色素细胞、蓝色素细胞和虹彩色素细胞6大类,其中黑色素细胞、白色素细胞、虹彩色素细胞可介导鱼类白色、灰色及透明体色的形成[6-8]。研究鱼类透明体色性状形成机制对提升鱼类观赏性具有一定意义。【前人研究进展】神仙鱼(Pterophyllum scalare)属于脊索动物门鲈形目丽鱼科(慈鲷科)天使鱼属[9-10],也有部分学者将其归为鲈形目髭鲷科叶鳍鱼属[11],原产于南美洲亚马逊河流域,享有“热带鱼皇后”“观赏鱼皇后”的美称[10]。经过长期的人工选择,神仙鱼已发展成黑神仙、金头神仙、红眼神仙、玻璃神仙、三色神仙等多个品种,其中黑神仙和三色神仙最为名贵。神仙鱼可根据其是否透明进行细分,例如白色玻璃神仙鱼全身透明,可见鱼骨,为观赏上品[12]。转录组学技术在水产动物代谢、发育和进化等研究方面发挥着重要作用,通过该技术可以精确测定特定基因的表达水平,分析基因和通路的富集差异,以解决许多与生物相关的问题[13-15]。因此基于转录组学技术开展神仙鱼透明性状相关研究具有极大的经济价值和科研价值。目前国内外对于神仙鱼的研究较少,主要集中于繁殖技术[16-18]、病虫害防治[19]、鱼类种类鉴定[20-21]方面。国内外对于神仙鱼透明性状相关的研究暂未见报道,但已有部分透明性状相关研究在其他鱼类[2, 22-26]中开展。Wakamatsu等[2]基于青鳉鱼构建了一种改进的透明青鳉鱼模型,该模型通过转基因技术生成与虹彩色素细胞相关的基因突变体,该突变体可在成鱼阶段和胚胎阶段均保持透明,且透明性状可遗传至后代,为鱼类胚胎后阶段的器官发生、生长、衰老,以及其他疾病的发生等研究提供了基础。李凯彬等[22]研究发现了一个突变的鲫鱼种群,该突变种群全身均透明,推测透明性状可通过后代遗传。Xu等[23]通过观察透明鲫鱼和红草金鱼分别杂交和自交的子代体色发现,透明性状为显性遗传。史东杰等[24]首次对红白锦鲤皮肤进行转录组学测序分析,发现红白锦鲤皮肤主要受132条代谢通路影响。黎学友等[25]对湖栖鳍虾虎鱼(Gobiopterus lacustris)黑色素聚集的眼睛和透明皮肤进行转录组学差异表达分析,共筛选到8 113个差异基因,黑色素合成通路中的tyr、tyrp1、tyrp2基因被筛选为透明体色直接相关的基因,为开展鱼类透明皮肤的机制研究提供了数据支持。【本研究切入点】近年我们在神仙鱼研究中发现鳃盖透明的表型(肉眼可直接通过腮盖观察到鱼体透明的鳃丝),这种神仙鱼可育且透明性状可通过子代遗传;透明鳃盖和不透明鳃盖的神仙鱼生殖周期无明显区别;从可清楚观察到鳃盖器官开始(第4周),直至观察结束(2年),鱼体鳃盖均表现为透明,透明程度不随时间的增长而变化。【拟解决的关键问题】本研利用转录组学测序技术,以神仙鱼鳃盖组织为研究材料,寻找影响神仙鱼透明鳃盖的相关基因,探究这些基因所涉及的生物途径,可为后续神仙鱼透明鳃盖性状相关基因的挖掘和功能研究提供基础数据。
1 材料与方法 1.1 试验材料及样本采集供试的神仙鱼为红顶三色品系,包括透明鳃盖(Transparent gill cover,TGC)个体和不透明鳃盖(Opaque gill cover,OGC)个体,均由上海蓝海水产发展有限公司观赏鱼养殖基地提供。2022年12月15日从同一养殖池随机挑选1龄性成熟的TGC、OGC神仙鱼各6尾作为试验材料,这些神仙鱼生长环境及时间、喂养条件均一致,大小形态均相近(图 1)。使用鱼类麻醉剂MS-222麻醉神仙鱼,从鳃部取下鳃盖后置于液氮中迅速冷冻后放于-80 ℃冰箱中保存备用。

|
| 图 1 神仙鱼鳃盖取样示意图 Fig. 1 Diagram of gill cover of Pterophyllum scalare sampling |
1.2 测序样本制备
使用Trizol Reagent试剂盒(赛默飞世尔科技公司)提取神仙鱼腮盖总RNA,使用Nanodrop2000仪器检测RNA的浓度和纯度,使用Agilent2100仪器检测RNA的降解情况,使用琼脂糖凝胶电泳检测RNA的完整性。上机样本质控标准:RNA总量≥ 1 μg,浓度≥ 35 ng/μL,OD260/280 ≥ 1.8,OD260/230 ≥ 1.0。
1.3 RNA测序、序列拼接和功能注释测序样本使用Illumina Novaseq 6000平台(上海美吉生物医药科技有限公司)进行测序,测序原始数据使用Fastp软件(Version 0.19.5,https://github.com/OpenGene/fastp)进行质控,质控原则为:(1)去除接头序列(Adapter),去除因接头自连等原因导致没有插入片段的Reads;(2)将序列末端(3' 端)质量值小于20的碱基修剪掉;(3)去除含N(模块碱基)的Reads;(4)去掉经过去除Adapter及质量修剪后长度小于30 bp的序列。利用Trinity软件(Version 2.8.5,https://github.com/trinityrnaseq/trinityrnaseq/wiki)对质控后的数据进行转录本从头拼接和组装,将组装后最长的转录本(Transcript)作为单基因(Unigene)。利用CD-HIT软件(Version 4.5.7,https://github.com/weizhongli/cdhit)和BUSCO软件(Version 3.0.2,https://busco.ezlab.org/)分别对初始组装序列进行去冗余和评估。
基于序列同源性将过滤评估后所得的Unigene分别与以下数据库进行比对,以获得Unigene的功能注释信息:GO(Version 2022.09.15,http://www.geneontology.org/)、KEGG(Version 2022.10,http://www.genome.jp/kegg/)、NR(Version 2022.10,https://www.ncbi.nlm.nih.gov/public/)、Pfam(Version 35.0,http://pfam.xfam.org/)、Swiss-Prot(Version 2022.10,ftp://ftp.uniprot.org/pub/databases/uniprot/current_release/knowledgebase/complete/uniprot_sprot.fasta.gz)、eggNOG(Version 2020.06,http://eggnogdb.embl.de/#/app/home)。
1.4 基因差异表达分析使用RSEM软件(Version 1.3.1,http://deweylab.github.io/RSEM/)计算每个基因的TPM(样本中平均每一百万个转录本中对应特定基因或转录本的数量)表达值;使用DESeq2软件(Version 1.24.0,http://bioconductor.org/packages/stats/bioc/DESeq2/)筛选TGC和OGC之间的差异表达基因(DEG)。差异表达基因筛选标准为:经错误发现率(False discovery rate,FDR)方法多重校正后P<0.05且|log2(fold change)| > 1。分别绘制差异表达基因的火山图和热图,对差异表达分析结果进行可视化展示。
1.5 差异表达基因富集分析分别使用Goatools软件(Version 0.6.5,https://github.com/tanghaibao/GOatools)和KOBAS软件(Version 3.0,http://kobas.cbi.pku.edu.cn/home.do)对差异表达基因进行GO和KEGG富集分析,寻找差异表达基因参与的生化信号途径。GO富集分析将基因按照参与的生物学过程、细胞组分和分子功能进行分类。GO和KEGG富集分析使用方法均为Fisher精确检验。为控制计算的假阳性率,使用FDR多重检验方法对P值进行校正,Padjust<0.5则视为存在显著富集情况。
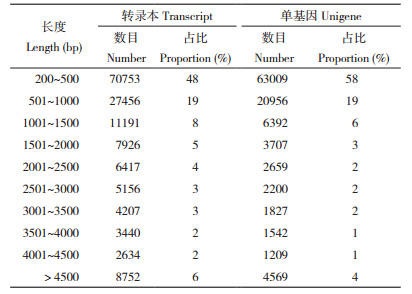
2 结果与分析 2.1 神仙鱼透明鳃盖与不透明鳃盖转录组测序注释分析2.1.1 测序数据质控 使用Illumina Novaseq 6000测序平台分别对12个神仙鱼鳃盖样本(OGC、TGC)进行测序,分别统计每1个样本的原始序列数,以及质控后的序列数、错误率、Q20、Q30、GC含量(表 1)。12个样本共获得82.69 Gb Clean data,各样品Clean data均达到6.22 Gb以上。由表 1可知,12个样本的测序原始序列数均超过42 275 394条Reads;对原始序列数过滤掉接头、低质量reads和短序列Reads后,每个样本质控后的序列数均超过41 904 944条高质量的Reads,每个样本对应的测序碱基平均错误率均低于0.1%,测序质量Q20碱基百分比≥ 97.78%,Q30碱基百分比≥ 93.58%,GC含量为47.04%~48.90%,表明测序数据质量可靠,可进行下游的组装分析。
|
2.1.2 组装结果统计 使用Trinity软件对得到的Clean reads进行从头组装后共获得200 303个Transcrip和317 938 823个碱基,对应123 178个Unigene和111 300 861个碱基。初始组装对应的最大序列长度为24 631 bp,最小测序长度为201 bp,Transcrip和Unigene的平均长度为1 587.29、903.58 bp,分别对应的N50长度为3 681、2 292 bp,E90N50长度分别为3 580和4 627 bp。所有样本的Clean reads合并后与组装后的Transcript比对,获得的片段匹配率为79.436%,Transcript对应的Unigene片段匹配率为69.128%,Transcript和Unigene对应的GC百分比分别为44.94% 和43.77%。将初始组装的Transcript和Unigene分别去冗余后共获得147 932个Transcript和108 070个Unigene,对应的总碱基数目分别为191 633 294、107 188 786个。去冗余后Transcript、Unigene最大长度和最小长度分别为24 543、201 bp;Transcript和Unigene的平均长度分别为1 295.41、991.85 bp,N50长度分别为2 928、2 430 bp,E90N50长度分别为3 827、4 571 bp。片段匹配率分别为82.18% 和69.164%,GC百分比分别为44.47% 和43.84%(表 2)。不论是初始组装还是去冗余优化后的片段匹配率和GC百分比均在正常范围内,说明组装结果可信。
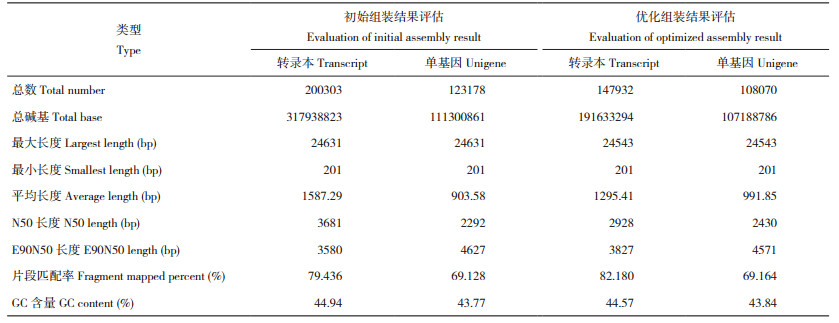
对组装去冗余后的Unigene、Transcript序列长度进行统计,结果(表 3)显示,序列长度<500 bp的Transcript数目最多、为70 753个,占总体48%;Transcript对应63 009个基因,占总体58%。综上表明转录拼接较好,利于后续分析。
2.1.3 Unigene功能注释 将转录组测序获得的Unigene信息与6个数据库(NR、Swiss-Prot、Pfam、eggNOG、GO和KEGG)进行注释比对,分别统计Unigene在各数据库中注释的数目。结果(图 2A)显示,6个数据库中NR数据库注释基因数目最多、为39 738个,占数据库总体基因比例37.56%;Pfam数据库注释基因数目最少、为19 423个,占数据库总体基因比例18.36%;6个数据库总体注释基因40 180个,占总体基因比例37.98%。对6个数据库对应的注释基因取交集,结果(图 2B)显示,10 747个基因同时在6个数据库中被注释,占总体数据库比例26.75%。
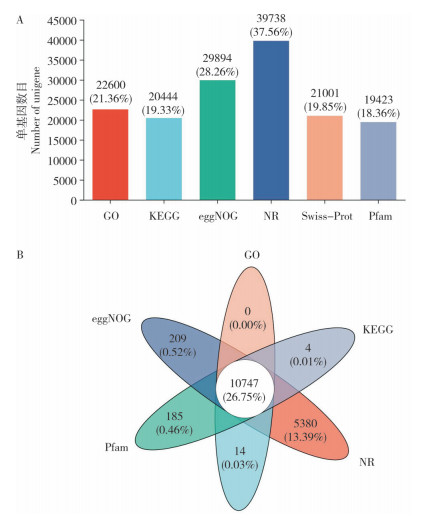
|
| A:6个数据库中Unigene的功能注释;B:6个数据库注释基因Venn图A: Functional annotation of unigene in 6 databases; B: Venn diagrams of annotated gene in 6 databases 图 2 Unigene功能注释统计 Fig. 2 Statistical result of unigene functional annotation |
2.2 神仙鱼透明鳃盖与不透明鳃盖基因相对表达量分析
为了比较OGC和TGC基因相对表达量的差异,对OGC和TGC两组鉴定到的基因数目进行统计,结果(图 3A)显示,OGC组共注释到32 248个基因,TGC组共注释到36 476个基因,其中24 211个基因均在两组中被注释,8 037个基因为OGC组特有,12 265个基因为TGC组特有。基于RSEM软件,利用比对至基因组的结果和基因组注释文件,获得每个样本基因的Read counts,对其进行TPM转换得到12个样本标准化后的基因表达水平。结果(图 3B)显示,12个样本的总体表达量分布相似。此外,两组12个样本基因表达量的主成分分析(PCA)散点图结果(图 3C)显示,TGC4样本与TGC组其他样本距离较远,OGC与TGC个体能获得有效区分,鉴于测序质控数据结果优良,说明此为TGC4本身转录组带来的差异。另外TGC组样本中,TGC2、TGC6距离较近,TGC1、TGC3、TGC5距离较近,说明TGC可能存在不同亚型。以上结果表明OGC和TGC两组基因的表达量存在一定程度的差异。
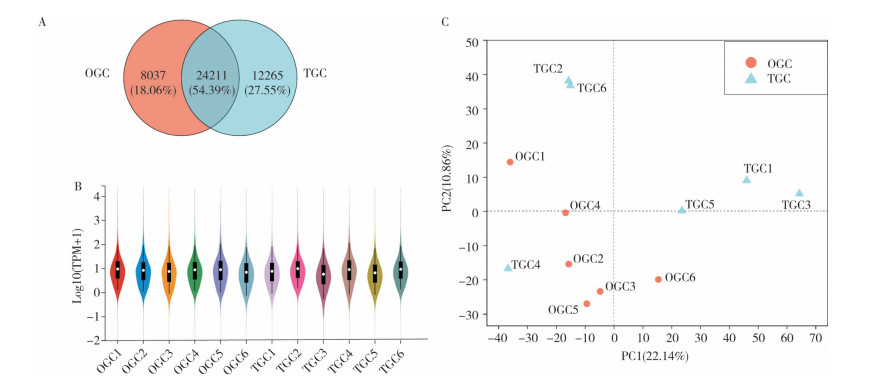
|
| A:OGC和TGC中鉴定的基因数目比较;B:样本总体表达量比较;C:基于OGC和TGC基因相对表达量的主成分分析散点图 A: Comparison of identified genes number in OGC and TGC; B: Comparison of sample total expressions; C: Scatter plot of PCA based on OGC and TGC gene relative expression 图 3 OGC和TGC基因注释及表达量分析 Fig. 3 Gene annotation and expression analysis of OGC and TGC |
2.3 神仙鱼透明鳃盖与不透明鳃盖差异表达分析
为了分析与OGC、TGC表型相关的具体基因,基于OGC和TGC的基因表达量使用DESeq2软件进行差异表达分析,分别绘制基因火山图和基因表达量散点图。使用Fold change ≥ 2或Fold change ≤ 1/2且Padjust<0.05作为DEG的筛选标准,最终筛选到prtfdc1、pfas、atic、ada2、(gda)、gucy2g、impdh2、nme、as3mt等432个在OGC和TGC中显著差异表达的基因,其中OGC相对于TGC显著上调基因165个、下调基因267个(图 4A、4B)。将432个DEG在不同样本间进行聚类分析,结果(图 4C)显示,OGC和TGC间的DEG可明显分为2个聚类,且OGC、TGC的样本各自聚类在一起,表明OGC和TGC样本间的转录组确实存在显著差异,推测432个DEG与TGC表型相关。
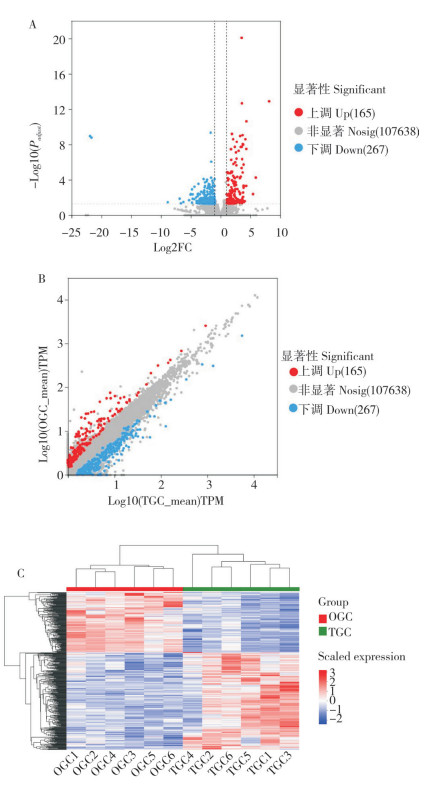
|
| A:火山图;B:散点图;C:聚类热图 A: Volcano plot; B: Scatter plot; C: Heatmap 图 4 OGC和TGC差异表达分析结果 Fig. 4 Result of OGC and TGC differential expression analysis |
2.4 神仙鱼透明鳃盖与不透明鳃盖差异表达基因富集分析
2.4.1 GO富集分析 将OGC和TGC中432个DEG进行GO富集,其中207个DEG在GO数据库中被匹配,涉及217个GO条目,各个GO条目按生物学过程、细胞组分和分子功能3大类别进行分类,按Padjust<0.05筛选标准显示Top20的富集结果。其中,DEG根据生物过程主要富集在肌苷酸生物合成过程、肌苷酸代谢过程、“从头”肌苷酸生物合成过程,均与嘌呤代谢相关;从分子功能角度主要富集在氨基酸结合通路、修饰氨基酸结合通路,均与氨基酸代谢相关(图 5)。
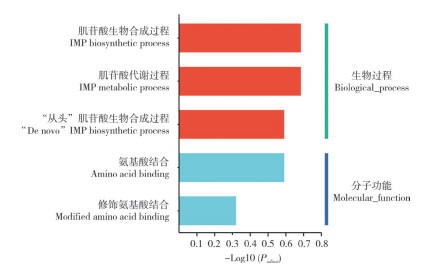
|
| 图 5 OGC和TGC差异表达基因GO富集分析结果 Fig. 5 GO enrichment analysis result of OGC and TGC differentially expressed genes |
2.4.2 KEGG富集分析 将OGC和TGC中432个DEG进行KEGG富集,其中203个DEG被富集到210个KEGG条目中,按Padjust<0.05筛选标准显示Top20的富集结果。从图 6可知,DEG主要富集在神经活性配体- 受体相互作用、酪氨酸代谢、嘌呤代谢、1个叶酸碳池、苯丙氨酸代谢、核苷酸代谢、泛醌和其他萜类化合物- 醌生物合成通路中。
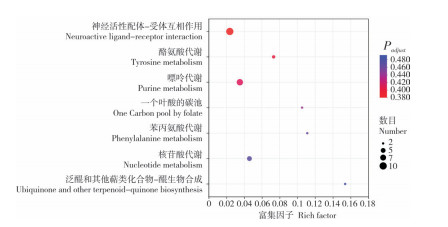
|
| 图 6 OGC和TGC差异表达基因KEGG富集分析结果 Fig. 6 KEGG enrichment analysis result of OGC and TGC differentially expressed genes |
3 讨论 3.1 神仙鱼透明鳃盖与不透明鳃盖转录组测序注释分析
本研究基于Illumina Novaseq 6000测序平台对神仙鱼透明鳃盖和不透明鳃盖组织样本进行转录组测序分析,在经过数据过滤后,12个样本共获得82.69 Gb Clean data,各样品Clean data均可达到6.22 Gb以上,12个样本对应的测序碱基平均错误率均低于0.1%,测序质量Q20碱基百分比在97.78% 以上,Q30碱基百分比在93.58% 以上,GC含量为47.04%~48.9%,说明测序质量高,数据可信。
本研究将转录组测序获得的所有转录本分别与六大数据库(NR、Swiss-Prot、Pfam、eggNOG、GO和KEGG)进行比对,结果显示,6个数据库中基因注释比例最高的是NR数据库、为39 738个,占数据库总体比例的37.56%;6个数据库总体注释基因40 180个,占总体的37.98%,该结果高于红白锦鲤转录组的注释比例(35.62%)[24]和花斑裸鲤的注释比例(31.32%)[27],但低于岩原鲤的注释比例(44.63%)[28]。推测这些差异来源于不同取样区域,同时也说明开展神仙鱼转录组学研究十分重要,剩下未被注释的基因也可能为非编码RNA、非功能基因或者为调控透明鳃盖的关键基因,随着研究技术的发展深入探究这些基因十分有必要。
3.2 神仙鱼透明鳃盖与不透明鳃盖差异表达基因及通路富集分析对神仙鱼透明鳃盖与不透明鳃盖的基因表达水平进行比较,得到432个在两种表型鳃盖中显著差异表达的基因,其中透明鳃盖相对于不透明鳃盖显著下调基因165个、上调267个。对432个差异表达基因分别进行GO和KEGG富集分析,其中GO富集结果显示差异表达基因主要富集在IMP生物合成过程、IMP代谢过程、“从头”IMP生物合成过程、氨基酸结合、修饰氨基酸结合;KEGG富集结果显示差异表达基因主要富集在神经活性配体- 受体相互作用、酪氨酸代谢、嘌呤代谢、一个叶酸碳池、苯丙氨酸代谢、核苷酸代谢、泛醌和其他萜类化合物- 醌生物合成通路中。这些通路除了神经活性配体- 受体相互作用为环境信息处理大类之外,其余通路均为代谢相关通路,说明调控神仙鱼透明鳃盖性状主要与代谢相关通路相关,但也受环境因素影响。
已有脊椎动物的相关研究表明酪氨酸代谢通路与黑色素合成相关,其调控机制为小眼畸形相关转录因子(Microphthalmia transcription factor,MITF)通过调控酪氨酸酶(Tyr)、酪氨酸酶相关蛋白1(Tyrp-1)、酪氨酸酶相关蛋白2(Tyrp-2)3种限制性酶基因的表达,从而影响黑色素细胞的分化[29-31]。在本研究中3个显著差异表达基因对应的乙醇脱氢酶(adh5)、4-羟基苯丙酮酸双加氧酶(hgd)、酪氨酸氨基转移酶(tat)富集在酪氨酸通路代谢中,但3个限制酶基因并未显著差异表达,而调控3个限制酶的基因mitf在透明鳃盖中显著下调,这说明控制神仙鱼透明鳃盖性状和酪氨酸代谢通路有关,但mitf不直接通过调控黑色素合成的酶调控该性状。另有相关研究证据表明hgd和adh5基因的单核苷酸多态性产生错义突变导致黑色素沉着缺乏,以及酪氨酸代谢通路中的差异基因在白乌鳢皮肤中表达量下降导致黑色素沉着量降低,这两种机制可共同促进乌鳢白化[32]。本研究中adh5基因在神仙鱼透明鳃盖中显著下调,因此酪氨酸代谢通路中adh5的下调很有可能与神仙鱼透明鳃盖性状相关,但其具体作用机制需要通过基因组测序技术进一步阐明。另外两个基因hgd和tat在神仙鱼透明鳃盖中显著上调,其中hgd可降解苯丙氨酸和酪氨酸分子[33],但两种基因在透明性状上的研究尚未有相关文献报道。
本研究中差异表达基因的GO富集结果显示与肌苷酸(次黄嘌呤)代谢相关,KEGG富集结果也富集在嘌呤代谢通路上,这些通路涉及含磷酸核糖转移酶结构域1(prtfdc1)、磷酸核糖甲酰甘氨脒合酶(pfas)、肌苷酸环水解酶(atic)、腺苷脱氨酶2(ada2)、鸟嘌呤脱氨酶(gda)、鸟苷酸环化酶2G(gucy2g)、肌苷单磷酸脱氢酶2(impdh2)、核苷二磷酸激酶(nme)、亚砷酸甲基转移酶(as3mt)的9个基因。虹彩细胞影响鱼类体色的机制为鱼体内的呈色物质遇水与其结合为晶体形式的鸟嘌呤,反射不同波长的光从而呈现出白色、蓝色和紫红3种色彩的结晶体[34]。以上结果与刘肖莲等[35]和Bian等[36]的研究结果一致,但刘肖莲等[35]采用的是皮肤组织,Bian等[36]使用的是皮肤和腹膜组织,而本研究中使用的神仙鱼鳃盖属于骨骼组织,因此只能推测与嘌呤代谢通路相关的基因共同参与调节鸟嘌呤合成,导致神仙鱼透明鳃盖中虹彩细胞和鸟嘌呤的缺失,使其呈现透明性状,后续仍需对比不同组织中的转录组学差异和进一步验证通路以得到具体结论。
鱼类的体色受很多复杂机制影响,除了神经内分泌系统调控色素细胞的影响外,也受遗传、营养、环境等多个外在因素影响[37-38]。本研究中富集到神经活性配体- 受体相互作用通路,该通路属于环境信息处理大类。此外,其他差异表达基因也富集在氨基酸代谢、一个叶酸碳池、苯丙氨酸代谢、核苷酸代谢、泛醌和其他萜类化合物- 醌生物合成各个通路中,但这些通路目前在鱼类透明性状相关研究中涉及较少,后续将进一步开展相关研究。
4 结论本研究首次基于转录组学测序技术对神仙鱼透明鳃盖性状展开研究。12个神仙鱼鳃盖样本的测序数据质量可靠、稳定。通过对神仙鱼透明鳃盖和不透明鳃盖转录组学数据进行差异表达分析,结果显示432个基因与神仙鱼透明鳃盖性状相关;经过GO和KEGG通路富集,结果显示432个基因涉及酪氨酸代谢、嘌呤代谢、氨基酸代谢等多条生物通路,这些基因和通路可为后续筛选调控透明神仙鱼性状的关键基因以及开展相关机制研究等提供数据参考。与嘌呤代谢和酪氨酸代谢通路相关的基因因其参与黑色素细胞和虹彩细胞的合成与降解过程,可以作为后续优先考虑的研究对象。
| [1] |
冯建伟, 王春植, 徐承旭. 中国休闲渔业发展监测报告发布[J]. 水产科技情报, 2021, 48(6): 313. FENG J W, WANG C Z, XU C X. Release of monitoring report on the development of recreational fishery in China[J]. Fisheries Science & Technology Information, 2021, 48(6): 313. |
| [2] |
WAKAMATSU Y, PRISTYAZHNYUK S, KINOSHITA M, TANAKA M, OZATO K. The see-through medaka: A fish model that is transparent throughout life[J]. Proceedings of the National Academy of Sciences, 2001, 98(18): 10046-10050. DOI:10.1073/pnas.181204298 |
| [3] |
MARUVADA S, HYNYNEN K, Optical monitoring of ultrasound interaction with blood vessels in transparent fish after injection with contrast agents[C]. 2002 IEEE Ultrasonics Symposium, 2002: 1985-1988. DOI: 10.1109/ULTSYM.2002.1192690.
|
| [4] |
贺国龙, 刘立鹤. 鱼类体色成因及其调控技术研究进展(上)[J]. 水产科技情报, 2010, 37(2): 88-91. HE G L, LIU L H. Research progress on the causes of body color and its regulation technology in fish (Part Ⅰ)[J]. Fisheries Science & Technology Information, 2010, 37(2): 88-91. |
| [5] |
冯幼, 许合, 刘定, 陈子腾, 许进奉, 黎相广, 蔡汉秋. 鱼类体色研究现状[J]. 水产养殖, 2014(2): 49-52. DOI:10.3969/j.issn.1001-0084.2014.02.013 FENG Y, XU H, LIU D, CHEN Z T, XU J F, LI X G, CAI H Q. Research status of skin color on fish[J]. Aquaculture, 2014(2): 49-52. DOI:10.3969/j.issn.1001-0084.2014.02.013 |
| [6] |
刘晓东, 陈再忠. 鱼类色素细胞及体色调控[J]. 水产科技情报, 2008, 35(1): 13-18. LIU X D, CHEN Z Z. Regulation of fish pigment cells and body color[J]. Fisheries Science & Technology Information, 2008, 35(1): 13-18. |
| [7] |
林翔, 周桂炫, 陈菲, 黄炜娟, 乐志操. 色素细胞发育分化的分子调控机制[J]. 福州大学学报(自然科学版), 2011, 39(5): 649-657. LIN X, ZHOU G X, CHEN F, HUANG W J, LE Z C. The molecular mechanism of melanocyte development and differentiation[J]. Journal of Fuzhou University (Natural Science Edition), 2011, 39(5): 649-657. |
| [8] |
SCHARTL M, LARUE L, GODA M, BOSENBERG M W, HASHIMOTO H, KELSH R N. What is a vertebrate pigment cell?[J]. Pigment Cell & Melanoma Research, 2015, 29(1): 8-14. DOI:10.1111/pcmr.12409 |
| [9] |
李建华, 江辉, 柳国军, 秦溱, 刘庆鹤. 三色神仙鱼人工繁殖技术措施探讨[J]. 养殖与饲料, 2014(10): 11-14. DOI:10.13300/j.cnki.cn42-1648/s.2014.10.030 LI J H, JIANG H, LIU G J, QIN Z, LIU Q H. Discussion on artificial reproduction dechniques and measures of three color anglefish[J]. Animals Breeding and Feed, 2014(10): 11-14. DOI:10.13300/j.cnki.cn42-1648/s.2014.10.030 |
| [10] |
张月星, 邵留. 金头红眼神仙鱼苗种繁育初探[J]. 科学养鱼, 2001(10): 25. DOI:10.3969/j.issn.1002-6681.2001.09.048 ZHANG Y X, SHAO L. A preliminary study on the breeding of golden-headed red-eyed Pterophyllum eimekei seedlings[J]. Scientific Fish Farming, 2001(10): 25. DOI:10.3969/j.issn.1002-6681.2001.09.048 |
| [11] |
海盗, 刘宇, 白明, 陆书亮. 永恒的经典——神仙鱼[M]. 北京: 中国科学技术文献出版社, 2012: 24-36. HAI D, LIU U, BAI M, LU S L. Eternal classic-Angle fish[M]. Beijing: China Science and Technical Documentation Press, 2012: 24-36. |
| [12] |
鱼融水, 白飞. 神仙鱼的种类和繁殖方法[J]. 水族世界, 2005(1): 84-88. YU R S, BAI F. Types and propagation methods of angelfish[J]. Aquarium, 2005(1): 84-88. |
| [13] |
CUI J, XU J, ZHANG S H, WANG K, JIANG Y, MAHBOOB S, GHANIM K, XU P. Transcriptional profiling reveals differential gene expression of Amur ide (Leuciscus waleckii) during spawning migration[J]. International Journal of Molecular Sciences, 2015, 16(6): 13959-13972. DOI:10.3390/ijms160613959 |
| [14] |
MININNI A N, MILAN M, FERRARESSO S, PETOCHI T, MARCO P D, MARINO G, LIVI S, ROMUALDI C, BARGELLONI L, PATARNELLO T. Liver transcriptome analysis in gilthead sea bream upon exposure to low temperature[J]. BMC Genomics, 2014, 15(1): 765-777. DOI:10.1186/1471-2164-15-765 |
| [15] |
董忠典, 黎学友, 黄承勤, 张海瑞, 黄顺楷, 张宁, 郭昱嵩, 王中铎. 湖栖鳍虾虎鱼性腺转录组比较分析[J]. 水产学报, 2021, 45(3): 365-380. DOI:10.11964/jfc.20200312214 DONG Z D, LI X Y, HUANG C Q, ZHANG H R, HUANG S K, ZHANG N, GUO Y S, WANG Z D. Comparative transcriptome analysis of the gonad of the lacustrine goby (Gobiopterus lacustris)[J]. Journal of Fisheries of China, 2021, 45(3): 365-380. DOI:10.11964/jfc.20200312214 |
| [16] |
吝思琪. 卤虫无节幼体添加对淡水黑、白神仙稚鱼生长、生理及体色的影响[D]. 海口: 海南大学, 2021. DOI: 10.27073/d.cnki.ghadu.2021.000508. LIN S Q. Effects of feeding Artemia sp. nauplii as complement on growth, physiology and skin color of white and black fresh water angel fish[D]. Haikou: Hainan University, 2021. DOI: 10.27073/d.cnki.ghadu.2021.000508. |
| [17] |
林涛, 周仁杰, 庄宏儒, 黄斌. 神仙鱼规模化繁育技术研究[J]. 福建水产, 2013, 35(1): 53-57. DOI:10.14012/j.cnki.fjsc.2013.01.013 LIN T, ZHOU R J, ZHUANG H R, HUANG B. Studies on angelfish large-scale breeding technology[J]. Journal of Fujian Fisheries, 2013, 35(1): 53-57. DOI:10.14012/j.cnki.fjsc.2013.01.013 |
| [18] |
SUSHILA N, DAS B K, MIR I N, ROUT A K, PANI P K, TRIPATHI G. Cloning, characterization and ontogenetic expression profile of RAG-2 and IgM in angelfish (Pterophyllum scalare)[J]. Gene, 2020. DOI:10.1016/j.gene.2020.144496 |
| [19] |
马家好, 张俊辉, 王玉平. 神仙鱼细菌性败血病病原及防治的研究[J]. 水产科学, 2000, 19(2): 11-13. DOI:10.16378/j.cnki.1003-1111.2000.02.003 MA J H, ZHANG J H, WANG Y P. Study on pathogen, prevention and cure of bacterial septicemia disease in angelfish (Pterophyllum eimekei)[J]. Fisheries Science, 2000, 19(2): 11-13. DOI:10.16378/j.cnki.1003-1111.2000.02.003 |
| [20] |
陈信忠, 袁芳, 许奕宸, 郭书林, 龚艳清, 杨俊萍. 七彩神仙鱼和神仙鱼的DNA条形码研究[J]. 检验检疫学刊, 2016, 26(6): 1-6. CHEN X Z, YUAN F, XU Y C, GUO S L, GONG Y Q, YANG J P. Study on the DNA barcode of the discuses and angelfish[J]. Journal of Inspection and Quarantine, 2016, 26(6): 1-6. |
| [21] |
MADIREDDY I. First ever whole genome sequencing and de novo assembly of the freshwater angelfish, Pterophyllum scalare[J]. Micropub Biology, 2022. DOI:10.17912/micropub.biology.000654 |
| [22] |
李凯彬, 汪学杰, 常藕琴, 刘春, 王芳, 马必勇, 梁慧丽, 吴淑勤. 透明鲫——一个适合活体研究的鱼类模型[J]. 中国实验动物学报, 2011, 19(4): 316-319, 378-379. DOI:10.3969/j.issn.1005-4847.2011.04.010 LI K B, WANG X J, CHAN O Q, LIU C, WANG F, MA B Y, LIANG H L, WU S Q. Transparent crucian carp Carassius auratus, a fish model suitable for study in vivo[J]. Acta Laboratorium Animalis Scientia Sinica, 2011, 19(4): 316-319, 378-379. DOI:10.3969/j.issn.1005-4847.2011.04.010 |
| [23] |
XU W, TONG G X, GENG L W, JIANG H F. Body color development and genetic analysis of hybrid transparent crucian carp (Carassius auratus)[J]. Genetics and Molecular Research, 2015, 14(2): 4399-4407. DOI:10.4238/2015.April.30.13 |
| [24] |
史东杰, 胡金有, 王赛赛, 朱华, 张欣. 红白锦鲤皮肤转录组测序分析[J]. 大连海洋大学学报, 2019, 34(4): 475-481. DOI:10.16535/j.cnki.dlhyxb.2019.04.003 SHI D J, HU J Y, WANG S S, ZHU H, ZHANG X. Transcriptome analysis of skin of red and white koi carp Cyprinus carpio haematopterus[J]. Journal of Dalian Ocean University, 2019, 34(4): 475-481. DOI:10.16535/j.cnki.dlhyxb.2019.04.003 |
| [25] |
黎学友, 谢明花, 黄承勤, 汪淳, 王中铎, 郭昱嵩, 董忠典. 湖栖鳍虾虎鱼皮肤和眼睛转录组比较[J]. 水产学报, 2021, 45(8): 1317-1326. DOI:10.11964/jfc.20200912401 LI X Y, XIE M H, HUANG C Q, WANG C, WANG Z D, GUO Y S, DONG Z D. Comparative analysis of skin and eye transcriptome in the Gobiopterus lacustris[J]. Journal of Fisheries of China, 2021, 45(8): 1317-1326. DOI:10.11964/jfc.20200912401 |
| [26] |
郝世鑫. 新西兰黑金鲍(Haliotis iris) 外套膜与腹足部黑色性状的初步研究[D]. 上海: 上海海洋大学, 2018. HAO S X. Preliminary research of black mantle and muscle of the New Zealand (Haliotis iris)[D]. Shanghai: Shanghai Ocean University, 2018. |
| [27] |
赵永利, 夏明哲, 吴蓉蓉, 晁燕, 祁得林. 基于高通量测序的花斑裸鲤转录组及功能分析[J]. 青海大学学报, 2018, 36(1): 1-8. DOI:10.13901/j.cnki.qhwxxbzk.2018.01.001 ZHAO Y L, XIA M Z, WU R R, CHAO Y, QI D L. High-throughput transcriptome sequencing and functional analysis of Gymncypris eckloni[J]. Journal of Qinghai University, 2018, 36(1): 1-8. DOI:10.13901/j.cnki.qhwxxbzk.2018.01.001 |
| [28] |
赵刚, 龚全, 刘亚, 赖见生, 陈叶雨, 杜军. 基于Illumina高通量测序的岩原鲤转录组分析[J]. 西南农业学报, 2016, 29(7): 1743-1749. DOI:10.16213/j.cnki.scjas.2016.07.043 ZHAO G, GONG Q, LIU Y, LAI J S, CHEN Y Y, DU J. Transcriptome analysis of Procypris rabaudi using Illumina high-throughput sequencing approach[J]. Southwest China Journal of Agricultural Sciences, 2016, 29(7): 1743-1749. DOI:10.16213/j.cnki.scjas.2016.07.043 |
| [29] |
王天柱, 吴庆, 张宁, 王东杰, 许洲. 鱼类黑色素合成及信号通路的研究进展[J]. 中国生物工程杂志, 2020, 40(5): 84-93. DOI:10.13523/j.cb.1912020 WANG T Z, WU Q, ZHANG N, WANG D J, XU Z. Advances in research on melanin synthesis and signaling pathway in fish[J]. China Biotechnology, 2020, 40(5): 84-93. DOI:10.13523/j.cb.1912020 |
| [30] |
FANG D, SETARLURI V. Role of microphthalmia transcription factor in regulation of melanocyte differentiation marker TRP-1[J]. Biochemical and Biophysical Research Communications, 1999, 256(3): 657-663. DOI:10.1006/bbrc.1999.0400 |
| [31] |
张艳苹. 红鳍笛鲷皮肤转录组分析及体色相关基因克隆、表达与进化研究[D]. 长沙: 湖南师范大学, 2016. ZHANG Y P. Study on skin transcriptome of crimson snapper (Lutjanus erythropterus), and their pigment genes related cloning, expression and evolution analysis[D]. Changsha: Hunan Normal University, 2016. |
| [32] |
李岩, 周燕, 雷骆, 苏建, 樊威. 基于转录组测序探究乌鳢皮肤白化的分子机制[J]. 水产科学, 2022, 41(5): 715-726. DOI:10.16378/j.cnki.1003-1111.21223 LI Y, ZHOU Y, LEI L, SU J, FAN W. Exploration of molecular mechanism of skin albinism in Albino Northern Snakehead Channa argus var. (Teleostei: Channidae) based in transcriptome sequencing[J]. Fisheries Science, 2022, 41(5): 715-726. DOI:10.16378/j.cnki.1003-1111.21223 |
| [33] |
FERNANDEZ-CANON J M, GRANADINO B, DE BERNA D B V, RENEDO M, FERNANDEZ-RUIZ E, PENALVA M A, DE CORDOBA S R. The molecular basis of alkaptonuria[J]. Nature Genetics, 1996, 14(1): 19-24. DOI:10.1038/ng0996-19 |
| [34] |
文胜, 刘锦辉, 张永勤, 肖亚梅. 色素细胞谱系及其发育调控研究进展[J]. 湖南师范大学自然科学学报, 2014, 37(6): 24-28. WEN S, LIU J H, ZHANG Y Q, XIAO Y M. Advances in the research of pigment cell lineage and its developmental mechanism[J]. Journal of Natural Science of Hunan Normal University, 2014, 37(6): 24-28. |
| [35] |
刘肖莲, 李春艳, 白晓慧, 郝爽, 刘国山. 透明草金鱼的皮肤转录组分析[J]. 水产科学, 2022, 41(1): 110-115. DOI:10.16378/j.cnki.1003-1111.20185 LIU X L, LI C Y, BAI X H, HAO S, LIU G S. Transcriptome analysis of transparent skin in grass gold fish Carassius auratus[J]. Fisheries Science, 2022, 41(1): 110-115. DOI:10.16378/j.cnki.1003-1111.20185 |
| [36] |
BIAN F, YANG X, OU Z, LUO J, TAN B, YUAN M, CHEN T, YANG R. Morphological characteristics and comparative transcriptome analysis of three different phenotypes of Pristella maxillaris[J]. Front Genet,, 2019, 10: 698-714. DOI:10.3389/fgene.2019.00698 |
| [37] |
王成辉, 项松平, 吕耀平, 王剑, 潘增云, 何小珍. 瓯江彩鲤红、白两种体色遗传关系的初步研究[J]. 上海水产大学学报, 2008, 17(4): 402-405. WANG C H, XIANG S P, LYU Y P, WANG J, PAN Z Y, HE X Z. Preliminary study on inheritance of red and white color phenotype in Qiujiang color Carp C. carpio var. color[J]. Journal of Shanghai Ocean University, 2008, 17(4): 402-405. |
| [38] |
何志刚, 王冬武, 曾国清, 邓时铭, 李绍明, 邹利. 不同饲料对红白锦鲤生长、体色和血液生化指标的影响[J]. 广东农业科学, 2015, 42(20): 107-111. DOI:10.16768/j.issn.1004-874X.2015.20.028 HE Z G, WANG D W, ZENG G Q, DENG S M, LI S M, ZOU L. Effects of different diets on growth, body pigmentation, serum biochemical indexes of ornamental carp (Cyprinus carpio L.)[J]. Guangdong Agricultural Sciences, 2015, 42(20): 107-111. DOI:10.16768/j.issn.1004-874X.2015.20.028 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51