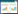文章信息
基金项目
- 上海市自然科学基金(20ZR1423600);国家自然科学基金(31902376)
作者简介
- 吉钰(1998—),女,硕士,研究方向为观赏鱼遗传育种,E-mail:VesperJ@qq.com.
通讯作者
- 陈再忠(1972—),男,博士,教授,研究方向为水产动物繁殖生物学、观赏鱼养殖,E-mail:chenzz@shou.edu.cn.
文章历史
- 收稿日期:2023-03-23
【研究意义】七彩神仙鱼(Symphysodon aequifasciatus)属于鲈形目(Perciformes)慈鲷科(Cichlidae)盘丽鱼属(Symphysodon)[1],主要来源于亚马孙河支流的野生水域[2],经多代反复杂交、回交、近交[3],形成了众多体色与花纹各异的人工选育品种[4]。然而,多数人工选育品种的遗传起源缺乏记录,目前仅通过外形特征进行分类,而表型背后的遗传基础尚不清楚,致使确定谱系十分困难[3, 5]。全基因组重测序的应用可为蓝色七彩神仙鱼品种的选育提供分子水平的数据支撑,有助于维持此类名优品种遗传特征的稳定性;同时,着眼于厘清品种间的系统发育关系,建立统一的分类标准和命名系统,制定合理的育种计划,可避免无计划杂交改良导致的近交萧条。【前人研究进展】近些年,对盘丽鱼属的研究聚焦于生长发育[6-7]、亲代抚育行为[8-9]、体色调控[10-11]等方面,群体遗传学方面的研究相对较少。利用传统分子标记方法,如随机扩增多态性DNA(RAPD)、扩增片段长度多态性(AFLP)和微卫星标记(SSR)等,均指向以下共同结果:首先,分子遗传相似性的分析结果往往与按照形态特征和地理分布产生的分类谱图不一致[3]。盘丽鱼属的分类学始终存在争议,较早研究中最常见的几个亚种为S. discus、S. aequifasciatus、S. harldi、S. axelrodi、S. tarzoo、S. willischwartzi[1],虽然Ready和Bleher重新将该属划分出3个亚种,但分类处理也大相径庭[12-13]。其次,两个形态不同的品种之间的分子遗传分化不明显[1, 5, 12],或指向不同的分类结果[5, 12-13]。这说明传统分子标记方法可以用于评价种间、种内的遗传变异和亲缘关系,但不足以区分亲缘关系过近的品种,也不能用于预测后代的表型[5],可见将形态学鉴定和分子手段互相补充具有必要性。这提示我们需要使用新一代测序技术或新一代分子标记等手段来获得更加准确全面的遗传学数据。目前对盘丽鱼属的研究未见重测序技术的应用,但SNP数据集已成熟应用于慈鲷科鱼类的物种进化研究。Olave等[14]基于SNP数据集重建了迈达斯慈鲷多物种的系统发育网络;Masonick等[15]分析了86种马拉维湖慈鲷的全基因组重测序数据,发现其肥大唇表型至少独立进化了两次;Albertson等[16]将定量性状位点分析与群体基因组学相结合,解析了慈鲷科鱼类在色素沉着水平和模式方面的遗传基础。SNP数据集也能在水产经济物种中实现养殖增益,例如,在尼罗罗非鱼(Oreochromis niloticus)中利用全基因组关联分析筛选与TiLV病毒抗性关联的位点[17]。【本研究切入点】群体的遗传多样性与适应能力及进化潜能之间存在正相关性,目前未见利用下一代测序技术(Next-generation sequencing,NGS)和第三代分子标记如SNP研究盘丽鱼属系统发育、群体结构和遗传多样性的相关报道。本研究团队前期获得了组装到染色体水平的七彩神仙鱼基因组数据(GenBank登录号:PRJNA863920),使得从基因组层面解析不同品种七彩神仙鱼的遗传基础成为可能。【拟解决的关键问题】本研究检测七彩神仙鱼野生型群体和两个人工选育蓝色群体中的SNP变异情况,从基因组水平分析七彩神仙鱼的遗传结构和多样性,筛选一些可能与蓝色品种形成相关的SNP,为建立七彩神仙鱼分类系统并制定基于遗传谱系的育种计划提供数据和参考。
1 材料与方法 1.1 试验材料进口获得的9尾野生型(WF)七彩神仙鱼(体表有蓝色、深绿色、棕褐色等花纹,身侧有纵向分布的棕黑色条带)来源于原生栖息地亚马逊盆地;人工选育型群体均取自上海海洋大学滨海基地,包含8尾全蓝型(BF)和10尾白化蓝型(ABF)七彩神仙鱼。剪取样本尾鳍1 cm置于液氮保存。
1.2 试验方法1.2.1 基因组DNA提取 使用改良的CTAB法[18]提取基因组DNA,用1% 的琼脂糖凝胶电泳检测DNA样品的质量,用超微量紫外分光光度计检测DNA样品的纯度。
1.2.2 基因组重测序和比对参考基因组 2020年5月由北京诺禾致源科技股份有限公司进行重测序。基因组DNA样本随机打断成长度为350 bp的片段,采用NEB Next® Ultra™ Ⅱ DNA Library Prep Kit构建双端测序文库,使用Illumina NovaseqTM平台进行PE150测序。用BWA将读取的高质量reads比对到基因组序列,并统计样本的匹配率和测序深度。
1.2.3 候选SNP目的片段的PCR扩增和测序 以野生型、全蓝型和白化蓝型群体中各5个不同个体的DNA为模板,使用设计的特异性引物进行PCR扩增,获得候选SNP所在区域长度为300~500 bp的目的核酸片段,测序确定SNP的碱基序列类型。PCR扩增反应体系为30 μL:Taq PCR Master Mix(2×)15 μL、模板DNA 2 μL和上下游引物各1 μL,补足ddH2O至30 μL。PCR扩增反应程序:95 ℃预变性3 min;94 ℃变性30 s、58~61 ℃退火30 s、72 ℃延伸1 min,40个循环;72 ℃延伸10 min。扩增引物的合成和扩增片段的测序由生工生物工程(上海)股份有限公司完成。
1.3 数据处理1.3.1 SNP鉴定和注释 采用SAMtools[19]和GATK[20]对BAM文件进行排序处理、去除重复序列及SNP鉴定,VCFtools[21]用于数据过滤,参数为Dp3-miss0.1-maf0.05。使用ANNOVAR[22]进行SNP注释。
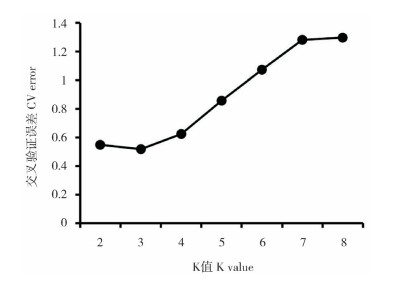
1.3.2 群体遗传多态性和群体遗传结构分析 使用VCFtools[21]和PLINK[23]计算3个七彩神仙鱼群体的核苷酸多态性(Pi)、期望杂合度(He)、观测杂合度(Ho)和多态性信息含量(PIC)以评估群体遗传多态性。在TreeBeST[24]中构建27个样本的系统发育进化树,引导值设置为1 000次。使用PLINK[23]进行主成分分析(Principal components analysis,PCA)。使用Admixture[25]分析3个群体的遗传结构,并通过计算最低交叉验证错误率(CV error)来判断最优聚类数(K)。
1.3.3 选择信号分析 使用PopLDdecay[26]计算3个七彩神仙鱼群体的连锁不平衡(Linkage disequilibrium,LD)衰减速度,使用VCFtools[21]以滑窗方法计算群体分化指数(Fixation indices,FST)和Pi,并进行基于FST和Pi的选择消除分析。筛选FST值和Pi的比值均位于前5% 的窗口作为两个群体间强选择性扫描下的候选区域。将这些染色体区域上的SNP利用DAVID数据库进行KEGG富集分析。
1.3.4 SNP筛选和验证 在进一步筛选SNP时,须特别关注导致非同义突变、剪切位点突变、基因调控元件突变的位点,并综合考虑根据核苷酸多态性测序深度计算的不同群体变异率(在体色变异群体中高于95%,在对比的非变异群体中低于10%),从而进一步缩小候选SNP范围。验证候选SNP的策略是PCR扩增和Sanger测序的结合,通过PCR扩增获得候选SNP所在区域长度为300~500 bp的目的核酸片段,测序比对以确定SNP所在位点的碱基类型,若与重测序结果相符且在变异群体中的不同个体测序结果一致则保留。候选位点的引物信息见表 1。
2 结果与分析 2.1 全基因组重测序和SNP变异检测结果
对27个七彩神仙鱼测序样本的产出数据进行统计,结果(表 2)表明,总的测序数据量为253.029 Gb,去除低质量序列后得到的clean data数据量为242.538 Gb,测序质量较高(Q20≥96.84%、Q30≥ 92.21%),GC含量为42.21%~44.64%,分布正常,此次建库测序成功。与七彩神仙鱼参考基因组的比对率超过98.88%,测序深度超过7.58×。

|
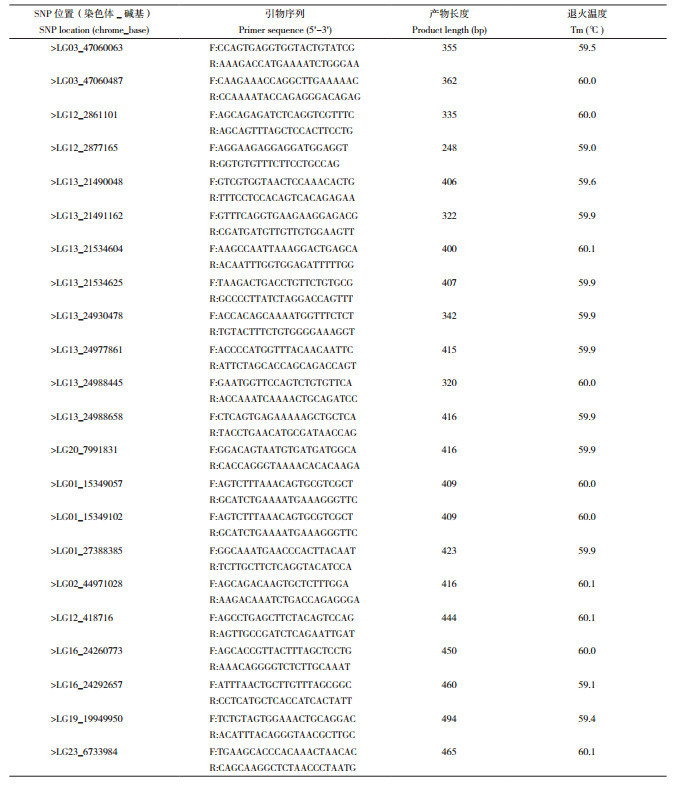
对3种不同体色的七彩神仙鱼群体进行SNP变异检测,共检测到1 360 293个SNP位点(表 3),通过ANNOVAR软件注释发现,SNP在基因间、内含子和外显子区域的比例分别为51.47%、37.00% 和4.32%。其中外显子区域中,小部分SNP为获得终止密码子的变异(280个)或失去终止密码子的变异(33个),大多数SNP导致同义突变(29 610个)或非同义突变(28 771个)。单碱基替换是SNP的主要类型,转换和颠换的比例为1.98︰1,存在碱基的转换/ 颠换偏差。
2.2 群体遗传多样性和群体遗传结构分析
2.2.1 群体遗传多样性比较 由表 4可见,3个七彩神仙鱼群体间存在遗传多样性差异,且3个群体的Pi值都较小,核苷酸多态性不高。根据Botstein等[27]的标准,野生型和全蓝型两群体的PIC值介于0.25~0.5,呈现中度多态性;白化蓝型的PIC值低于0.25,呈现低度多态性。全蓝型和白化蓝型群体内的Ho值低于He值,野生型群体内的Ho值高于He值,暗示群体内部交配方式的不同。两个人工选育群体相较于野生型群体呈现较低的Ho,群体内等位基因的分布更均匀。

|
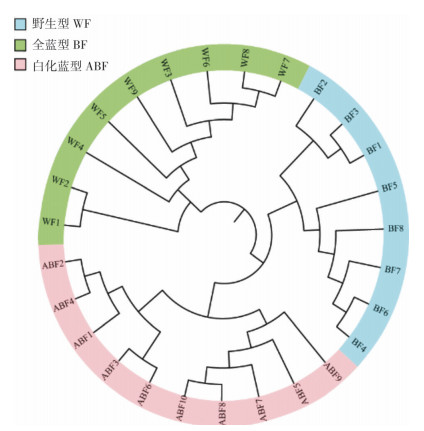
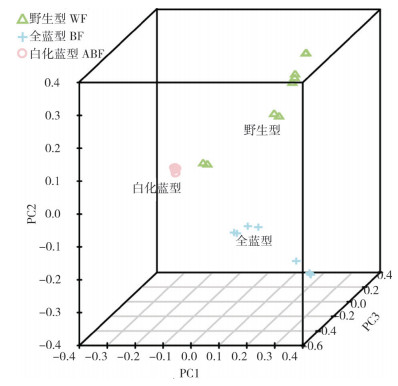
2.2.2 系统发育关系分析 对27个七彩神仙鱼个体检测出的SNP进行系统发育关系分析,从进化树(图 1)可见,3个群体各自聚集,亲缘关系存在一定距离,并显示了野生群体和人工选育群体的差异。野生型群体中的8个个体聚集成一个大分支,与全蓝型和白化蓝型相距较远;白化蓝型的10个个体和全蓝型的8个个体分别聚成一簇,随后汇集形成另一个大分支。同时,PCA三维聚类图(图 2)显示3个群体明显分开,白化蓝型群体内各点高度聚集,全蓝型群体内的点聚为两组,野生型群体内的点相对分散。聚类分析结果与进化树结果高度吻合。
|
| 图 1 3个七彩神仙鱼群体的系统发育进化树 Fig. 1 Phylogenetic tree of 3 populations of Symphysodon aequifasciatus |
|
| 图 2 3种七彩神仙鱼群体的SNP主成分分析 Fig. 2 Principal component analysis of SNPs in 3 populations of Symphysodon aequifasciatus |
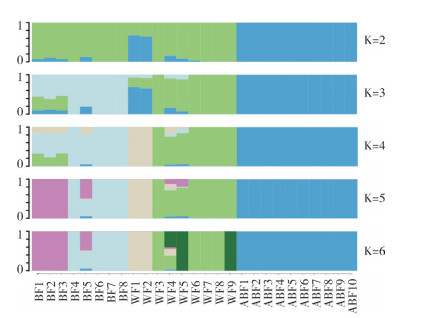
2.2.3 群体遗传结构分析 群体结构分析结果(图 3、图 4)显示,当K=2时,白化蓝型群体已明显独立出来,并且来自于一个祖先亚群;当K=3时,全蓝型和野生型能够明显区分,但有7个个体显示出3种组成成分,说明很有可能是由3个祖先亚群杂交而来;当K>3时,特殊的两个野生型个体分离出来,出现了新的祖先亚群;当K>4时,全蓝型群体中的BF1、BF2、BF3能够形成新的分类群,所属祖先亚群与其他种类完全不同。全蓝型群体中BF6、BF7、BF8和野生型群体中WF6、WF7、WF8、WF9的血统均比较纯净,而其他个体则存在不同程度的基因交流。
|
| 图 3 3种七彩神仙鱼群体结构分析 Fig. 3 Admixture analysis of population structure in 3 populations of Symphysodon aequifasciatus |
|
| 图 4 3种七彩神仙鱼群体的交叉验证 Fig. 4 Cross validation (CV) in 3 populations of Symphysodon aequifasciatus |
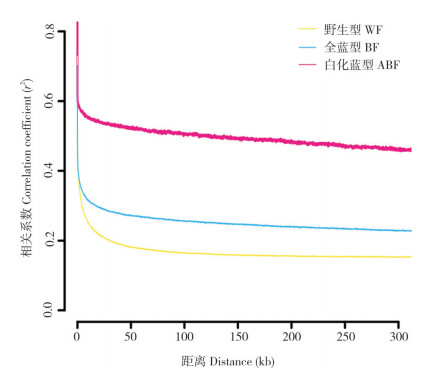
2.2.4 连锁不平衡衰减分析 比较3个群体的连锁不平衡衰减速度,结果(图 5)显示,相关系数r2值均随着遗传距离的增加而下降,野生型群体的衰减速度明显快于两个蓝色群体,而全蓝型群体的衰减速度比白化蓝型群体快。
|
| 图 5 3种七彩神仙鱼群体的连锁不平衡衰减曲线 Fig. 5 Linkage disequilibrium decay curve of 3 populations of Symphysodon aequifasciatus |
2.3 基于FST和Pi的群体选择消除分析
群体选择消除分析结果中,将FST和log2(Pi野生 /Pi全蓝)的值均排在最大前5% 的区域(图 6A)视为全蓝型七彩神仙鱼相对野生型七彩神仙鱼的受选择区域,筛选到202个染色体区域,这些区域主要集中在LG03、LG12、LG13、LG18和LG20染色体上(图 7A),参考基因组注释信息,在这些受到强烈选择的染色体区域上分布有529个编码蛋白基因;而计算FST和log2(Pi全蓝 /Pi白化),筛选到98个白化蓝型七彩神仙鱼相对于全蓝型七彩神仙鱼的受选择区域(图 6B),主要集中在LG01、LG16、LG18和LG19染色体上(图 7B),比对到335个编码蛋白基因。
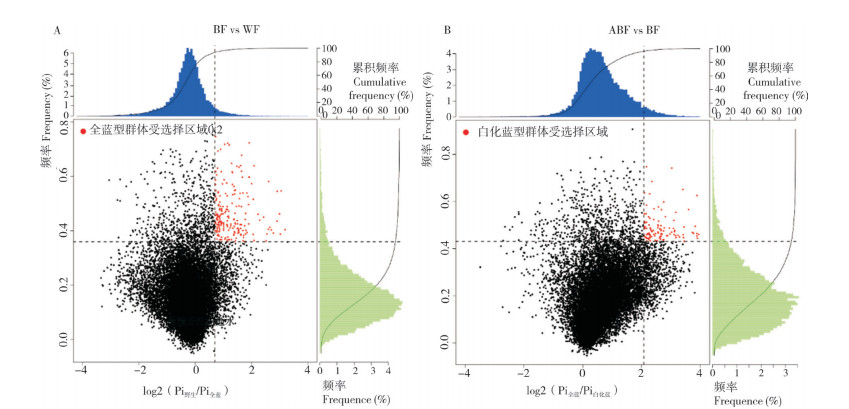
|
| 横坐标代表Pi比值,对应上侧的频率分布图,纵坐标代表FST值,对应右侧的频率分布图,图片中央的点图代表不同窗口内相应的FST和Pi比值。其中蓝色和绿色区域为Pi选择出来的Top 5% 区域,红色区域为FST选择出来的Top 5% 区域 The horizontal coordinate represents Pi ratio, corresponding to the frequency distribution diagram on the upper side; the vertical coordinate represents FST, corresponding to the frequency distribution diagram on the right side respectively; the dot plots in the center of the image represent corresponding FST and Pi ratio values in different windows.The blue and green areas are the Top 5% areas selected by Pi, and the red areas are the Top 5% areas selected by FST 图 6 全蓝型与野生型(A)、白化蓝型与全蓝型(B)七彩神仙鱼群体间基于FST和Pi信号的选择消除分析 Fig. 6 FST and Pi based selective sweep analysis between solid blue and wild (A), albino blue and solid blue (B) populations of Symphysodon aequifasciatus |
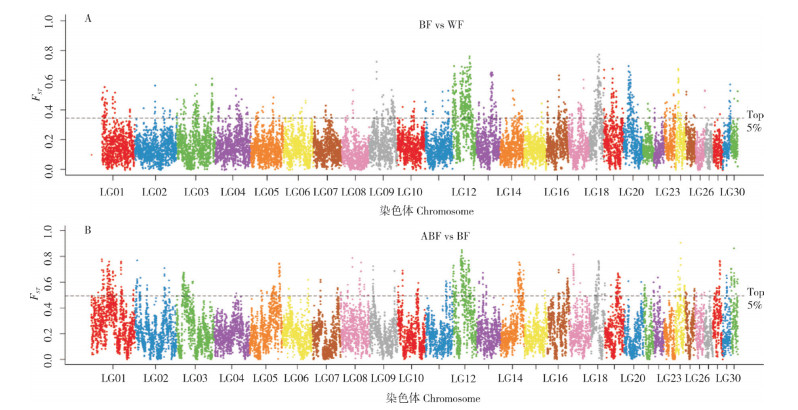
|
| 图中各点代表一个长度为50 000碱基的染色体区域 Each dot in the diagram represents a chromosomal region with a length of 50 000 bases 图 7 全蓝型与野生型(A)、白化蓝型与全蓝型(B)七彩神仙鱼群体的FST曼哈顿图 Fig. 7 Manhattan graph of FST values between solid blue and wild (A), albino blue and solid blue (B) populations of Symphysodon aequifasciatus |
使用KEGG数据分析两个人工选育七彩神仙鱼群体中受选择基因可能参与的代谢途径。在全蓝型群体中,通路主要涉及甘油脂和α-亚麻酸等脂类代谢、糖胺聚糖和硫酸皮肤素等聚糖合成和降解等新陈代谢类功能,信号分子转导等环境信息处理类功能,细胞内吞和紧密连接等细胞过程类功能。在白化蓝型群体中,通路主要集中在酪氨酸、赖氨酸等氨基酸合成和代谢,丙酮酸等碳水化合物代谢,糖胺聚糖和鞘糖脂等聚糖合成和代谢等新陈代谢大类(图 8)。
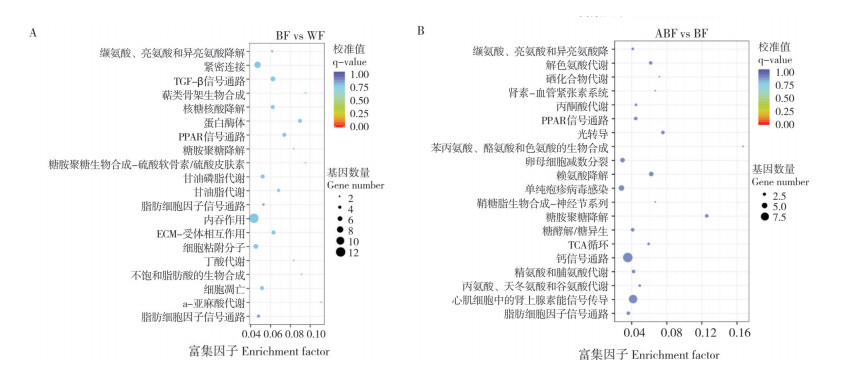
|
| 图 8 全蓝型(A)与白化蓝型(B)七彩神仙鱼群体中受选择SNP的KEGG富集通路 Fig. 8 KEGG enrichment pathway of selected SNPs in solid blue (A) and albino blue (B) populations of Symphysodon aequifasciatus |
2.4 SNP筛选和验证结果
结合ANNOVAR软件对高质量SNP位点的功能注释,以及不同群体中核苷酸碱基的出现频率,进一步缩小SNP候选范围。在全蓝型VS野生型和白化蓝型VS全蓝型中分别确定了50个和31个待验证的SNP。经PCR扩增和Sanger测序后,在全蓝型群体中确定了13个候选SNP,白化蓝型群体中确定了9个候选SNP,对应USP43、FGFR2、DENND4、MLL1等基因(表 5)。

|
3 讨论
野生棕/ 蓝盘丽鱼(S. haraldi)一直被认为是全蓝型七彩神仙鱼的起源。白化品种是由全蓝型七彩神仙鱼内交产生的突变品种,体表缺少黑色素细胞,颜色更加浅而明亮。普遍认为,七彩神仙鱼经历了从野生型到全蓝型再到白化蓝型的选育史,但尚未有可直接佐证的遗传学数据。本研究以这3种群体的全基因组重测序数据为基础,结合已组装到染色体水平的参考基因组,检测野生型群体和两个人工选育蓝色群体的SNP变异情况,分析群体的遗传结构和多样性,并利用基于FST和Pi的选择消除分析筛选出一些可能调控七彩神仙鱼蓝色体色的SNP,对应USP43、FGFR2、DENND4、MLL1等基因。位于3号染色体上的两个候选SNP可导致泛素特异性肽酶43(USP43)基因的非同义突变。许多研究提示USP43和上皮细胞的改变相关。在斑马鱼中,USP43被确定为驱动组织特异性Gal4表达的增强子,Gal4驱动的绿色荧光蛋白在色素细胞和表皮细胞等组织中均有强烈且可复制的表达[28]。另有大量肿瘤相关研究提出,USP43与角质层的形成、上皮细胞的改变相关[29-30],也可通过调控ZEB1的功能参与上皮细胞- 间质细胞的转化,影响黑色素瘤的发展[31]。位于12号染色体上的一个候选SNP导致了成纤维细胞生长因子受体2(FGFR2)基因的非同义突变。FGFR2与成纤维细胞生长因子配体结合后,作为受体酪氨酸激酶,激活RAS-MAPK和PI3K-AKT信号通路[32],全蓝型七彩神仙鱼体表黑色条纹的消失可能受到该基因调控。同时,推测FGFR2基因中的核苷酸变异与依赖鸟嘌呤晶体反射的结构性蓝色体色形成密切关联。FGFR2-IIIb在角质形成细胞中,作为角质形成细胞生长因子(KGF)的高亲和力受体,参与上皮修复过程[33-34],而参与嘌呤从头合成和补救合成途径的腺苷琥珀酸裂解酶编码基因ADSL与KGF的产生直接相关[35]。也有研究表明FGFR2在某些癌变组织中与AMPD2基因的外显子形成融合基因[36],而AMPD2编码的酶可将单磷酸腺苷转化为肌苷酸,从而参与嘌呤代谢。此外,通过氨基取代和乙酰胺取代产生的两种鸟嘌呤衍生物可作为包括FGFR2在内多种亚型FGFR的小分子抑制剂[37],这显示了鸟嘌呤和FGFR家族的潜在联系。位于1号染色体上的一个候选SNP导致DENND4基因的非同义突变。DENND4编码一类含DENN结构域的蛋白质,与Rab蛋白一起存在于具有极化上皮细胞层的多细胞生物中[38]。经证实,DENND4家族蛋白和Rab10在上皮细胞极化过程中发挥关键作用[38-39]。一些研究也暗示了DENND4可能与白化蓝型七彩神仙鱼中黑色素的消失有关,例如,DENND4B能够通过调控IGF-1信号通路的活性来调节酪氨酸酶的磷酸化,从而影响酪氨酸酶的活性和黑色素合成[40]。Rab27a参与黑色素小体的运输,在肌动蛋白细胞骨架轨道上运输黑素体[41]。而在硬骨鱼中,dennd4与编码Rab家族蛋白的许多基因是串联复制的[42]。位于16号染色体上的MLL1编码一种组蛋白甲基转移酶,该基因在黑色素瘤[43]、葡萄膜黑色素瘤[44]的发生、黑色素瘤的程序性死亡[45]中可能发挥重要作用。这些研究结果提示了该基因可能与七彩神仙鱼白化过程中黑色素细胞的缺失相关。
在完全随机、没有生物因素干扰的情况下,转换和颠换的理论比例为1︰2,但实际上转换比颠换更容易发生[46-47],这种偏差是一种常态,在七彩神仙鱼群体中存在碱基的转换/ 颠换偏差。3个群体的PIC值均处于中低度水平[27],推测可能由该物种自身的群体结构导致。Pi和PIC分别从核苷酸变异和DNA变异的角度反映了群体内部的多态性,在3个七彩神仙鱼群体中,其数值从野生型、全蓝型至白化蓝型递减的趋势显示了群体间选择压力的差异[48],符合当时选育品种不如野生品种遗传多样性高的结果[3],暗示全蓝型品种受到的强烈选择及其遗传特征的稳定性。Ho和He两者之间的平衡关系可用来推测群体内基因型的分布规律[49],通常两者越接近,说明等位基因在群体内的分布越均匀,符合Hardy-Weinberg平衡[50]。在3个群体内部Ho和He均存在显著差异:野生型群体的Ho>He,反映样本中存在更多杂合子;两个人工选育蓝色群体的Ho<He,反映其杂合子缺失状态,推断这两个群体中存在较高程度的近亲交配[51-52]。两个蓝色群体偏离Hardy-Weinberg平衡,很可能是因为群体中的个体是人工选择杂交的后代,人工选择作用会造成基因频率改变,影响基因组平均多样性水平[53];也可能是因为群体中的个体来自遗传上具有亲缘关系的同胞家系[54],以及种质资源的有效群体规模过小[55];遗传漂变对小群体的基因频率造成的随机波动也可能导致偏离Hardy-Weinberg平衡[56]。
本研究3个七彩神仙鱼群体的系统发育关系分析、主成分分析和群体遗传结构分析均呈现出与人工选育进程的一致性。系统发育进化树可将3个群体区分开来,它们的进化关系符合从野生品种到选育品种、从普通全蓝型到白化蓝型的选育史。在PCA结果中,白化蓝型个体的高度聚集说明群体内部的遗传多样性低、个体差异较小;在全蓝型群体中,点的分布表明可能存在两个品系;野生型群体内的点分布相对分散,表示具有多种不同血源。遗传群体结构分析结果显示,白化蓝型群体有较为纯净单一的血统,杂合度很低;全蓝型群体和野生型群体中的许多个体存在不同程度的基因交流,是不同祖先杂交产生的后代,提示蓝色体色的形成机制可能很复杂;当K=5时全蓝型群体中出现的新分类群与PCA结果中的分离呈现一致;WF1和WF2的存在也表明野生蓝棕群体中存在明显分化,可能是来自两种异质性祖先。鉴于21世纪初对自然水域中盘丽鱼属进行的几项分类研究未能得出统一结论,这提示利用第二代大通量基因组测序技术和方法对天然盘丽鱼属制定新的权威性分类和命名标准非常必要[1, 12-13]。连锁不平衡衰减曲线表明,全蓝型的LD衰减速度比白化蓝型快,说明白化蓝型群体受驯化程度较高,受选择强度较大。
综上所述,在蓝色系七彩神仙鱼中,全蓝型品系尤其是其中的白化类型,可以作为研究七彩神仙鱼体色变异的良好材料,也可以为水生动物中罕见的蓝色体色形成机制提供一个重要模型。基于FST和Pi的选择消除分析获得的候选基因列表对研究全蓝型突变产生的分子基础非常有价值。利用传统的分子标记评估系统发育关系和建立分类标准具有一定局限性[3, 57-59]。而下一代测序技术可一次性产生百万个序列,并获得包含所有遗传信息的完整基因组序列,作为第三代分子标记的SNP在慈鲷科鱼类中也常用于系统发育分析[14–16]。本研究是利用SNP标记研究七彩神仙鱼的系统发育关系和遗传多样性的首次尝试,为建立统一的分类标准提供了数据和方法基础。
遗传多态性在物种和基因组间均存在差异,已有许多研究证实了遗传多样性与群体的适应能力和进化潜力之间呈正相关[53, 56, 60],杂合子的大量存在能提高生物的适应能力,应对自然选择更具优势,群体的高杂合度也意味着更高的进化潜力。一般情况下,近交会导致基因型趋于纯和,杂合度迅速下降[55]。在个体数量小、养殖环境相对封闭的情况下,遗传漂变和近交衰退将导致群体对环境适应能力的持续下降,使种群面临退化风险[51, 61]。在观赏鱼产业中,养殖者追求体型、体色、花纹等特异性状的稳定性遗传,往往使用同一种源及其后代进行世代繁殖。目前,为了应对无计划育种实践导致的近交衰退,每年需要从原产地进口大量野生鱼种进行品系改良和种质资源复壮[62]。种质资源是水产养殖业可持续发展的核心要素[63],但在观赏水族产业中,野生资源经常被过度开发,而人工选育缺乏交流和指导,这使得优良种质难以科学保存[63-64]。对此提出以下建议,以利于观赏鱼产业的可持续发展:(1)使用不同来源的群体交配,提高养殖群体的遗传多样性,以保护各种独特品系间基因库的合理利用和保存;(2)合理使用基因组工具以加快水产养殖的可持续遗传增益;(3)应当为七彩神仙鱼制定能被广泛接受的分类标准和命名系统,利用全基因组重测序等技术阐明不同品种遗传基础,使现有的选育品种按照遗传相似性进行分类,并绘制较为全面的遗传图谱,便于制定基于谱系的育种计划。
4 结论本研究对野生型和人工选育全蓝型七彩神仙鱼群体的遗传多样性进行分析,鉴定了1 360 293个SNP位点。3个七彩神仙鱼群体的核苷酸多态性Pi介于0.201~0.326,多态性信息含量PIC介于0.201~0.326,期望杂合度He介于0.254~0.307,观测杂合度Ho介于0.190~0.350,总体来看遗传多样性处于中低度水平。野生型群体的Ho>He,杂合度高,两个人工选育蓝色群体的Ho<He,存在近亲交配。白化蓝型群体的LD衰减速度低于全蓝型群体,受驯化程度较高,受选择强度较大。27尾七彩神仙鱼的进化树、主成分分析和群体遗传结构分析结果均显示3个群体分离明显,遗传关系与人工选育进程具有一致性。在全蓝型VS野生型和白化蓝型VS全蓝型中分别确定了13个和9个候选SNP,USP43、FGFR2、DENND4、MLL1可能与体色形成密切相关。蓝色七彩神仙鱼尤其白化类型,可作为研究体色变异的良好模型,选择消除分析获得的候选基因可用于进一步研究全蓝型突变产生的分子基础。
| [1] |
FARIAS I P, HRBEK T. Patterns of diversification in the discus fishes (Symphysodon spp. Cichlidae) of the Amazon basin[J]. Molecular Phylogenetics and Evolution, 2008, 49(1): 32-43. DOI:10.1016/j.ympev.2008.05.033 |
| [2] |
CRAMPTON W G R. Ecology and life history of an Amazon floodplain cichlid: The discus fish Symphysodon (Perciformes: Cichlidae)[J]. Neotropical Ichthyology, 2008, 6(4): 599-612. DOI:10.1590/S1679-62252008000400008 |
| [3] |
KOH T L, KHOO G, FAN L Q. Genetic diversity among wild forms and cultivated varieties of discus (Symphysodon spp.) as revealed by Random Amplified Polymorphic DNA (RAPD) fingerprinting[J]. Aquaculture, 1999, 173(1): 486-497. DOI:10.1016/S0044-8486(98)00478-5 |
| [4] |
LIVENGOOD E J, OHS C L, CHAPMAN F A. Candidate species for Florida aquaculture: Discus Symphysodon spp., a profitable but challenging species for Florida aquaculture[Z]. Institute of Food and Agricultural Sciences (IFAS) US, 2009.
|
| [5] |
NG T T, SUNG Y Y, DANISH-DANIEL M, SORGELOOS P, PEER Y V D, WONG L L, TAN M P. Genetic variation of domesticated discus (Symphysodon spp.)[J]. AACL Biofl ux, 2021, 14(2): 832-840. |
| [6] |
刘颖, 刘怡南, 刘汉鹏, 温彬, 王南, 陈再忠, 高建忠. 饵料钙磷水平对七彩神仙鱼生长、体组成及消化酶活性的影响[J]. 上海海洋大学学报, 2018, 27(6): 833-843. DOI:10.12024/jsou.20180402271 LIU Y, LIU Y N, LIU H P, WEN B, WANG N, CHEN Z Z, GAO J Z. Effects of dietary phosphorus and calcium levels on growth, body composition and digestive enzyme activities of juvenile discus fish(Symphysodon haraldi)[J]. Journal of Shanghai Ocean University, 2018, 27(6): 833-843. DOI:10.12024/jsou.20180402271 |
| [7] |
ZHANG Y, WEN B, MENG L J, GAO J Z, CHEN Z Z. Dynamic changes of gut microbiota of discus fish (Symphysodon haraldi) at different feeding stages[J]. Aquaculture, 2021, 531: 735912. DOI:10.1016/j.aquaculture.2020.735912 |
| [8] |
FU Y, XU Z, WEN B, GAO J Z, CHEN Z Z. Gonad-specific transcriptomes reveal differential expression of gene and miRNA between male and female of the discus fish (Symphysodon aequifasciatus)[J]. Frontiers in Physiology, 2020, 11: 754. DOI:10.3389/fphys.2020.00754 |
| [9] |
周建桥, 欧阳明艳, 温彬, 陈再忠, 高建忠. 七彩神仙鱼繁殖间期与繁殖期体表黏液的部分组分分析[J]. 上海海洋大学学报, 2021, 30(1): 39-46. DOI:10.12024/jsou.20190302571 ZHOU J Q, OUYANG M Y, WEN B, CHEN Z Z, GAO J Z. Epidermal mucus components analysis of discus fish (Symphysodon haraldi) during breeding period and interval-breeding period[J]. Journal of Shanghai Ocean University, 2021, 30(1): 39-46. DOI:10.12024/jsou.20190302571 |
| [10] |
YANG B T, WEN B, JI Y, WANG Q, ZHANG H R, ZHANG Y, GAO J Z, CHEN Z Z. Comparative metabolomics analysis of pigmentary and structural coloration in discus fish (Symphysodon haraldi)[J]. Journal of Proteomics, 2021, 233: 104085. DOI:10.1016/j.jprot.2020.104085 |
| [11] |
TU N P C, HA N N, LINH N T T, TRI N N. Effect of astaxanthin and spirulina levels in black soldier fly larvae meal-based diets on growth performance and skin pigmentation in discus fish, Symphysodon spp.[J]. Aquaculture, 2022, 553: 738048. DOI:10.1016/j.aquaculture.2022.738048 |
| [12] |
READY J S, FERREIRA E J G, KULLANDER S O. Discus fishes: mitochondrial DNA evidence for a phylogeographic barrier in the Amazonian genus Symphysodon (Teleostei: Cichlidae)[J]. Journal of Fish Biology, 2006, 69(S): 200-211. DOI:10.1111/j.1095-8649.2006.01232.x |
| [13] |
BLEHER H, STÖLTING K N, SALZBURGER W, MEYER A. Revision of the genus Symphysodon Heckel, 1840 (Teleostei: Perciformes: Cichlidae) based on molecular and morphological characters[J]. Aqua, International Journal of Ichthyology, 2007, 12(4): 133-174. |
| [14] |
OLAVE M, MEYER A. Implementing large genomic single nucleotide polymorphism data sets in phylogenetic network reconstructions: a case study of particularly rapid radiations of cichlid fish[J]. Systematic Biology, 2020, 69(5): 848-862. DOI:10.1093/sysbio/syaa005 |
| [15] |
MASONICK P, MEYER A, HULSEY C D. Phylogenomic analyses show repeated evolution of hypertrophied lips among lake malawi cichlid fishes[J]. Genome Biology and Evolution, 2022, 14(4): evac051. DOI:10.1093/gbe/evac051 |
| [16] |
ALBERTSON R C, POWDER K E, HU Y, COYLE K P, ROBERTS R B, PARSONS K J. Genetic basis of continuous variation in the levels and modular inheritance of pigmentation in cichlid fishes[J]. Molecular Ecology, 2014, 23(21): 5135-5150. DOI:10.1111/mec.12900 |
| [17] |
BARRÍA A, TRỊNH T Q, MAHMUDDIN M, PEÑALOZA C, PAPADOPOULOU A, GERVAIS O, CHADAG V M, BENZIE J A H, HOUSTON R D. A major quantitative trait locus affecting resistance to Tilapia lake virus in farmed Nile tilapia (Oreochromis niloticus)[J]. Heredity, 2021, 127(3): 334-343. DOI:10.1038/s41437-021-00447-4 |
| [18] |
MURRAY M, THOMPSON W. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research, 1980, 8(19): 4321-4326. DOI:10.1093/nar/8.19.4321 |
| [19] |
LI H, HANDSAKER B, WYSOKER A, FENNELL T, RUAN J, HOMER N, MARTH G, ABECASIS G, DURBIN R. The sequence alignment map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp324 |
| [20] |
MCKENNA A, HANNA M, BANKS E, SIVACHENKO A, CIBULSKIS K, KERNYTSKY A, GARIMELLA K, ALTSHULER D, GABRIEL S, DALY M, DEPRISTO M A. The Genome Analysis Toolkit: A MapReduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research, 2010, 20(9): 1297-1303. DOI:10.1101/gr.107524.110 |
| [21] |
DANECEK P, AUTON A, ABECASIS G, ALBERS C A, BANKS E, DEPRISTO M A, HANDSAKER R E, LUNTER G, MARTH G T, SHERRY S T, MCVEAN G, DURBIN R. The variant call format and VCFtools[J]. Bioinformatics, 2011, 27(15): 2156-2158. DOI:10.1093/bioinformatics/btr330 |
| [22] |
WANG K, LI M, HAKONARSON H. ANNOVAR: functional annotation of genetic variants from high-throughput sequencing data[J]. Nucleic Acids Research, 2010, 38(16): e164-e164. DOI:10.1093/nar/gkq603 |
| [23] |
PURCELL S, NEALE B, TODD-BROWN K, THOMAS L, FERREIRA M A R, BENDER D, MALLER J, SKLAR P, BAKKER P I W D, DALY M, SHAM P C. PLINK: A tool set for whole-genome association and population-based linkage analyses[J]. The American Journal of Human Genetics, 2007, 81(3): 559-575. DOI:10.1086/519795 |
| [24] |
VILELLA A J, SEVERIN J, URETA-VIDAL A, HENG L, DURBIN R, BIRNEY E. Ensembl compara genetrees: complete, duplication-aware phylogenetic trees in vertebrates[J]. Genome Research, 2009, 19(2): 327-335. DOI:10.1101/gr.073585.107 |
| [25] |
ALEXANDER D H, NOVEMBRE J, LANGE K. Fast model-based estimation of ancestry in unrelated individuals[J]. Genome Research, 2009, 19(9): 1655-1664. DOI:10.1101/gr.094052.109 |
| [26] |
ZHANG C, DONG S S, XU J Y, HE W M, YANG T L. PopLDdecay: a fast and effective tool for linkage disequilibrium decay analysis based on variant call format files[J]. Bioinformatics, 2019, 35(10): 1786-1788. DOI:10.1093/bioinformatics/bty875 |
| [27] |
BOTSTEIN D, WHITE R L, SKOLNICK M, DAVIS R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314. DOI: 10.1.1.420.6196.
|
| [28] |
LEVESQUE M P, KRAUSS J, KOEHLER C, BODEN C, HARRIS M P. New tools for the identification of developmentally regulated enhancer regions in embryonic and adult zebrafish[J]. Zebrafi sh, 2013, 10(1): 21-29. DOI:10.1089/zeb.2012.0775 |
| [29] |
JACKSON N D, DYJACK N, GOLEVA E, BIN L, MONTGOMERY M T, RIOS C, EVERMAN J L, TAYLOR P, BRONCHICK C, RICHERS B N, LEUNG D Y, SEIBOLD M A. Atopic dermatitis complicated by recurrent eczema herpeticum is characterized by multiple, concurrent epidermal inflammatory endotypes[J]. BioRxiv, 2023. DOI:10.1101/2023.02.27.530316 |
| [30] |
PANDEY C K, LANDMAN N, NEIKES H K, HULSMAN D, LIEFTINK C, BEIJERSBERGEN R, KOLLURI K K, JANES S M, VERMEULEN M, BADHAI J, LOHUIZEN M V. Genetic screens reveal new targetable vulnerabilities in BAP1-deficient mesothelioma[J]. Cell Reports Medicine, 2023, 4(2): 100915. DOI:10.1016/j.xcrm.2022.100915 |
| [31] |
HUANG Z, ZHANG Z, ZHOU C W, LIU L, HUANG C H. Epithelial– mesenchymal transition: The history, regulatory mechanism, and cancer therapeutic opportunities[J]. MedComm, 2022, 3(2): e144. DOI:10.1002/mco2.144 |
| [32] |
TENHAGEN M, DIEST P J V, IVANOVA I A, WALL V D, GROEP P V D. Fibroblast growth factor receptors in breast cancer: Expression, downstream effects, and possible drug targets[J]. Endocrine-related Cancer, 2012, 19(4): R115-R129. DOI:10.1530/ERC-12-0060 |
| [33] |
BEER H D, GASSMANN M G, MUNZ B, STEILING H, ENGELHARDT F, BLEUEL K, WERNER S. Expression and function of keratinocyte growth factor and activin in skin morphogenesis and cutaneous wound repair[J]. Journal of Investigative Dermatology Symposium Proceedings, 2000, 5(1): 34-39. DOI:10.1046/j.1087-0024.2000.00009.x |
| [34] |
KELLER U A D, KRAMPERT M, KÜMIN A, BRAUN S, WERNER S. Keratinocyte growth factor: effects on keratinocytes and mechanisms of action[J]. European Journal of Cell Biology, 2004, 83(11/12): 607-612. DOI:10.1078/0171-9335-00389 |
| [35] |
JIANG T T, SÁNCHEZ-RIVERA F J, SOTO-FELICIANO Y M, YANG Q Y, SONG C Q, BHUATKAR A, HAYNES C M, HEMANN M T, XUE W. Targeting the de novo purine synthesis pathway through adenylosuccinate lyase depletion impairs liver cancer growth by perturbing mitochondrial function[J]. Hepatology, 2021, 74(1): 233-247. DOI:10.1002/hep.31685 |
| [36] |
KONGPETCH S, JUSAKUL A, LIM J Q, YOUNG C C, CHAN J Y, RAJASEGARAN V, LIM T H, LIM K H, CHOO S P, DIMA S, POPESCU I, DUDA D G, KUKONGVIRIYAPAN V, KHUNTIKEO N, PAIROJKUL C, ROZEN S G, TAN P, TEH B T. Lack of targetable FGFR2 fusions in endemic fluke-associated cholangiocarcinoma[J]. JCO Global Oncology, 2020, 6: 628-638. DOI:10.1200/GO.20.00030 |
| [37] |
YE F, CHEN L, HU L, XIAO T, YU S F, CHEN D, WANG Y, LIANG G, LIU Z G, WANG S C. Design, synthesis and preliminary biological evaluation of C-8 substituted guanine derivatives as small molecular inhibitors of FGFRs[J]. Bioorganic & Medicinal Chemistry Letters, 2015, 25(7): 1556-1560. DOI:10.1016/j.bmcl.2015.02.010 |
| [38] |
YOSHIMURA S, GERONDOPOULOS A, LINFORD A, RIGDEN D J, BARR F A. Family-wide characterization of the DENN domain Rab GDP-GTP exchange factors[J]. Journal of Cell Biology, 2010, 191(2): 367-381. DOI:10.1083/jcb.201008051 |
| [39] |
OCHI Y, YAMASHITA H, YAMADA Y, SATOH T, SATOH A K. Stratum is required for both apical and basolateral transport through stable expression of Rab10 and Rab35 in Drosophila photoreceptors[J]. Molecular Biology of the Cell, 2022, 33(10): 17. DOI:10.1091/mbc.E21-12-0596 |
| [40] |
REETOO N. Purification of protein phosphatases and 14-3-3-binding proteins and their regulation by the CAMP-PKA pathway[D]. Scotland: University of Dundee, 2012.
|
| [41] |
HAMMER Ⅲ J A, WU X. Slip sliding away with myosin V[J]. Proceedings of the National Academy of Sciences, 2007, 104(13): 5255-5256. DOI:10.1073/pnas.0701071104 |
| [42] |
NUCKELS R J. Differential selection pressure among duplicated genes in teleosts[D]. Texas: Texas State University, 2018.
|
| [43] |
KOYAUCHI T, NⅡDA H, MOTEGI A, SAKAI S, UCHIDA C, OHHATA T, IIJIMA K, YOKOYAMA A, SUDA T U, KITAGAWA M. Chromatin-remodeling factor BAZ1A/ACF1 targets UV damage sites in an MLL1-dependent manner to facilitate nucleotide excision repair[J]. Biochimica Et Biophysica Acta (BBA)-Molecular Cell Research, 2022, 1869(11): 119332. DOI:10.1016/j.bbamcr.2022.119332 |
| [44] |
PAN H, WANG H X, ZHANG X Y, YANG F, FAN X Q, ZHANG H. Chromosomal instability-associated MAT1 lncRNA insulates MLL1-guided histone methylation and accelerates tumorigenesis[J]. Cell Reports, 2022, 41(11): 111829. DOI:10.1016/j.celrep.2022.111829 |
| [45] |
ZHANG Z, HAN Y, SUN Q, WANG Y, SUN L. The DPY30-H3K4me3 axis-mediated PD-L1 expression in melanoma[J]. Journal of Inflammation Research, 2022, 15: 5595-5609. DOI:10.2147/JIR.S377678 |
| [46] |
WANG J, RASKIN L, SAMUELS D C, SHYR Y, GUO Y. Genome measures used for quality control are dependent on gene function and ancestry[J]. Bioinformatics, 2015, 31(3): 318-323. DOI:10.1093/bioinformatics/btu668 |
| [47] |
STOLTZFUS A, NORRIS R W. On the causes of evolutionary transition: transversion Bias[J]. Molecular Biology and Evolution, 2016, 33(3): 595-602. DOI:10.1093/molbev/msv274 |
| [48] |
钟丹丹, 林勇, 宾石玉, 余艳玲, 陈忠, 曾兰, 杜雪松, 张永德. 两个罗氏沼虾种群的遗传多样性研究[J]. 广东农业科学, 2015, 42(24): 140-145. DOI:10.16768/j.issn.1004-874X.2015.24.069 ZHONG D D, LIN Y, BIN S Y, YU Y L, CHEN Z, ZENG L, DU X S, ZHANG Y D. Genetic diversity of two giant freshwater prawn Macrobrachium rosenbergii populations[J]. Guandong Agricultural Sciences, 2015, 42(24): 140-145. DOI:10.16768/j.issn.1004-874X.2015.24.069 |
| [49] |
徐晓美, 李颖, 孙启迪, 徐小万, 衡周, 李涛, 王恒明. 辣椒种质材料疫病抗性鉴评及遗传多样性分析[J]. 广东农业科学, 2022, 49(10): 19-28. DOI:10.16768/j.issn.1004-874X.2022.10.003 XU X M, LI Y, SUN Q D, XU X W, HENG Z, LI T, WANG H M. Resistance evaluation and genetic diversity analysis of phytophthora disease in pepper germplasm materials[J]. Guandong Agricultural Sciences, 2022, 49(10): 19-28. DOI:10.16768/j.issn.1004-874X.2022.10.003 |
| [50] |
DONG Q F. Microsatellite analysis of genetic diversity and phylogenetic relationship of nine species of grouper in genus Epinephelus[J]. Hereditas, 2007, 29(7): 837. DOI:10.1360/yc-007-0837 |
| [51] |
DOSSYBAYEV K, ORAZYMBETOVA Z, MUSSAYEVA A, SAITOU N, ZHAPBASOV R, MAKHATOV B, BEKMANOV B. Genetic diversity of different breeds of Kazakh sheep using microsatellite analysis[J]. Archives Animal Breeding, 2019, 62(1): 305-312. DOI:10.5194/aab-62-305-2019 |
| [52] |
DEMIR E, BALCIOGLU M S. Genetic diversity and population structure of four cattle breeds raised in Turkey using microsatellite markers[J]. Czech Journal of Animal Science, 2019, 64(10): 411-419. DOI:10.17221/62/2019-CJAS |
| [53] |
余成晨, 沈玉帮, 徐晓雁, 王荣泉, 李家乐. 草鱼生长相关的微卫星标记在选育群体中的验证[J]. 水产学报, 2021, 45(3): 321-332. DOI:10.11964/jfc.20200212165 YU C C, SHEN Y B, XU X Y, WANG R Q, LI J L. Verification of microsatellite markers associated with growth traits in selected populations of grass carp (Ctenopharyngodon idella)[J]. Journal of Fisheries of China, 2021, 45(3): 321-332. DOI:10.11964/jfc.20200212165 |
| [54] |
王润风, 鲁清, 洪彦彬, 梁炫强, 陈小平, 刘国强, 洪成佳, 李少雄. 中国南方区试花生品种的遗传多样性分析[J]. 广东农业科学, 2021, 48(12): 44-53. DOI:10.16768/j.issn.1004-874X.2021.12.006 WANG R F, LU Q, HONG Y B, LIANG X Q, CHEN X P, LIU G Q, HONG C J, LI S X. Analysis on the genetic diversity of peanut varieties in regional tests of Southern China[J]. Guandong Agricultural Sciences, 2021, 48(12): 44-53. DOI:10.16768/j.issn.1004-874X.2021.12.006 |
| [55] |
马春艳, 马洪雨, 马凌波, 杨柯. 凡纳滨对虾引进群体和2个养殖群体遗传变异的微卫星分析[J]. 海洋渔业, 2011, 33(1): 1-8. DOI:10.13233/j.cnki.mar.fish.2011.01.011 MA C Y, MA H Y, MA L B, YANG K. Microsatellite analysis on genetic variation of imported Litopenaeus vannamei population and cultured stocks[J]. Marine Fisheries, 2011, 33(1): 1-8. DOI:10.13233/j.cnki.mar.fish.2011.01.011 |
| [56] |
DEWOODY J A, HARDER A M, MATHUR S, WILLOUGHBY J R. The long‐standing significance of genetic diversity in conservation[J]. Molecular Ecology, 2021, 30(17): 4147-4154. DOI:10.1111/mec.16051 |
| [57] |
DEGANI G. Genetic variation in strains of discus (Symphyodon aequifaciata) using RAPD PCR[J]. Israeli Journal of Aquaculture-Bamidgeh, 2003, 55: 86-93. DOI:10.46989/001c.20344 |
| [58] |
AMADO M V, HRBEK T, GRAVENA W, FANTIN C, ASSUNÇÃO E N D, ASTOLFI-FILHO S, FARIAS I P. Isolation and characterization of microsatellite markers for the ornamental discus fish Symphysodon discus and cross-species amplification in other Heroini cichlid species[J]. Molecular Ecology Resources, 2008, 8(6): 1451-1453. DOI:10.1111/j.1755-0998.2008.02200.x |
| [59] |
AMADO M V, FARIAS I P, HRBEK T. A molecular perspective on systematics, taxonomy and classification amazonian discus fishes of the genus Symphysodon[J]. International Journal of Evolutionary Biology, 2011, 1-16. DOI:10.4061/2011/360654 |
| [60] |
梁业松, 张维炜, 宋飞彪, 张娴, 刘伊凡, 刘晓春, 林浩然, 骆剑. 虎龙杂交斑养殖群体遗传多样性与遗传结构的微卫星分析[J]. 水产学报, 2022, 46(1): 31-40. DOI:10.11964/jfc.20210212621 LIANG Y S, ZHANG W W, SONG F B, ZHANG X, LIU Y F, LIU X C, LIN H R, LUO J. Genetic diversity and genetic structure analysis of aquaculture groups of hybrid grouper 〔Epinephelus fuscoguttatus (♀)×E.lanceolatus (♂)〕 using microsatellite markers[J]. Journal of Fisheries of China, 2022, 46(1): 31-40. DOI:10.11964/jfc.20210212621 |
| [61] |
REED D H, FRANKHAM R. Correlation between fitness and genetic diversity[J]. Conservation Biology, 2003, 17(1): 230-237. DOI:10.1046/j.1523-1739.2003.01236.x |
| [62] |
王磊, 孙培英, 陈再忠, 罗小溪, 高建忠, 李仲璞, 韩灵莉. 黑格尔七彩神仙鱼摩氏摩根菌分离鉴定及耐药性分析[J]. 南方农业学报, 2016, 47(10): 1772-1777. DOI:10.3969/jissn.2095-1191.2016.10.1772 WANG L, SUN P Y, CHEN Z Z, LUO X X, GAO J Z, LI Z P, HAN L L. Isolation, identification and drug resistance analysis of Morganella morganii from Symphysodon discus[J]. Journal of Southern Agriculture, 2016, 47(10): 1772-1777. DOI:10.3969/jissn.2095-1191.2016.10.1772 |
| [63] |
DAR R A, AHMAD M, KUMAR S, RESHI M. Agriculture germplasm resources: a tool of conserving diversity[J]. Scientific Research and Essays, 2015, 10(9): 326-338. DOI:10.5897/SRE2015.6206 |
| [64] |
PURDY P H, WILSON C S, SPILLER S F, BLACKBURN H D. Biobanking genetic resources: Challenges and implementation at the USDA National Animal Germplasm Program[J]. Reproduction, Fertility and Development, 2016, 28(8): 1072-1078. DOI:10.1071/RD15399 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51