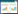文章信息
基金项目
- 国家自然科学基金(32370359)
作者简介
- 刘思嘉(1998—),女,在读硕士生,研究方向为热带园艺植物,E-mail: 1379174809@qq.com.
通讯作者
- 杨荣超(1979—),博士,讲师,研究方向为热带园艺植物,E-mail: yangrc2021@163.com.
文章历史
- 收稿日期:2023-12-05
【研究意义】我国是番茄(Lycopersicum esculentum)种植面积最大、产量最高的国家[1]。番茄因其果实酸甜可口、风味独特,且富含多种营养物质,深受人们喜爱[2]。贾俊婷等[3]指出,组蛋白修饰基因家族在植物生长发育中起着至关重要的作用。但目前,研究人员对重要园艺作物番茄中的组蛋白修饰(Histone modification,HM)基因家族却知之甚少。通过全面分析番茄HM的基因结构、功能蛋白结构域、与拟南芥和水稻同源基因的进化关系等生物特征,为深入研究番茄HM基因的功能提供理论参考,并为加快优良番茄品种分子设计育种奠定基础。【前人研究进展】大量研究表明,HM可以通过改变染色质结构、调节细胞分裂、分化、迁移等方式来影响植物的形态和生理变化[4-8]。组蛋白修饰过程由4种不同的HM基因家族成员调控,包括组蛋白甲基转移酶(Histone methyltransferase,HMT)、组蛋白去甲基化酶(Histone demethylase,HDM)、组蛋白乙酰化酶(Histone acetylase,HAT)和组蛋白去乙酰化酶(Histone deacetylase,HDAC)。4个HM基因家族均由不同数量的亚家族组成,且具有不同的结构域。HMT包含SDG(Set domain group)和PRMT(Protein arginine methyltransferases)亚家族;HDM包含HDMA(SWIRM、C-terminal domain)和JMJ(JmjC结构域蛋白)亚家族;HAT包含HAG(GCN5-、ELP3-和HAT1-样组蛋白乙酰化酶结构域)、HAM(MOZ-YBF2结构域)、HAC(p300/creb结合蛋白结构域)和HAF(TATA-结合蛋白相关因子-TAFⅡ250)亚家族;HDAC包含HDA(RPD3/HDA1superfamily)、SRT(Silent information regulator 2)和HDT(HD2 family)亚家族[9-12]。已有研究表明,不同植物中含有不同数量的HM基因成员,如苹果、香橙、荔枝、油棕榈中分别含有198、136、87、109个HM[13-17]。【本研究切入点】番茄是重要的园艺作物,同时也是一种模式植物,主要用于果实发育等研究。虽然番茄中已见HDAC家族基因的分离鉴定[18],但关于番茄HM全基因家族的研究尚未见报道。【拟解决的关键问题】本研究拟通过番茄全基因组分析,鉴定番茄中该基因家族的成员,并详细分析各成员的染色体定位、基因结构和蛋白保守基序特征,揭示各基因家族成员与拟南芥(Arabidopsis thaliana)和水稻(Oryza sativa)同源基因的亲缘关系,同时明确各基因的组织器官表达特征,为深入研究番茄HM各基因的功能提供依据。
1 材料与方法 1.1 HM家族成员的鉴定番茄基因组数据来源于Sol Genomics Network(https://solgenomics.net/)网站。通过已知的拟南芥、水稻基因组及其HM基因信息,使用E value < 1e-10的BLSATP程序对番茄基因组进行查询。从Pfam数据库(https://pfam.xfam.org/)下载HM家族HMM模型文件(附件S1),使用HMMER软件进一步确定假定序列。
1.2 番茄HM基因结构分析根据番茄基因组注释信息,通过GSDS(http://gsds.cbi.pku.edu.cn/)网站在线分析并对番茄HM基因结构进行可视化绘图。
1.3 番茄HM蛋白系统进化树构建从拟南芥基因数据网站(https://www.arabidopsis.org/index.jsp)获取拟南芥HM蛋白序列,从水稻基因组数据库(http://rice.uga.edu/index.shtml)获取水稻HM蛋白序列,利用茄科数据库(http://palmxplore.mpob.gov.my/palmxplore/)查找下载番茄HM蛋白序列。使用ClustalW Muscle默认参数对番茄、拟南芥、水稻的HM蛋白序列进行多序列比对,使用MEGA 7.0软件[19-20]以最大似然法分别构建番茄、拟南芥、水稻三者HMT、HDM、HAT和HDAC成员的系统发育树,bootstrap值为1 000。
1.4 番茄HM基因染色体定位根据番茄全基因组注释数据,获取番茄各染色体长度及HM基因在染色体上的位置,并利用TBtools软件对其进行可视化绘图[21]。
1.5 番茄HM基因在不同组织中的表达分析从Tomexpress(http://tomexpress.toulouse.inra.fr/)获取HM基因的RNA-seq数据,采用Tbtools绘制热图,研究HM基因的时空表达模式。
2 结果与分析 2.1 番茄HM基因家族的鉴定在番茄中共鉴定到148个组蛋白修饰基因(HM),其中编码组蛋白甲基转移酶(HMT)的基因50个、编码组蛋白去甲基化酶(HDM)的基因55个、编码组蛋白乙酰化酶(HAT)和组蛋白去乙酰化酶(HDAC)的基因分别有32和11个。番茄HM基因家族成员根据其蛋白结构域被分为11个亚家族(SDG、PRMT、HDMA、JMJ、HAG、HAM、HAC、HAF、HDA、SRT和HDA)。HMT家族包含SDG和PRMT;HDM家族包含HDMA和JMJs;HAT家族包括HAGs、HAMs、HACs和HAF;HDAC家族包括SRT、HDA和HDA亚家族。番茄HM基因家族成员的所有基因ID见附件S2。
2.2 番茄HM基因家族成员染色体定位分析对148个番茄HM成员的染色体定位进行分析,结果(图 1)显示,番茄HM基因家族成员不均匀地分布在12条染色体上。其中,3号染色体上分布最多、有21个基因,12号染色体分布最少、有4个基因。

|
| 图 1 番茄中HM基因的染色体定位 Fig. 1 Chromosomal localization of HM genes in tomato |
2.3 番茄HM蛋白结构预测与基因结构分析
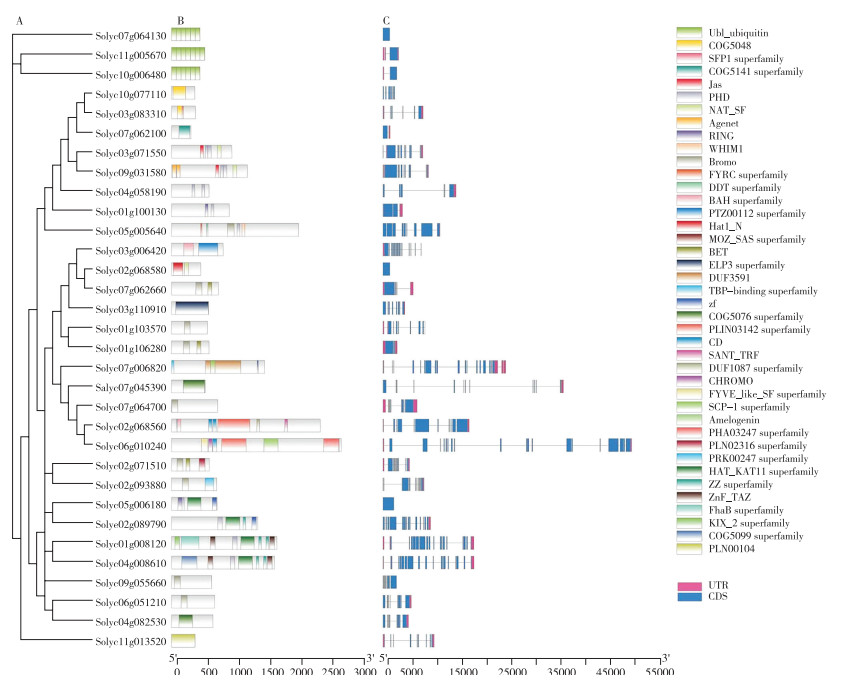
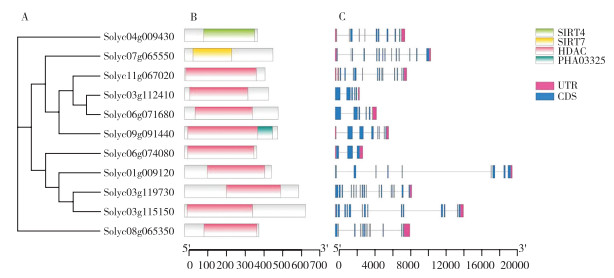
HM家族成员蛋白结构预测结果(图 2B、图 3B、图 4B、图 5B)表明,Solyc07g064130、Solyc11g005670、Solyc10g006480仅存在Ubl结构域;Solyc07g062100、Solyc10g077110和Solyc03g083310仅含有Zn-finger结构域;Solyc03g110910包含ELP3结构域、N端自由基SAM结构域和C端GNAT乙酰转移酶结构域,其中ELP3结构域是RNA聚合酶Ⅱ全酶的组成部分。Solyc05g005640、Solyc07g062660、Solyc01g103570、Solyc01g106280、Solyc07g064700、Solyc02g068560、Solyc02g071510、Solyc02g093880、Solyc09g055660、Solyc06g051210等10个成员含有Bromo结构域。Solyc04g009430和Solyc07g065550含有SIRT结构域。Solyc05g024100、Solyc07g020990、Solyc05g024040、Solyc11g030760、Solyc11g030780、Solyc05g055590、Solyc05g016300、Solyc05g051150、Solyc05g051153、Solyc05g024070、Solyc05g040010、Solyc11g008320和Solyc05g051157成员含有PLN03000结构域。Solyc10g049410、Solyc05g006250、Solyc07g063500、Solyc07g063450蛋白存在PLN02529结构域,该结构域与赖氨酸特异性组蛋白去甲基化有关。负责组蛋白甲基化的酶类有3种,其中含赖氨酸特异性SET结构域的组蛋白甲基化酶负责组蛋白H的4位(H3K4)、9位(H3K9)、20位、27位(H3K27)赖氨酸的甲基化。此外,Solyc04g081100存在PLN02976结构域,Solyc08g005970具有PRMT5结构域,其他49个基因均存在SET结构域。
|
| A:系统进化树;B:蛋白结构域;C:基因结构 A: Phylogenetic tree; B: Protein structure domain; C: Gene structure 图 2 番茄HAT家族的蛋白结构域和基因结构分析 Fig. 2 Analysis of protein structure domain and gene structure of the HAT family in tomato |
|
| A:系统进化树;B:蛋白结构域;C:基因结构 A: Phylogenetic tree; B: Protein structure domain; C: Gene structure 图 3 番茄HDAC家族的蛋白结构域和基因结构分析 Fig. 3 Analysis of protein structure domain and gene structure of the HDAC family in tomato |
|
| A:系统进化树;B:蛋白结构域;C:基因结构 A: Phylogenetic tree; B: Protein structure domain; C: Gene structure 图 4 番茄HDM家族的蛋白结构域和基因结构分析 Fig. 4 Analysis of protein structure domain and gene structure of the HDM family in tomato |
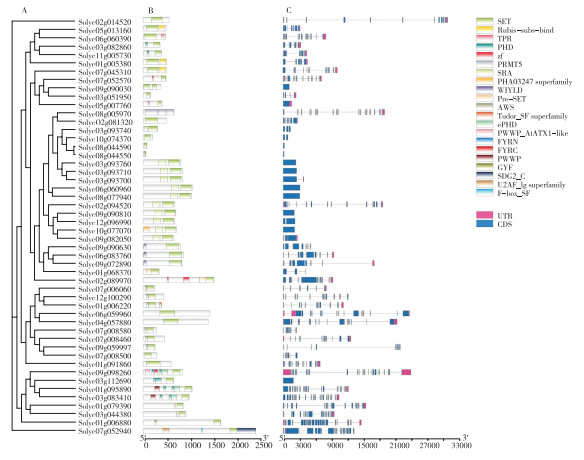
|
| A:系统进化树;B:蛋白结构域;C:基因结构 A: Phylogenetic tree; B: Protein structure domain; C: Gene structure 图 5 番茄HMT家族的蛋白结构域和基因结构分析 Fig. 5 Analysis of protein structure domain and gene structure of the HMT family in tomato |
HM基因家族结构特征分析结果(图 2C)表明,HM基因家族成员中外显子数量不同。在HAT家族中,Solyc07g064130、Solyc02g068580和 Solyc05g006180均只有1个外显子,而Solyc07g006820基因却有21个外显子。在HDM家族中,Solyc04g028580、Solyc05g0024100、Solyc05g024040、Solyc12g008850、Solyc05g018880、Solyc07g039310、Solyc11g030760、Solyc05g051150、Solyc05g051153、Solyc10g04910均只有1个外显子,而Solyc08g081000基因外显子多达34个。在HDAC家族中,Solyc06g074080基因仅有3个外显子,而 Solyc03g119730基因有17个外显子。在HMT家族中,Solyc09g090030、Solyc06g007760、Solyc08g044590、Solyc08g044550、Solyc03g093760、Solyc03g093710、Solyc06g060960、Solyc08g077940、Solyc09g090810、Solyc10g077070、Solyc03g112690均仅有1个外显子,Solyc09g098260基因外显子数量多达25个(图 2C、图 3C、图 4C、图 5C)。
2.4 番茄HM家族成员系统发育分析由图 6、图 7可知,番茄中HDAC、HDM家族的蛋白序列与拟南芥和水稻中的HM蛋白同源关系均较远。但整体来看,与水稻相比,番茄HM蛋白序列与拟南芥HM蛋白序列在进化关系上更为相近。
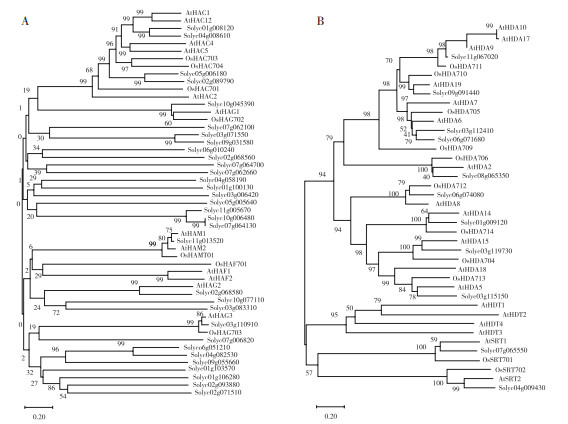
|
| 图 6 番茄、拟南芥和水稻HAT(A)、HDAC(B)家族成员的系统发育关系 Fig. 6 Phylogenetic relationship of HAT (A) and HDAC (B) members in tomato, Arabidopsis thaliana and Oryza sativa |

|
| 图 7 番茄、拟南芥和水稻HMT(A)、HDM(B)成员的系统发育关系 Fig. 7 Phylogenetic relationship of HMT (A) and HDM (B) members in tomato, Arabidopsis thaliana and Oryza sativa |
2.5 番茄HM基因家族的共线性分析
为了解番茄基因组中HM基因家族的扩张情况,本研究利用Circos生成一个重复区块的基因复制事件图(图 8)。通过比较12条番茄染色体上的148个番茄HM基因,共鉴定出13对HM基因存在连锁。HDM家族所含数量最多(6对),分别为Solyc02g081390和Solyc03g031880、Solyc03g097090和Solyc08g076390、Solyc03g097090和Solyc08g005240、Solyc05g055590和Solyc11g008320、Solyc07g039310和Solyc12g008850、Solyc07g043590和Solyc12g006370,其中Solyc03g097090为重复基因;HMT家族共有5对,分别为Solyc01g095890和Solyc03g083410、Solyc01g006220和Solyc12g100290、Solyc06g060960和Solyc08g077940、Solyc06g083760和Solyc09g072890、Solyc09g072890和Solyc09g090630;HAT家族共有1对,Solyc07g064130和Solyc10g006480;HDAC家族共有1对,Solyc03g112410和Solyc06g071680。配对的HM重复基因均位于番茄基因组的不同染色体块中(图 8)、如3号染色体,以上结果暗示番茄HM基因家族扩张可能是通过这些重复区域发生。
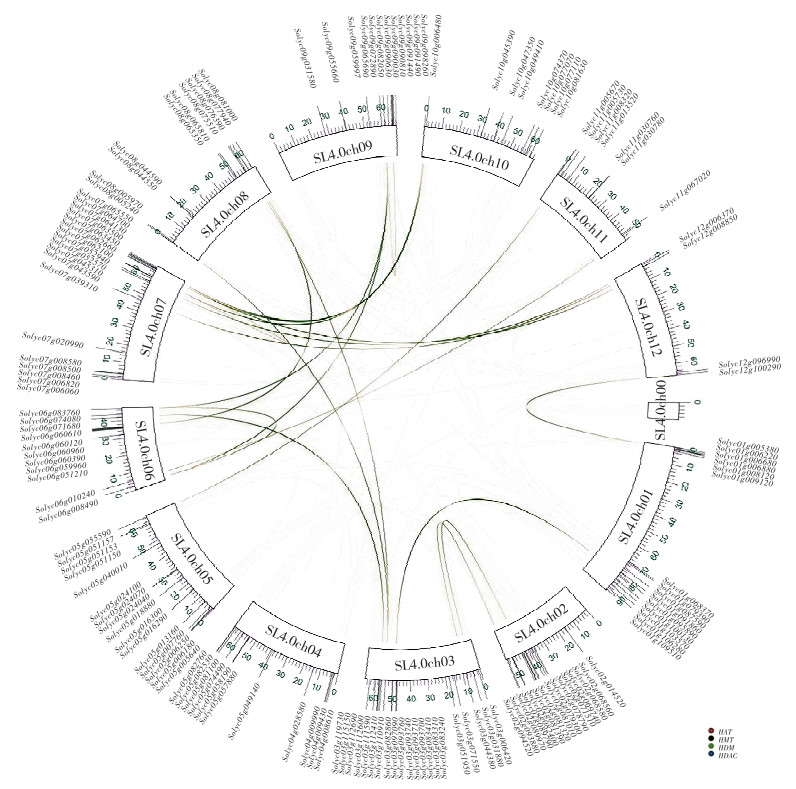
|
| 图 8 番茄HM基因复制事件及染色体间关系 Fig. 8 HM gene replication events and interchromosomal relationships in tomato |
2.6 番茄HM基因家族的组织表达分析
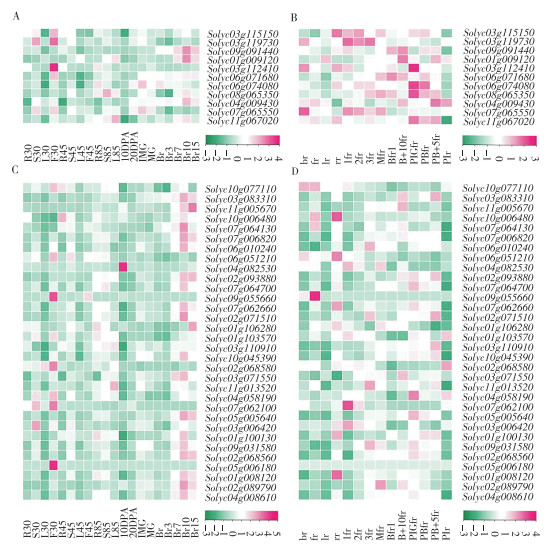
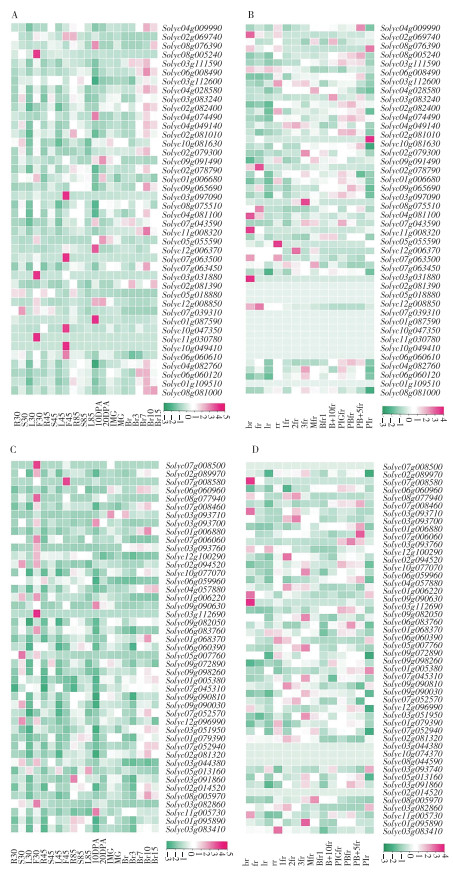
为分析148个番茄HM基因家族成员在不同组织的表达水平,本研究利用公开的转录组数据分析番茄不同发育组织中HM的转录丰度,结果如图 9、图 10所示,番茄HM基因成员在3个不同品种(‘Micro-Tom’‘pimp’‘Heinz’)不同发育阶段的组织中差异表达。
|
| ‘Micro Tom’品种-芽期:发芽后30 d(R30:根,S30:茎,L30:叶,F30:花);开花期:全株50% 的花开放时(R45:根,S45:茎,L4:叶,F45:花);破色期:第1个果实达到破色期时(R85:根,S85:茎,L85:叶);10 DPA、20 DPA:开花后10、20 d的果实;IMG、MG:未成熟、成熟绿色果实;Br、Br3、Br7、Br10、Br15:果实破色第1、3、7、10、15 d.‘Heinz’品种-br:花蕾;fr:花;lr:叶;rr:根;1fr:1 cm果实;2fr:2 cm果实;3fr:3 cm果实;Mfr:成熟果实;Bfr1、Bfr2:绿熟期果实;B+10fr1、B+10fr:果实破色第10 d.‘Pimp’品种-Plgfr:未成熟果实;PB+5fr:未成熟果实生长到5cm时;Plf:叶 'Micro Tom' variety-Bud stage: 30 days after germination (R30: Root, S30: Stem, L30: Leaf, F30: Flower); Anthesis: When 50 % of the whole plant flowers open (R45: Root, S45: Stem, L4: Leaf, F45: Flower); Breaker: When the first fruit reaches color break (R85: Root, S85: Stem, L85: Leaf); 10 DPA, 20 DPA: Fruits at ten, twenty days post-anthesis; IMG, MG: Immature, Mature green fruit; Br, Br3, Br7, Br10, Br15: Fruit color breaking day 1, 3, 7, 10, 15. 'Heinz' variety-br: Buds; fr: Flower; lr: Leaf; rr: Root; 1fr: 1 cm fruit; 2fr: 2 cm fruit; 3fr: 3 cm fruit; MFr: Mature fruit; Bfr1, Bfr2: Green ripe fruit; B+10fr1, B+10fr: Fruit color breaking day 10. 'Pimp' variety-Plgfr: Immature fruit; PB+5fr: When immature fruit grows to 5cm fruit; Plf: Leaf 图 9 番茄HDAC(A、B)与HAT(C、D)家族基因在不同组织不同发育时期的表达分析 Fig. 9 Expression analysis of tomato HDAC (A, B) and HAT (C, D) family genes in different tissues at different developmental stages |
|
| ‘Micro Tom’品种-芽期:发芽后30 d(R30:根,S30:茎,L30:叶,F30:花);开花期:全株50% 的花开放时(R45:根,S45:茎,L4:叶,F45:花);破色期:第1个果实达到破色期时(R85:根,S85:茎,L85:叶);10 DPA、20 DPA:开花后10、20 d的果实;IMG、MG:未成熟、成熟绿色果实;Br、Br3、Br7、Br10、Br15:果实破色第1、3、7、10、15 d.‘Heinz’品种-br:花蕾;fr:花;lr:叶;rr:根;1fr:1 cm果实;2fr:2 cm果实;3fr:3 cm果实;Mfr:成熟果实;Bfr1、Bfr2:绿熟期果实;B+10fr1、B+10fr:果实破色第10 d.‘Pimp’品种-Plgfr:未成熟果实;PB+5fr:未成熟果实生长到5cm时;Plf:叶 'Micro Tom' variety-Bud stage: 30 days after germination (R30: Root, S30: Stem, L30: Leaf, F30: Flower); Anthesis: When 50 % of the whole plant flowers open (R45: Root, S45: Stem, L4: Leaf, F45: Flower); Breaker: When the first fruit reaches color break (R85: Root, S85: Stem, L85: Leaf); 10 DPA, 20 DPA: Fruits at ten, twenty days post-anthesis; IMG, MG: Immature, Mature green fruit; Br, Br3, Br7, Br10, Br15: Fruit color breaking day 1, 3, 7, 10, 15. 'Heinz' variety-br: Buds; fr: Flower; lr: Leaf; rr: Root; 1fr: 1 cm fruit; 2fr: 2 cm fruit; 3fr: 3 cm fruit; MFr: Mature fruit; Bfr1, Bfr2: Green ripe fruit; B+10fr1, B+10fr: Fruit color breaking day 10. 'Pimp' variety-Plgfr: Immature fruit; PB+5fr: When immature fruit grows to 5cm fruit; Plf: Leaf 图 10 番茄HDM(A、B)与HMT(C、D)家族基因在不同组织不同发育时期的表达分析 Fig. 10 Expression analysis of tomato HDM (A, B) and HMT (C, D) family genes in different tissues at different developmental stages |
HDAC家族成员(图 9A、图 9B),如‘Micro-Tom’品种中,Solyc03g112410基因在发芽后30 d的花(F30)中高表达;‘Pimp’品种中,Solyc03g112410、Solyc06g074080、Solyc08g065350、Solyc11g067020等4个基因在未成熟果实(Plgfr)中高表达。
HAT家族成员(图 9C、图 9D),如‘Micro-Tom’品种中,Solyc09g055660、Solyc05g006180在发芽后30 d的花(F30)中高表达,Solyc04g082530基因在开花后10 d的果实中(10 DPA)高表达;‘Heinz’品种中,Solyc09g055660基因在花(fr)中高表达,Solyc10g006480、Solyc06g051210、Solyc01g008120基因在根(rr)中高表达,Solyc04g058190基因在未成熟果实(Plgfr)中高表达。
HDM成员(图 10A、图 10B),如‘Micro-Tom’品种中,Solyc08g005240、Solyc03g031880、Solyc11g030780等3个基因在发芽后30 d的花(F30)中高表达,Solyc03g097090、Solyc07g063500、Solyc10g047350、Solyc10g049410、Solyc06g060610基因在全株50% 的花开放时的花(F45)中高表达,Solyc12g006370、Solyc01g087590等基因在开花后10 d的果实中(10 DPA)高表达;‘Heinz’品种中,Solyc02g069740、Solyc07g063500、Solyc03g031880、Solyc11g030780等4个基因在花蕾(br)中高表达,Solyc03g097090基因在花(fr)中高表达,Solyc05g018880基因在根(rr)中高表达,Solyc05g055590基因在3 cm果实(3fr)中高表达,Solyc09g091490基因在未成熟果实生长到5 cm时的叶片中(Plr)中高表达。
HMT成员(图 10C、图 10D),如‘Micro-Tom’品种中,Solyc07g008500、Solyc03g112690基因在发芽后30 d的花(F30)中高表达,Solyc07g008580基因在全株50% 的花开放时的花(F45)中高表达;‘Heinz’品种中,Solyc07g008580、Solyc03g112690基因在花蕾(br)中高表达,Solyc01g095890基因在3 cm果实(3fr)中高表达;‘Pimp’品种中,Solyc07g006060基因在未成熟5 cm果实(PB+5fr)中高表达。
3 讨论组蛋白修饰酶(HM)通过修饰调节组蛋白的状态,在植物不同的生长和发育过程中发挥重要作用。目前,已有研究在拟南芥、水稻、苹果等多种植物中分析了组蛋白修饰酶基因家族,但番茄中尚未对该家族基因进行鉴定,且其具体生物学功能也不清楚。
本研究共鉴定出148个番茄HM成员,分别为32个组蛋白乙酰转移酶(HAT)、11个组蛋白去乙酰化酶(HDAC)、50个组蛋白甲基转移酶(HMT)和55个组蛋白去甲基化酶(HDM),并分析了它们在染色体上的位置、基因结构、保守基序、基因复制事件及系统发育关系,揭示了该类基因与拟南芥和水稻同源基因亲缘关系的远近。同时,检测分析了全部基因在不同组织的表达模式。番茄HM基因家族成员数量与已鉴定物种成员的数量相近。例如,在油棕中共鉴定到109个HM基因家族成员,其中HMT 48个,HDM 27个[15],同时发现其基因组上存在13种组蛋白乙酰化酶亚基(简称H型)与21种组蛋白去羧化酶亚基(简称C型)。Peng等[17]从荔枝基因组上共鉴定出87个HM家族成员,其中HAT 6个、HDAC 11个、HMT 48个和HDM 22个。这些结果表明,在不同植物中该类基因家族成员的数量存在很大差异,进一步体现了植物在进化过程中的多样性。
利用已知的转录组数据库,本研究分析了番茄HM基因家族成员在不同组织的表达水平。HDAC家族中,Solyc01g009120在叶组织中高表达,且有报道指出该基因定位于叶绿体[22],暗示该基因可能在番茄叶片叶绿体形成和发育调控中起着重要作用。Solyc06g071680在果实发育的未成熟阶段高表达,并随着果实发育其表达量下降,表明该基因可能在番茄果实发育调控中起作用,这与已报道的结果[23]一致。水稻中,编码HDM家族的OsJMJ706成员通过对组蛋白H3赖氨酸9(H3K9)的二甲基和三甲基的去甲基化作用,参与调节水稻花器官的发育,影响其外壳开放性[24]。系统进化分析显示,番茄中的Solyc08g076490和Solyc08g0005240与OsJMJ706高度同源。此外,Solyc08g076490和Solyc08g0005240在不同番茄品种花组织中的表达水平均较高,这暗示着二者也极有可能参与番茄花器官的发育。编码HMT家族的Solyc04g057880和Solyc06g059960均在果实发育期高表达,且系统进化分析显示二者与拟南芥AtSDG8亲缘关系最近,而研究表明AtSDG8可通过调节CRTISO(胡萝卜素异构酶)上的H3K4三/二甲基化来影响拟南芥中类胡萝卜素的生物合成[25]。类胡萝卜素作为番茄果实重要的组成成分,推测Solyc04g057880和Solyc06g059960也可能参与了番茄果实中类胡萝卜素的生物合成,但这些基因的具体功能及分子调控机制有待进一步验证。
4 结论本研究共鉴定出148个番茄HM基因(32个HAT、11个HDAC、50个HMT和55个HDM),不均匀地分布在12条染色体上。不同基因其结构差异较大,均包含多种功能结构域。番茄HM与拟南芥和水稻的同源关系较远,且在植物不同生长发育阶段及不同组织中均有一定的表达,暗示其可能参与了番茄多种生长发育过程。其中,部分基因在特定的组织中高表达,有望进一步开展分子研究验证其功能,为番茄遗传改良奠定基础。
| [1] |
李君明, 项朝阳, 王孝宣, 国艳梅, 黄泽军, 刘磊, 李鑫, 杜永臣. "十三五"我国番茄产业现状及展望[J]. 中国蔬菜, 2021(2): 13-20. DOI:10.19928/j.cnki.1000-6346.2021.2009 LI J M, XIANG C Y, WANG X X, GUO Y M, HUANG Z J, LIU L, LI X, DU Y C. Current situation of tomato industry in China during "The Thirteenth Five-Year Plan" period and future prospect[J]. Chinese Vegetables, 2021(2): 13-20. DOI:10.19928/j.cnki.1000-6346.2021.2009 |
| [2] |
陈金香, 王艳娣, 梁健文, 谢振斌, 覃敏, 杨小龙, 陈日远, 张轶婷. 华南连栋温室基质袋培模式下6个樱桃番茄品种的综合鉴评[J]. 广东农业科学, 2023, 50(9): 142-154. DOI:10.16768/j.issn.1004-874X.2023.09.015 CHEN J X, WANG Y D, LIANG J W, XIE Z B, QIN M, YANG X L, CHEN R Y, ZHANG Y T. Comprehensive evaluation of 6 cherry tomato varieties under the substrate bag cultivation mode in multi-span greenhouse in south china[J]. Guangdong Agricultural Sciences, 2023, 50(9): 142-154. DOI:10.16768/j.issn.1004-874X.2023.09.015 |
| [3] |
贾俊婷, 陈兵先, 吴柔贤, 戴彰言, 钟明生, 解昊, 刘双幸, 刘军. 水稻组蛋白去甲基化酶基因OsJMJ719的克隆及表达分析[J]. 广东农业科学, 2022, 49(11): 152-161. DOI:10.16768/j.issn.1004-874X.2022.11.016 JIA J T, CHEN B X, WU R X, DAI Z Y, ZHONG M S, XIE H, LIU S X, LIU H. Analysis on cloning and expression of histone d'emethylase gene OsJMJ719 in rice[J]. Guangdong Agricultural Sciences, 2022, 49(11): 152-161. DOI:10.16768/j.issn.1004-874X.2022.11.016 |
| [4] |
ANDREW J B, TONY K. Regulation of chromatin by histone modifications[J]. Cell Research, 2011, 21(3): 381-395. DOI:10.1038/cr.2011.22 |
| [5] |
TONY K. Chromatin modifications and their function[J]. Cell, 2007, 128(4): 693-705. DOI:10.1016/j.cell.2007.02.005 |
| [6] |
LU T, FONG M P, WANG J Y, NING E W, HONG M J, DOERGE R W, CHEN Z J. Reversible histone acetylation and deacetylation mediate genome-wide, promoter-dependent and locus-specific changes in gene expression during plant development[J]. Genetics, 2005, 169(1): 337-345. DOI:10.1534/genetics.104.033142 |
| [7] |
FUCHS J, DEMIDOV D, HOUBEN A, SCHUBERT I. Chromosomal histone modification patterns-from conservation to diversity[J]. Trends in Plant Science, 2006, 11(4): 199-208. DOI:10.1016/j.tplants.2006.02.008 |
| [8] |
卢晶霞, 杨松光, 吴克强. 番茄组蛋白去乙酰化酶HD2家族生物信息学及表达模式分析[J]. 广东农业科学, 2014, 41(12): 138-143. DOI:10.16768/j.issn.1004-874X.2014.12.004 LU J X, YANG S G, WU K Q. Bioinformatics and expression pattern analysis of histonedeacetylase HD2 subfamily in tomato[J]. Guangdong Agricultural Sciences, 2014, 41(12): 138-143. DOI:10.16768/j.issn.1004-874X.2014.12.004 |
| [9] |
AQUEA F, VEGA A, TIMMERMANN T, POUPIN M J, JOHNSON P A. Genome-wide analysis of the SET domain group family in grapevine[J]. Plant Cell Reports, 2011, 30(6): 1087-1097. DOI:10.1007/s00299-011-1015-0 |
| [10] |
JIANG D H, YANG W N, HE Y H, AMASINO R M, NOTERS A. Arabidopsis relatives of the human lysine-specific demethylase1 repress the expression of FWA and FLOWERING LOCUS C and thus promote the floral transition[J]. The Plant Cell, 2007, 19(10): 2975-2987. DOI:10.1105/tpc.107.052373 |
| [11] |
PANDEY R, MULLER A, NAPOLI C A, SELINGER D A, PIKAARD C S, RICHARDS E J, BENDER J, MOUNT D W, JORGENSEN R A. Analysis of histone acetyltrans-ferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes[J]. Nucleic Acids Research, 2002, 30(23): 5036-5055. DOI:10.1093/nar/gkf660 |
| [12] |
FENG Q, WANG H B, NG H H, BROMAGE H E, TEMPST P, STRUHL K, ZHANG Y. Methylation of H3-lysine 79 is mediated by a new family of HMT ases without a SET domain[J]. Current Biology, 2002, 12(12): 1052-1058. DOI:10.1016/S0960-9822(02)00901-6 |
| [13] |
RICCARDO A C, WALTER S, GAETANA C MARIA R E, FEAERICA M C, FEDERICA M C. Genome-wide analysis of histone modifiers in tomato: Gaining an insight into their developmental roles[J]. BMC Genomics, 2013, 14(1): 57. DOI:10.1186/1471-2164-14-57 |
| [14] |
XU J D, XU H D, LIU Y L, WANG X, XU Q, DENG X X. Genome-wide identification of sweet orange (Citrus sinensis) histone modification gene families and their expression analysis during the fruit development and fruit-blue mold infection[J]. Frontiers in Plant Science, 2015, 6: 607. DOI:10.3389/fpls.2015.00607 |
| [15] |
ZHOU L X, RAJESH Y, JIN L F, YANG Y D, CAO H X, ZHAO Z H. Identification and expression analysis of histone modification gene (HM) family during somatic embryogenesis of oil palm[J]. BMC Genomics, 2022, 23(1): 11. DOI:10.1186/s12864-021-08245-2 |
| [16] |
FAN S, WANG J, LEI C, GAO C, YANG Y, LI Y M, AN N, ZHANG D, HAN M Y. Identification and characterization of histone modification gene family reveal their critical responses to flower induction in apple[J]. BMC Plant Biology, 2018, 18(1): 173. DOI:10.1186/s12870-018-1388-0 |
| [17] |
PENG M J, YING P Y, LIU X C, LI C Q, XIA R, LI J G, ZHAO M L. Genome-wide identification of histone modifiers and their expression patterns during fruit abscission in litchi[J]. Frontiers in Plant Science, 2017, 8: 639. DOI:10.3389/fpls.2017.00639 |
| [18] |
ZHAO L M, LU J X, ZHANG J X, WU P W, YANG S G, WU K Q. Identification and characterization of histone deacetylases in tomato (Solanum lycopersicum[J]. Frontiers in Plant Science, 2015, 5: 760. DOI:10.3389/fpls.2014.00760 |
| [19] |
THOMPSON J D, HIGGINS D G, GIBSON T J. CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 1994, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673 |
| [20] |
TAMURA K, PETERSON D, PETERSON N, STECHER G, NEI M, KUMAR S, NOTES A. MEGA 5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. DOI:10.1093/molbev/msr121 |
| [21] |
CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [22] |
ZHOU C H, LABBE H, SRIDHA S, WANG L, TIAN L N, GREEN M L, YANG Z, BROWN D, MIKI B, WU K Q. Expression and function of HD2-type histone deacetylases in Arabidopsis development[J]. The Plant Journal: for Cell and Molecular Biology, 2004, 38(5): 715-724. DOI:10.1111/j.1365-313X.2004.02083.x |
| [23] |
GOU J E, HU Z L, LI F F, ZHANG L C, YU X H, TANG B Y, CHEN G P. Silencing of histone deacetylase SlHDT3 delays fruit ripening and suppresses carotenoid accumulation in tomato[J]. Plant Science, 2017, 265: 29-38. DOI:10.1016/j.plantsci.2017.09.013 |
| [24] |
LIU H H, GOU S Y, XU Y Y, LI C H, ZHANG Z Y, ZHANG D J, XU S J, ZHANG C, CHONG K, NOTES A. OsmiR396d-regulated OsGRFs function in floral organogenesis in rice through binding to their targets OsJMJ706 and OsCR4[J]. Plant Physiology, 2014, 165(1): 160-174. DOI:10.1104/pp.114.235564 |
| [25] |
CAZZONELLI C I, CUTTRISS A J, COSSETTO S B, PYE W, CRISP P, WHELAN J, FINNEGAN E J, TURNBUL C, POGSON B J, NOTES A. Regulation of carotenoid composition and shoot branching in arabidopsis by a chromatin modifying histone methyltransferase, SDG8[J]. The Plant Cell, 2009, 21(1): 39-53. DOI:10.1105/tpc.108.063131 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51