文章信息
基金项目
- 国家重点研发计划项目(2018YFC1706400);广东省林业局生态林业发展项目(2019-42091278)
作者简介
- 陈志晖(1995—),男,在读博士生,研究方向为系统发育区系学,E-mail:chenzhh93@mail2.sysu.edu.cn.
通讯作者
- LEE Shiou Yih(1985—),男,博士,教授,研究方向为森林生物科技,E-mail:shiouyih.lee@newinti.edu.my.
文章历史
- 收稿日期:2023-11-28
2. 深圳市坪山规划和自然资源研究中心,广东 深圳 518000;
3. 广西师范大学生命科学学院,广西 桂林 541004;
4. 中国医学科学院 & 北京协和医学院药用植物研究所海南分所/海南省南药资源保护与开发重点实验室/中药质量国际联合研究中心,海南 海口 570311;
5. 英迪国际大学健康与生命科学学院,马来西亚 森美兰 汝来 71800
2. Pingshan Research Center for Planning and Natural Resources in Shenzhen, Shenzhen 518000, China;
3. School of Life Sciences, Guangxi Normal University, Guilin 541004, China;
4. Hainan Branch Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College/Hainan Key Laboratory of Resources Conservation and Development of Southern Medicine/International Joint Research Center for Quality of Traditional Chinese Medicine, Haikou 570311, China;
5. Faculty of Health and Life Sciences, INTI International University, Nilai 71800, Malaysia
沉香(Agarwood)为瑞香科(Thymelaeaceae)沉香属(Aquilaria Lam.)植物,是名贵的传统药材,也是熏香、香水等香料的世界性原料,在生长过程中沉香木质部受到外伤或被菌类感染后逐渐形成含树脂的木材[1]。全世界记录的沉香有21种[2],主要分布于东南亚和南亚地区,包括中国、缅甸、泰国、越南、老挝、柬埔寨、马来西亚、文莱、印度尼西亚、印度和不丹等地。其中,马来西亚和菲律宾的沉香属物种生物多样性最为丰富,约占沉香属1/2物种,其次为印度尼西亚和泰国[3]。在我国仅分布土沉香〔Aquilaria sinensis(Lour.)Spreng.〕及云南沉香(A. yunnanensis S. C. Huang)两种,但近年在越南发现云南沉香的新记录[4]。沉香种质相关产业经历了大规模种苗繁育及种植阶段,期间也曾出现过种源混乱的局面。在种苗繁育的推动下,各地陆续建立了大规模的沉香种植基地,随着沉香原料的增多,各类的沉香衍生产品也开始在市场上出现。目前国际上尚未形成沉香产品加工中心,印度尼西亚、马来西亚、越南、泰国等东南亚国家仅有零散的沉香精油加工作坊。而我国沉香产业加工体系则较为完善,加工产品主要分为4大类300多种,福建省是我国主要的进口沉香粗加工基地,广东省是沉香加工及贸易的集散地,而海南省现有医药、健康养生、宗教、日化等不同业态的沉香产品百余款,其中文化饰品、熏香类产品等初级加工类商品占90% 以上。
濒危野生动植物种国际贸易公约(CITES)数据显示,东北亚(日本、韩国及中国台湾)和西亚(中东地区)为沉香最主要的两大终端市场,且沉香进出口量逐年增加,世界沉香年贸易额达200亿~300亿美元,主要来自印度尼西亚、马来西亚和越南,主要销往地为沙特阿拉伯、阿联酋、日本和中国。以马来西亚、印度尼西亚、越南、缅甸等为主的东南亚国家,占据全球90% 的沉香贸易出口份额,中东国家进口印度尼西亚沉香比例最高、占60%~70%。自2005年开始,为确保沉香属物种在可持续体系下开展贸易,CITES附录Ⅱ将沉香属全部列入贸易管制物种。尽管沉香的人工种植和结香技术在不断发展,但由于市场对天然沉香的高需求,野生种群的砍伐偷盗现象难以杜绝[5]。2012—2018年,我国土沉香的野生资源锐减1/4以上,可能仅存8万株[6]。已有13个沉香属物种列入世界自然保护联盟(IUCN)红色名录,其中极危(CR)4种、濒危(EN)1种,易危(VU)8种,还有7种评估数据不足[7]。在我国,土沉香和云南沉香均被列为国家Ⅱ级重点保护野生植物[8]。目前对于沉香属植物的研究多集中在沉香种质资源及其开发利用[9-10]、栽培育种[11]、生理生态学[12-13]、化学成分[14]、人工结香技术及结香机理[15-17]、质量评价[18-19]、药理药效[20-21]及贸易[22-23]等方面,但沉香的质量受基原物种、产地、结香方式及遗传多样性等因素的影响较大。遗传多样性是生物多样性的基础,物种的稳定性和进化潜力依赖于遗传多样性,其关系到物种多样性的保护,可影响生态环境的稳态与经济价值[24]。对于药用价值而言,遗传多样性更是决定其次生代谢产物的重要因素[25]。此外,遗传多样性也是种质资源利用的关键,可为育种和遗传改良提供依据。目前,随着分子生物学技术的发展,沉香属植物的遗传多样性呈现出由宏观的植物表型特性向微观分子水平发展的趋势。因此,本文对近年来国内外沉香属物种的遗传多样性和分子鉴定相关研究进行综述,以期为沉香属物种的遗传多样性保护、种质资源的筛选、鉴定和遗传育种等提供参考。
1 沉香种质资源的开发利用 1.1 普通沉香的种植现状全世界野生沉香主要出产于中国、东南亚、南亚和东亚等10余个国家和地区,沉香属植物全世界约有21种,拟沉香属(Gyrinopsis)植物有9种[3],其中野生植物资源中分布较广的有白木香(A. sinensis)、马来沉香(A. malaccensis)、贝卡利沉香(A. beccariana)、柯拉斯那沉香(A. crassna)、丝沉香(A. fi laria)、毛叶沉香(A. hirta)和小果沉香(A. microcarpa),国际上种植的沉香树种主要来源于野生资源(表 1)。目前,全球沉香树种植面积100多万hm2,其中印度尼西亚、马来西亚、越南、泰国等是沉香树主要种植国。我国仅有沉香属白木香和云南沉香2个特有乡土树种。白木香是最为主要的乡土基原树种,主要分布于海南、广东、广西、云南等地,四川、重庆、江西、贵州等地也有零星种植[10];云南沉香仅分布在云南与缅甸、老挝交界区域[26]。
1.2 奇楠沉香的种植现状
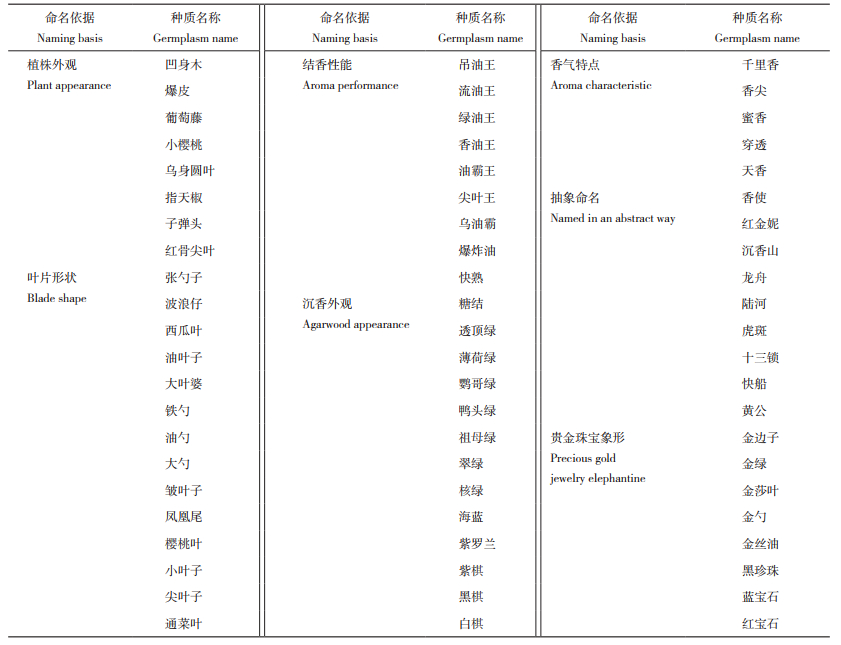
奇楠沉香(A. malaccensis)种质来自于我国广东、海南等地的野生沉香资源,通过DNA条形码初步分析显示,奇楠沉香种质与白木香亲缘关系最近[27]。目前,市面上所谓的“奇楠”只是一个统称,商户们根据不同种质的特征比对筛选出奇楠沉香的多个特性,并以此来自行命名,共分为66个种质7大类(表 2)。奇楠沉香种质种植以广东省茂名市电白区为中心,向四周扩散,随着嫁接技术在奇楠沉香种质繁育中取得成功,广东及周边省区如海南、广西、云南、福建等地已大规模种植。
|
2 沉香属植物遗传多样性研究 2.1 表型性状的遗传多样性研究
植物的表型性状可受遗传因素和生长环境的共同作用发生变化,常作为遗传变异的重要表征和研究线索。叶片作为植物光合作用的器官之一,具有可用于系统研究属种结构的稳定性[28]。赵翾等[29]分析了白木香群体表型多样性,发现居群内外叶片表型性状均存在广泛的变异性,居群内变异大于居群外,且海拔、年均气温和年降雨量等生长环境对表型性状的影响较大。林丹[30]分析了不同居群白木香叶片表型多样性,发现居群内的叶片表型变异是白木香表型总变异的主要来源。周元元等[31]分析了白木香种质叶片数量和质量性状的表型遗传多样性,发现可以依据叶的表型性状划分种质类群,类群间具有不同的形态特征和丰富的遗传多样性。
2.2 基于核基因组分子标记的遗传多样性研究DNA分子标记是指能反映生物个体或种群间基因组中某种差异的特异性DNA片段,能够从DNA水平直接反映生物的遗传本质,不受环境因素、采收加工、生长年限、组织器官发育等影响,具有灵敏性好、准确性高等特点[32-33]。因叶绿体DNA序列为母系遗传,不经历重组、易于扩增,可用于沉香属植物系统发育关系的推测[34]。Lee等[35]利用trnL-trnF序列重建了印度尼西亚4个沉香属物种的系统发育关系,但其结果不能较好地反映沉香属物种间的关系。Farah等[36]尝试利用matK、rbcL、trnL、trnL-trnF和psbC-trnS 5条序列的组合重建12个沉香属物种的系统发育树,结果显示多态性位点比例较低,种间分辨率不足。
在大多数被子植物中,与单亲遗传的质体DNA相比,双亲遗传的核糖体DNA保守性较差,进化速度较快,因此有助于区分种内差异;且双亲遗传的核基因也能为植物的网状进化、杂交和多倍体现象提供参考[37]。ITS(包含ITS1、5.8S和ITS2)在协同进化的过程中,其序列变异有同质化的趋势,在包括沉香属内的大多数物种中均容易进行扩增。利用ITS序列对沉香属种群多样性的研究尚少,目前仅见于大叶沉香(A. beccariana Teigh.)[38]、土沉香[39]和马来沉香[40] 3种。其中Pern等[38]研究了大叶沉香的6个居群(47个个体),将其划分为马来半岛和婆罗洲地区2个主要分化组。申彦晶等[39]指出,由于云开山脉从东北延伸至西南,形成了广东、广西两地的地理隔离屏障,将土沉香第一系(福建、广东和海南)的基因流动与第二系(广西和云南)分隔,同时发现与广东和海南两省分布的土沉香相比,分布于广西和云南的土沉香与云南沉香的遗传关系更近。Lee等[40]利用ITS序列检测了马来西亚9个马来沉香种群,发现种群间无明显分化,增加来自印度尼西亚和印度的居群样品后发现,来自3个国家的种群均各自聚类为不同组,表明地理边界可能起到缓冲区的作用,从而避免自然种群间的远交。此外,Lee等[41]利用ITS评估了包括来自马来西亚和泰国的7个沉香属物种之间的系统发育关系。之后,Farah等[36]将沉香属样品增加到12种,得到的ITS与叶绿体多序列组合的分析结果不完全一致。
基于对基因组进行PCR扩增的DNA指纹鉴定技术,则是通过扩增条带指纹的差异进行鉴定。目前,此项技术应用于沉香属物种分子鉴定的手段主要有随机扩增引物多态性DNA(Random amplified polymorphic DNA,RAPD)、简单重复序列(Simple sequence repeats,SSR)和简单重复序列区间(Inter simple sequence repeats,ISSR)。RAPD是通过设计随机引物对基因组DNA进行扩增的共显性分子标记技术。Lee等[42]针对马来西亚的马来沉香、毛叶沉香和未命名物种Aquilaria sp.,采用RAPD技术筛序得到23条引物,成功扩增出368个特异性条带,根据多种群的描述性统计,发现这3个种群之间有一定的相似性。随后,Qipitiyah等[43]对分布于印度尼西亚加里曼丹的两个小果沉香自然种群进行遗传多样性研究,共扩增出9对RAPD引物,检测到49个多态性位点,且发现东西部两个种群表现出遗传分化的性状。微卫星标记(SSR)主要是基于生物基因组序列中的单拷贝序列,通过序列两端设计特异引物,对重复序列进行扩增[44],目前针对沉香属的马来沉香、土沉香分别开发出了对55对和54对SSR引物[45-46]。Lee等[47]运用14个SSR引物,对马来西亚半岛35个野生马来沉香居群进行研究,利用SSR特有的等位基因对两个主要区域进行区分。Pern等[38]成功开发了厚叶沉香(A. crassna)、马来沉香和土沉香(A. sinensis)的30个微卫星标记,且在大叶沉香、毛叶沉香、小果沉香、锥果沉香(A. rostrata)、皱果沉香(A. rugosa)、泰国沉香(A. subintegra)和云南沉香等7个沉香属物种上进行评估,结果显示共有18个微卫星标记展现出良好效果,这18个标记所构建的UPGMA聚类结果也在多数物种的试验中表现出一致性。
ISSR则是利用真核生物基因组广泛存在的简单重复序列来设计引物,无需预先克隆和测序[47]。申彦晶等[39]首次采用ISSR标记,对我国不同地区的土沉香和云南沉香开展亲缘关系研究,结果显示广东和海南的土沉香同属一支,广西和云南的土沉香则构成另一支,外类群云南沉香独立分支,表明主要是地理因素造成了谱系分化。Zou等[48]分析了来自广东和海南的112个样品,结果发现这些样品共来自5个不同的居群。Banu等[49]使用ISSR技术针对马来沉香7个居群进行研究,发现印度居群内的遗传多样性较低,推测此类居群早期可能经历了远距离的人为迁移。Lee等[50]针对马来西亚半岛15个野生马来沉香居群进行ISSR标记,结果表明在柔佛州和彭亨州之间,沿着Titiwangsa山脉往西延伸至Keledang山脉和Bintang山脉的南部,是野生马来沉香基因的分界线,这条线形成了该地区两个野生沉香居群间基因流动的地理障碍,导致基因碎片化。为此,基于ISSR和SSR[42]结果的不一致性,恢复沉香属物种的基因池成为维持其野生居群遗传多样性的关键[48]。
2.3 基于细胞器基因组分子标记的遗传多样性研究高通量测序技术的发展为基因组规模数据的获取带来了便利,大大提高了分辨物种系统发育关系的能力。根据首个沉香属叶绿体基因组的结果,Wang等[51]利用82个蛋白编码基因数据将土沉香认定为锦葵目(Malvales)中膝柱木属拉敏木(Gonystylus bancanus)的近缘种。目前,NCBI GenBank上共有16种沉香属物种的叶绿体全基因序列,包括土沉香[52-53]、印度沉香(A. agallocha Roxb.,异名A. malaccensis)[54]、大叶沉香[55]、奎明沉香〔A. cumingiana(Decne.)Ridl.〕 [54]、毛叶沉香[55]、小果沉香[55]、锥果沉香[56]、皱纹沉香(A. rugosa K. Le-Cong & Kessler)[54]、泰国沉香[55]、厚叶沉香[55]、马来沉香[56]和云南沉香[57]。基于精确的组装策略,已明确沉香属叶绿体基因组的平均大小约为174 000 bp[58],但Deng等[52]报道的土沉香叶绿体基因组大小差异较大,为159 565 bp,这可能是由于当时缺乏高组装精度的基因组组装工具造成的误差。与Farah等[36]对5个叶绿体基因序列的联合数据集分析结果相比,利用叶绿体全基因组序列重建的系统发育树在物种水平上具有更好的分辨率。植物线粒体基因组与叶绿体基因组相似,均为母系遗传。虽然线粒体是参与呼吸和代谢的重要细胞器,但线粒体基因组的进化速率较慢、基因保守性高,与叶绿体基因组相比,线粒体基因组在系统发育重建中的效果较差[59]。目前仅有土沉香的线粒体全基因组记录[55],与锦葵目下的其他14个姐妹类群相比,线粒体基因组存在341 829 bp的长度差异,在基因组中所预测的32个蛋白编码基因中,只有7个是土沉香与木棉属(Bombax)、黄麻属(Corchorus)、棉属(Gossypium)、木槿属(Hibiscus)和豇豆属(Vigna)这些近缘类群所共有的[60]。
随着测序技术的发展,越来越多研究者开展了食用、药用和观赏性植物全基因组测序工作[61]。印度沉香是瑞香科首次报道基因组草图的物种,为沉香属在核基因组水平上的分子定位提供了开创性的认识[62]。此外还有3个土沉香的全基因组信息被报道,分别为1个基因组草图[63]和2个具有较高完整性的参考基因组[64]。基于全基因组序列的分化时间估算,土沉香与锦葵目下的陆地棉(Gossypium hirsutum)、可可(Theobroma cacao)拥有共同祖先,其分化时间约在8 214万年之前[64]。除全基因组测序外,基于NGS开发的RAD-seq、Ion Torrent PGM等二代测序技术可以使研究人员以较低的成本获取基因组水平的大量数据[65],但目前仅应用于SSR引物的开发。通过基因组水平测序,可以获取大量SNP位点[66],Law等[67]基于WGRS和RAD-seq的SNP数据,对来自中国香港和内地的土沉香分别进行了群体基因组分析,结果表明这些样品至少存在2个遗传分化组。Wang等[65]对分布于广西、海南、广东及香港的土沉香进行研究并最终确定了两个主要集群,表明了我国香港的土沉香居群在遗传上明显与我国内地其他地区的土沉香居群存在差异。
3 沉香属植物分子鉴定沉香目前主要以木材或其他产品的形式进行交易,尽管许多国家都有着严格的跨境贸易监测,但由于沉香木常被制成粉末或木块等各种形态,与其他物种混合运输而导致检测困难,因此非法的国际贸易及跨境走私案件常有发生,同时也存在不法商人混杂具有类似沉香木材解剖特征的伪品进行贸易的现象[68],因此如何有效区分这些物种已成为打击沉香伪品贸易环节中最重要的一环。沉香属物种的鉴定主要依靠花和果实这类宏观形态进行识别,但形态特征的高度相似性和不完整性对于区分真假而言具有较大的挑战。传统的木材解剖技术一般也只能识别到属[69],而其他的识别方法,如生化标志物等,因为涉及到各种复杂的化学物质,其技术的可行性仍需要进一步深入探索。当前,为准确识别沉香产品的来源,研究者们主要通过分子标记的方法对沉香属植物进行区别与鉴定。
3.1 DNA指纹图谱的分子鉴定Eurlings等[70]开发了4个SSR引物,建立了厚叶沉香的DNA指纹(DNA fingerprinting)图谱库,利用等位基因片段长度溯源沉香产品(木片、粉末、油、香及香水等),此方法在区分厚叶沉香和马来沉香上较为有效,因此可以作为鉴别沉香的良好候选标记之一。Deng等[52]使用SSR标记对广东省非法贸易的土沉香进行鉴定,发现电白的非法贸易土沉香居群与东莞的沉香居群聚成一支。此外,SNP位点分析发现非法贸易的土沉香并没有形成单独的聚类居群,其在遗传上与栽培种相同,同时根据SNP以外的基因位点显示,非法贸易的土沉香种群具有较低的种子流,因此,SNP标记被认为更容易受选择压力影响,具有更高的准确性。
3.2 核质基因片段的DNA条形码分子鉴定DNA条形码是利用基因组中一段公认的标准短序列进行物种鉴定的分子诊断技术,目前已广泛应用于生物分类学研究[71]。Eurling等[72]通过9个沉香属和拟沉香属(Gyrinops)质体间隔区的trnL~trnF鉴定了一批沉香样品,发现这批样品中含有拟沉香属的物种。Jiao等[73]使用了trnL~trnF和ITS1对土沉香的物种进行鉴定。Lee等[74]评估了7个沉香属物种的8个候选DNA条形码区域,发现trnL~trnF+ITS或trnL~trnF+ITS2均能分辨多数的沉香属物种,而ITS2的序列相对于ITS,其序列更短,更有利于木材DNA的扩增,因此trnL~trnF+ITS2的组合更适合用于识别沉香制品的物种来源。Pern等[75]利用trnL~trnF+ITS2组合证明了至少10个沉香属物种的分类学地位。尽管近年来已有不少关于沉香属DNA条形码候选位点开发的报道[76-78],但与Lee等[74]的研究相比,这些报道中物种鉴定的分辨率和数量并未增加。Lin等[79]基于机器学习方法开发了沉香属植物DNA条形码,在物种鉴定水平上与传统方法(如距离法和聚类树)相比,其准确性更高。此外,Hishamuddin等[80]从8个沉香属植物的叶绿体基因组高变区中筛选出的psbJ~petA+trnT~trnL组合可有效鉴定所有沉香属物种。而高分辨率熔化(HRM)是一种监测DNA解离动力学的新兴方法,样品间单一碱基的变化也容易被检测和识别,具有灵敏性高和时效性短等优点[81]。Lee等[82]开发了5个150~300 bp的mini-barcode用于HRM分析,但该技术需要依赖于物种已知的DNA序列信息,且在分析前还需要提供参考物种的DNA模板。综上,Bar-HRM在沉香属的物种检测和鉴定上仍具有广阔的应用前景。
4 结语与展望截至目前,尽管国内外学者在沉香属物种的遗传多样性和分子鉴定研究上取得了一定进展,但其研究多限于本土物种,在世界上仍有部分沉香物种未得到充分研究,尤其是分布于菲律宾的沉香物种。因此,针对沉香属植物物种研究仍然存在一些亟待解决的问题:(1)缺乏全面了解沉香属物种生存状况、种质资源及潜在基因库等信息。在学术界和行业内应更为全面地了解属内物种的状况,有针对性地制定保护策略,这是濒危物种在野外保护的一个重要步骤。(2)沉香属内物种间的系统发育关系解析不全面。以往研究多依赖于古老的凭证标本,并利用短基因序列进行系统发育关系解析。但随着第二代和第三代测序技术的发展,如叶绿体基因组、简化基因组、转录组等在解决沉香属物种间的系统关系上显示出一定潜力。利用基因组规模的数据重建其系统发育关系将具有重要意义,可以更准确地解决沉香属内物种的分子位置和系统学关系。由于基因组数据的测序需要高质量的DNA模板以达到最大的基因组覆盖率,需要对沉香属尚未研究的物种进行研究。(3)缺乏快速、准确沉香属物种的分子鉴定方法。尽管现有沉香物种分子鉴定技术具有很大潜力,但其实际应用的效果仍需要现场评估。从不同的沉香属物种的叶绿体基因组中可以识别出高变异位点,与现有的沉香属DNA条形码相比,有望开发出具有更高识别率的DNA条形码,为海关、CITES对非法沉香贸易执法提供更有利的工具。(4)加强沉香分布区研究人员的合作与交流,共同推动沉香属物种的遗传多样性研究,是实现对珍稀濒危资源保护与研究的重要发展方向。随着国际贸易的升温和东南亚国家经济的发展提速,沉香国际市场环境总体向好,人们生活水平和健康观念的提升,也加快促使沉香需求量的增加,在发展沉香种质相关产业的同时要树立“绿水青山就是金山银山”的生态理念,坚持“高质量发展”与“可持续发展”相结合,一方面,在环境可持续发展的前提下适度增加种植规模,推动产业之间协调发展;另一方面,在推动产业规模扩大的同时,注重产品质量的把控和提升。
| [1] |
MOHAMED R, LEE S Y. Keeping up appearances: Agarwood grades and quality//MOHAMED R. Agarwood[M]. Singapore: Springer, Tropical Forestry, 2016.
|
| [2] |
POWO. Plants of the world online[DB/OL]. (2023-02-16). https://powo.science.kew.org/.
|
| [3] |
高志晖, 赵文婷, 孙佩文, 魏建和. 世界各国(地区)沉香资源与保护[J]. 中国现代中药, 2017, 19(8): 1057-1063. DOI:10.13313/j.issn.1673-4890.2017.8.002 GAO Z H, ZHAO W T, SUN P W, WEI J H. Species and conservation status of the endangered agarwood-producing genus Aquilaria[J]. Modern Chinese Medicine, 2017, 19(8): 1057-1063. DOI:10.13313/j.issn.1673-4890.2017.8.002 |
| [4] |
SAM H V, NHA N T, CHU T V, TUAN N T, THO N T, TAM D T, THANH L B, HAI T N, HUAN H V, HANG H T, HIEU D T, CERBONCINI C, LEXANDRA O. Aquilaria yunnanensis SC Huang (Thymelaeaceae), a new record for the flora of Vietnam[J]. Forest and Society, 2019, 3(2): 202-208. DOI:10.24259/fs.v3i2.6696 |
| [5] |
蒋谦才, 谭宗健, 廖浩斌, 刘盼盼, 孙红梅, 王瑞江, 曹洪麟, 黄向阳, 赖思茹, 梁丹, 王铮峰. 广东省中山市五桂山土沉香遗传多样性[J]. 广西植物, 2018, 38(6): 804-811. DOI:10.11931/guihaia.gxzw201709027 JIANG Q C, TAN Z J, LIAO H B, LIU P P, SUN H M, WANG R J, CAO H L, HUANG Q Y, LAI S R, LIANG D, WANG Z F. Genetic diversity of Aquilaria sinensis in Wuguishan, Zhongshan, Guangdong Province[J]. Guihaja, 2018, 38(6): 804-811. DOI:10.11931/guihaia.gxzw201709027 |
| [6] |
IUCN. The IUCN red list of threatened species[EB/OL]. [2024-02-18]. https://www.iucnredlist.org/.
|
| [7] |
BARDEN A, ANAK N A, MULLIKEN T, SONG M. Heart of the matter: Agarwood use and trade and CITES implementation for Aquilaria malaccensis[J]. Cambridge: Traffic International, 2000. |
| [8] |
国家林业和草原局农业农村部. 国家重点保护野生植物名录[EB/OL]. [2023-10-30]. http://www.forestry.gov.cn/c/www/gkml/11057.jhtml. National Forestry and Grassland Administration Ministry of Agriculture and Rural Affairs. List of national key protected wild plants in China[EB/OL]. [2023-10-30]. http://www.forestry.gov.cn/c/www/gkml/11057.jhtml. |
| [9] |
晏飞. 全国沉香标准制定在五桂山开启[N]. 中山日报, 2018-09-27. YAN F. The national aloes standard was established in Wugui Mountain [N]. Chung shan daily news, 2018-09-27. |
| [10] |
张小霞. 土沉香开发利用研究进展[J]. 防护林科技, 2020(4): 63-66. DOI:10.13601/j.issn.1005-5215.2020.04.024 ZHANG X X. Research progress on development and utilization of Aquilaria sinensis agarwood[J]. Protection Forest Science and Technology, 2020(4): 63-66. DOI:10.13601/j.issn.1005-5215.2020.04.024 |
| [11] |
黄文政. 土沉香的栽培技术[J]. 绿色科技, 2017(7): 156-157. DOI:10.16663/j.cnki.lskj.2017.07.058 HUANG W Z. Cultivation techniques of Aquilaria sinensis agarwood[J]. Journal of Green Science and Technology, 2017(7): 156-157. DOI:10.16663/j.cnki.lskj.2017.07.058 |
| [12] |
寿海洋. 土沉香(瑞香科)的分布和生物学特性研究[D]. 北京: 北京林业大学, 2010. SHOU H Y. The distribution and biological characteristics of Aqualaria sinensis (Lour.) Spreng. (Thymelaeaceae)[D]. Beijing: Beijing Forestry University, 2010. |
| [13] |
钟铭隆. 一氧化氮对酸铝胁迫下土沉香幼苗生长生理特性的影响[D]. 南宁: 广西大学, 2016. ZHONG M L. The affection of NO under aluminum stress to Aquilaria sinensis seedings growth and physiology index[D]. Nanning: Guangxi University, 2016. |
| [14] |
田程飘. 广西沉香的化学成分分析及生物活性研究[D]. 南宁: 广西民族大学, 2020. TIAN C P. Analysis of chemical constituents and biological activities of Guangxi agarwood[D]. Nanning: Guangxi University Nationalities, 2020. |
| [15] |
江浩. 茉莉酸甲酯诱导白木香结香技术研究[D]. 海口: 海南大学, 2018. JIANG H. A study on the agarwood-inducing technique of Aquilaria sinensis induced by methyl jasmonate[D]. Haikou: Hainan University, 2018. |
| [16] |
潘质洪. 白木香植物激素诱导结香解剖学及化学成分的研究[D]. 哈尔滨: 东北林业大学, 2019. DOI: 10.27009/d.cnki.gdblu.2019.000016. PAN Z H. Research of anatomy and chemical component of Aquilaria sinensis induced by plant hormones[D]. Harbin: Northeast Forestry University, 2019. DOI: 10.27009/d.cnki.gdblu.2019.000016. |
| [17] |
张争, 杨云, 魏建和, 孟慧, 隋春, 陈怀琼. 白木香结香机制研究进展及其防御反应诱导结香假说[J]. 中草药, 2010, 41(1): 156-159. ZHANG Z, YANG Y, WEI J H, MENG H, SUI C, CHEN H Q. Advances in studies on mechanism of agarwood formation in Aquilaria sinensis and its hypothesis of agarwood formation induced by defense response[J]. Chinese Traditional and Herbal Drug, 2010, 41(1): 156-159. |
| [18] |
刘洋洋. 通体结香技术产沉香的质量特征研究[D]. 北京: 北京协和医学院, 2018. LIU Y Y. Research on quality characteristics of agarwood produced by whole-tree agarwood-inducing technique[D]. Beijing: Peiking Union Medical College, 2018. |
| [19] |
陈凤鸣. 奇楠沉香化学成分及质量评价研究[D]. 北京: 中国中医科学院, 2023. CHEN F M. Study on chemical constituents and quality evaluation of Kynam agarwood[D]. Beijing: China Academy of Chinese Medical Sciences, 2023. |
| [20] |
马家乐. 沉香对胆汁酸诱导胃粘膜损伤的保护作用及机制研究[D]. 北京: 北京中医药大学, 2022. MA J L. Protective effect of agarwood on bile acid-induced gastric mucosal injury and its mechanism[D]. Beijing: Beijing University of Chinese Medicine, 2022. |
| [21] |
KANKAYNAR M, CEYHUN H A, BARAN A, SULUKAN E, YILDIRIM F, BOLAT I, TORAMAN E, NADAROGLU H, ARSLAN M, CEYHUN S B. The anxiolytic and circadian regulatory effect of agarwood water extract and its effects on the next generation; zebrafish modelling[J]. Comparative Biochemistry and Physiology. Toxicology and Pharmacology: CBP, 2023, 269: 109621. DOI:10.1016/j.cbpc.2023.109621 |
| [22] |
董梦妤, 焦立超, 姜笑梅, 张淑娟, 李改云, 殷亚芳. 沉香的资源分布、识别与贸易现状[J]. 木材工业, 2016, 30(4): 20-24. DOI:10.19455/j.mcgy.2016.04.005 DONG M Y, JIAO L C, JIANG X M, ZHANG S J, LI G Y, YIN Y F. Resources, identification and utilization of agarwood[J]. China Wood Industry, 2016, 30(4): 20-24. DOI:10.19455/j.mcgy.2016.04.005 |
| [23] |
LÓPEZ-SAMPSON A, PAGE T. History of use and trade of agarwood[J]. Economic botany, 2018, 72(1): 107-129. DOI:10.1007/s12231-018-9408-4 |
| [24] |
葛淑俊, 孟义江, 李广敏, 马峙英. 我国药用植物遗传多样性研究进展[J]. 中草药, 2006(10): 1584-1589. DOI:10.3321/j.issn:0253-2670.2006.10.051 GE S J, MENG Y J, LI G M, MA Z Y. Research progress on genetic diversity in Chinese medicinal plants[J]. Chinese Traditional and Herbal Drugs, 2006(10): 1584-1589. DOI:10.3321/j.issn:0253-2670.2006.10.051 |
| [25] |
刘涛. 国产沉香属植物遗传结构研究[D]. 南昌: 南昌大学, 2016. LIU T. Study on genetic structure of aquilaria in China[D]. Nanchang: Nanchang University, 2016. |
| [26] |
中国科学院中国植物志编辑委员会. 中国植物志[M]. 北京: 科学出版社, 1977: 386. Editorial Committee of Flora of China, Chinese Academy of Sciences. Flora of China[M]. Beijing: Science Press, 1977: 386. |
| [27] |
KANG Y, LIU P W, LYU F F, ZHANG Y X, YANG Y, WEI J H. Genetic relationship and source species identification of 58 Qi-Nan germplasms of Aquilaria species in China that easily form agarwood[J]. PloS One, 2022, 17(6): e0270167-e0270167. DOI:10.1371/journal.pone.0270167 |
| [28] |
孙启高, 宋书银, 王宇飞, 李承森. 介绍双子叶植物叶结构分类术语[J]. 植物分类学报, 1997, 35(3): 275-288. SUN Q G, SONG S Y, WANG Y F, LI C S. Introduction to terminology of classification of dicotyledonous leaf architecture[J]. Acta Phytotaxonomica Sinica, 1997, 35(3): 275-288. |
| [29] |
赵翾, 赵树进. 白木香群体的表型多样性分析[J]. 华南理工大学学报(自然科学版), 2007, 35(4): 117-122. DOI:10.3321/j.issn:1000-565X.2007.04.025 ZHAO X, ZHAO S J. Phenotypic diversity of different Aquilairia sinensis (Lour.) Spreng populations[J]. Journal of South China University of technology (Natural Science Edition), 2007, 35(4): 117-122. DOI:10.3321/j.issn:1000-565X.2007.04.025 |
| [30] |
林丹. 白木香遗传多样性分析及核心种质的初步构建[D]. 东莞: 广东药科大学, 2018. LIN D. Analysis of genetic diversity and preliminary construction of core collection of Aquilaria sinensis (Lour.) Spreng[D]. Dongguan: Guangdong Pharmaceutical University, 2018. |
| [31] |
周元元, 王军, 杨照剑, 王德立, 徐诗涛. 不同白木香种质叶表型遗传多样性分析[J]. 分子植物育种, 2022, 20(21): 7281-7294. DOI:10.13271/j.mpb.020.007281 ZHOU Y Y, WANG J, YANG Z J, WANG D L, XU S T. Genetic diversity on leaf phenotypes of different Aquilaria sinensis germplasms[J]. Molecular Plant Breeding, 2022, 20(21): 7281-7294. DOI:10.13271/j.mpb.020.007281 |
| [32] |
郭艳丽, 鞠爱霞. DNA分子标记技术在中药鉴定学中的应用[J]. 黑龙江医药, 2012, 25(4): 545-547. DOI:10.14035/j.cnki.hljyy.2012.04.018 GUO L Y, JU A X. Application of DNA molecular marker technology on identification of medicinal plants[J]. Heilongjiang Medicine Journal, 2012, 25(4): 545-547. DOI:10.14035/j.cnki.hljyy.2012.04.018 |
| [33] |
朱伟豪, 杜伟锋, 洪浩, 洪智慧, 葛卫红. 聚合酶链式反应技术在中药鉴定研究中的应用进展[J]. 中华中医药杂志, 2020, 35(10): 5091-5094. ZHU W H, DU W F, HONG H, HONG Z H, GE W H. Progress in the application of PCR technology in the identification traditional Chinese medicine research[J]. China Journal of Traditional Chinese Medicine and Pharmacy, 2020, 35(10): 5091-5094. |
| [34] |
LI X, YANG Y, HENRY R J, ROSSETTO M, WANG Y, CHEN S. Plant DNA barcoding: From gene to genome[J]. Biological Reviews, 2015, 90(1): 157-166. DOI:10.1111/brv.12104 |
| [35] |
LEE S Y, TURJAMAN M, MOHAMED R. Phylogenetic relatedness of several agarwood-producing taxa (Thymelaeaceae) from Indonesia[J]. Tropical Life Sciences Research, 2018, 29(2): 13-28. DOI:10.21315/tlsr2018.29.2.2 |
| [36] |
FARAH A H, LEE S Y, GAO Z H, YAO T L, MADON M, MOFAMED R. Genome size, molecular phylogeny, and evolutionary history of the tribe Aquilarieae (Thymelaeaceae), the natural source of agarwood[J]. Frontiers in Plant Science, 2018, 9: 712. DOI:10.3389/fpls.2018.00712 |
| [37] |
ÁLVAREZ I, WENDEL J F. Ribosomal ITS sequences and plant phylogenetic inference[J]. Molecular Phylogenetics and Evolution, 2003, 29(3): 417-434. DOI:10.1016/S1055-7903(03)00208-2 |
| [38] |
PERN Y C, LEE S Y, NG W L, MOHAMED R. Cross-amplification of microsatellite markers across agarwood-producing species of the Aquilarieae tribe (Thymelaeaceae)[J]. 3 Biotech, 2020, 10(3): 1-9. DOI:10.1007/s13205-020-2072-2 |
| [39] |
申彦晶, 严萍, 赵翾, 庞启华, 赵树进. ISSR和ITS标记在白木香遗传变异研究中的应用[J]. 华南理工大学学报(自然科学版), 2008, 36(12): 128-132. DOI:10.3321/j.issn:1000-565X.2008.12.025 SHEN Y J, YAN P, ZHAO X, PANG Q H, ZHAO S J. Application of ISSR marker and ITS sequence to investigation of genetic variation of Aquilaria sinensis[J]. Journal of South China University of Technology (Natural Science Edition), 2008, 36(12): 128-132. DOI:10.3321/j.issn:1000-565X.2008.12.025 |
| [40] |
LEE S Y, MOHAMED R, FARIDAH-HANUM I, LAMASUDIN D L. Utilization of the internal transcribed spacer (ITS) DNA sequence to trace the geographical sources of Aquilaria malaccensis Lam. populations[J]. Plant Genetic Resources, 2018, 16(2): 103-111. DOI:10.1017/S1479262117000016 |
| [41] |
LEE S Y, MOHAMED R. Rediscovery of Aquilaria rostrata (Thymelaeaceae), a species thought to be extinct, and notes on Aquilaria conservation in Peninsular Malaysia[J]. Blumea: Biodiversity, Evolution and Biogeography of Plants, 2016, 61(1): 13-19. DOI:10.3767/000651916X691196 |
| [42] |
LEE S Y, WEBER J, MOHAMED R. Genetic variation and molecular authentication of selected Aquilaria species from natural populations in Malaysia using RAPD and SCAR markers[J]. Asian Journal of Plant Sciences, 2011, 10(3): 202-211. DOI:10.3923/ajps.2011.202.211 |
| [43] |
QIPTIYAH M, WIDYATMOKO A Y P B C, NURTJAHJANINGSIH I L G, PRIHATINI I. Genetic diversity of Aquilaria microcarpa Baill. in Kalimantan using RAPD Markers[J]. IOP Conference Series: Earth and Environmental, 2021, 914(1): 12039. DOI:10.1088/1755-1315/914/1/012039 |
| [44] |
王玲玲, 陈东亮, 黄丛林, 邢震. SSR分子标记技术在植物研究中的应用[J]. 安徽农业科学, 2017, 45(36): 123-126. DOI:10.13989/j.cnki.0517-6611.2017.36.039 WANG L L, CHEN D L, HUANG C L, XING Z. Application of SSR molecular marker technique in plant research[J]. Journal of Anhui Agricultural Sciences, 2017, 45(36): 123-126. DOI:10.13989/j.cnki.0517-6611.2017.36.039 |
| [45] |
NANDA, P, SINGH, A K, NE GI, M S, TRPATHI S B. Rapid development and characterization of fifteen novel microsatellite loci in vulnerable tree Aquilaria malaccensis using next generation ion torrent PGM™ sequencing[J]. Indian Journal of Genetics and Plant Breeding, 2017, 77(1): 145-152. DOI:10.5958/0975-6906.2017.00019.0 |
| [46] |
WANG Z F, CAO H L, CAI C X, GUO Y, WANG Z M. Microsatellites development and cross-amplification for Aquilaria sinensis, an endangered agarwood-producing tree[J]. Journal of Genetics, 2018, 97(1): 139-45. DOI:10.1007/s12041-018-1010-0 |
| [47] |
LEE S L, ZAKARIA N F, TNAH L H, NG C H, NG K K S, LEE C T, LAU K H, CHUA L S L. DNA databases of a CITES listed species Aquilaria malaccensis (Thymelaeaceae) as the tracking tools for forensic identification and chain of custody certification[J]. Forensic Science International: Genetics, 2022, 57: 102658. DOI:10.1016/j.fsigen.2021.102658 |
| [48] |
ZOU M L, XIA Z Q, LU C, WANG H, JI J, WANG W. Genetic diversity and differentiation of Aquilaria sinensis (Lour.) Gilg revealed by ISSR and SRAP markers[J]. Crop Science, 2012, 52(5): 2304-2313. DOI:10.2135/cropsci2011.10.0576 |
| [49] |
BANU S, BARUAH D, BHAGWAT R M, SARKAR P, BHOWMICK A, KADOOL N Y. Analysis of genetic variability in Aquilaria malaccensis from Bramhaputra valley, Assam, India using ISSR markers[J]. Flora, 2015, 217: 24-32. DOI:10.1016/j.flora.2015.09.007 |
| [50] |
LEE S Y, NG W L, LAMASUDIN, D U, MOHAMED R. Inter-simple sequence repeat markers reveal genetic relatedness between natural Aquilaria populations in Peninsular Malaysia[J]. Chiang Mai Journal Science, 2018, 45(3): 1307-1317. |
| [51] |
WANG Y, ZHAN D F, JIA X, MEI W L, DAI H F, CHEN X T, PENG S Q. Complete chloroplast genome sequence of Aquilaria sinensis (Lour.) Gilg and evolution analysis within the Malvales order[J]. Frontiers in Plant Science, 2016, 7: 280. DOI:10.3389/fpls.2016.00280 |
| [52] |
DENG X, JIANG Z X, JIANG Q B, GUO W, LI Y Q, ZHANG X Z. Characterization of the complete chloroplast genome of Aquilaria sinensis, an endangered agarwood-producing tree[J]. Mitochondrial DNA Part B, 2020, 5(1): 422-423. DOI:10.1080/23802359.2019.1703593 |
| [53] |
LEE S Y, TURJAMAN M, CHAVEERACH A, SUBASINGHE S, FAN Q, LIAO W. Phylogenetic relationships of Aquilaria and Gyrinops (Thymelaeaceae) revisited: Evidence from complete plastid genomes[J]. Botanical Journal of the Linnean Society, 2022, 200(3): 344-359. DOI:10.1093/botlinnean/boac014 |
| [54] |
HISHAMUDDIN M S, LEE S Y, NG W L, RANLEE S I, LAMASUDIN D U, MOHAMED R. Comparison of eight complete chloroplast genomes of the endangered Aquilaria tree species (Thymelaeaceae) and their phylogenetic relationships[J]. Scientific Reports, 2020, 10(1): 13034. DOI:10.1038/s41598-020-70030-0 |
| [55] |
LEE S Y, NG W L, MOHAMED R, TERHEM R. The complete chloroplast genome of Aquilaria malaccensis Lam. (Thymelaeaceae), an important and threatened agarwood-producing tree species[J]. Mitochondrial DNA Part B, 2018, 3(2): 1120-1121. DOI:10.1080/23802359.2018.1519382 |
| [56] |
ZHANG Y H, HUANG Y, LI Z M, ZHANG S D. Characterization of the complete chloroplast genome of the vulnerable agarwood tree, Aquilaria yunnanensis (Thymelaeaceae)[J]. Conservation Genetics Resources, 2019, 11(2): 161-164. DOI:10.1007/s12686-018-0989-0 |
| [57] |
FREUDENTHAL J A, PFAFF S, TERHOEVEN N, KORTE A, ANKENBRAND M J, FÖRSTER F. A systematic comparison of chloroplast genome assembly tools[J]. Genome Biology, 2020, 21: 254. DOI:10.1186/s13059-020-02153-6 |
| [58] |
NG W L, TAN S G. Inter-simple sequence repeat (ISSR) markers: Are we doing it right[J]. ASM Science Journal, 2015, 9(1): 30-39. |
| [59] |
ANDREWS K R, LUIKART G. Recent novel approaches for population genomics data analysis[J]. Molecular Ecology, 2014, 23: 1661-1667. DOI:10.1111/mec.12686 |
| [60] |
MACKENZIE S, MCINTOSH L. Higher plant mitochondria[J]. The Plant Cell, 1999, 11(4): 571-585. DOI:10.1105/tpc.11.4.571 |
| [61] |
SHAHRAJABIAN M H, SUN W, CHENG Q. A review on plant genomes of some important traditional chinese fruits and herbs[J]. Cercetări Agronomice în Moldova, 2020, 53(2): 217-232. DOI:10.46909/cerce-2020-019 |
| [62] |
CHEN C H, KUO T C Y, YANG M H, CHIEN T Y, CHU M J, HUANG L C, CHEN C Y, LO H S, JENG S T, CHEN L F O. Identification of cucurbitacins and assembly of a draft genome for Aquilaria agallocha[J]. BMC Genomics, 2014, 15: 578. DOI:10.1186/1471-2164-15-578 |
| [63] |
DING X P, MEI W L, LIN Q, WANG H, WANG J, PENG S Q, LI H L, ZHU J H, LI W, WANG P, CHEN H Q, DONG W H, GUO D, CAI C H, HUANG S Z, CUI P, DAI H F. Genome sequence of the agarwood tree Aquilaria sinensis (Lour.) Spreng: The first chromosome-level draft genome in the Thymelaeceae family[J]. GigaScience, 2020, 9(3): giaa013. DOI:10.1093/gigascience/giaa013 |
| [64] |
NONG W Y, LAW S T S, WONG A Y P, BARIL T, SWALE T, CHU L M, Hayward A, LAU D T W, HUI J H L. Chromosomal‐level reference genome of the incense tree Aquilaria sinensis[J]. Molecular Ecology Resources, 2020, 20(4): 971-979. DOI:10.1111/1755-0998.13154 |
| [65] |
WANG Z F, CAO H L, CAI C X, WANG Z M. Using genetic markers to identify the origin of illegally traded agarwood-producing Aquilaria sinensis trees[J]. Global Ecology Conservation, 2020, 22: e00958. DOI:10.1016/j.gecco.2020.e00958 |
| [66] |
PARIHAR P. Bioinformatics in plant genomics for next-generation plant breeding//AHLAWAT J. Plant Genomics for Sustainable Agriculture[M]. Singapore: Springer Nature Singapore, 2022: 165-190.
|
| [67] |
LAW S T S, NONG W Y, YIP H Y, LIU E K Y, NG T P T, TSANG R H L, XIA N H, SHAW P C, LAM H M, LAU D T W, HUI J H L. Population genomic analyses of protected incense trees Aquilaria sinensis reveals the existence of genetically distinct subpopulations[J]. Frontiers in Ecology and Evolution, 2023, 11: 1216370. DOI:10.3389/fevo.2023.1216370 |
| [68] |
YIN Y F, JIAO L C, DONG M Y. Wood resources, identification, and utilization of agarwood in China//MOHAMED R. Agarwood[M]. Singapore: Springer, Tropical Forestry, 2016: 21-38.
|
| [69] |
GASSON P. How precise can wood identification be? Wood anatomy's role in support of the legal timber trade, especially CITES[J]. IAWA Journal, 2011, 32(2): 137-154. DOI:10.1163/22941932-90000049 |
| [70] |
EURLINGS M C M, VAN BEEK H H, GRAVENDEEL B. Polymorphic microsatellites for forensic identification of agarwood (Aquilaria crassna)[J]. Forensic Science International, 2010, 197(1/3): 30-34. DOI:10.1016/j.forsciint.2009.12.017 |
| [71] |
陈士林, 宋经元, 姚辉, 石林春, 罗焜, 韩建萍. 药用植物DNA条形码鉴定策略及关键技术分析[J]. 中国天然药物, 2009, 7(5): 322-327. DOI:10.3724/SP.J.1009.2009.00322 CHEN S L, SONG J Y, YAO H, SHI L C, LOU K, HAN J P. Strategy and key technique of identification of Chinese herbal medicine using DNA barcoding[J]. Chinese Journal of Natural Medicines, 2009, 7(5): 322-327. DOI:10.3724/SP.J.1009.2009.00322 |
| [72] |
EURLINGS M C M, GRAVENDEEL B. TrnL-trnF sequence data imply paraphyly of Aquilaria and Gyrinops (Thymelaeaceae) and provide new perspectives for agarwood identification[J]. Plant Systematics and Evolution, 2005, 254(1): 1-12. DOI:10.1007/s00606-005-0312-x |
| [73] |
JIAO L C, YIN Y F, CHENG Y M, JIANG X M. DNA barcoding for identification of the endangered species Aquilaria sinensis: Comparison of data from heated or aged wood samples[J]. Holzforschung, 2014, 68(4): 487-494. DOI:10.1515/hf-2013-0129 |
| [74] |
LEE S Y, NG W L, MAHAT M N, NAZRE M, MOHAMED R. DNA barcoding of the endangered Aquilaria (Thymelaeaceae) and its application in species authentication of agarwood products traded in the market[J]. PloS One, 2016, 11(4): e0154631. DOI:10.1371/journal.pone.0154631 |
| [75] |
PERN Y C, LEE S Y, KAMARUDIN N, MOHAMED R. Genetic variation and DNA barcoding of the endangered agarwood-producing Aquilaria beccariana (Thymelaeaceae) populations from the Malesia region[J]. Forestist, 2020, 70(2): 85-94. DOI:10.5152/forestist.2020.20009 |
| [76] |
FENG T T, LI Q W, WANG Y S, QIU S M, HE M L, ZHANG W M, DONG J, ZHANG S. Phylogenetic analysis of Aquilaria Lam. (Thymelaeaceae) based on DNA barcoding[J]. Holzforschung, 2019, 73(6): 517-523. DOI:10.1515/hf-2018-0127 |
| [77] |
TANAKA S, ITO M. DNA barcoding for identification of agarwood source species using trnL-trnF and matK DNA sequences[J]. Journal of Natural Medicines, 2020, 74(1): 42-50. DOI:10.1007/s11418-019-01338-z |
| [78] |
KANG Y. Molecular identification of Aquilaria species with distribution records in China using DNA barcode technology[J]. Mitochondrial DNA Part B, 2021, 6(4): 1525-1535. DOI:10.1080/23802359.2021.1914210 |
| [79] |
LIN Y, FENG T, DAI J, LIU Q, CAI J, KUANG J, WANG Z. DNA barcoding identification of IUCN Red listed threatened species in the genus Aquilaria (Thymelaeaceae) using machine learning approaches[J]. Phytochemistry Letters, 2023, 55: 105-111. DOI:10.1016/j.phytol.2023.04.007 |
| [80] |
HISHAMUDDIN M S, LEE S Y, SYAZWAN S A, RAMLEE S I, LAMASUDIN D U, MOHAMED R. Highly divergent regions in the complete plastome sequences of Aquilaria are suitable for DNA barcoding applications including identifying species origin of agarwood products[J]. 3 Biotech, 2023, 13(3): 78. DOI:10.1007/s13205-023-03479-1 |
| [81] |
SUESATPANIT T, OSATHANUNKUL K, MADESIS P, OSATHANUNKUL M. Should DNA sequence be incorporated with other taxonomical data for routine identifying of plant species?[J]. BMC Complementary and Alternative Medicine, 2017, 17(1): 1-10. DOI:10.1186/s12906-017-1937-3 |
| [82] |
LEE S Y, LAMASUDIN D U, MOHAMED R. Rapid detection of several endangered agarwood-producing Aquilaria species and their potential adulterants using plant DNA barcodes coupled with highresolution melting (Bar-HRM) analysis[J]. Holzforschung, 2019, 73(5): 435-444. DOI:10.1515/hf-2018-0149 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51