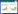文章信息
基金项目
- 国家自然科学基金(U22A20464);广东省重点领域研发计划项目(2022B0202060002);广东省乡村振兴战略专项资金种业振兴项目(2022-NPY-00-013);广东省农业科学院“十四五”新兴学科团队项目(202111TD)
作者简介
- 陈静(1997—),女,在读硕士生,研究方向为水稻育种,E-mail:chenjing19970906@163.com.
通讯作者
- 王重荣(1980—),男,博士,研究员,研究方向为水稻分子育种,E-mail:wangcr1980@163.com.
文章历史
- 收稿日期:2024-02-10
2. 广东省农业科学院水稻研究所/广东省水稻育种新技术重点实验室/广东省水稻工程实验室,广东 广州 510640
2. Rice Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory of New Technology in Rice Breeding/Guangdong Rice Engineering Laboratory, Guangzhou 510640, China
【研究意义】水稻(Oryza sativa L.)是世界上最重要的粮食作物之一,50% 以上的人口以稻米为主食。由稻瘟病菌(Magnapothe oryzae)引起的稻瘟病是对水稻生产为害最严重的病害之一,每年可造成水稻大量减产,严重时减产可高达50%[1-3]。由于稻瘟病病菌生理小种致病性变异频繁,导致水稻单一抗性品种的抗性会在该品种大面积种植3~5年后逐渐丧失,培育持久抗病品种必须聚合多个广谱抗病基因才能达到育种目的[4-5]。从环境保护与农业可持续发展的角度考虑,选育与种植抗稻瘟病品种是防治稻瘟病最安全有效的方法[6]。开发抗稻瘟病基因Pi2和Pita的功能基因标记,有利于建立高效、简便、环保的检测体系,对促进抗性基因在商业化育种中的应用以及促进广东省水稻品种资源利用具有重要意义[7-8]。【前人研究进展】单核苷酸多态性(Single nucleotide polymorphism,SNP)主要是由单个核苷酸变异引起的DNA序列多态性,包括单个碱基的转换、颠换等。SNP占所有已知多态性的90% 以上,由于SNP分布广泛,它也被应用在分子育种大规模快速筛查中[9-10]。使用SNP检测最常用的方法有Taqman探针法、直接测序法、ARMS-PCR法以及竞争性等位基因PCR(Kompetitive Allele Specific PCR,KASP)[11-14]。其中,KASP是基于引物末端碱基的特异性匹配对SNP进行检测的方法,该方法在引物和探针设计上与常规荧光PCR不同,首先需针对SNP的等位基因设计两条对应的上游引物以及一条通用下游引物,并且设计与特异性上游引物接头序列相同的荧光探针及互补的淬灭探针,通过计算PCR过程中产生的荧光信号,实现对变异位点的检测。【本研究切入点】传统依靠表型鉴定的抗性品种选育方法往往因为环境和人为因素的影响对表型把握不准,需要加大杂交群体和增加表型鉴定试验批次、数量,这极大地增加了育种工作量与成本。而利用已克隆稻瘟病抗性基因的连锁标记或功能基因标记,从遗传基础上跟踪目标性状,选择含有目标抗性基因的单株进行杂交(回交),不但能准确地进行目标性状方向的育种,而且能减小杂交(回交)群体规模、节省成本[15]。Pi2和Pita是对稻瘟病生理小种具有广谱抗性的基因,对水稻的稻瘟病抗性育种具有非常重要的应用价值[16-17]。在第三代分子标记技术出现之前,许多技术需要依靠凝胶电泳和测序等,导致操作繁杂、效率低下,且这些标记检测过程中使用的化学试剂(如EB、聚丙烯酰胺、甲醛等)易对环境造成污染,对人体产生危害[18]。KASP以常规PCR和荧光检测为基础,能够在普通实验室操作的基础上满足低、中、高通量基因分型的要求,检测过程无需电泳,减少了实验操作过程对环境的污染和人体的伤害,并且该技术采用的是通用探针,可以与各种不同的基因特异引物配合使用,而不需要针对每个特定的位点进行探针合成,极大降低了实验成本[19-21]。【拟解决的关键问题】本研究开发了两个抗稻瘟病基因Pi2和Pita的特异性KASP标记,并且对培育的41份水稻新品种材料进行标记检测,筛选出11个抗稻瘟病水稻品种。利用本研究开发的KASP功能分子标记,可在早期(种子或苗期)检测育种材料的稻瘟病抗性基因Pi2和Pita的等位基因型,预测育种材料的稻瘟病抗性,对育种材料进行准确筛选,无需将育种材料种植到病圃鉴定,从而促进抗稻瘟病水稻品种的遗传改良、提高品种培育效率。
1 材料与方法 1.1 试验材料标记开发所用标准对照基因型材料‘黄广油占’是目前生产上应用的高抗稻瘟病栽培品种,‘广陆矮4号’是20世纪80年代培育的高产品种,目前对华南稻区的稻瘟病生理小种已不具备抗性;Tetep为来自越南的高抗稻瘟病地方种质资源,丽江新团黑谷是易感稻瘟病的地方种质资源,这两份种质资源已在国家水稻数据中心公开。
标记测试材料为广东省农业科学水稻研究所2022年晚造品种比较试验的新品种,包括14个常规稻品种、8个香稻品种和19个杂交水稻组合。
14个常规稻品种:新2221、新2222、新2223、新2224、粤抗2221、粤抗2222、粤抗2223、粤抗2224、广超2221、广超2223、广超2224、功2221、功2222、源2221。
8个香稻品种:华香丝苗、黄两优1223、广超22香、昇香两优喷香、昇香两优钰香、香禾优3083、康香占、富香丝苗。
19个杂交水稻:扬泰优5750、金颖优327、南泰优5308、台两优粤农丝苗、粤秀优粤福占、75S/2944,香禾优5662、香禾优9号、五两优4978、贵优2958、贵优817、贵优2874、南泰优1697、昇香两优517、沃两优粤芽丝苗、欣田优5619、吉优336、广10优336、垣优2677。
田间试验采用随机排列组合设计,3次重复,每个重复种植3行、每行7株,插植规格为20 cm×17 cm,自然诱发病圃设在广州市从化区吕田镇水稻实验田,全生育期每667 m2施用纯氮肥9.15 kg,N∶P2O5∶K2O=1∶0.5∶0.7,试验期间防虫不防病。应用W-Pi2、W-Pita两个标记对41个品比待测水稻品种进行KASP检测,稻瘟病由广东省农业科学院植物保护研究所鉴定,对供试材料的叶瘟、穗瘟综合分析进行稻瘟病抗性鉴定,按国际水稻研究所(IRRI)0~9级的分级标准进行记录:0级无病、1级抗、3级中抗、5级中感、7级感、9级高感。
1.2 KASP分子标记开发本团队前期研究发现,Pi2基因的第787、788位密码子上存在核苷酸变异GCA GGA/GTG TTA,高抗稻瘟病品种‘黄广油占’与感稻瘟病品种‘广陆矮4号’在该位点存在多态性差异,其中‘黄广油占’为GCA GGA基因型,‘广陆矮4号’为GTG TTA基因型[22],提取该位点左右两侧各100 bp的序列,开发标记W-Pi2,所述标记序列如表 1中SEQ ID No.1、SEQ ID No.2、SEQ ID No.3所示。
Wang等[23]研究表明,在Pita基因的第二外显子6 640位碱基上存在核苷酸变异G/T,导致Pita基因蛋白LRD区域的单个氨基酸变换,导致水稻稻瘟病抗性产生变化:如果第918氨基酸是丙氨酸(A),则表现为抗病;如果第918氨基酸是丝氨酸(S),则表现为感病。
根据文献[24-25]报道,高抗稻瘟病种质资源Tetep与高感稻瘟病种质资源丽江新团黑谷在Pita基因位点存在多态性差异,其中Tetep为G基因型、丽江新团黑谷为T基因型。本研究从Ricevarmap网站(http://ricevarmap.ncpgr.cn/v2/)上检索到该突变位点vg1210607554,并下载该突变位点上下游各100 bp的序列,设计KASP标记引物,开发成Pita基因的KASP功能标记W-Pita,所述标记序列如表 1中SEQ ID No.1、SEQ ID No. 2、SEQ ID No. 3所示。
1.3 分子标记基因分型应用1.3.1 分子标记基因分型步骤 以植物基因组DNA为模板,采用所述分子标记的引物进行PCR,待反应完成后,借助荧光信号采集仪Bio-Rad CFX Connect获取对应PCR扩增产物荧光信号值,最终完成基因分型。PCR反应体系5 µL,包括:20~30 ng/µL DNA 2 µL,分子标记引物0.14 µL(由2条正向引物、1条反向通用引物按照1∶1∶2混合),2×KASP Master mixture 2 µL,最后补充ddH2O 0.86 µL。反应条件为:94 ℃ 15 min;94 ℃ 20 s,63~55 ℃ 1 min,每个循环降低0.8 ℃,共10个循环;94℃ 20 s,55 ℃ 1 min,共26个循环。荧光信号值的读取条件为16 ℃、20 s。提取感病基因、抗病基因、杂合DNA样本进行KASP检测,以ddH 2O作阴性对照(CK),各3次重复。
1.3.2 荧光信号结果判断方法 (1)Pi2等位基因型判断方法:若只检测到FAM荧光信号,则待测水稻品种为Pi2抗性等位基因型CAG;若只检测到HEX荧光信号,则待测水稻品种为Pi2感病等位基因型TGT;若同时检测到两种荧光信号,则待测水稻品种为Pi2杂合基因型。
(2)Pita等位基因型判断方法:若只检测到FAM荧光信号,则待测水稻品种为Pita抗性等位基因型G;若只检测到HEX荧光信号,则待测水稻品种为Pita感病等位基因型T;若同时检测到两种荧光信号,则待测水稻品种为Pita杂合基因型。
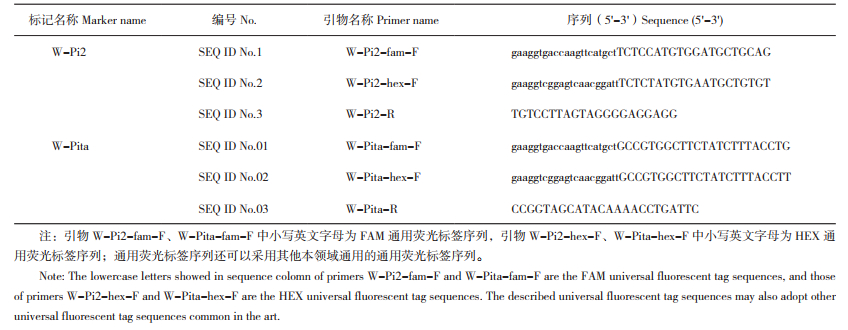
2 结果与分析 2.1 抗稻瘟病标记开发应用W-Pi2进行检测需提取‘黄广油占’和‘广陆矮4号’的DNA,分别作为抗性等位基因型和感病等位基因型的标准DNA样本,取两个品种的等量DNA混合作为杂合基因型的标准DNA样本;W-Pita则提取Tetep和丽江新团黑谷的DNA,分别作为抗性等位基因型和感病等位基因型的标准DNA样本,取两个品种的等量DNA混合作为杂合基因型的标准DNA样本。检测结果(图 1)表明,这两个标记可以准确区分抗性等位基因型、感病等位基因型和杂合基因型。
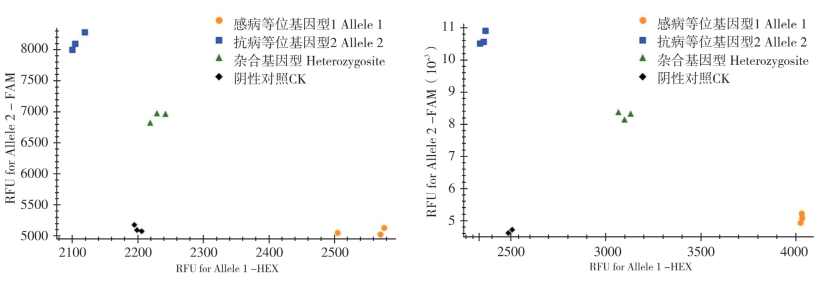
|
| 图 1 W-Pi2、W-Pita标记分型结果 Fig. 1 Genotyping results of W-Pi2 and W-Pita |
2.2 稻瘟病抗性鉴定
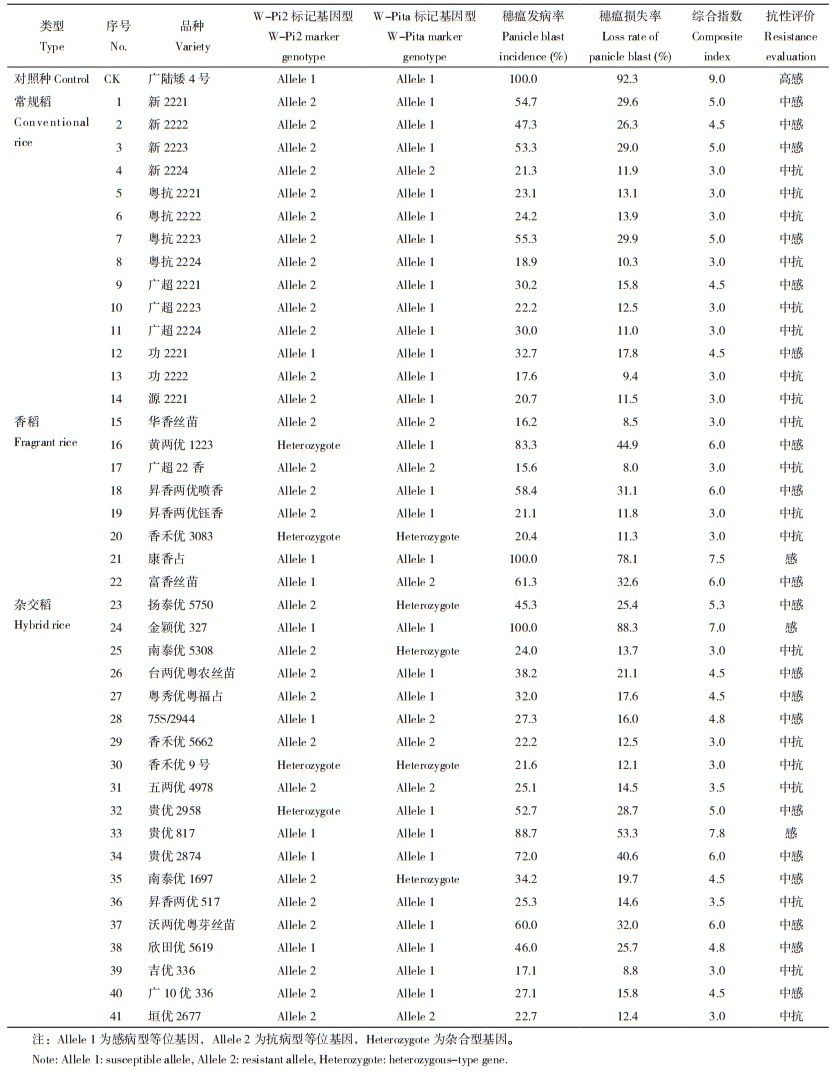
应用W-Pi2、W-Pita标记对41个水稻品种进行KASP检测的结果如图 2所示,两种标记能够准确地将感病基因、抗病基因和杂合基因分离开来。水稻品种抗性评价结果(表 2)显示,41个品种中,感病品种3个、中感品种19个、中抗品种19个。
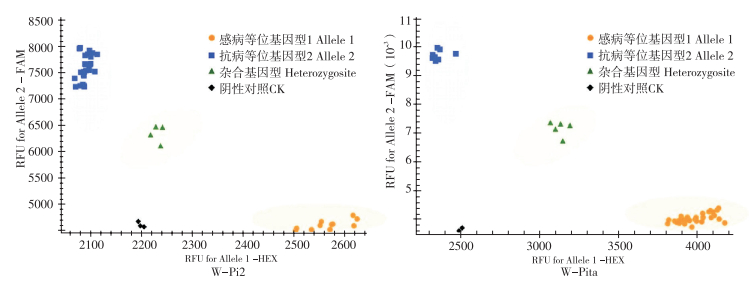
|
| 图 2 41个水稻品种W-Pi2、W-Pita标记分型结果 Fig. 2 W-Pi2 and W-Pita marker typing results of 41 rice varieties |
|
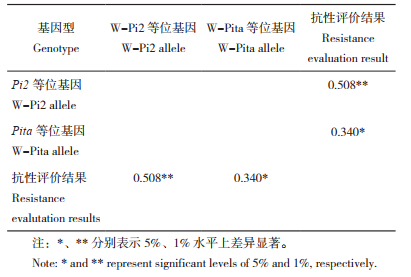
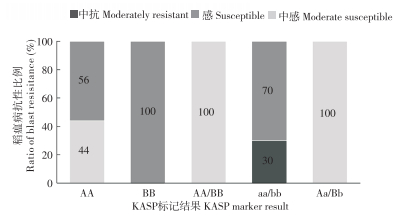
为探明水稻品种携带稻瘟病抗性基因类型或抗性等位基因数量与植株抗病性之间的相关性,本研究统计了41个品种的W-Pi2、W-Pita标记基因型结果与抗性评价结果的相关性和不同等位基因类型的抗病品种频率。相关性分析结果(表 3)显示,W-Pi2与抗性表型呈极显著正相关、相关系数0.508(P<0.01),W-Pita与抗性表型呈显著正相关、相关系数0.340(P<0.05)。由图 3可知,只携带Pi2抗病等位基因的水稻品种有25个,抗性评价结果为中抗的有11个、占44%,中感有14个、占56%;只携带Pita抗病等位基因的品种有2个,且抗性评价结果均为中感,感病品种比例100%;2个基因均为抗病等位基因型的品种有6个,2个抗性基因均为杂合基因型的品种有2个,且二者的抗性评价均为中抗,抗病比例100%;分子标记检测2个基因均为感病等位基因型的品种有6个,这6个品种在抗性评价中均表现为中感和感病,感病品种比例100%。综上,不同稻瘟病抗性基因对提升水稻品种稻瘟病抗性频率的贡献不同,携带Pi2抗性基因的品种的频率高于携带Pita抗性基因的品种。此外,含有2个抗性基因的水稻品种不论基因型为纯合还是杂合,其抗病品种频率均显著高于只有单一抗性基因的品种,抗病等位基因的聚合有利于提高稻瘟病抗性。
|
|
| AA:Pi2抗病,BB:Pita抗病,AA/BB:Pi2与Pita均抗病,aa/bb:Pi2与Pita均感病,Aa/Bb:Pi2与Pita均为杂合 AA: Pi2 disease resistance; BB: Pita disease resistance; AA/BB: Pi2 and Pita are both disease resistant; aa/bb: Pi2 and Pita are both susceptible; Aa/Bb: Pi2 and Pita are both heterozygous 图 3 41个水稻品种不同抗性等位基因的分布与稻瘟病抗性比例 Fig. 3 Distribution and ratio of blast resisitance of alleles with different resistance in 41 varieties |
3 讨论 3.1 抗稻瘟病基因标记的开发与应用
自1992年起,研究人员陆续从各种植物中克隆出多个抗性基因。在水稻基因组中,通过图位克隆、等位基因挖掘、转座子标签技术等方式挖掘出多个抗稻瘟病基因,目前已经鉴定了102个稻瘟病抗性基因和500多个抗稻瘟病QTL,其中39个稻瘟病抗性基因被成功克隆[3, 26-28]。
开发抗稻瘟病分子标记辅助选择,可提高育种效率。杨好等[29]通过分析Pik位点11个等位基因间编码区核苷酸的多态位点,利用155个水稻资源基因组分析其特异性,筛选出Pikp-1、Pikh-1、Pik-1、Pikm-1和Pi1-1并开发成分子标记,鉴定出多个含有Pik-1、Pikh-1、Pikp-1、Pi1-1和Pikm-1基因的材料。赵传超等[30]以593份水稻自然种质资源作为材料,通过对该群体的基因组测序信息筛选SNP位点,利用H-74和H-78开发KASP标记,并且利用病圃自然诱发进行验证,证实标记具有可靠性。何娜等[31]对piz位点1个主效应抗稻瘟病基因Piz-t的等位基因Pi2、Pi9、Pigm、Pi50的序列差异,开发了一套基于PARMS检测技术的荧光分子标记,并利用该标记筛选到抗稻瘟病的水稻品种,同时对其后代进行检测验证,结果表明该标记具有可靠性。
3.2 稻瘟病抗性基因Pi2、Pita特异KASP标记的应用广东高温高湿的气候条件特别容易引起稻瘟病的大面积发生,培育抗病新品种是最经济、安全和有效的方法。陆展华等[32]、汪文娟等[33]研究表明,已克隆的主效稻瘟病抗性基因中,Pi1、Pi2、Pib、Pita是广泛存在于华南籼稻品种中的有效抗性基因。了解水稻品种抗病基因的构成是利用抗病品种控制稻瘟病的基础。应用本研究开发的Pi2、Pita特异KASP标记,能有效筛选含有不同抗性基因的品种,有目的地进行基因聚合和利用。本研究对水稻新品种的检测结果表明,聚合了Pi2、Pita两个抗性基因的品种在自然发病条件下具有较强抗性,即对当前广东优势稻瘟病菌群具有较强抗性。结合前人已发表的其他抗性基因标记结果表明,聚合多个抗性基因将能培育出具有持久抗性的抗病品种,为发展环境友好的水稻产业提供有力支撑。
3.3 KASP技术的应用前景分子标记是以个体间遗传物质内核苷酸序列变异为基础的一种遗传标记,是DNA水平遗传多态性的直接反应。分子标记主要分为3类:一是以分子标记杂交为基础的DNA标记技术;二是以聚合酶链式(PCR反应)为基础的DNA指纹技术;三是以DNA序列为核心的分子标记。与其他两种分子标记相比,第三类分子标记的表现更为优秀,其中最常用的是SNP检测方法。SNP检测方法包括TaqMan探针法、直接测序法和ARMS-PCR法,而KASP技术也属于SNP分子检测技术,是TaqMan的替代品,在原理上与TaqMan类似,也是基于终端荧光读取判断,不同之处在于KASP技术采用的是通用探针,可与各种基因特异引物配合使用,而无需针对每个特定位点进行探针合成,可降低约80% 的实验试剂成本。此外,KASP技术不依赖于凝胶电泳和专门设备,可使用常规qPCR仪器进行SNP基因分型,具有良好的兼容性,且操作更简便、实验材料更环保。目前在农业育种中,KASP技术在种质资源鉴定、亲缘关系研究、分子标记辅助育种、遗传图谱构建与基因定位、种子纯度鉴定等方面应用越来越广泛。例如,梁雪雨等[34]利用KASP技术对南方稻区的香稻品种进行香味基因Badh2的鉴定,结果表明KASP分子标记可准确地对香味基因Badh2进行基因分型,并对香稻种质资源进行鉴定。
4 结论本研究开发了两个稻瘟病抗性基因的功能位点KASP标记W-Pi2、W-Pita,可对抗稻瘟病基因Pi2、Pita进行准确分型。利用这两个标记对41份新培育的水稻材料进行双标记鉴定,筛选出抗性品种11个。双标记可在早期(种子或苗期)检测育种材料的稻瘟病抗性基因Pi2和Pita的等位基因型,预测育种材料的稻瘟病抗性,无需将育种材料种植到病圃鉴定,即可对育种材料进行准确筛选,从而促进抗稻瘟病水稻品种的遗传改良、提高品种培育效率。
| [1] |
RALPH D, JAN A L V K, ZACHARIAS A P, KIN E H K, ANTONIO D P, PIETRO D S, JASON J R, MARTY D, REGINE K, JEFF E, GARY D F. The top 10 fungal pathogens in molecular plant pathology[J]. Molecular Plant Pathology, 2012, 13(4): 414-430. DOI:10.1111/j.1364-3703.2012.00822.x |
| [2] |
ELIZAB P. Armed and dangerous[J]. Science, 2010, 327(5967): 804-805. DOI:10.2307/40509889 |
| [3] |
杨婕, 杨长登, 曾宇翔, 侯雨萱, 陈天晓, 梁燕. 水稻稻瘟病抗性基因挖掘与利用研究进展[J]. 中国水稻科学, 2024, 1-17. DOI:10.2307/40509889 YANG J, YANG C D, ZENG Y X, HOU Y X, CHEN T X, LIANG Y. Research progress on mining and utilization of rice blast resistance genes[J]. Chinese Journal of Rice Science, 2024, 1-17. DOI:10.2307/40509889 |
| [4] |
黄衍焱, 李燕, 王贺, 王文明. 水稻小种特异性抗稻瘟病基因的等位性变异研究进展[J]. 植物病理学报, 2023, 53(5): 753-792. DOI:10.13926/j.cnki.apps.000866 HUANG Y Y, LI Y, WANG H, WANG W M. Allelic variation in the race-specific blast resistance genes in rice[J]. Acta Phytopathologica Sinica, 2023, 53(5): 753-792. DOI:10.13926/j.cnki.apps.000866 |
| [5] |
曹妮, 陈渊, 季芝娟, 曾宇翔, 杨长登. 水稻抗稻瘟病分子机制研究进展[J]. 中国水稻科学, 2019, 33(6): 489-498. DOI:10.16819/j.1001-7216.2019.8126 CAO N, CHEN Y, JI Z J, ZENG Y X, YANG C D. Recent progress in molecular mechanism of rice blast resistance[J]. Chinese Journal of Rice Science, 2019, 33(6): 489-498. DOI:10.16819/j.1001-7216.2019.8126 |
| [6] |
郝鲲, 马建, 程治军, 王帅, 赵沙沙. 水稻抗稻瘟病基因资源与分子育种策略[J]. 植物遗传资源学报, 2013, 14(3): 479-485. DOI:10.13430/j.cnki.jpgr.2013.03.013 HAO K, MA J, CHENG Z J, WANG S, ZHAO S S. Potential gene resources and molecular strategies for improving rice blast resistance[J]. Journal of Plant Genetic Resources, 2013, 14(3): 479-485. DOI:10.13430/j.cnki.jpgr.2013.03.013 |
| [7] |
党小景, 吴爽, 张爱芳, 张瑛, 江建华. 稻瘟病抗性差异亲本配组对杂交水稻抗性和产量的影响[J]. 分子植物育种, 2024, 1-18. DANG X J, WU S, ZHANG A F, ZHANG Y, JIANG J H. Effects of cross-breeding of different parents with blast resistance on the resistance and yield of F1 hybrid rice[J]. Molecular Plant Breeding, 2024, 1-18. |
| [8] |
WANG F J, CHUN L, LIU P Q, LEI C L, HAO W. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J]. PloS One, 2016, 11(4): e0154027. DOI:10.1371/journal.pone.0154027 |
| [9] |
DOU T Y, WANG C C, MA Y L, CHEN Z Y, ZHANG J. CoreSNP: An efficient pipeline for core marker profile selection from genome-wide SNP datasets in crops[J]. BMC Plant Biology, 2023, 23(1): 580-581. DOI:10.1186/s12870-023-04609-w |
| [10] |
ROLLY K N, SIMON A, YOUNGHO K, MYEONG L S, WON O K. Genome-wide association study (GWAS) with high-throughput SNP chip DNA markers identified novel genetic factors for mesocotyl elongation and seedling emergence in rice (Oryza sativa L.) using multiple GAPIT models[J]. Frontiers in Genetics, 2023, 14: 1282620-1282620. DOI:10.3389/FGENE.2023.1282620 |
| [11] |
QUEDNAU M, SIV A, PETERSSON A C, MOLIN G. Identification of clinically important species of Enterococcus within 1 day with randomly amplified polymorphic DNA (RAPD)[J]. Current Microbiology, 1998, 36(6): 332-336. DOI:10.1007/s002849900318 |
| [12] |
王晓涛, 姚志刚, 瓮巧云. AFLP分子标记技术及其应用[J]. 农技服务, 2010, 27(8): 1099-1100. DOI:10.3969/j.issn.1004-8421.2010.08.083 WANG X T, YAO Z G, WENG Q Y. AFLP molecular labeling technology and its application[J]. Agricultural Technology Service, 2010, 27(8): 1099-1100. DOI:10.3969/j.issn.1004-8421.2010.08.083 |
| [13] |
KIM S, MISRA A. SNP genotyping: Technologies and biomedical applications[J]. Annual Review of Biomedical Engineering, 2007, 289-320. DOI:10.1146/annurev.bioeng.9.060906.152037 |
| [14] |
罗怀容, 施鹏, 张亚平. 单核苷酸多态性的研究技术[J]. 遗传, 2001(5): 471-476. DOI:10.3321/j.issn:0253-9772.2001.05.021 LUO H R, SHI P, ZHANG Y P. Detection for single nucleotide polymorphisms[J]. Hereditas (Beijing), 2001(5): 471-476. DOI:10.3321/j.issn:0253-9772.2001.05.021 |
| [15] |
易怒安, 李魏, 戴良英. 水稻抗稻瘟病基因的克隆及其分子育种研究进展[J]. 分子植物育种, 2015, 13(7): 1653-1659. DOI:10.13271/j.mpb.013.001653 YI N A, LI W, DAI L Y. Advances in the cloning of rice blast resistance gene and its molecular breeding[J]. Molecular Plant Breeding, 2015, 13(7): 1653-1659. DOI:10.13271/j.mpb.013.001653 |
| [16] |
PIECK M L, AMY R, FARMAN M K, PETERSON G, STACK J P, VALENT B, PEDLEY K F. Genomics-based marker discovery and diagnostic assay development for wheat blast[J]. Plant Disease, 2017, 101(1): 103-109. DOI:10.1094/PDIS-04-16-0500-RE |
| [17] |
YAN X, TALBOT N J. Investigating the cell biology of plant infection by the rice blast fungus Magnaporthe oryzae-ScienceDirect[J]. Current Opinion in Microbiology, 2016, 34: 147-153. DOI:10.1016/j.mib.2016.10.001 |
| [18] |
AHMAR S, GILL R A, JUNG K H, FAHEEM A, QASIM M U, MUBEEN M, ZHOU W J. Conventional and molecular techniques from simple breeding to speed breeding in crop plants: Recent advances and future outlook[J]. International Journal of Molecular Science, 2020, 21(7): 2590. DOI:10.3390/ijms21072590 |
| [19] |
赵越, 孙宇峰, 徐磊, 王盼, 边境, 王晓楠. KASP标记技术在作物基因定位中的应用进展[J]. 北方园艺, 2023(19): 122-127. DOI:10.11937/bfyy.20231080 ZHAO Y, SUN Y F, XU L, WANG P, BIAN J, WANG X N. KASP marker technology in crop gene mapping[J]. Northern Horticulture, 2023(19): 122-127. DOI:10.11937/bfyy.20231080 |
| [20] |
杨青青, 唐家琪, 张昌泉, 高继平, 刘巧泉. KASP标记技术在主要农作物中的应用及展望[J]. 生物技术通报, 2022, 38(4): 58-71. DOI:10.13560/j.cnki.biotech.bull.1985.2021-1378 YANG Q Q, TANG J Q, ZHANG C Q, GAO J P, LIU Q Q. Application and prospect of KASP Marker Technology in main crops[J]. Biotechnology Bulletin, 2022, 38(4): 58-71. DOI:10.13560/j.cnki.biotech.bull.1985.2021-1378 |
| [21] |
SEMAGN K, BABU R, HEARNE S, OLSEN M. Single nucleotide polymorphism genotyping using Kompetitive Allele Specific PCR (KASP): Overview of the technology and its application in crop improvement[J]. Molecular Breeding, 2014, 33(1): 1-14. DOI:10.1007/s11032-013-9917-x |
| [22] |
杨义强, 朱林峰, 李晓芳, 付杰, 黄道强. 抗稻瘟病基因Pi2的基因特异性KASP标记开发与应用[J]. 植物遗传资源学报, 2021, 22(5): 1314-1321. DOI:10.13430/j.cnki.jpgr.20210327001 YANG Y Q, ZHU L F, LI X F, FU J, HUANG D Q. Development and application of KASP marker specific for rice blast resistance Pi2 gene[J]. Journal of Plant Genetic Resources, 2021, 22(5): 1314-1321. DOI:10.13430/j.cnki.jpgr.20210327001 |
| [23] |
WANG X, FJELLSTROM R, JIA Y, SCHEFFLER B E, WU D, SHU Q, MCCLUNG A. Characterization of Pi-ta blast resistance gene in an international rice core collection[J]. Plant Breeding, 2010, 129(5): 491-501. DOI:10.1111/j.1439-0523.2009.01706.x |
| [24] |
NING X, YUNYU W, AIHONG L. Strategy for use of rice blast resistance genes in rice molecular breeding[J]. Rice Science, 2020, 27(4): 263-277. DOI:10.1016/j.rsci.2020.05.003 |
| [25] |
ZHOU S X, ZHU Y, WANG L F, ZHENG Y P, CHEN J F. OsamiR1873 fine-tunes rice immunity against Magnaporthe oryzae and yield traits[J]. Journal of Integrative Plant Biology, 2020, 62(8): 1213-1226. DOI:10.1111/jipb.12900 |
| [26] |
COULSON A R, SULSTON J E, BRENNER S, KARN J. Towards a physical map of the genome of the nematode Caenorhabditis elegans[J]. Proceedings of the National Academy of Sciences, 1986, 83(20): 7821-7825. DOI:10.1073/pnas.83.20.7821 |
| [27] |
DENG Y, ZHAI K, XIE Z, YANG D Y, ZHU X D. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance[J]. Science, 2017, 355(6328): eaai8898. |
| [28] |
TAKAHASHI A, HAYASHI N, MIYAO A, HIROCHIKA H. Unique features of the rice blast resistance Pish locus revealed by large scale retrotransposon-tagging[J]. BMC Plant Biology, 2010, 10(1): 175. DOI:10.1186/1471-2229-10-175 |
| [29] |
杨好, 黄衍焱, 易春霖, 谭褚湉, 任文芮. 水稻Pik位点五个等位基因特异分子标记的开发及应用[J]. 植物病理学报, 2024, 1-11. DOI:10.13926/j.cnki.apps.000897 YANG H, HUANG Y Y, YI C L, TAN Z T, REN W R. Development and application of specific molecular markers for five allelc rice resistance in Pik gene site in rice[J]. Acta Phytopathologica Sinica, 2024, 1-11. DOI:10.13926/j.cnki.apps.000897 |
| [30] |
赵传超, 丛森, 梁思怡, 谢华斌, 陆文宇. 水稻抗稻瘟病基因Pi2的KASP标记开发与应用[J]. 华南农业大学学报, 2023, 44(5): 725-734. DOI:10.7671/j.issn.1001-411X.202304026 ZHAO C C, CONG S, LIANG S Y, XIE H B, LU W Y. Development and application of KASP marker for rice blast resistance gene Pi2[J]. Journal of South China Agricultural University, 2023, 44(5): 725-734. DOI:10.7671/j.issn.1001-411X.202304026 |
| [31] |
何娜, 王丽丽, 高虹, 张丽颖, 唐志强, 付亮, 王昌华, 隋国民, 郑文静, 马作斌. 基于PARMS技术的抗稻瘟病基因Piz-t分子标记的开发与利用[J]. 辽宁农业科学, 2023(5): 20-25. DOI:10.3969/j.issn.1002-1728.2023.05.004 HE N, WANG L L, GAO H, ZHANG L Y, TAANG Z Q, FU L, WANG C H, SUI G M, ZHENG W J, MA Z B. Development and application of molecular marker of rice blast resistance gene Piz-t on based of PARMS Technology[J]. Liaoning Agricultural Sciences, 2023(5): 20-25. DOI:10.3969/j.issn.1002-1728.2023.05.004 |
| [32] |
陆展华, 付魏魏, 刘维, 卢东柏, 王晓飞. 广东省主栽水稻品种稻瘟病主效抗性基因的鉴定及分析[J]. 植物病理学报, 2020, 50(6): 711-722. DOI:10.13926/j.cnki.apps.000505 LU Z H, FU W W, LIU W, LU D B, WANG X F. Identification and analysis of major resistance genes to Magnaporthe oryzae in main rice varieties in Guangdong Province[J]. Acta Phytopathologica Sinica, 2020, 50(6): 711-722. DOI:10.13926/j.cnki.apps.000505 |
| [33] |
汪文娟, 周继勇, 汪聪颖, 苏菁, 封金奇. 八个抗稻瘟病基因在华南籼型杂交水稻中的分布[J]. 中国水稻科学, 2017, 31(3): 299-306. DOI:10.16819/j.1001-7216.2017.6114 WANG W J, ZHOU J Y, WANG C Y, SU J, FENG J Q. Distribution of eight rice blast resistance genes in indica hybrid rice in China[J]. Chinese Journal of Rice Science, 2017, 31(3): 299-306. DOI:10.16819/j.1001-7216.2017.6114 |
| [34] |
梁雪雨, 郭敏, 欧旭华, 陈莹, 周丹华. 南方稻区主栽香稻品种香味基因Badh2基因型分析[J]. 广东农业科学, 2023, 50(12): 104-111. DOI:10.16768/j.issn.1004-874X.2023.12.010 LIANG X Y, GUO M, OU X H, CHEN Y, ZHOU D H. Genotypic analysis of aroma gene Badh2 in aromatic rice cultivars in South China[J]. Guangdong Agricultural Sciences, 2023, 50(12): 104-111. DOI:10.16768/j.issn.1004-874X.2023.12.010 |
(责任编辑 张辉玲)
 2024, Vol. 51
2024, Vol. 51