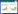文章信息
基金项目
- 国家自然科学基金(32102703);广东省基础与应用基础研究联合基金(2019A1515111186);肇庆学院农村科技特派员工作站项目(2022N001);肇庆大学大学生创新创业培训计划项目(X202310580123)
作者简介
- 区炳明(1989—),男,博士,讲师,研究方向为预防兽医学,E-mail: obm2018@zqu.edu.cn.
文章历史
- 收稿日期:2023-12-10
2. 广东工业大学生物医药学院,广东 广州 510006;
3. 华南师范大学体育科学学院,广东 广州 510006
2. School of Biomedical and Pharmaceutical Sciences, Guangdong University of Technology, Guangzhou 510006, China;
3. School of Physical Education and Sports Science, South China Normal University, Guangzhou, 510006, China
【研究意义】近年来,畜禽肠道益生菌资源备受关注,它可作为兽用抗生素和生长刺激剂的替代品[1]。通过在畜禽饲料中添加益生菌以改变肠道菌群、刺激免疫系统、减少炎症、防止病原体定殖、促进肠道消化和氨或尿素的排泄,从而提高畜禽生长性能。该方法已在养殖业中得到广泛应用,效果良好[2]。然而,实验室开发的典型益生菌通常是供人类使用的,针对动物特异性的益生菌种类极少。此外,这些典型常用的实验室益生菌菌株,虽然已经被基因工程改造并应用于动物宿主,但效果不尽人意,其原因为实验室益生菌菌株未能有效适应动物肠道环境,在与那些已经过数千代繁衍并适应了其特定生态位的原有微生物的竞争中处于劣势地位[3]。因此,这些典型的实验室益生菌菌株难以在动物肠道中长时间定殖,这也使得它们难以在宿主体内发挥应有的疫苗或治疗功效[4]。
【前人研究进展】对畜禽肠道内常驻菌群的调查可以为畜牧业动物肠道疾病的预防和抗菌治疗提供创新策略,目前已取得丰硕成果。近年来,一些研究人员从畜禽中分离出益生菌,然后将这些经过分离鉴定的益生菌直接进行基因改造,放入宿主动物(或模型动物)体内进行试验,取得良好效果。例如,Zhang等[5]从健康猪粪便样品中分离出一株优质的原生植物乳杆菌,并对其进行基因改造,使其具有猪甲型流感病毒血凝性,经口服或鼻内免疫后,可有效保护老鼠免受H1N1病毒感染,这说明从宿主动物体内分离出来的原生细菌可在其宿主动物体内定殖,从而发挥积极作用。因此,利用原生益生菌作为基础菌种开发猪疫病防治疫苗或开发生物制剂极具前景。
【本研究切入点】大肠杆菌(Escherichia coli)是研究最透彻的微生物之一,也是最容易被基因改造的微生物之一。据报道,从宿主动物体内分离出的原生大肠杆菌菌株可在宿主动物肠道内长期定殖,而经过基因工程改造的原生大肠杆菌可达到长期调控宿主生理性状和防治疾病的目的[6]。在对动物源大肠杆菌的早期研究中,鼠源大肠杆菌菌株(MP-1[6-7]和NGF-1[8-9])可以在经过抗生素预处理且无特异病原体(SPF)条件下饲养的鼠肠道腔内环境中长期定殖并持续存在70~200 d。然而,关于猪肠道原生大肠杆菌的报道甚少。猪原生大肠杆菌是一种长期定殖在猪肠道内的共生菌株,可调节猪的生理状况。近10年来,对猪大肠杆菌的研究大多集中在流行病学调查、耐药性、致病性或致病机制等方面,而对猪肠道原生大肠杆菌的非致病性、无抗生素耐药等生物学特性分析及其开发应用的报道甚少。一些关于猪肠道中原生大肠杆菌(或肠杆菌属)[10-12]的早期报道并未涉及其生长性能与系统发育关系的分析。此外,对这种在猪肠道中普遍存在的原生大肠杆菌生物学特性分析的报道也极少且不完整,这无疑增加了利用猪原生大肠杆菌作为微生态制剂开发的难度。
【拟解决的关键问题】鉴于此,本研究旨在从健康猪的粪便中分离出优质猪源性非致病性且无抗生素耐药性的大肠杆菌,并分析其生长性能及遗传进化关系,以期为后续猪用微生态制剂的开发提供优质猪大肠杆菌菌种资源。
1 材料与方法 1.1 试验材料试验所用动物和菌株样本来源为从广东省肇庆市封开县莲都镇生态养殖猪场随机选取11头成年健康的美系三元杂交商品猪(2022年9月),以及从广东省佛山市南海区西樵镇一个小型常规养殖猪场随机选取15头成年健康的美系三元杂交商品猪(2022年10月)。
1.2 试验方法1.2.1 粪便收集和初步筛查 用无菌棉签戳入猪的肛门收集粪便样本,并储存在保温箱中,在3 h内送到实验室进行处理。将粪便样本转移至无菌EP管中,并加入适量PBS溶液,充分振荡摇匀,以使粪便样本溶解。将肇庆市封开县莲都镇生态养殖猪场11头猪对应的粪便样品,按照顺序分别编号为1~11,将佛山市南海区西樵镇养猪场对应的15份粪便样本按照字母顺序编号为A~O,共得到26份粪便样本溶液。
将26份粪便样本原液分别进行倍比稀释至10-3、10-4、10-5、10-6倍,将每个浓度梯度的稀释液涂布于麦康凯琼脂板,在37 ℃下孵育过夜。菌落形成后,从含有适当菌落数量的平板上随机挑选20个经乳糖发酵后呈红色湿润状的单菌落(疑似大肠杆菌菌株),随后用平板划线的方法将挑选出来的单菌落重新接种至新的麦康凯培养基中,并进行编号(如样品1中的菌落标记为1-1、1-2等,样品A中的菌落标记为A-1、A-2等),将培养皿置于37 ℃下培养。培养出明显菌落后,再次划线纯化菌株培养,以此获得纯度最高的疑似大肠杆菌菌株并保存菌种。
1.2.2 16S rRNA基因序列分析 采用煮沸法[13]粗略提取疑似大肠杆菌菌株的基因组DNA,使用细菌16S rRNA基因通用引物(27F:5'-AGAGTTTGATCCTGGCTCAG-3' 和1492R:5'-GGTTACCTTGTTACGACTT-3')[14-15]对该菌株分离的16S rRNA基因片段进行PCR扩增,扩增子大小约为1 500 bp。使用艾科瑞生物科技有限公司的ApexHF HS DNA聚合酶预混液-FS的PCR试剂盒进行PCR扩增。16S rRNA基因PCR产物经1% 琼脂糖凝胶电泳检测,并从琼脂糖凝胶中收集目标16S rRNA基因片段,送至苏州金唯智生物科技有限公司测序。利用NCBI中的BLAST工具,将测序得到的16S rRNA基因的核苷酸序列与NCBI数据库中的序列比对,鉴定其是否为大肠杆菌;如果是大肠杆菌,则可选择用于后续实验。
1.2.3 毒力基因检测 根据Zhang等[13]研究,重新设计部分引物,扩增猪致病性大肠杆菌中常见的13个毒力基因(K88、F18、F41、987P、K99、LT、STa、STb、Stx2e、EAST1、AIDA-I、paa、EAE,表 1),并通过PCR扩增验证每株大肠杆菌是否含有常见的13个毒力基因。采用艾科瑞生物科技有限公司的Taq DNA聚合酶(Mg2+ free和dNTPs+)的PCR试剂盒进行PCR扩增毒力基因,PCR产物在2% 琼脂糖凝胶上电泳检测。

|
1.2.4 药敏试验 采用Kirby-Bauer纸片扩散法[16-17]测定无毒力基因的大肠杆菌分离株对7类共14种常用抗生素的敏感性,包括青霉素类(羧苄青霉素、氨苄西林、阿莫西林)、头孢菌素类(头孢噻肟、头孢唑啉)、氯霉素类(氯霉素)、氨基糖苷类(卡那霉素、庆大霉素、壮观霉素、新霉素、链霉素)、复合抗生素类(复方新诺明)、氟喹诺酮类(恩诺沙星)、四环素类(四环素)。
首先,将选择的大肠杆菌菌株在液体LB培养基中培养约8 h,使其光密度(OD600)达到0.5。之后取100 μL菌悬液涂布于LB琼脂平板上,用镊子将药敏纸片(杭州微生物试剂有限公司)置于培养基表面,在37 ℃培养箱中培养12 h。观察菌株在LB琼脂平板上是否存在抑菌圈,如有抑菌圈则用游标卡尺测量抑制圈大小。抑菌圈大小参照杭州微生物试剂有限公司《药敏试验单片法抑菌范围解释标准》进行检测,以鉴别和评估大肠杆菌的抗生素敏感性。
1.2.5 生长曲线绘制 将过夜培养的菌液OD600调整至1.0,按1∶1 000的比例接种于200 mL LB培养基中,在37 ℃、200 r/min的摇床中连续培养16 h。培养后4 h内,每隔0.5 h测定一次菌悬液OD600;4~16 h内,每隔1 h测定一次菌悬液OD600。以培养时间为横坐标、OD600为纵坐标,绘制生长曲线。
1.2.6 胃液环境耐受性分析 配制PBS溶液(pH值6.68)和人工胃液(pH值3.00),分别吸取1 mL于已高压灭菌的2 mL EP管中,再分别接种100 μL提前培养好且OD600为1.000的25株非致病性无抗的大肠杆菌及4株标准菌(E. coli ATCC 25922、E. coli Nissle 1917、E. coli BL 21、E. coli MG1655)的菌液,37 ℃培养3 h。培养结束后取出,连续稀释到合适梯度,再从稀释液中取100 µL涂布在LB琼脂平板上,于37℃下过夜培养,次日计算活菌数、求存活率。

|
1.2.7 全基因组系统进化树分析 采用生工生物(上海)股份有限公司的Ezup柱式细菌基因组DNA提取试剂盒对分离株的全基因组DNA进行提取,再将其送至上海美吉生物医药科技有限公司,通过Illumina平台进行测序。获得测序结果后,批量化使用Trimmomatic程序去除接头序列和低质量碱基。使用SPAdes程序对改进后的基因组序列进行基因组草图组装,最后使用Quast程序对组装后的基因组效果进行评估[18]。
此外,从GeneBank中下载5株典型非致病性大肠杆菌〔即E. coli Nissle 1917(NZ_CP007799.1)、E. coli ATCC 25922(NZ_CP009072. 1)、E. coli BL21(NZ_CP053601.1)、E. coli EcAZ01(OX341604.1)和E. coli MG1655(NZ_CP025268.1)〕,以及4株猪致病性大肠杆菌〔即E. coliUMNK88(CP002729.1)、E. coli UMNF18(NZ_AGTD01000001)[19]、E. coli PCN061(NZ_CP006636.1)和E. coli PCN033(NZ_CP006632.1)[20]〕的全基因组序列,并作为参考菌株,对25株非致病性、无抗生素耐药性大肠杆菌分离菌株和已报道的典型大肠杆菌菌株进行遗传进化分析。
以E. coli MG1655(NZ_CP025268.1)为参考基因组,构建25株大肠杆菌分离株和9株典型大肠杆菌的进化树。在Linux系统中,通过Parsnp程序[21]构建基于核心基因组SNP(cgSNP)进化树,采用8个线程,且强制包含所有基因组。构建进化树后,利用iTOL工具[22]对遗传进化结果进行美化。
2 结果与分析 2.1 16S rRNA基因序列分析对260株菌株的16S rRNA基因样本进行DNA测序后,再通过BLAST序列比对,最终鉴定出206株菌株为大肠杆菌,将其用于后续实验。
2.2 大肠杆菌所含毒力基因检测鉴定出的206株大肠杆菌中有107株不含13种常见猪致病性大肠杆菌毒力基因。因此,这107株大肠杆菌被视为非致病性猪大肠杆菌菌株,编号如下:1-4、1-8、1-9、2-4、2-5、2-6、2-7、2-9、2-10、2-11、3-1、3-2、3-4、3-7、4-6、4-7、5-1、5-2、5-3、5-5、5-7、5-8、5-9、5-11、6-1、6-2、6-4、6-9、6-10、7-1、7-2、7-4、7-6、7-7、7-8、7-9、7-10、8-2、8-8、8-9、9-8、9-11、10-2、10-5、11-1、11-2、11-3、11-7、11-8、11-9、A-2、B-4、B-6、B-7、B-10、D-7、D-10、E-2、E-3、E-5、E-6、E-7、E-8、E-9、E-10、F-1、F-3、F-5、F-6、F-7、F-8、F-10、G-1、G-2、G-3、G-4、G-5、G-6、G-7、G-8、G-9、G-10、I-1、I-3、I-5、I-6、I-8、J-1、J-4、J-6、J-8、K-2、K-3、K-4、K-5、K-6、K-8、L-6、L-8、L-10、M-4、N-2、N-5、N-6、O-1、O-8、O-9。
2.3 非致病性大肠杆菌常规抗生素耐药性分析在107株非致病性大肠杆菌中,有25株对7类14种抗生素无抗性(表 2),编号如下:2-9**、3-2***、3-4***、5-1****、5-2****、6-1****、6-2****、6-4***、6-9***、6-10****、8-2***、8-9***、10-5****、B-4****、B-6***、B-7***、B-10**、D-10***、E-2****、E-10***、J-1****、J-4****、K-6***、L-6*、O-9*****。其中5-1、5-2、6-1、6-2、6-10、10-5、B-4、E-2、J-1、J-4、O-9对抗生素较为敏感。
2.4 生长曲线绘制
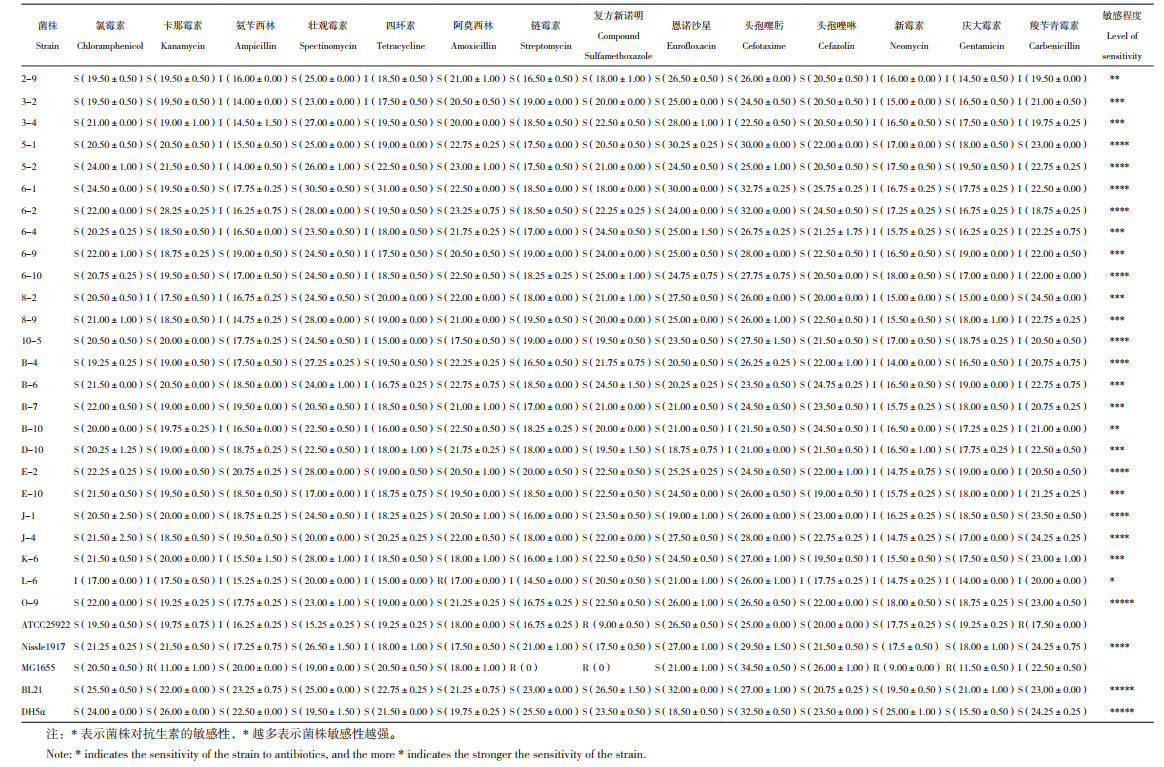
生长曲线结果(图 1)显示,25株非致病性、无抗生素耐药性菌株的生长速率与典型大肠杆菌Nissle 1917、MG1655无显著差异,其中,6-4和5-2的生长速率较其他分离菌株高。此外,与典型基因工程菌株大肠杆菌DH5α和大肠杆菌BL21相比,本研究分离到的大肠杆菌菌株的生长速率更高。
|
| 图 1 30株大肠杆菌分离株的生长曲线 Fig. 1 Growth curves of 30 Escherichia coli isolates |
2.5 胃液环境耐受性分析
以菌株在PBS环境下生存1.5 h或3 h为对照,计算25株非致病性且无抗生素耐药性的大肠杆菌及4株标准菌(E. coli ATCC 25922、E. coli Nissle 1917、E. coli BL 21和E. coli MG1655)的存活率。由图 2可知,E-10、E-2、6-4、6-9、8-2、O-9有较高的存活率。在25株非致病性无抗的大肠杆菌中,除3-2、6-2、6-4、8-2、J-1、K-6外,其他菌株均随着酸性环境时间延长存活率降低。在人工胃液下有较高存活率菌株可能比较适合定殖于猪的肠胃中,不易死亡。此外,在胃液模拟环境中,随着时间的延长,部分菌株数量增长,表明其能在酸性环境下生存并繁殖。综上,本研究推断菌株E-10、E-2、6-4、6-9、8-2、O-9、3-2、6-2、J-1、K-6的耐酸性能较好,其中6-4、8-2是猪胃液环境耐酸性能最强的2个菌株,可能适合长期定殖于猪的肠道内。

|
| 图 2 大肠杆菌菌株在人工胃液中的存活率 Fig. 2 Survival rates of Escherichia coli strains in artificial gastric juices |
2.6 全基因组系统进化树分析
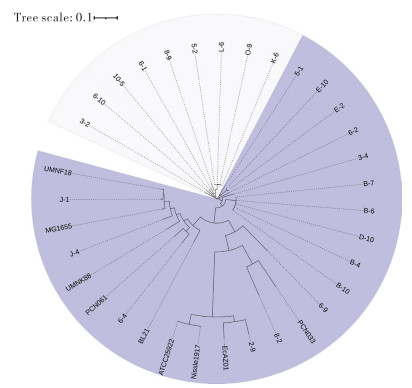
本研究从NCBI中下载了5株经典非致病性大肠杆菌和4株猪源性致病性大肠杆菌的全基因组序列,并结合25株非致病性且无抗生素耐药性大肠杆菌的全基因组序列,利用核心基因组SNP(cgSNP)对34株菌进行全基因组进化树构建(图 3)。在25株非致病性且无抗生素耐药性大肠杆菌分离株中,有9株(即3-2、6-10、10-5、6-1、8-9、5-2、L-6、O-9和K-6)在进化树的起始处属于一个大类群,表明它们很可能属于猪原生大肠杆菌的祖先群,但仍需要进一步研究验证。2-9与人源大肠杆菌Nissle 1917(NZ_CP007799.1)、ATCC 25922(NZ_CP009072.1)及鼠源大肠杆菌EcAZ01(OX341604.1)的进化距离较为接近,暗示其可能是一株具有优良性状的大肠杆菌菌株。此外,J-1、6-4和8-2分别与猪致病性大肠杆菌UMNF18(NZ_AGTD01000001)、PCN061(NZ_CP006636.1)和PCN033(NZ_CP006632.1)[20]同属一个分支,表明它们可能具有致病性。
|
| 图 3 核心基因组遗传进化树 Fig. 3 Genetic evolution tree of the core genome |
2.7 综合分析判断结果
采用维恩图法对这些菌株进行综合评判与筛选,结果(图 4)显示,在对抗生素敏感性的集合中,本研究选取11株菌株(即5-1、5-2、6-1、6-2、6-10、10-5、B-4、E-2、J-1、J-4和O-9);在生长性能集合中,将25株分离菌株全部视为生长动力较强株;在对胃液环境耐受性能的集合中,本研究选取10株菌株(即E-10、E-2、6-4、6-9、8-2、O-9、3-2、6-2、J-1、K-6)。在系统进化树集合中,选择9株位于系统进化树开端属同一分支的菌株(即3-2、6-10、10-5、6-1、8-9、5-2、L-6、O-9、K-6)。通过对抗生素敏感性、生长性能、进化树和胃液环境耐受性能的交集分析,有1株菌株(O-9)在交集分析中表现出优势。
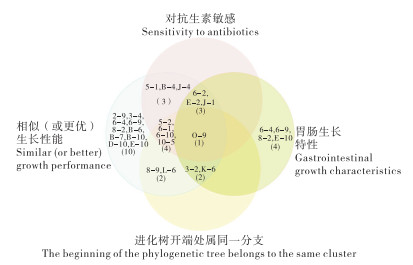
|
| 图 4 维恩图分析 Fig. 4 Venn diagram analysis |
3 讨论
本研究共分离出25株非致病性且无抗生素耐药性的猪原生大肠杆菌,在生长动力学研究中,这25株大肠杆菌的生长速率与典型菌株大肠杆菌Nissle 1917和大肠杆菌MG1655无显著差异。与大多数大肠杆菌分离株相比,6-4和5-2的生长速率更高,表明二者的代谢负荷比其他菌株小,但这25株菌株的生长曲线并无显著差异。本研究认为这25株猪大肠杆菌菌株的代谢负荷可能与典型大肠杆菌Nissle 1917和大肠杆菌MG1655相似。
系统进化树分析结果显示,3-2、6-10、10-5、6-1、8-9、5-2、L-6、O-9和K-6位于系统进化树的起始端,推测它们可能是猪原生大肠杆菌的祖先种,具有较大的研究价值,有待进一步探讨。考虑到8-2与大肠杆菌菌株PCN033(GenBank:NZ_CP006632.1)亲缘关系密切,6-4与大肠杆菌PCN061(NZ_CP006636.1)、J-1与大肠杆菌UMNF18(NZ_AGTD01000001.1)亲缘关系较近,因此推断8-2、6-4与J-1可能具有致病性,尚需进一步研究。此外,2-9与鼠源益生菌大肠杆菌EcAZ01、人源大肠杆菌Nissle 1917和ATCC 25922的进化距离较为接近,且同属一个分支,说明2-9极有可能是优良的猪原生大肠杆菌菌株,这为后续研究提供了重要参考。整体上看,进化树的起始亚群(即3-2、6-10、10-5、6-1、8-9、5-2、L-6、O-9、K-6)和2-9(靠近大肠杆菌EcAZ01、大肠杆菌Nissle 1917和大肠杆菌ATCC 25922)可能是优质猪肠道原生大肠杆菌菌种,均具有极大的研究潜力。
在系统进化树上,上游聚类3-2、6-10、10-5、6-1、8-9、5-2、L-6、O-9和K-6与其他聚类明显分离,推测这些可能是猪原生大肠杆菌的祖先群。此外,2-9与经典的优质大肠杆菌菌株(EcAZ01、Nissle 1917和ATCC 25922)进化距离较近,可能具有较大的开发应用潜力。结合上述结果和常规抗生素耐药性分析结果,分离得到的大肠杆菌菌株(即5-2、6-1、6-10、10-5)可能是优质猪肠道原生大肠杆菌的良好资源,这些菌株可以为开发猪口服疫苗或开发生物制剂提供重要基础,值得进一步分析和研究。
然而,有报道称猪原生大肠杆菌是IS26相关Ⅰ类整合子的基因储存库,可能会对人类健康和公共卫生安全构成潜在风险[23]。因此,本研究对经过常规毒力基因PCR检测和7类(14种)常规抗生素药敏试验所筛选出的非致病性、无抗生素耐药性的原生大肠杆菌还有待进一步验证和发掘,尚可能存在一些隐藏的毒力基因(或致病因素)及其他耐药因素未被检测出,因此仍然存在潜在风险。类似地,据报道广泛使用的人源益生菌菌株大肠杆菌Nissle 1917,其含有1个能编码产生毒素蛋白的pks基因簇[24]。大量研究均在寻找具有益生菌活性的替代安全菌株。因此,未来可以利用ResFinder、VirulenceFinder或其他生物信息学工具[25]对这些大肠杆菌分离株进行耐药性和毒力基因鉴定,以确定这些菌株是否具有进一步分析的潜力。本研究分离出的菌株2-9与前面提到的大肠杆菌Nissle 1917关系密切,有待展开深入分析。本研究可为猪原生大肠杆菌的分离筛选提供初步参考、为探索猪微生态制剂的优质菌株资源奠定重要基础,未来还需要对这批微生物的基因组进行全面鉴定和解释,充分挖掘其生物功能。
4 结论本试验从猪肠道中成功分离出无毒力基因且无抗生素耐药性的大肠杆菌,并分析其生长性能、胃液环境耐受性能和遗传进化规律。综合对大肠杆菌的抗生素敏感性、生长性能、胃液环境耐受性能和全基因组进化树分析,其中有1株分离株(即O-9)在这4个方面同时具备优势,且该菌株与大肠杆菌Nissle 1917和大肠杆菌MG1655的生长动力学相似甚至更优,表明其在抗生素耐药性较弱的同时拥有良好的生长特性,该菌株可能是优质的猪肠道原生大肠杆菌,具有极大研究潜力。
| [1] |
SHOKRYAZDAN P, FASELEH JAHROMI M, LIANG J B, HO Y W. Probiotics: From isolation to application[J]. Journal of the American College of Nutrition, 2017, 36(8): 666-676. DOI:10.1080/07315724.2017.1337529 |
| [2] |
JHA R, DAS R, OAK S, MISHRA P. Probiotics (direct-fed microbials) in poultry nutrition and their effects on nutrient utilization, growth and laying performance, and gut health: A systematic review[J]. Animals, 2020, 10(10): 1863. DOI:10.3390/ani10101863 |
| [3] |
HUANG S, JIANG S, HUO D, ALLABAND C, ESTAKI M, CANTU V, BELDA-FERRE P, VáZQUEZ-BAEZA Y, ZHU Q, MA C. Candidate probiotic Lactiplantibacillus plantarum HNU082 rapidly and convergently evolves within human, mice, and zebrafish gut but differentially influences the resident microbiome[J]. Microbiome, 2021, 9(1): 1-17. DOI:10.1186/s40168-021-01102-0 |
| [4] |
OU B, JIANG B, JIN D, YANG Y, ZHANG M, ZHANG D, ZHAO H, XU M, SONG H, WU W. Engineered recombinant Escherichia coli probiotic strains integrated with F4 and F18 fimbriae cluster genes in the chromosome and their assessment of immunogenic efficacy in vivo[J]. ACS Synthetic Biology, 2020, 9(2): 412-426. DOI:10.1021/acssynbio.9b00430 |
| [5] |
ZHANG Y, YANG L, ZHANG J, HUANG K, SUN X, YANG Y, WANG T, ZHANG Q, ZOU Z, JIN M. Oral or intranasal immunization with recombinant Lactobacillus plantarum displaying head domain of Swine Influenza A virus hemagglutinin protects mice from H1N1 virus[J]. Microbial Cell Factories, 2022, 21(1): 185. DOI:10.1186/s12934-022-01911-4 |
| [6] |
RUSSELL B J, BROWN S D, SIGUENZA N, MAI I, SARAN A R, LINGARAJU A, MAISSY E S, MACHADO A C D, PINTO A F, SANCHEZ C. Intestinal transgene delivery with native E. coli chassis allows persistent physiological changes[J]. Cell, 2022, 185(17): 3263-3277.e15. DOI:10.1016/j.cell.2022,06.050 |
| [7] |
LASARO M, LIU Z, BISHAR R, KELLY K, CHATTOPADHYAY S, PAUL S, SOKURENKO E, ZHU J, GOULIAN M. Escherichia coli isolate for studying colonization of the mouse intestine and its application to two-component signaling knockouts[J]. Journal of Bacteriology, 2014, 196(9): 1723-1732. DOI:10.1128/jb.01296-13 |
| [8] |
RIGLAR D T, GIESSEN T W, BAYM M, KERNS S J, NIEDERHUBER M J, BRONSON R T, KOTULA J W, GERBER G K, WAY J C, SILVER P A. Engineered bacteria can function in the mammalian gut long-term as live diagnostics of inflammation[J]. Nature Biotechnology, 2017, 35(7): 653-658. DOI:10.1038/nbt.3879 |
| [9] |
KOTULA J W, KERNS S J, SHAKET L A, SIRAJ L, COLLINS J J, WAY J C, SILVER P A. Programmable bacteria detect and record an environmental signal in the mammalian gut[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(13): 4838-4843. DOI:10.1073/pnas.1321321111 |
| [10] |
KATOULI M, LUND A, WALLGREN P, KüHN I, SÖDERLIND O, MÖLLBY R. Phenotypic characterization of intestinal Escherichia coli of pigs during suckling, postweaning, and fattening periods[J]. Applied and Environmental Microbiology, 1995, 61(2): 778-783. DOI:10.1128/aem.61.2.778-783.1995 |
| [11] |
SCHIERACK P, RÖMER A, JORES J, KASPAR H, GUENTHER S, FILTER M, EICHBERG J, WIELER L H. Isolation and characterization of intestinal Escherichia coli clones from wild boars in Germany[J]. Applied and Environmental Microbiology, 2009, 75(3): 695-702. DOI:10.1128/AEM.01650-08 |
| [12] |
SCHIERACK P, WALK N, REITER K, WEYRAUCH K D, WIELER L H. Composition of intestinal Enterobacteriaceae populations of healthy domestic pigs[J]. Microbiology, 2007, 153(11): 3830-3837. DOI:10.1099/mic.0.2007/010173-0 |
| [13] |
ZHANG W, ZHAO M, RUESCH L, OMOT A, FRANCIS D. Prevalence of virulence genes in Escherichia coli strains recently isolated from young pigs with diarrhea in the US[J]. Veterinary Microbiology, 2007, 123(1/3): 145-152. DOI:10.1016/j.vetmic.2007.02.018 |
| [14] |
WANG M, WEI P, CAO M, ZHU L, LU Y. First report of rice seedling blight caused by Burkholderia plantarii in North and Southeast China[J]. Plant Disease, 2016, 100(3): 645. DOI:10.1094/PDIS-07-15-0765-PDN |
| [15] |
金正雨, 邓永军, 杨泽敏, 李双, 杨颖. 瑶鸡粪便中乳酸菌的分离鉴定及筛选[J]. 中国生物制品学杂志, 2022, 35(4): 413-417. DOI:10.13200/j.cnki.cjb.003586 JIN Z Y, DENG Y J, YANG Z M, LI S, YANG Y. Isolation, identification and screening of lactobacillus from Yao chicken feces[J]. Chinese Journal of Biologicals, 2022, 35(4): 413-417. DOI:10.13200/j.cnki.cjb.003586 |
| [16] |
QAMAR M U, HANNAN A, ARSHAD M U, ARSHAD M. Metallo-β-lactamase producing Enterobacter cloacae: An emerging threat in neonates[J]. African Journal of Microbiology Research, 2014, 8(6): 525-528. DOI:10.5897/AJMR2013.6348 |
| [17] |
巩新廷, 石岩, 苏玉贤. 河南省部分地区鸡源沙门菌分离鉴定与耐药性分析[J]. 中国动物传染病学报, 2022, 30(6): 153-157. DOI:10.19958/j.cnki.cn31-2031/s.20220411.002 GONG X T, SHI Y, SU Y X. Isolation, identifi cation and drug resistance of Salmonella strains from chickens in some areas of Henan Province[J]. Chinese Journal of Animal Infectious Diseases, 2022, 30(6): 153-157. DOI:10.19958/j.cnki.cn31-2031/s.20220411.002 |
| [18] |
CORTESE I J, CASTRILLO M L, ONETTO A L, BICH G Á, ZAPATA P D, LACZESKI M E. De novo genome assembly of Bacillus altitudinis 19RS3 and Bacillus altitudinis T5S-T4, two plant growth-promoting bacteria isolated from Ilex paraguariensis St. Hil. (yerba mate)[J]. PloS One, 2021, 16(3): e0248274. DOI:10.1371/journal.pone.0248274 |
| [19] |
SHEPARD S M, DANZEISEN J L, ISAACSON R E, SEEMANN T, ACHTMAN M, JOHNSON T J. Genome sequences and phylogenetic analysis of K88-and F18-positive porcine enterotoxigenic Escherichia coli[J]. Journal of Bacteriology, 2011. DOI:10.1128/jb.06225-11 |
| [20] |
LIU C, ZHENG H, YANG M, XU Z, WANG X, WEI L, TANG B, LIU F, ZHANG Y, DING Y. Genome analysis and in vivo virulence of porcine extraintestinal pathogenic Escherichia coli strain PCN033[J]. BMC Genomics, 2015, 16(1): 1-18. DOI:10.1186/s12864-015-1890-9 |
| [21] |
WANG Z, JIANG Y, XU H, JIAO X, WANG J, LI Q. Poultry production as the main reservoir of ciprofloxacin-and tigecycline-resistant extended-spectrum β-lactamase (ESBL)-producing Salmonella enterica serovar Kentucky ST198. 2-2 causing human infections in China[J]. Applied and Environmental Microbiology, 2023, 89(9): e0094423. DOI:10.1128/aem.00944-23 |
| [22] |
SHANG Z, LIU S, DUAN Y, BAO C, WANG J, DONG B, CAO Y. Complete genome sequencing and investigation on the fiber-degrading potential of Bacillus amyloliquefaciens strain TL106 from the tibetan pig[J]. BMC Microbiology, 2022, 22(1): 186. DOI:10.1186/s12866-022-02599-7 |
| [23] |
REID C J, WYRSCH E R, ROY CHOWDHURY P, ZINGALI T, LIU M, DARLING A E, CHAPMAN T A, DJORDJEVIC S P. Porcine commensal Escherichia coli: A reservoir for class 1 integrons associated with IS 26[J]. Microbial Genomics, 2017, 3(12): e000143. DOI:10.1099/mgen.0.000143 |
| [24] |
MASSIP C, BRANCHU P, BOSSUET-GREIF N, CHAGNEAU C V, GAILLARD D, MARTIN P, BOURY M, SECHER T, DUBOIS D, NOUGAYRèDE J P. Deciphering the interplay between the genotoxic and probiotic activities of Escherichia coli Nissle 1917[J]. PLoS Pathogens, 2019, 15(9): e1008029. DOI:10.1371/journal.ppat.1008029 |
| [25] |
KLEINHEINZ K A, JOENSEN K G, LARSEN M V. Applying the ResFinder and VirulenceFinder web-services for easy identification of acquired antibiotic resistance and E. coli virulence genes in bacteriophage and prophage nucleotide sequences[J]. Bacteriophage, 2014, 4(2): e27943. DOI:10.4161/bact.27943 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51