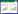文章信息
基金项目
- 国家自然科学基金(31801800);广东省自然科学基金(2020A151501960);广东省农业科学院协同创新中心项目(XTXM202209);国家荔枝龙眼产业技术体系专项(CARS-32);广东省农业科学院乡村振兴战略专项(2024TS-2-2);广州市科技计划项目(2024E04J1261)
作者简介
-
姚琼,博士,研究员,广东省农业科学院植物保护研究所果树害虫防控课题组负责人,主要从事荔枝龙眼虫害诊断及绿色防控新技术研究。主持国家自然科学基金、广东省自然科学基金、广东省公益研究与能力建设专项和广州市科技创新人才专项等13项。发表学术论文40余篇,参与制定团体标准2项、出版著作1部。获广州市“珠江科技新星”称号;获广东省科学技术二等奖、广东省农业技术推广奖等4项;获授权国家发明专利20余件。现任国家荔枝龙眼产业技术体系虫害防控岗位专家、中国昆虫学会农业昆虫委员会委员、广东省地理标志专家库专家、深圳市农业科技促进中心专家库专家、《广东农业科学》青年编委。担任本期“果树病虫害研究”专题的客座主编。
姚琼(1984—),女,博士,研究员,研究方向为农业昆虫与害虫防治,E-mail:joanyao_0603@163.com.
通讯作者
- 李文景(1987—),男,博士,副研究员,研究方向为农业昆虫与害虫防治,E-mail:lwj4631221@126.com.
文章历史
- 收稿日期:2024-04-27
2. 华南师范大学生命科学学院,广东 广州 510631
2. School of Life Sciences, South China Normal University, Guangzhou 510631, China
【研究意义】荔枝蒂蛀虫(Conopomorpha sinesis Bradley)属鳞翅目细蛾科,是荔枝、龙眼危害最为严重的害虫之一,主要分布于中国、尼泊尔、印度、东南亚等地[1]。其幼虫蛀食寄主的花穗、嫩梢及果实,危害严重时蛀果率可高达90%,大大降低了荔枝和龙眼的品质,给荔枝、龙眼产业造成巨大的经济损失[2]。在当前的荔枝、龙眼栽培领域,荔枝蒂蛀虫的防控仍主要依赖化学防治,其单一的防控方式导致果实农药残留过高,对环境和消费者健康造成潜在威胁[3]。因此,亟需转变防控思路,积极探索绿色防控措施。海藻糖作为昆虫血淋巴的主要糖类,不仅是昆虫应激代谢的重要产物,还是一种贮藏性糖类,为昆虫各组织器官活动提供能量[4]。海藻糖在昆虫体内唯一的代谢途径是在海藻糖酶(Trehalase,Tre)的作用下专一性地分解为葡萄糖[5]。海藻糖酶在昆虫的多个生理过程中发挥着重要作用,可调控昆虫几丁质的合成[6],而几丁质作为昆虫表皮的主要成分,其合成与降解对昆虫的生长发育具有举足轻重的意义。因此,掌握荔枝蒂蛀虫海藻糖酶基因的功能及表达模式,可为今后开发该虫防控新靶标提供重要基础支撑。【前人研究进展】在昆虫体内,海藻糖酶是海藻糖代谢过程中不可或缺的一环, 最早由Frerejacaque在1941年发现。随着研究的深入,科学家们发现昆虫体内存在两种类型的海藻糖酶:可溶型(Tre1)和膜结合型(Tre2)[7]。前者主要存在于昆虫的消化和循环系统中,后者主要存在于昆虫的肌肉中[8]。Takiguchi等[9]在黄粉虫(Tenebrio molitor)中首先克隆出第一个可溶型海藻糖酶基因,而膜结合型海藻糖酶基因由Mitsumasu等[10]在家蚕(Bombyx mori)中首次克隆获得。研究表明,大多数的昆虫体内均存在着1个可溶型和1个膜结合型海藻糖酶基因,少数昆虫(如褐飞虱、异色瓢虫)发现多个可溶型海藻糖酶基因[11]。研究显示,分别对甜菜夜蛾(Spodoptera exigua)可溶型和膜结合型海藻糖酶基因进行RNA干扰,可抑制其几丁质合成酶1和2基因的表达,导致表皮和中肠几丁质的合成显著减少[12]。【本研究切入点】尽管目前针对昆虫海藻糖酶已开展了大量的研究,但荔枝蒂蛀虫海藻糖酶基因的分子特性及功能仍然未知。【拟解决的关键问题】本研究从荔枝蒂蛀虫中克隆海藻糖酶基因CsTre1和CsTre2,研究其基因序列、编码蛋白的分子特征和基因表达模式,以期为研究荔枝蒂蛀虫海藻糖酶基因的生物学功能和能量代谢相关分子网络的研究提供一定的理论依据。
1 材料与方法 1.1 供试昆虫供试荔枝蒂蛀虫:采集广东省农业科学院白云基地荔枝园的落果,在养虫室等待其化蛹,收集虫蛹,待成虫羽化后,置于养虫笼内,以10% 的蜂蜜水饲养。养虫室饲养条件:温度25(±1)℃,相对湿度75(±1)%,光周期14∶10(L∶D)。
1.2 总RNA的提取与反转录利用Trizol试剂分别提取荔枝蒂蛀虫蛹、雌性成虫、雄性成虫,以及雌性成虫的头、胸、腹、卵巢、腿和雄性成虫的头、胸、腹、腿的总RNA,采用2%的琼脂糖凝胶电泳进行检测,采用PrimeScript RT Reagent试剂盒反转录合成cDNA第一链,置于-20℃备用。
1.3 基因克隆和分析以荔枝蒂蛀虫转录组(数据待发表)测序结果为基础,搜索注释好的unigene中关于“trehalase”的基因序列,将其对应的蛋白序列与NCBI中的NR数据库进行blastp,验证是否为海藻糖酶。使用Primer 5.0软件根据目的基因片段设计引物(表 1),用于扩增荔枝蒂蛀虫CsTre1、CsTre2基因全长序列。分别以上述的特异性引物进行5' 和3' RACE扩增,RACE试剂盒使用SMARTer® RACE 5'/3'Kit(Takara)。5' RACE反应程序为:94℃ 3 min;94℃ 30 s,65℃ 30 s,72℃ 1 min,32个循环;72℃ 10min;4℃保存。3' RACE反应程序为:94℃ 3 min;94℃ 30 s,58℃ 30 s,72℃ 1 min,32个循环;72℃ 10 min;4℃保存。最后通过DNAstar软件将所得的序列片段拼接为完整的序列。
使用NCBI的ORF Finder网站查找序列的开放阅读框(ORF);使用DNAstar软件预测其编码氨基酸序列;使用ProtParam软件预测蛋白的分子量、等电点等理化性质;利用SignalP 4.1 Server来预测信号肽;使用NetPhos2.0 Server软件来进行磷酸化位点分析;使用ProtScale和Protparam软件进行疏水性分析;利用SWISSMODEL在线网站来进行序列二级结构分析;使用Phyre2和pymol软件来构建并绘制出序列的三维分子结构图;系统进化树使用IQ-TREE和ITOL进行绘制。
1.4 荧光定量检测荔枝蒂蛀虫CsTre1、CsTre2实时荧光定量PCR(RT-qPCR)引物由Primer 5.0软件设计(表 1),采用GoTaq qPCR Master Mix试剂盒(Promega,美国)进行RT-qPCR。反应体系:10 μL GoTaq qPCR Master Mix,1 μL cDNA模板,0.5 μL正向引物,0.5 μL反向引物,8 μL DEPC水。反应程序:95℃预变性2 min;95℃变性15 s;60℃退火60 s,40个循环。以Csβ-actin基因作为内参基因计算各样本的相对表达量,使用比较CT值方法来进行数据分析[13]。
2 结果与分析 2.1 CsTre1和CsTre2基因克隆与序列分析根据荔枝蒂蛀虫转录组数据中CsTre1和CsTre2基因的序列信息(图 1),设计针对二者的特异性引物,以荔枝蒂蛀虫成虫cDNA为模板进行PCR扩增,得到CsTre1和CsTre2基因ORF的长度为1 701 bp和1 821 bp;分别编码566个和606个氨基酸;蛋白质分子量分别为64.53、69.08 kD;等电点分别为5.34和5.39。

|
| A: CsTre1; B: CsTre2 图 1 CsTre开放阅读框及其氨基酸序列 Fig. 1 Open reading frame and amino acid sequences of CsTre |
2.2 CsTre1和CsTre2蛋白的信号肽预测
信号肽是一种在新合成多肽链中,参与指导蛋白质跨膜转移(定位)的N- 末端氨基酸残基序列[14]。使用SignalP 4.1 Server软件进行信号肽预测,依据S-score接近1的部分为信号肽和C-score越高越可能为剪切位点,发现CsTre1和CsTre2均具有信号肽,位点分别在1-16和1-17(图 2)。
|
| A: CsTre1; B: CsTre2 图 2 CsTre蛋白信号肽预测 Fig. 2 Signal peptide prediction of CsTre proteins |
2.3 CsTre1和CsTre2蛋白的磷酸化位点分析
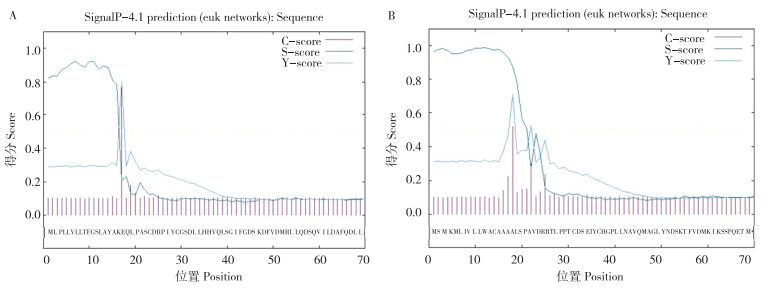
蛋白质磷酸化是一种调节蛋白质功能和活性的最基本、最普遍、最重要的机制[15]。依据NetPhos-3.1软件,超过阈值0.5的为潜在磷酸化位点。由 图 3可知,CsTre1有24个Ser、15个Tyr、10个Thr可能成为蛋白激酶的结合位点;CsTre2有27个Ser、10个Tyr、13个Thr可能成为蛋白激酶的结合位点。蛋白激酶将三磷酸腺苷上的γ- 磷酸转移到这些结合位点上(Ser、Thr、Tyr),通过添加磷酸基团到底物蛋白,可调节多种蛋白的活动、定位及总体功能[16]。
|
| A: CsTre1; B: CsTre2 图 3 CsTre蛋白磷酸化位点分析 Fig. 3 Phosphorylation site analysis of CsTre proteins |
2.4 CsTre1和CsTre2蛋白的疏水性分析
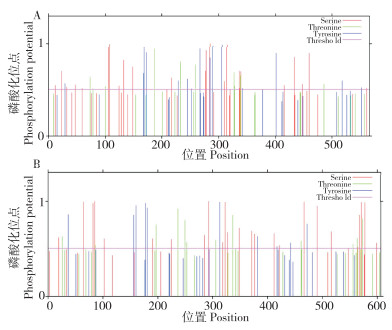
由图 4可知,CsTre1氨基酸序列的第6号缬氨酸(Val)分值最高(2.633),疏水性最强。第406号天冬酰胺(Asn)分值最低(-3.622),亲水性最强;CsTre2氨基酸序列的第593号亮氨酸(Leu)分值最高(3.033),疏水性最强。第493号天冬氨酸(Asp)分值最低(-2.733),亲水性最强。CsTre1的平均疏水性为-0.380,CsTre2的平均疏水性为-0.311,整体亲水性均大于疏水性,表明二者都为亲水蛋白。
|
| A: CsTre1; B: CsTre2 图 4 CsTre蛋白疏水性分析 Fig. 4 Hydrophobicity analysis of CsTre proteins |
2.5 CsTre1和CsTre2编码蛋白二级结构预测
蛋白质的二级结构是空间结构的基础,在空间上形成α螺旋、β折叠、β转角和无规则卷曲等结构[17]。采用SOPMA在线工具分析CsTre1和CsTre2蛋白的二级结构,由图 5可知,CsTre1主要由α- 螺旋和无规则卷曲组成:α螺旋占46.47%,β折叠占8.83%,无规则卷曲占44.70%。CsTre2也主要由α- 螺旋和无规则卷曲组成:α螺旋占46.04%,β折叠占8.75%,无规则卷曲占45.21%。
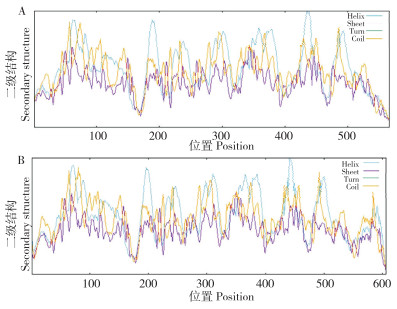
|
| A: CsTre1; B: CsTre2 图 5 CsTre蛋白二级结构预测 Fig. 5 Prediction of secondary structure of CsTre proteins |
2.6 CsTre1和CsTre2编码蛋白三级结构模拟
使用Phyre2软件进行CsTre1和CsTre2蛋白同源建模,并在pymol软件中可视化。Phyre2软件的结果显示,CsTre1的同源模型为拟南芥酸敏感型突变体的海藻糖酶(c7e9xA),可信度达100%,覆盖度为94%;CsTre2的同源模型是拟南芥酸敏感型突变体海藻糖复合体的海藻糖酶(c7eawA),可信度达100%,覆盖度为88%(图 6)。
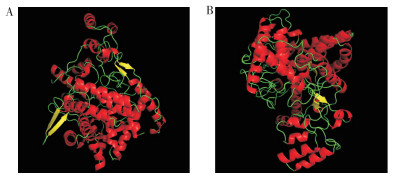
|
| A: CsTre1; B: CsTre2 图 6 CsTre蛋白三级结构模拟 Fig. 6 Prediction of tertiary structure of CsTre proteins |
2.7 CsTre1和CsTre2系统进化树分析
根据已知昆虫海藻糖酶氨基酸全长序列构建系统进化树,结果(图 7)显示可溶型和膜结合型两种昆虫海藻糖酶基因各自聚成一支,CsTre1位于可溶型鳞翅目昆虫海藻糖酶的分支,而CsTre2位于膜结合型鳞翅目昆虫海藻糖酶的分支。荔枝蒂蛀虫海藻糖酶与竹蠹螟(Omphisa fuscidentalis)的海藻糖酶亲缘关系最近,与其他非鳞翅目昆虫海藻糖酶的亲缘关系较远。系统进化树表明,海藻糖酶在鳞翅目昆虫中具有相对保守的进化特性。
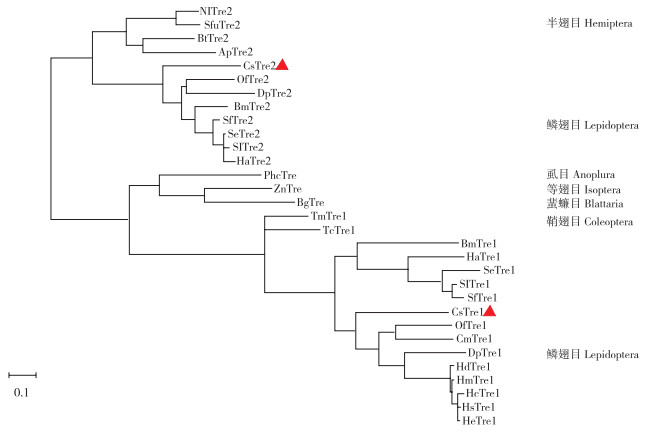
|
| 图 7 基于已知昆虫海藻糖酶氨基酸序列构建的系统进化树 Fig. 7 Phylogenetic analysis of amino acid sequences based on known trehalases from insects |
2.8 CsTre1和CsTre2基因表达模式分析
由不同发育阶段表达结果可知,成虫期CsTre1和CsTre2的相对表达量高于蛹期,且成虫期CsTre1的相对表达量要高于成虫期CsTre2。然而,CsTre1在雄成虫第2 d和第5 d的相对表达量陡然下降,在雌成虫中则保持稳定高表达(图 8A)。由不同组织部位表达结果可知,CsTre1在雌成虫的腹部和卵巢及雄成虫的腹部的相对表达量比较高,而CsTre2的相对表达量则与之相反(图 8B)。结合不同发育阶段和不同组织部位表达结果,发现CsTre1的相对表达量普遍高于CsTre2,推测CsTre1对荔枝蒂蛀虫的影响更为显著。
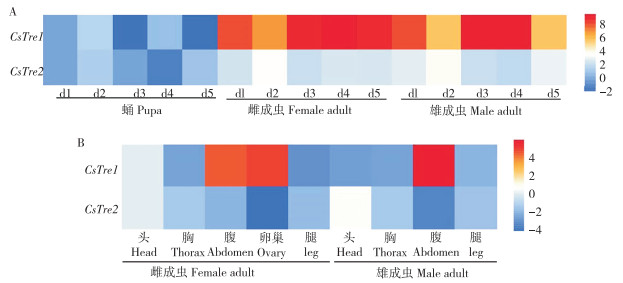
|
| 图 8 荔枝蒂蛀虫不同发育阶段(A)和不同组织部位(B)CsTre的相对表达量 Fig. 8 Relative expression level of CsTre in different developmental stages (A) and tissues (B) of Conopomorpha Sinensis Bradley |
3 讨论
本研究从荔枝蒂蛀虫的转录组数据中鉴定到2个unigene——CsTre1和CsTre2,其编码的蛋白质与昆虫海藻糖酶具有较高的相似性,因此推测二者为荔枝蒂蛀虫的海藻糖酶基因。使用RACE方法克隆得到CsTre1和CsTre2的全长cDNA序列,通过系统进化树、同源建模等生物信息学方法,进一步验证了CsTre1和CsTre2分别为可溶型海藻糖酶基因和膜结合型海藻糖酶基因。CsTre1的ORF长1 701 bp,编码566个氨基酸,蛋白分子量为64.53 kD。CsTre2的ORF长1 821 bp,编码606个氨基酸,蛋白分子量为69.08 kD。二者整体亲水性均大于疏水性,二级结构均主要由α- 螺旋和无规则卷曲组成。CsTre1和CsTre2的编码蛋白在N端预测有信号肽,推测该两个蛋白可能为分泌型蛋白,可跨膜转运到其他组织部位中发挥作用[18]。
昆虫海藻糖酶的分子量一般在60~140 kD,根据分子大小、动力学特性、分布位置,将该酶划分为两种:可溶型海藻糖酶和膜结合型海藻糖酶。可溶型海藻糖酶已被科学家们在昆虫的卵的匀浆液、血淋巴、中肠杯状细胞腔中纯化得到[19],而膜结合型海藻糖酶则在昆虫的精囊、卵巢细胞、飞行肌、中肠、脑等组织中有被研究[20]。前人研究发现,昆虫海藻糖酶的活性能够被激素调节,20- 羟基蜕皮酮能够增强竹蠹螟Tre1的活性,但对Tre2的影响不大[21]。海藻糖可为昆虫生理活动提供稳定的葡萄糖作为能量来源,并在调节昆虫几丁质合成、耐寒、迁飞等活动中发挥着决定性的作用[22]。
本研究采用RT-qPCR技术,对CsTre1和CsTre2在荔枝蒂蛀虫不同发育阶段和不同组织部位的表达进行分析,结果显示2种海藻糖酶在成虫期的表达水平要比蛹期要高,推测是由于成虫需要更多的能量用于补充飞行运动中的消耗,促使产生更多海藻糖酶用于分解海藻糖为葡萄糖;而蛹期表达相对较少,可能是蛹期这个阶段不需要太多的能量。这一结果与田宇等[23]的研究结果一致。另外,比较CsTre1与CsTre2各发育阶段的表达水平,发现CsTre1的相对表达量普遍高于CsTre2(图 8A),表明CsTre1可能是荔枝蒂蛀虫主要调控海藻糖代谢的基因,为昆虫飞行和肌肉剧烈运动提供能量。有研究表明昆虫体内可溶型海藻糖酶的主要分布区域为消化道和血淋巴[24]。本研究通过荔枝蒂蛀虫不同组织部位CsTre的相对表达量分析中,观察到在雌雄成虫的腹部,CsTre1的表达水平相对较高,进一步验证了前人的研究结果。除此之外,CsTre1在卵巢的表达也呈现出相对较高的水平,卵巢的高表达可能是为了诱导更多的海藻糖水解为葡萄糖,以供荔枝蒂蛀虫卵巢发育所需的能量。
近年来,越来越多的科学家开始深入探索海藻糖酶基因的具体功能。对褐飞虱中的海藻糖酶基因进行干扰,抑制各个海藻糖酶基因的表达后,几丁质合成酶等基因也相应发生了下调[26]。赤拟谷盗的单个Tre基因沉默后,同家族其他Tre基因会发生补充和连锁抑制的效果[27]。异色瓢虫的Tre2-like和Tre2基因被干扰后,不仅导致异色瓢虫死亡,还引发了蜕皮障碍,进而推测抑制Tre2-like和Tre2基因的表达,可能下调了相关基因在胰岛素通路上的表达,进而调控海藻糖等糖类的物质代谢[28]。鉴于荔枝蒂蛀虫海藻糖酶基因的功能和机制尚未清楚,后续研究还需通过RNAi、基因编辑等技术来干扰CsTre1和CsTre2,进一步揭示其在荔枝蒂蛀虫中的作用机制。大力开展利用RNAi技术沉默农业害虫海藻糖酶基因的技术研发,破坏海藻糖等糖类物质代谢的调控系统,最终导致害虫的生长发育障碍甚至死亡,从而实现对害虫的有效防控,这为害虫的绿色防控提供了新思路和新靶标。
4 结论荔枝蒂蛀虫海藻糖酶基因CsTre1的ORF长1 701 bp,编码566个氨基酸,蛋白分子量为64.53 kD;CsTre2的ORF长1 821 bp,编码606个氨基酸,蛋白分子量为69.08 kD。二者整体亲水性均大于疏水性,二级结构均主要由α- 螺旋和无规则卷曲组成。CsTre1和CsTre2的编码蛋白在N端预测有信号肽,两个蛋白可能为分泌型蛋白。成虫期CsTre1和CsTre2的相对表达量是高于蛹期,且CsTre1的相对表达量普遍高于CsTre2,推测CsTre1可能是荔枝蒂蛀虫主要调控海藻糖代谢的基因。本研究结果为探明荔枝蒂蛀虫海藻糖酶基因的具体生理功能和昆虫能量代谢相关分子网络提供了一定的数据基础。
| [1] |
李文景, 董易之, 姚琼, 陈炳旭. 荔枝蒂蛀虫研究进展[J]. 昆虫学报, 2018, 61(6): 721-732. DOI:10.16380/j.kcxb.2018.06.011 LI W J, DONG Y Z, YAO Q, CHEN B X. Research progress in the litchi fruit borer, Conopomorpha sinensis (Lepidoptera: Gracillariidae)[J]. Acta Entomologica Sinica, 2018, 61(6): 721-732. DOI:10.16380/j.kcxb.2018.06.011 |
| [2] |
YAO Q, LIANG Z, CHEN B. Evidence for the participation of chemosensory proteins in response to insecticide challenge in Conopomorpha sinensis[J]. Journal of Agricultural and Food Chemistry, 2023, 71(3): 1360-1368. DOI:10.1021/acs.jafc.2c05973 |
| [3] |
YAO Q, DONG Y, LI W, CHEN B. The effects of non-host plant extracts on the oviposition deterrent and ovicidal activity of Conopomorpha sinensis Bradley (Lepidoptera: Gracillariidae) [J]. Florida Entomologist, 2019, 102: 298-302, 5. DOI:10.1653/024.102.0202 |
| [4] |
TELLIS M B, KOTKAR H M, JOSHI R S. Regulation of trehalose metabolism in insects: From genes to the metabolite window[J]. Glycobiology, 2023, 33(4): 262-273. DOI:10.1093/glycob/cwad011 |
| [5] |
ARG ELLES J C. Physiological roles of trehalose in bacteria and yeasts: a comparative analysis[J]. Archives of Microbiology, 2000, 174(4): 217-24. DOI:10.1007/s002030000192 |
| [6] |
MERZENDORFER H, ZIMOCH L. Chitin metabolism in insects: structure, function and regulation of chitin synthases and chitinases[J]. Journal of Experimental Biology, 2003, 206: 4393-4412. DOI:10.1242/jeb.00709 |
| [7] |
唐斌, 魏苹, 陈洁, 王世贵, 张文庆. 昆虫海藻糖酶的基因特性及功能研究进展[J]. 昆虫学报, 2012, 55(11): 1315-1321. DOI:10.16380/j.kcxb.2012.11.008 TANG B, WEI P, CHEN J, WANG S G, ZHANG W Q. Progress in gene features and functions of insect trehalases[J]. Acta Entomologica Sinica, 2012, 55(11): 1315-1321. DOI:10.16380/j.kcxb.2012.11.008 |
| [8] |
ZHANG B, ZHANG Y, GUAN R, DU M, YIN X, ZHAO W, AN S. Trehalase is required for sex pheromone biosynthesis in Helicoverpa armigera[J]. Insect Molecular Biology, 2022, 31(3): 334-345. |
| [9] |
TAKIGUCHI M, NIIMI T, SU Z H, YAGINUMA T. Trehalase from male accessory gland of an insect, Tenebrio molitor. cDNA sequencing and developmental profile of the gene expression[J]. Biochemical Journal, 1992, 288: 19-22. DOI:10.1042/bj2880019 |
| [10] |
MITSUMASU K, AZUMA M, NⅡMI T, YAMASHITA O, YAGINUMA T. Membrane-penetrating trehalase from silkworm Bombyx mori. Molecular cloning and localization in larval midgut[J]. Insect Molecular Biology, 2005, 14(5): 501-508. DOI:10.1111/j.1365-2583.2005.00581.x |
| [11] |
SHUKLA E, THORAT L J, NATH B B, GAIKWAD S M. Insect trehalase: Physiological significance and potential applications[J]. Glycobiology, 2015, 25(4): 357-367. DOI:10.1093/glycob/cwu125 |
| [12] |
CHEN J, TANG B, CHEN H, YAO Q, HUANG X, CHEN J, ZHANG D, ZHANG W. Different functions of the insect soluble and membrane-bound trehalase genes in chitin biosynthesis revealed by RNA interference[J]. PLoS One, 2010, 5(4): e10133. DOI:10.1371/journal.pone.0010133 |
| [13] |
SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative C(T) method[J]. Nature Protocols, 2008, 3(6): 1101-1108. DOI:10.1038/nprot.2008.73 |
| [14] |
YAO Q, XU S, DONG Y, LU K, CHEN B. Identification and characterisation of two general odourant-binding proteins from the litchi fruit borer, Conopomorpha sinensis Bradley[J]. Pest Management Science, 2016, 72: 877-887. DOI:10.1002/ps.4062 |
| [15] |
ALLEN M C, KARPLUS P A, MEHL R A, COOLEY R B. Genetic encoding of phosphorylated amino acids into proteins[J]. Chemical Reviews, 2024, 124(10): 6592-6642. DOI:10.1021/acs.chemrev.4c00110 |
| [16] |
SHI Y, WU C, SHI J, GAO T, MA H, LI L, ZHAO Y. Protein phosphorylation and kinases: Potential therapeutic targets in necroptosis[J]. European Journal of Pharmacology, 2024, 970: 176508. DOI:10.1016/j.ejphar.2024.176508 |
| [17] |
BRUNAK S, ENGELBRECHT J. Protein structure and the sequential structure of mRNA: Alpha-helix and beta-sheet signals at the nucleotide level[J]. Proteins, 1996, 25(2): 237-252. DOI:10.1002/SICI1097-0134 |
| [18] |
OKAKPU O K, DILLMAN A R. Review of the role of parasitic nematode excretory/secretory proteins in host immunomodulation[J]. Journal of Parasitology, 2022, 108(2): 199-208. DOI:10.1645/21-33 |
| [19] |
SILVA M C, RIBEIRO A F, TERRA W R, FERREIRA C. Sequencing of Spodoptera frugiperda midgut trehalases and demonstration of secretion of soluble trehalase by midgut columnar cells[J]. Insect Molecular Biology, 2009, 18(6): 769-784. DOI:10.1111/j.1365-2583.2009.00920.x |
| [20] |
SHUKLA E, THORAT L J, NATH B B, GAIKWAD S M. Insect trehalase: Physiological significance and potential applications[J]. Glycobiology, 2015, 25(4): 357-367. DOI:10.1093/glycob/cwu125 |
| [21] |
TATUN N, SINGTRIPOP T, SAKURAI S. Dual control of midgut trehalase activity by 20 -hydroxyecdysone and an inhibitory factor in the bamboo borer Omphisa fuscidentalis Hampson[J]. Journal of Insect Physiology, 2008, 54(2): 351-357. DOI:10.1016/j.jinsphys.2007.10.006 |
| [22] |
CHEN J, TANG B, CHEN H, YAO Q, HUANG X, CHEN J, ZHANG D, ZHANG W. Different functions of the insect soluble and membrane-bound trehalase genes in chitin biosynthesis revealed by RNA interference[J]. PLoS One, 2010, 5(4): e10133. DOI:10.1371/journal.pone.0010133 |
| [23] |
田宇, 杜娟, 李尚伟, 李娇, 王爽. 稻纵卷叶螟海藻糖酶基因的克隆、分子特性和表达分析[J]. 昆虫学报, 2016, 59(6): 602-612. DOI:10.16380/j.kcxb.2016.06.003 TIAN Y, DU J, LI S W, LI J, WANG S. Molecular cloning, characterization and expression analysis of trehalase genes in the rice leaf folder, Cnaphalocrocis medinalis (Lepidoptera: Pyralidae)[J]. Acta Entomologica Sinica, 2016, 59(6): 602-612. DOI:10.16380/j.kcxb.2016.06.003 |
| [24] |
YANG H, WANG Y, ZHANG W, ZHANG X, WANG S, CUI M, ZHAO X, FAN D, DAI C. Knockdown of the expression of two trehalase genes with RNAi disrupts the trehalose and chitin metabolism pathways in the oriental armyworm, Mythimna separata[J]. Insects, 2024, 15(3): 142-150. DOI:10.3390/insects15030142 |
| [25] |
ORTOL B, DAR S J A. RNA Interference in Insects: from a natural mechanism of gene expression regulation to a biotechnological crop protection promise[J]. Biology, 2024, 13(3): 137-144. DOI:10.3390/biology13030137 |
| [26] |
ZHAO L, YANG M, SHEN Q, LIU X, SHI Z, WANG S, TANG B. Functional characterization of three trehalase genes regulating the chitin metabolism pathway in rice brown planthopper using RNA interference[J]. Scientifi c Reports, 2016, 6: 27841. DOI:10.1038/srep27841 |
| [27] |
唐斌, 肖仲久, 曾伯平, 邱玲玉, 潘碧莹, 李昆, 张道伟. 赤拟谷盗TRE基因家族特性及RNAi抑制表达效果分析[J]. 环境昆虫学报, 2019, 41(6): 1311-1320. TANG B, XIAO Z J, ZENG B P, QIU L Y, PAN B Y, LI K, ZHANG D W. Characteristics analysis of TRE gene and RNAi suppression expression effect of Tribolium castaneum[J]. Journal of Environmental Entomology, 2019, 41(6): 1311-1320. |
| [28] |
张道伟, 李燕, 张萌, 王莎莎, 肖仲久, 曾伯平, 倪晓丽, 唐斌. 海藻糖酶基因TRE2-like和TRE2在异色瓢虫羽化过程中的表达与功能[J]. 昆虫学报, 2019, 62(6): 663-671. DOI:10.16380/j.kcxb.2019.06.002 ZHANG D W, LI Y, ZHANG M, WANG S S, XIAO Z J, ZENG B P, NI X L, TANG B. Expression and function of trehalase genes TRE2-like and TRE2 during adult eclosion in Harmonia axyridis (Coleoptera: Coccinellidae)[J]. Acta entomologica sinica, 2019, 62(6): 663-671. DOI:10.16380/j.kcxb.2019.06.002 |
(责任编辑 陈丽娥)
 2024, Vol. 51
2024, Vol. 51