文章信息
基金项目
- 国家自然科学基金(U22A20497,32172564);广东省重点领域研发计划项目(2022B0202080001);广东省自然科学基金(2023A1515012694)
作者简介
-
程蛟文,农学博士,华南农业大学副教授,硕士生导师。主要从事辣椒和苦瓜等蔬菜作物重要农艺性状分子标记开发、形成分子机理以及分子遗传育种相关应用技术研究。主持国家自然科学基金2项、广东省自然科学基金2项、广州市科技计划项目2项,参与国家自然科学基金3项、省市级各类项目多项;以第一作者(含共同)或通信作者(含共同)在《 Plant Journal》《 Theoretical and Applied Genetics》《 Horticultural Plant Journal》和《园艺学报》等期刊发表学术论文20余篇;获授权国家发明专利2件;选育农业农村部登记辣椒新品种2个、广东省审定苦瓜新品种1个。
程蛟文(1987—),男,博士,副教授,研究方向为蔬菜遗传育种与生物技术,E-mail:jiaolong1015@126.com.
通讯作者
- 胡开林(1963—),男,博士,教授,研究方向为蔬菜遗传育种与生物技术,E-mail:hukailin@scau.edu.cn.
文章历史
- 收稿日期:2024-04-19
2. 仲恺农业工程学院园艺园林学院,广东 广州 510225;
3. 遵义职业技术学院现代农业系/贵州省遵义辣椒种质资源保护及繁育种植工程研究中心,贵州 遵义 563006;
4. 广东省农业科学院蔬菜研究所,广东 广州 510640;
5. 广东和利农生物种业股份有限公司,广东 汕头 515800;
6. 佛山大学农业与生物工程学院,广东 佛山 528225
2. College of Horticulture and Landscape Architecture, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;
3. Modern Agriculture Department, Zunyi Vocational and Technical College / Engineering Research Center of Zunyi Pepper Germplasm Resources Conservation and Breeding Cultivation of Guizhou Province, Zunyi 563006, China;
4. Vegetable Research Institute, Guangdong Academy of Agricultural Sciences, Guangzhou 510640, China;
5. Guangdong Helinong Biological Seed Industry Co., Ltd., Shantou 515800, China;
6. School of Agricultural and Biological Engineering, Foshan University, Foshan 528225, China
辣椒(Capsicum spp.)又名番椒、海椒、辣子和辣茄等,属于茄科(Solanaceae)茄亚族(Solaninae Dunal)辣椒属(Capsicum)一年生或多年生植物。化石和考古学证据表明,辣椒起源于中南美洲热带和亚热带地区[1-2],且最早在该地区被人工驯化,距今已有6000多年的栽培历史[3-5]。截至2022年,辣椒属中已发现的辣椒种的数量超过40个,其中绝大多数仍处于野生或半野生状态[6-7]。辣椒属主要栽培种有5个,分别是一年生辣椒(C. annuum L.)、灌木辣椒(C. frutescens L.)、中国辣椒(C. chinense Jacq.)、下垂辣椒(C. baccatum L.)和绒毛辣椒(C. pubescens Ruiz & Pavon),前3个在世界范围内广泛栽培,后2个则主要在中南美洲地区种植[7-8]。
辣椒因其果实含有特有的辣椒素和二氢辣椒素等辣味物质以及丰富的营养物质而闻名,现已成为世界范围内重要蔬菜作物之一。据联合国粮食及农业组织统计,2022年全球辣椒种植面积超过366万hm2,总产量超过4 100万t。辣椒相关产品已被广泛用于各色菜肴烹饪、调味品加工、临床医药、生物防治和军事等领域[9-11]。辣椒大约于明朝末期(16世纪)传入中国[12-13],经过400多年发展,目前已成为我国人民日常食谱的重要蔬菜和调味品,对保障我国蔬菜周年均衡供应、丰富人民生活饮食文化发挥了重要作用[8]。据我国农业农村部大宗蔬菜产业技术体系统计,近年来我国辣椒年播种面积已超过200万hm2,约占我国蔬菜总播种面积的10%,居各类蔬菜作物首位[14]。
基因组(Genome)是生物体所有遗传物质的总和。基因组学(Genomics)是研究与解读生物基因组蕴藏的所有遗传信息的一门学科。辣椒参考基因组草图于2014年首次公布,标志辣椒正式进入基因组学研究时代[15-16]。近10年(2014—2023)来,随着测序技术的快速发展和完善以及测序成本的不断降低,越来越多辣椒材料的全基因组被测序、组装和解读,极大推动了辣椒基因组学相关基础和应用研究。本文主要回顾和总结过去近10年辣椒基因组研究领域取得的进展,并对构建更高质量的辣椒参考基因组和泛基因组以及完善辣椒基因功能研究体系进行展望,以期为辣椒基因组学及其应用研究提供参考。
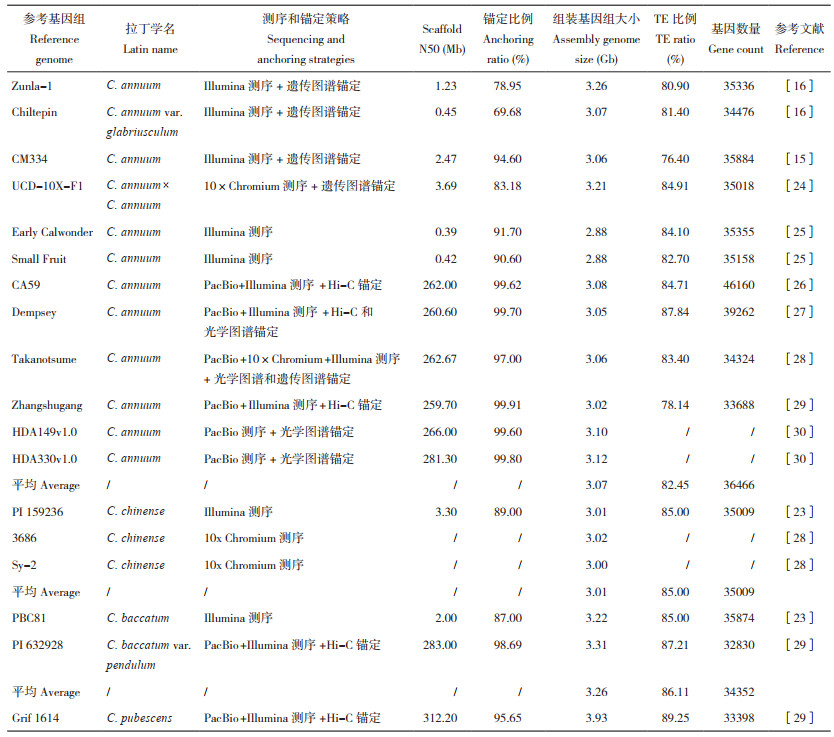
1 辣椒全基因组测序进展 1.1 一年生辣椒栽培种一年生辣椒(C. annuum L.)是世界范围内主要种植的辣椒栽培种。2014年,由四川农业大学、遵义市农业科学研究院、华南农业大学、深圳华大基因股份有限公司和墨西哥CINVESTAV研究所等单位组成的国际研究团队,采用二代Illumina测序和遗传图谱锚定的策略,完成‘遵辣1号’(‘Zunla-1’)和‘Chiltepin’的全基因组测序和组装[16-18]。‘Zunla-1’是遵义市农业科学研究院经多年系统选育而成的常规朝天椒品种[19]。‘Chiltepin’则是从墨西哥中北部Queretaro省采集的一年生辣椒的一个近缘野生变种(C. annuum var. glabriusculum),当地俗称‘Tepín’[20-21]。同年,韩国首尔国立大学等单位采用相同策略,完成‘CM334’的全基因组测序和组装[15]。‘CM334’是墨西哥Morelos省的一个地方品种,其对辣椒疫病、斑驳病毒病和根结线虫等表现出较高抗性[22]。2017年,韩国首尔国立大学研究团队进一步对‘CM334’的基因组组装和注释进行了完善更新[23]。
2018年,美国加州大学研究团队将10× Chromium技术应用于一年生辣椒种内杂交种‘UCD-10X-F1’的全基因组测序,该杂交种以‘CM334’为母本、1个甜椒自交系为父本通过人工杂交获得,最终通过phased组装获得2个单倍型全基因组序列[24]。2021年,韩国首尔国立大学研究团队采用二代Illumina测序和从头组装的方法,分别获得甜椒‘Early Calwonder’和辣椒‘Small Fruit’的全基因组序列[25]。2022年,华南农业大学和美国加州大学研究团队采取三代PacBio + 二代Illumina测序+ 从头组装+ 高通量染色体构象捕获(High-throughput chro-mosome conformation capture,Hi-C)锚定策略,获得‘CA59’的高质量全基因组序列[26]。同年,韩国首尔国立大学研究团队采取三代PacBio + 二代Illumina测序+ 从头组装+Hi-C + 光学图谱锚定策略,获得甜椒‘Dempsey’的高质量全基因组序列[27]。日本Kazusa DNA研究所团队综合运用三代PacBio+10×Chromium+ 二代Illumina测序+ 从头组装+ 光学图谱和遗传图谱锚定手段,获得日本辣椒品种‘Takanotsume’的高质量全基因组序列[28]。2023年,湖南农业大学和美国博伊斯汤普森植物研究所领衔的研究团队采取三代PacBio+ 二代Illumina测序+ 从头组装+Hi-C锚定策略,绘制完成湖南省辣椒地方品种‘樟树港’(‘Zhangshugang’)的高质量全基因组图谱[29]。同年,美国农业部农业研究局和北卡罗来纳州立大学领衔的研究团队以一年生辣椒种内杂交种(‘HDA149’בHDA330’)为材料,利用三代PacBio测序+trio-binning组装+ 光学图谱锚定的策略,获得两个辣椒单倍型全基因组序列[30]。
综上所述,过去近10年前人共计完成11份一年生辣椒材料的全基因组测序和从头组装,获得12个一年生辣椒全基因组序列(表 1)。除上述通过从头组装策略获得全基因组序列外,前人还基于已有的辣椒参考基因组,通过基因组重构(Genome reconstruction)或有参辅助基因组组装(Reference-guided genome assembly)途径,获得一年生辣椒一系列材料的全基因组序列。如2020年,意大利都灵大学研究团队以‘CM334’全基因组序列为参照,通过高深度二代Illumina重测序和基因组重构,获得4个意大利甜椒品种的全基因组序列[31];2022年,韩国首尔国立大学研究团队以甜椒‘Dempsey’全基因组序列为参考,利用10×Chromium和Nanopore技术进行高深度重测序,通过有参辅助组装获得10份一年生辣椒材料的全基因组序列[27]。
1.2 其他辣椒栽培种
除一年生辣椒(C. annuum)外,前人还完成了其他3个辣椒栽培种的全基因组测序。包括2017年,韩国首尔国立大学研究团队利用二代Illumina测序+ 从头组装的方法,获得中国辣椒(C. chinense)材料‘PI159236’和下垂辣椒(C. baccatum)材料‘PBC81’的全基因组序列[23];2022年,日本Kazusa DNA研究所团队利用10×Chromium测序+ 从头组装方法,获得两个中国辣椒(C. chinense)材料‘3686’和‘Sy-2’的全基因组序列[28];2023年,湖南农业大学和美国博伊斯汤普森植物研究所领衔的研究团队采取三代PacBio+ 二代Illumina测序+ 从头组装+Hi-C锚定策略,完成下重辣椒变种(C. baccatum var. pendulum)材料‘PI 632928’和绒毛辣椒(C. pubescens)材料‘Grif 1614’的全基因组测序和组装[29]。至此,对于5个主要辣椒栽培种,除栽培种灌木辣椒(C. frutescens)以外,其余4个均已完成全基因组测序和组装(表 1)。
2 辣椒基因组基本特征 2.1 染色体核型研究发现,辣椒属染色体组成主要有2n=24(x=12)和2n=26(x=13)两种类型。其中,5个主要辣椒栽培种均具有2n=24(x=12)染色体组成,而一些辣椒野生种如C. campylopodium、C. cornutum和C. hunzikerianum等则具有2n=26 (x=13)染色体组成。具有2n=24(x=12)染色体组成的核型更对称,一般由11对中间着丝粒(Metacentric)染色体和1对亚中间着丝粒(Submetacentric)染色体构成,而具有2n=26 (x=13)染色体组成的核型则包含更多对亚中间着丝粒(Submetacentric)染色体,甚至出现1对 端着丝粒(Telocentric)染色体[32]。
近年来,随着越来越多新的辣椒种被鉴别,经统计发现上述两种染色体组成的辣椒种的数量已大致相同[7]。有关上述两种染色体组成的演化关系,由于2n=24(x=12)染色体核型更具对称性,因此大部分人认为2n=26(x=13)染色体组成是由2n=24(x=12)染色体核型衍生而来[32-33]。然而,也有人提出相反观点,认为2n=26(x=13)才是辣椒属的祖先染色体组成,是由于第13对小的染色体丢失才衍生出2n=24(x=12)类型[34]。
2.2 基因组大小基因组大小(Genome size)在辣椒种间和种内均存在差异[35-36]。由表 1可知,一年生辣椒(C. annuum)栽培种基因组大小介于2.88~3.26 Gb,平均为3.07 Gb;3个中国辣椒(C. chinense)和2个下垂辣椒(C. baccatum)材料的平均基因组大小分别为3.01 Gb和3.26 Gb。绒毛辣椒(C. pubescens)材料‘Grif 1614’的基因组是目前已组装辣椒基因组中最大的(3.93 Gb),这与早期通过细胞流式技术测定的基因组含量结果[37-38]基本吻合。在茄科植物中,辣椒基因组显著大于番茄[39]、马铃薯[40]和茄子[41]的基因组,造成这种现象的原因主要是辣椒基因组积累更高比例(均大于70%)的转座元件(Transposable elements,TE,表 1),其中又以LTR(Long terminal repeat)类反转座子贡献最大[15-16, 42]。
2.3 基因数量由表 1可知,一年生辣椒栽培种基因组注释的蛋白编码基因数量最多的是‘CA59’(46 160个),最少的是‘Zhangshugang’(33 688个),但是‘CA59’基因注释的BUSCO(Benchmarking Universal Single-Copy Orthologs)评估完整度(79.4%)低于‘Zhangshugang’(97.7%)、‘CM334’(86.7%)和‘Zunla-1’(84.1%),比‘Dempsey’(77.0%)和‘Chiltein’(78.5%)略高[29]。中国辣椒(C. chinense)材料‘PI 159236’基因组注释的蛋白编码基因数为35 009个。下垂辣椒(C. baccatum)材料‘PBC81’和‘PI 632928’基因组注释的蛋白编码基因数分别为35 874个和32 830个。有意思的是,尽管绒毛辣椒(C. pubescens)材料‘Grif 1614’基因组最大,但其注释的蛋白编码基因数(33 398个)低于一年生辣椒基因组的平均值(36 466个)和中国辣椒(C. chinense)材料‘PI 159236’(35 009个)。
2.4 三维结构特征三维基因组学(Three-dimensional genomics)是基因组三维空间结构与功能研究的简称,是对基因组序列在细胞核内的三维空间构象及其在基因复制、重组、表达等生物过程中发挥的功能进行研究的新兴学科[43]。2022年,华南农业大学研究团队利用Hi-C技术,整合表观组、转录组以及遗传变异数据,首次探索了辣椒三维基因组结构的基本特征及其与基因组功能和进化的关系。结果表明,辣椒基因组虽然具有与哺乳动物类似的拓扑结合域(Topologically associating domains,TADs),但其形成机制与潜在功能并不完全一样。辣椒TADs的边界是染色体重排的热点区域,该区域的序列高度保守,说明其结构完整性对基因组功能有益,受到进化正选择。该研究还证实染色质构象虽然能够广泛预测转录上的量化差异,但并不能直接解释组织间基因差异表达,暗示基因组构象一定程度上是基因组功能的映射。辣椒染色质三维结构主要由转录工厂模型介导的异染色质折叠驱动,且这种结构有潜在功能性,普遍适用于其他大型植物基因组[26]。
3 辣椒泛基因组研究进展泛基因组(Pangenome)是指某一物种的全部基因组信息的集合。相较于单一个体参考基因组,泛基因组可以更全面揭示某一物种的遗传信息,对于挖掘新的功能基因和加深对物种遗传多样性的认识具有重要意义[44-46]。近年来,辣椒在泛基因组构建和应用方面也取得一些进展。
2018年,湖南省农业科学院蔬菜研究所研究团队首先利用包含4个主要辣椒栽培种的383份辣椒种质资源重测序数据进行从头组装,然后以‘Zunla-1’基因组为参考进行比对分析,构建了第一个辣椒泛基因组[47]。该辣椒泛基因组一共注释51 757个高可信度(High quality,HQ)基因,其中包含28 840个核心基因(Core gene,即4个主要栽培种共有)。然后,该研究团队进一步基于基因PAV(Presence–absence variations)进行全基因组关联分析,成功鉴定引起辣椒果色和辣味变化的重要候选基因[47]。
2022年,韩国首尔国立大学研究团队以甜椒‘Dempsey’全基因组序列为参照,将另外11个辣椒材料的全基因组序列与之比对,构建了辣椒首个图形泛基因组[27]。该辣椒图形泛基因组共鉴定42 972个基因家族,包括12个辣椒基因组共有的19 662个核心基因家族以及23 115个非核心基因家族。同时,该团队基于PAV频率分析,鉴定了引起辣椒和甜椒分化的重要变异位点[27]。
2023年,湖南农业大学和美国博伊斯汤普森植物研究所研究团队以‘Zhangshugang’(樟树港)全基因组序列为参考,将下垂辣椒(C. baccatum)‘PI 632928’和绒毛辣椒(C. pubescens)‘Grif 1614’全基因组序列与之比对,构建了1个跨种间的辣椒图形泛基因组。该图形泛基因组揭示的变异信息为进一步研究辣椒属系统发育、群体结构、种群分化以及种群历史等群体遗传学等奠定了重要基础[29]。
4 辣椒基因组学应用 4.1 探究辣椒种间分化和基因渗入对来源于7个不同辣椒种的500份核心种质资源进行重测序和系统发育分析发现,属于Pubescens分支的绒毛辣椒(C. pubescens)最早从辣椒属分化,其次是属于Baccatum分支的C. chacoense、C. baccatum var. baccatum和C. baccatum var. pendulum,最后分化的Annuum分支包含C. galapagoense、灌木辣椒(C. frutescens)、中国辣椒(C. chinense)、C. annuum var. glabriusculum和C. annuum var. annuum[29]。基于水稻、拟南芥、葡萄、番茄、土豆和辣椒6个物种的8个基因组中鉴定的2 540个单拷贝直系同源基因进行系统发育分析,推断绒毛辣椒(C. baccatum)大约在170万年前从辣椒属中分化,一年生辣椒(C. annuum)和中国辣椒(C. chinense)的分化时间则大约在110万年前[23]。
前人还利用固定指数(FST)分析5个驯化辣椒种群(即绒毛辣椒C. pubescens、C. baccatum var. pendulum、灌木辣椒C. frutescens、中国辣椒C. chinense和C. annuum var. Annuum)以及3个野生辣椒种群(即C. chacoense、C. baccatum var. baccatum和C. annuum var. glabriusculum)之间的基因组分化情况,结果表明下垂辣椒(C. baccatum)的两个变种(即C. baccatum var. baccatum和C. baccatum var. Pendulum)之间的分化水平最低(FST = 0.29),所有其他比较组的FST值均大于0.37,表明辣椒种间存在显著的遗传分化[29]。
除分化以外,辣椒不同种群之间也存在基因渗入现象,包括野生辣椒到栽培辣椒以及不同栽培种间的基因渗入。如群体结构分析发现,栽培辣椒品种‘CM334’、‘ZJ14’、‘ZJ19’、‘11c1363’、‘ZJ18’和‘ZJ12’中存在明显来自野生辣椒‘Chiltepin’的基因渗入[16]。此外,通过TreeMix分析也发现中国辣椒(C. chinense)和灌木辣椒(C. frutescens)基因组中存在下垂辣椒(C. baccatum)某些基因的渗入,在这些渗入的基因中,部分可能为这两个辣椒种提供了对各种生物(如病原体、害虫)和非生物(如干旱、盐碱、高温)胁迫的耐受性[29]。
4.2 解析辣椒重要性状进化机制辣味是辣椒特有性状,主要由辣椒素等辣味物质引起。全基因组序列的获得为全面系统鉴定辣椒素生物合成基因奠定了基础[15-16]。系统进化分析表明,辣味物质的生物合成与辣椒特有的基因复制事件,以及这些基因拷贝之间的序列分歧导致新功能分化有关[15-16]。通过对辣椒和番茄之间的比较转录组学分析还发现,某些基因如BCAT(Branched-chain amino acid aminotransferase)、Kas(Ketoacyl-ACP synthase)和CS(Capsaicin synthase)的差异表达也可能有助于辣味物质的合成[15]。此外,还发现辣椒素合成关键基因AT3(Acyltransferase 3,等同于CS)的2个拷贝(即AT3-D2和AT3-D1)的剂量补偿效应可能塑造了辣椒的辣味程度差别[16]。
核苷酸结合和富含亮氨酸重复(Nucleotide- binding and leucine-rich repeat,NLR)基因是植物进化出来的最大的一类抗病基因家族,对于植物的健康和抵御病原体侵害至关重要。相比于番茄和马铃薯,NLR在辣椒中发生了显著扩张[48]。通过基因组分析发现,辣椒属NLR基因的扩张主要是由LTR型反转子介导的反转录复制(Retroduplication)引起[23]。一年生辣椒(C. annuum)种内的遗传变异进一步导致种内NLR基因的多样化[25]。此外,通过比较分析下垂辣椒(C. baccatum)、中国辣椒(C. chinense)和一年生辣椒(C. annuum)3个辣椒栽培种的基因组发现,反转录复制、串联复制(Tandem duplications)和染色体片段复制(Segmental duplications)很可能共同导致下垂辣椒(C. baccatum)抗炭疽病基因的分化[23]。
4.3 阐明辣椒遗传多样性特征通过全基因组测序对来自全球的、代表 14个不同辣椒种(变种)的10 038份种质资源进行基因分型和遗传多样性分析,结果表明,从辣椒种来看,一年生辣椒(C. annuum)具有最丰富的遗传多样性,其次是绒毛辣椒(C. pubescens),中国辣椒(C. chinense)遗传多样性最低[49]。从地理分布来看,中美洲、东亚/ 南亚/ 东南亚和非洲地区的辣椒遗传多样性都较为丰富且在遗传上高度分化,表明上述地区应该都是辣椒多样化中心[49]。
通过对311份来源不同的一年生辣椒(C.annuum)种质资源进行重测序和遗传多样性分析发现,包含早期驯化品种的祖先品种群、古老的农家品种群和后来的栽培品种群均具有较为丰富的遗传多样性,说明一年生辣椒(C. annuum)早期驯化过程中没有出现显著的遗传瓶颈效应,抑或是存在多样化选择。在后期驯化过程中形成的栽培品种群中,包含甜椒在内的块状果型辣椒品种群体遗传多样性最低。来自中国的3个栽培品种群中,高海拔地区(包括云南、贵州、四川和西藏)辣椒种质遗传多样性高于西北/ 华北以及华中地区辣椒种质[50]。
4.4 揭示辣椒人工驯化特征从辣椒栽培种和近缘野生种之间的形态差异来看,辣椒的人工驯化涉及果实的辣味、大小、形状、朝向、脱落,以及株型、种子休眠、抗病和抗逆等诸多性状。尽管C. annuum var. annuum和C. baccatum var. Pendulum的果实大小、形状和辣味等性状均受到人工选择影响,但是全基因组选择分析表明这两个栽培种受选择的基因组区域大部分是不同的,表明C. annuum var. annuum和C. baccatum var. pendulum很可能是独立驯化的[29]。
对从全球范围收集的7 848份一年生辣椒种质资源进行群体结构分析发现,不同地区间的地理差异确实可在一定程度上解释一年生辣椒的遗传多样性。然而,不同地区收集的一年生辣椒之间的遗传重叠比其遗传分离更为显著,如欧洲来源的一年生辣椒样本位于一个渐变群(Cline)的一端,而亚洲来源的一年生辣椒样本则位于另一端,美洲收集的一年生辣椒样本则相当广泛地分布在这个渐变群上。另外,还存在一个主要由中美洲来源的一年生辣椒样本组成的亚群(Deme),在大多数地区均发现位于这个亚群和上述渐变群之间的中间样本。这意味着来自不同地区的辣椒在遗传上并不是完全独立的,而是有一定的相似性。这些结果揭示了一年生辣椒驯化模型的一个整体轮廓,即源自中美洲的具有广泛自然多样性的辣椒被大量驯化,并且在驯化过程中频繁地进行长距离运输和种间杂交,导致现有的辣椒在遗传上的复杂性和多样性[49]。
通过对311份不同来源的一年生辣椒种质资源进行全基因组重测序和系统发育分析,中国农业科学院研究团队提出一年生辣椒果实驯化和多样化的演化模型:早期驯化过程中,对辣椒果实的辣味、形状进行强烈的多样化选择,而果实朝向则受到普遍一致的朝下选择;后期驯化过程中,对辣味的选择在不同品种群中表现出不同趋势,如在农家品种中辣味略微增加,而在部分栽培品种中辣味降低,尤其对pun1的选择和固定导致甜椒类型的形成。但是,来源于中国西北、西南和中部地区的辣椒种质资源辣味又有所增加,这与中国对增加辣椒素水平的二次选择相一致,因为辛辣食物在中国越来越受欢迎。果型方面,窄果型辣椒的早期演化过程涉及对大量果实辣味和形状相关候选基因的选择,块状果实辣椒的形成则可能与两个来源于野生辣椒的基因组片段渗入有关[50]。
4.5 追溯辣椒全球扩张传播历史辣椒起源于中南美洲,作为一种非常受欢迎且具有交易价值的商品,其全球扩张传播深受人类贸易活动和文化交流的影响。历史、考古和民族植物学证据表明,辣椒的全球传播存在多条途径,其中包括美洲的纵向迁移、3条美洲外迁途径(通过西班牙/ 葡萄牙贸易路线直接进入欧亚大陆、通过非洲和印度洋的葡萄牙和阿拉伯贸易路线进入欧亚大陆、通过哥伦布时代前后的跨太平洋贸易路线直接进入南亚/ 东南亚)、欧亚大陆的陆路迁移,以及连接美洲、欧洲和非洲的跨大西洋奴隶贸易等。基于全球范围收集的7 848份一年生辣椒种质资源的全基因组重测序和遗传变异分析,采用ReMIXTURE方法对一年生辣椒的全球传播途径进行系统深入剖析,结果表明,除亚洲至美洲直接的传播路径尚无法证实以外,上述所有已知的传播路径都与各地区所收集辣椒样本间的主要遗传相似性相吻合[49]。
4.6 发掘辣椒重要性状形成基因全基因组序列的获得极大地促进了传统QTL(Quantitative trait locus)定位、全基因组关联分析(Genome-wide association study,GWAS)和混池分离分析结合全基因组重测序(Bulk segregant analysis with whole-genome resequencing,BSA-seq)定位等正向遗传学研究手段在辣椒中的应用。结合基于重组体分析的精细定位以及病毒诱导基因沉默(Virus-Induced Gene Silencing,VIGS)功能验证等,前人已经成功鉴定辣椒许多重要性状形成关键或候选基因,如始花节位基因[51];质核互作雄性不育(Cytoplasmic male sterility,CMS)主效恢复基因[52]、微效恢复基因Rf2[53]、不稳定恢复基因Rfu[54];细胞核雄性不育基因msc-1[55]、ms1[56]、msc-3[57];花粉育性降低基因rpf1[58];果实脱落基因S[59];果实朝向基因up[60];果实形状基因fs10[61];果实颜色基因CaPP2C35[62]、CaAN3[63]、ly[64];果实辣味程度基因[65];抗疫病基因[66];抗CMV(Cucumber mosaic virus)基因[67];抗炭疽病基因[68];抗蓟马基因[69]等,为上述辣椒性状的分子标记辅助选择及形成的分子机理解析奠定了重要基础。
5 结语与展望近10年来,辣椒基因组学研究取得显著进展。多个研究团队采用不同的测序和组装策略,完成4个主要辣椒栽培种包括一年生辣椒(C. annuum)、中国辣椒(C. chinense)、下垂辣椒(C. baccatum)和绒毛辣椒(C. pubescens)的全基因组测序。通过对辣椒基因组的深入研究,系统揭示了其染色体核型、基因组大小、基因数量以及三维基因组结构等基本特征。通过综合利用不同辣椒的基因组信息,初步构建了辣椒泛基因组。辣椒基因组学应用方面也已经取得显著进展,探究了辣椒种间分化和基因渗入、解析了辣椒重要性状进化机制、阐明了辣椒遗传多样性特征、揭示了辣椒人工驯化特征、追溯了辣椒全球扩张传播历史、以全基因组序列为基础采用多种正向遗传学手段成功发掘了辣椒诸多重要性状形成的关键或候选基因。
随着测序技术的不断进步和测序成本的降低,可以预见未来将有更多的辣椒栽培种和野生种被测序,这将进一步丰富辣椒的基因组资源。同时,我们也期待将来能够获得更高准确性、连续性和完整性的如T2T(Telomere-to-Telomere)水平的辣椒参考基因组,以更全面准确地探究辣椒种间分化和基因渗入特征、重要性状进化机制、遗传多样性和人工驯化特征以及全球扩张传播历史。此外,通过整合转录组、蛋白质组、代谢组和表型组等多组学数据,搭建完善的基因功能研究平台,如VIGS、转基因、基因编辑等技术体系,我们将能够更深入地阐释辣椒基因组的功能与调控机制,进而指导更精准和高效地对辣椒进行遗传改良,创制出更多具有优良性状和广适性的辣椒新品种,满足不同地区和消费者的多样化需求。最后,作为重要的蔬菜与调味品,辣椒基因组学研究还将有助于我们深入探索其营养成分与生物活性物质的合成途径及调控机制,为辣椒的功能性开发与利用开辟新的思路与方向。
| [1] |
KRAFT K H, BROWN C H, NABHAN G P, LUEDELING E, LUNA RUIZ J D J, COPPENS D'EECKENBRUGGE G, HIJMANS R J, GEPTS P. Multiple lines of evidence for the origin of domesticated chili pepper, Capsicum annuum, in Mexico[J]. Proceedings of the National Academy of Sciences of the United Staes of America, 2014, 111(17): 6165-6170. DOI:10.1073/pnas.1308933111 |
| [2] |
MONGKOLPORN O, TAYLOR P J. Capsicum. In: KOLE C. Wild crop relatives: Genomic and breeding resources[M]. Springer Berlin Heidelberg, 2011: 43-57.
|
| [3] |
CARRIZO G C, BARFUSS M H, SEHR E M, BARBOZA G E, SAMUEL R, MOSCONE E A, EHRENDORFER F. Phylogenetic relationships, diversification and expansion of chili peppers (Capsicum, Solanaceae)[J]. Annals of Botany, 2016, 118(1): 35-51. DOI:10.1093/aob/mcw079 |
| [4] |
PERRY L, DICKAU R, ZARRILLO S, HOLST I, PEARSALL D M, PIPERNO D R, BERMAN M J, COOKE R G, RADEMAKER K, RANERE A J, RAYMOND J S, SANDWEISS D H, SCARAMELLI F, TARBLE K, ZEIDLER J A. Starch fossils and the domestication and dispersal of chili peppers (Capsicum spp. L.) in the Americas[J]. Science, 2007, 315(5814): 986-988. DOI:10.1126/science.1136914 |
| [5] |
PICKERSGILL B. Domestication of plants in the Americas: Insights from Mendelian and molecular genetics[J]. Annals of Botany, 2007, 100(5): 925-940. DOI:10.1093/aob/mcm193 |
| [6] |
BARBOZA G E, GARCíA C C, SCALDAFERRO M, BOHS L. An amazing new Capsicum (Solanaceae) species from the Andean-Amazonian Piedmont[J]. PhytoKeys, 2020, 167: 13-29. DOI:10.3897/phytokeys.167.57751 |
| [7] |
BARBOZA G E, CAROLINA CARRIZO G, LUCIANO DE BEM B, ROMERO M V, SCALDAFERRO M. Monograph of wild and cultivated chili peppers (Capsicum L., Solanaceae)[J]. PhytoKeys, 2022, 200: 1-423. DOI:10.3897/phytokeys.200.71667 |
| [8] |
邹学校, 马艳青, 戴雄泽, 李雪峰, 杨莎. 辣椒在中国的传播与产业发展[J]. 园艺学报, 2020, 47(9): 1715-1726. DOI:10.16420/j.issn.0513-353x.2020-0103 ZOU X X, MA Y Q, DAI X Z, LI X F, YANG S. Spread and industry development of pepper in China[J]. Acta Horticulturae Sinica, 2020, 47(9): 1715-1726. DOI:10.16420/j.issn.0513-353x.2020-0103 |
| [9] |
BAENAS N, BELOVIC M, ILIC N, MORENO D A, GARCIA-VIGUERA C. Industrial use of pepper (Capsicum annum L.) derived products: Technological benefits and biological advantages[J]. Food Chemistry, 2019, 274: 872-885. DOI:10.1016/j.foodchem.2018.09.047 |
| [10] |
ZOU Z, ZOU X. Geographical and ecological differences in pepper cultivation and consumption in China[J]. Frontiers in Nutrition, 2021, 8: 718517. DOI:10.3389/fnut.2021.718517 |
| [11] |
KHAN F A, MAHMOOD T, ALI M, SAEED A, MAALIK A. Pharmacological importance of an ethnobotanical plant: Capsicum annuum L.[J]. Natural Product Research, 2014, 28(16): 1267-1274. DOI:10.1080/14786419.2014.895723 |
| [12] |
覃成, 程蛟文, 吴智明, 罗希榕, 胡开林. 辣椒基因组学的研究进展[J]. 农业生物技术学报, 2015, 23(7): 953-966. DOI:10.3969/j.issn.1674-7968 QIN C, CHENG J W, WU Z M, LUO X R, HU K L. Research advances in the genomics of pepper (Capsicum spp.)[J]. Journal of Agricultural Biotechnology, 2015, 23(7): 953-966. DOI:10.3969/j.issn.1674-7968 |
| [13] |
蒋慕东, 王思明. 辣椒在中国的传播及其影响[J]. 中国农史, 2005(2): 17-27. JIANG M D, WANG S M. The spread of pepper and its influence in China[J]. Agricultural History of China, 2005(2): 17-27. |
| [14] |
王立浩, 张宝玺, 张正海, 曹亚从, 于海龙, 冯锡刚. "十三五"我国辣椒育种研究进展、产业现状及展望[J]. 中国蔬菜, 2021(2): 21-29. DOI:10.19928/j.cnki.1000-6346.2021.0004 WANG L H, ZHANG B X, ZHANG Z H, CAO Y C, YU H L, FENG X G. Status in breeding and production of Capsicum spp. in China during 'The Thirteenth Five-year Plan' Period and future prospect[J]. China Vegetables, 2021(2): 21-29. DOI:10.19928/j.cnki.1000-6346.2021.0004 |
| [15] |
KIM S, PARK M, YEOM S I, KIM Y M, LEE J M, LEE H A, SEO E, CHOI J, CHEONG K, KIM K T, JUNG K, LEE G W, OH S K, BAE C, KIM S B, LEE H Y, KIM S Y, KIM M S, KANG B C, JO Y D, YANG H B, JEONG H J, KANG W H, KWON J K, SHIN C, LIM J Y, PARK J H, HUH J H, KIM J S, KIM B D, COHEN O, PARAN I, SUH M C, LEE S B, KIM Y K, SHIN Y, NOH S J, PARK J, SEO Y S, KWON S Y, KIM H A, PARK J M, KIM H J, CHOI S B, BOSLAND P W, REEVES G, JO S H, LEE B W, CHO H T, CHOI H S, LEE M S, YU Y, DO CHOI Y, PARK B S, VAN DEYNZE A, ASHRAFI H, HILL T, KIM W T, PAI H S, AHN H K, YEAM I, GIOVANNONI J J, ROSE J K, SORENSEN I, LEE S J, KIM R W, CHOI I Y, CHOI B S, LIM J S, LEE Y H, CHOI D. Genome sequence of the hot pepper provides insights into the evolution of pungency in Capsicum species[J]. Nature Genetics, 2014, 46(3): 270-278. DOI:10.1038/ng.2877 |
| [16] |
QIN C, YU C, SHEN Y, FANG X, CHEN L, MIN J, CHENG J, ZHAO S, XU M, LUO Y, YANG Y, WU Z, MAO L, WU H, LING-HU C, ZHOU H, LIN H, GONZALEZ-MORALES S, TREJO-SAAVEDRA D L, TIAN H, TANG X, ZHAO M, HUANG Z, ZHOU A, YAO X, CUI J, LI W, CHEN Z, FENG Y, NIU Y, BI S, YANG X, LI W, CAI H, LUO X, MONTES-HERNANDEZ S, LEYVA-GONZALEZ M A, XIONG Z, HE X, BAI L, TAN S, TANG X, LIU D, LIU J, ZHANG S, CHEN M, ZHANG L, ZHA ANG Y, YANG S, WANG X, XU J, LI X, LI S, WANG J, PALLOIX A, BOSLAND P W, LI Y, KROGH A, RIVERA-BUSTAMANTE R F, HERRERA-ESTRELLA L, YIN Y, YU J, HU K, ZHANG Z. Whole-genome sequencing of cultivated and wild peppers provides insights into Capsicum domestication and specialization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(14): 5135-5140. DOI:10.1073/pnas.1400975111 |
| [17] |
CHENG J, CHEN Y, HU Y, ZHOU Z, HU F, DONG J, CHEN W, CUI J, WU Z, HU K. Fine mapping of restorer-of-fertility gene based on high-density genetic mapping and collinearity analysis in pepper (Capsicum annuum L.)[J]. Theoretical and Applied Genetics, 2020, 133(3): 889-902. DOI:10.1007/s00122-019-03513-y |
| [18] |
CHENG J, QIN C, TANG X, ZHOU H, HU Y, ZHAO Z, CUI J, LI B, WU Z, YU J, HU K. Development of a SNP array and its application to genetic mapping and diversity assessment in pepper (Capsicum spp.)[J]. Scientific Reports, 2016, 6: 33293. DOI:10.1038/srep33293 |
| [19] |
余常水, 令狐昌英, 李岸桥, 唐相群, 龙昌伟, 杨秀伟. 遵辣1号新品种的选育[J]. 种子, 2009, 28(12): 100-101. DOI:10.16590/j.cnki.1001-4705.2009.12.070 YU C S, LIN HU C Y, LI A Q, TANG X Q, LONG C W, YANG X W. Breeding of 'Zunla No. 1' hot pepper[J]. Seed, 2009, 28(12): 100-101. DOI:10.16590/j.cnki.1001-4705.2009.12.070 |
| [20] |
GONZALEZ-JARA P, MORENO-LETELIER A, FRAILE A, PINERO D, GARCIA-ARENAL F. Impact of human management on the genetic variation of wild pepper, Capsicum annuum var. glabriusculum[J]. PLoS One, 2011, 6(12): e28715. DOI:10.1371/journal.pone.0028715 |
| [21] |
AGUILAR-MELÉNDEZ A, MORRELL P L, ROOSE M L, KIM S-C. Genetic diversity and structure in semiwild and domesticated chiles (Capsicum annuum; Solanaceae) from Mexico[J]. American Journal of Botany, 2009, 96(6): 1190-1202. DOI:10.3732/ajb.0800155 |
| [22] |
PEGARD A, BRIZZARD G, FAZARI A, SOUCAZE O, ABAD P, DJIAN-CAPORALINO C. Histological characterization of resistance to different root-knot nematode species related to phenolics accumulation in Capsicum annuum[J]. Phytopathology, 2005, 95(2): 158-165. DOI:10.1094/phyto-95-0158 |
| [23] |
KIM S, PARK J, YEOM S I, KIM Y M, SEO E, KIM K T, KIM M S, LEE J M, CHEONG K, SHIN H S, KIM S B, HAN K, LEE J, PARK M, LEE H A, LEE H Y, LEE Y, OH S, LEE J H, CHOI E, CHOI E, LEE S E, JEON J, KIM H, CHOI G, SONG H, LEE J, LEE S C, KWON J K, LEE H Y, KOO N, HONG Y, KIM R W, KANG W H, HUH J H, KANG B C, YANG T J, LEE Y H, BENNETZEN J L, CHOI D. New reference genome sequences of hot pepper reveal the massive evolution of plant disease-resistance genes by retroduplication[J]. Genome Biology, 2017, 18(1): 210. DOI:10.1186/s13059-017-1341-9 |
| [24] |
HULSE-KEMP A M, MAHESHWARI S, STOFFEL K, HILL T A, JAFFE D, WILLIAMS S R, WEISENFELD N, RAMAKRISHNAN S, KUMAR V, SHAH P, SCHATZ M C, CHURCH D M, VAN DEYNZE A. Reference quality assembly of the 3.5-Gb genome of Capsicum annuum from a single linked-read library[J]. Horticulture Research, 2018, 5(1): 4. DOI:10.1038/s41438-017-0011-0 |
| [25] |
KIM M S, CHAE G Y, OH S, KIM J, MANG H, KIM S, CHOI D. Comparative analysis of de novo genomes reveals dynamic intraspecies divergence of NLRs in pepper[J]. BMC Plant Biology, 2021, 21(1): 247. DOI:10.1186/s12870-021-03057-8 |
| [26] |
LIAO Y, WANG J, ZHU Z, LIU Y, CHEN J, ZHOU Y, LIU F, LEI J, GAUT B S, CAO B, EMERSON J J, CHEN C. The 3D architecture of the pepper genome and its relationship to function and evolution[J]. Nature Communications, 2022, 13(1): 3479. DOI:10.1038/s41467-022-31112-x |
| [27] |
LEE J H, VENKATESH J, JO J, JANG S, KIM G W, KIM J M, HAN K, RO N, LEE H Y, KWON J K, KIM Y M, LEE T H, CHOI D, VAN DEYNZE A, HILL T, KFIR N, FREIMAN A, DAVILA OLIVAS N H, ELKIND Y, PARAN I, KANG B C. High-quality chromosome-scale genomes facilitate effective identification of large structural variations in hot and sweet peppers[J]. Horticulture Research, 2022, 9: uhac210. DOI:10.1093/hr/uhac210 |
| [28] |
SHIRASAWA K, HOSOKAWA M, YASUI Y, TOYODA A, ISOBE S. Chromosome-scale genome assembly of a Japanese chili pepper landrace, Capsicum annuum 'Takanotsume'[J]. DNA Research, 2022, 30(1): dsac052. DOI:10.1093/dnares/dsac052 |
| [29] |
LIU F, ZHAO J, SUN H, XIONG C, SUN X, WANG X, WANG Z, JARRET R, WANG J, TANG B, XU H, HU B, SUO H, YANG B, OU L, LI X, ZHOU S, YANG S, LIU Z, YUAN F, PEI Z, MA Y, DAI X, WU S, FEI Z, ZOU X. Genomes of cultivated and wild Capsicum species provide insights into pepper domestication and population differentiation[J]. Nature Communications, 2023, 14(1): 5487. DOI:10.1038/s41467-023-41251-4 |
| [30] |
DELOREAN E E, YOUNGBLOOD R C, SIMPSON S A, SCHOONMAKER A N, SCHEFFLER B E, RUTTER W B, HULSEKEMP A M. Representing true plant genomes: Haplotype-resolved hybrid pepper genome with trio-binning[J]. Frontiers in Plant Science, 2023, 14: 1184112. DOI:10.3389/fpls.2023.1184112 |
| [31] |
ACQUADRO A, BARCHI L, PORTIS E, NOURDINE M, CARLI C, MONGE S, VALENTINO D, LANTERI S. Whole genome resequencing of four Italian sweet pepper landraces provides insights on sequence variation in genes of agronomic value[J]. Scientific Reports, 2020, 10(1): 9189. DOI:10.1038/s41598-020-66053-2 |
| [32] |
MOSCONE E A, SCALDAFERRO M A, GRABIELE M, CECCHINI N M, SÃNCHEZ GARCȊA Y, JARRET R, DAVIÑA J R, DUCASSE D A, BARBOZA G E, EHRENDORFER F. The evolution of chili peppers (Capsicum- Solanaceae): A cytogenetic perspective[C]. 745 edn. International Society for Horticultural Science (ISHS), Leuven, Belgium, 2007: 137-170.
|
| [33] |
SCALDAFERRO M A, DA CRUZ M V R, CECCHINI N M, MOSCONE E A. FISH and AgNor mapping of the 45S and 5S rRNA genes in wild and cultivated species of Capsicum (Solananceae)[J]. Genome, 2015, 59(2): 95-113. DOI:10.1139/gen-2015-0099 |
| [34] |
POZZOBON M T, SCHIFINO-WITTMANN M T, DE BEM BIANCHETTI L. Chromosome numbers in wild and semidomesticated Brazilian Capsicum L. (Solanaceae) species: do x= 12 and x= 13 represent two evolutionary lines?[J]. Botanical Journal of the Linnean Society, 2006, 151(2): 259-269. DOI:10.1111/j.1095-8339.2006.00503.x |
| [35] |
程蛟文, 吴智明, 崔竣杰, 李卫鹏, 谭澍, 胡开林. 主要蔬菜作物基因组含量统计与比较分析[J]. 园艺学报, 2013, 40(1): 135-144. DOI:10.16420/j.issn.0513-353x.2013.01.017 CHENG J W, WU Z M, CUI J J, LI W P, TAN S, HU K L. Statistical and comparative analysis of vegetable genome size[J]. Acta Horticulturae Sinica, 2013, 40(1): 135-144. DOI:10.16420/j.issn.0513-353x.2013.01.017 |
| [36] |
SCALDAFERRO M A, MOSCONE E A. Cytology and DNA content variation of Capsicum genomes. In: Ramchiary N, Kole C (eds) The Capsicum genome[M]. Springer International Publishing, 2019: 57-84.
|
| [37] |
BELLETTI P, MARZACHì C, LANTERI S. Flow cytometric measurement of nuclear DNA content in Capsicum (Solanaceae)[J]. Plant Systematics and Evolution, 1998, 209(1/2): 85-91. |
| [38] |
MOSCONE E A, BARANYI M, EBERT I, GREILHUBER J, EHRENDORFER F, HUNZIKER A T. Analysis of nuclear DNA content in Capsicum (Solanaceae) by flow cytometry and Feulgen densitometry[J]. Annals of Botany, 2003, 92(1): 21-29. DOI:10.1093/aob/mcg105 |
| [39] |
CONSORTIUM T T G. The tomato genome sequence provides insights into fleshy fruit evolution[J]. Nature, 2012, 485(7400): 635-641. DOI:10.1038/nature11119 |
| [40] |
XU X, PAN S, CHENG S, ZHANG B, MU D, NI P, ZHANG G, YANG S, LI R, WANG J, ORJEDA G, GUZMAN F, TORRES M, LOZANO R, PONCE O, MARTINEZ D, DE LA CRUZ G, CHAKRABARTI S K, PATIL V U, SKRYABIN K G, KUZNETSOV B B, RAVIN N V, KOLGANOVA T V, BELETSKY A V, MARDANOV A V, DI GENOVA A, BOLSER D M, MARTIN D M, LI G, YANG Y, KUANG H, HU Q, XIONG X, BISHOP G J, SAGREDO B, MEJIA N, ZAGORSKI W, GROMADKA R, GAWOR J, SZCZESNY P, HUANG S, ZHANG Z, LIANG C, HE J, LI Y, HE Y, XU J, ZHANG Y, XIE B, DU Y, QU D, BONIERBALE M, GHISLAIN M, HERRERA MDEL R, GIULIANO G, PIETRELLA M, PERROTTA G, FACELLA P, O'BRIEN K, FEINGOLD S E, BARREIRO L E, MASSA G A, DIAMBRA L, WHITTY B R, VAILLANCOURT B, LIN H, MASSA A N, GEOFFROY M, LUNDBACK S, DELLAPENNA D, BUELL C R, SHARMA S K, MARSHALL D F, WAUGH R, BRYAN G J, DESTEFANIS M, NAGY I, MILBOURNE D, THOMSON S J, FIERS M, JACOBS J M, NIELSEN K L, SONDERKAER M, IOVENE M, TORRES G A, JIANG J, VEILLEUX R E, BACHEM C W, DE BOER J, BORM T, KLOOSTERMAN B, VAN ECK H, DATEMA E, HEKKERT B, GOVERSE A, VAN HAM R C, VISSER R G. Genome sequence and analysis of the tuber crop potato[J]. Nature, 2011, 475(7355): 189-195. DOI:10.1038/nature10158 |
| [41] |
WEI Q, WANG J, WANG W, HU T, HU H, BAO C. A high-quality chromosome-level genome assembly reveals genetics for important traits in eggplant[J]. Horticulture Research, 2020, 7(1): 153. DOI:10.1038/s41438-020-00391-0 |
| [42] |
PARK M, PARK J, KIM S, KWON J K, PARK H M, BAE I H, YANG T J, LEE Y H, KANG B C, CHOI D. Evolution of the large genome in Capsicum annuum occurred through accumulation of single‐type long terminal repeat retrotransposons and their derivatives[J]. The Plant Journal, 2012, 69(6): 1018-1029. DOI:10.1111/j.1365-313X.2011.04851.x |
| [43] |
张富涵, 沈宗毅, 喻长远, 杨昭. 三维基因组学研究进展[J]. 生物工程学报, 2020, 36(12): 2791-2812. DOI:10.13345/j.cjb.200197 ZHANG F H, SHEN Z Y, YU C Y, YANG Z. Advances in three-dimensional genomics[J]. Chinese Journal of Biotechnology, 2020, 36(12): 2791-2812. DOI:10.13345/j.cjb.200197 |
| [44] |
MORGANTE M, DE PAOLI E, RADOVIC S. Transposable elements and the plant pan-genomes[J]. Current Opinion in Plant Biology, 2007, 10(2): 149-155. DOI:10.1016/j.pbi.2007.02.001 |
| [45] |
SHI J, TIAN Z, LAI J, HUANG X. Plant pan-genomics and its applications[J]. Molecular Plant, 2023, 16(1): 168-186. DOI:10.1016/j.molp.2022.12.009 |
| [46] |
江彪, 闫晋强, 晏石娟, 谢大森, 刘文睿, 王敏. 葫芦科作物基因组学研究进展[J]. 广东农业科学, 2023, 50(4): 1-12. DOI:10.16768/j.issn.1004-874X.2023.04.001 JIANG B, YAN J Q, YAN S J, XIE D S, LIU W R, WANG M. Research progresses on the genomics of Cucurbitaceae crops[J]. Guangdong Agricultural Sciences, 2023, 50(4): 1-12. DOI:10.16768/j.issn.1004-874X.2023.04.001 |
| [47] |
OU L, LI D, LYU J, CHEN W, ZHANG Z, LI X, YANG B, ZHOU S, YANG S, LI W, GAO H, ZENG Q, YU H, OUYANG B, LI F, LIU F, ZHENG J, LIU Y, WANG J, WANG B, DAI X, MA Y, ZOU X. Pan-genome of cultivated pepper (Capsicum) and its use in gene presence-absence variation analyses[J]. New Phytologist, 2018, 220(2): 360-363. DOI:10.1111/nph.15413 |
| [48] |
SEO E, KIM S, YEOM S I, CHOI D. Genome-wide comparative analyses reveal the dynamic evolution of nucleotide-binding leucine-rich repeat gene family among Solanaceae plants[J]. Frontiers in Plant Science, 2016, 7. DOI:10.3389/fpls.2016.01205 |
| [49] |
TRIPODI P, RABANUS-WALLACE M T, BARCHI L, KALE S, ESPOSITO S, ACQUADRO A, SCHAFLEITNER R, VAN ZONNEVELD M, PROHENS J, DIEZ M J, BöRNER A, SALINIER J, CAROMEL B, BOVY A, BOYACI F, PASEV G, BRANDT R, HIMMELBACH A, PORTIS E, FINKERS R, LANTERI S, PARAN I, LEFEBVRE V, GIULIANO G, STEIN N. Global range expansion history of pepper (Capsicum spp.) revealed by over 10, 000 genebank accessions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(34). DOI:10.1073/pnas.2104315118 |
| [50] |
CAO Y, ZHANG K, YU H, CHEN S, XU D, ZHAO H, ZHANG Z, YANG Y, GU X, LIU X, WANG H, JING Y, MEI Y, WANG X, LEFEBVRE V, ZHANG W, JIN Y, AN D, WANG R, BOSLAND P, LI X, PARAN I, ZHANG B, GIULIANO G, WANG L, CHENG F. Pepper variome reveals the history and key loci associated with fruit domestication and diversification[J]. Molecular Plant, 2022, 15(11): 1744-1758. DOI:10.1016/j.molp.2022.09.021 |
| [51] |
TAN S, CHENG J W, ZHANG L, QIN C, NONG D G, LI W P, TANG X, WU Z M, HU K L. Construction of an interspecific genetic map based on InDel and SSR for mapping the QTLs affecting the initiation of flower primordia in pepper (Capsicum spp.)[J]. PLoS One, 2015, 10(3). DOI:10.1371/journal.pone.0119389 |
| [52] |
ZHANG Z, ZHU Y, CAO Y, YU H, BAI R, ZHAO H, ZHANG B, WANG L. Fine mapping of the male fertility restoration gene CaRf032 in Capsicum annuum L.[J]. Theoretical and Applied Genetics, 2020, 133(4): 1177-1187. DOI:10.1007/s00122-020-03540-0 |
| [53] |
ZHANG Z, AN D, YU H, SUN L, CAO Y, ZHANG B, WANG L. Fine mapping of Rf2, a minor Restorer-of-fertility (Rf) gene for cytoplasmic male sterility in chili pepper G164 (Capsicum annuum L.)[J]. Theoretical and Applied Genetics, 2022, 135(8): 2699-2709. DOI:10.1007/s00122-022-04143-7 |
| [54] |
KANG M C, KANG H J, JUNG S Y, LEE H Y, KANG M Y, JO Y D, KANG B C. The Unstable Restorer-of-fertility locus in pepper (Capsicum annuum L.) is delimited to a genomic region containing PPR genes[J]. Theoretical and Applied Genetics, 2022, 135(6): 1923-1937. DOI:10.1007/s00122-022-04084-1 |
| [55] |
CHENG Q, WANG P, LIU J, WU L, ZHANG Z, LI T, GAO W, YANG W, SUN L, SHEN H. Identification of candidate genes underlying genic male-sterile msc-1 locus via genome resequencing in Capsicum annuum L.[J]. Theoretical and Applied Genetics, 2018, 131(9): 1861-1872. DOI:10.1007/s00122-018-3119-1 |
| [56] |
CHENG Q, LI T, AI Y, LU Q, WANG Y, WU L, LIU J, SUN L, SHEN H. Phenotypic, genetic, and molecular function of msc-2, a genic male sterile mutant in pepper (Capsicum annuum L.)[J]. Theoretical and Applied Genetics, 2020, 133(3): 843-855. DOI:10.1007/s00122-019-03510-1 |
| [57] |
DONG J, HU F, GUAN W, YUAN F, LAI Z, ZHONG J, LIU J, WU Z, CHENG J, HU K. A 163-bp insertion in the Capana10g000198 encoding a MYB transcription factor causes male sterility in pepper (Capsicum annuum L.)[J]. The Plant Journal, 2023, 113(3): 521-535. DOI:10.1111/tpj.16064 |
| [58] |
ZHANG Z, LIU Y, YUAN Q, XIONG C, XU H, HU B, SUO H, YANG S, HOU X, YUAN F, PEI Z, DAI X, ZOU X, LIU F. The bHLH1-DTX35/DFR module regulates pollen fertility by promoting flavonoid biosynthesis in Capsicum annuum L.[J]. Horticulture Research, 2022, 9: uhac172. DOI:10.1093/hr/uhac172 |
| [59] |
HU F, DONG J, ZHANG S, SONG Z, GUAN W, YUAN F, ZHONG J, LIU J, HU K, CHENG J. Fine mapping and gene silencing pinpoint Capana10g002229 as a strong candidate gene regulating the deciduous character of ripe pepper fruit (Capsicum spp.)[J]. Theoretical and Applied Genetics, 2023, 136(5): 107. DOI:10.1007/s00122-023-04355-5 |
| [60] |
HU F, CHENG J, DONG J, ZHONG J, ZHOU Z, HU K. Fine mapping and candidate gene analysis of the up locus determining fruit orientation in pepper (Capsicum spp.)[J]. Theoretical and Applied Genetics, 2021, 134(9): 2901-2911. DOI:10.1007/s00122-021-03867-2 |
| [61] |
BOROVSKY Y, RAZ A, DORON-FAIGENBOIM A, ZEMACH H, KARAVANI E, PARAN I. Pepper fruit elongation is controlled by Capsicum annuum Ovate Family Protein 20[J]. Frontiers in Plant Science, 2022, 12: 815589. DOI:10.3389/fpls.2021.815589 |
| [62] |
WU L, WANG H, LIU S, LIU M, LIU J, WANG Y, SUN L, YANG W, SHEN H. Mapping of CaPP2C35 involved in the formation of light- green immature pepper (Capsicum annuum L.) fruits via GWAS and BSA[J]. Theoretical and Applied Genetics, 2021, 135(2): 591-604. DOI:10.1007/s00122-021-03987-9 |
| [63] |
BYUN J, KIM T G, LEE J H, LI N, JUNG S, KANG B C. Identification of CaAN3 as a fruit-specific regulator of anthocyanin biosynthesis in pepper (Capsicum annuum)[J]. Theoretical and Applied Genetics, 2022, 135(7): 2197-2211. DOI:10.1007/s00122-022-04106-y |
| [64] |
SONG Z, ZHONG J, DONG J C, HU F, ZHANG B G, CHENG J W, HU K L. Mapping immature fruit colour-related genes via bulked segregant analysis combined with whole-genome re-sequencing in pepper (Capsicum annuum)[J]. Plant Breeding, 2022, 141(2): 277-285. DOI:10.1111/pbr.12997 |
| [65] |
ZHU Z, SUN B, CAI W, ZHOU X, MAO Y, CHEN C, WEI J, CAO B, CHEN C, CHEN G, LEI J. Natural variations in the MYB transcription factor MYB31 determine the evolution of extremely pungent peppers[J]. New Phytologist, 2019, 223(2): 922-938. DOI:10.1111/nph.15853 |
| [66] |
SIDDIQUE M I, LEE H Y, RO N Y, HAN K, VENKATESH J, SOLOMON A M, PATIL A S U, CHANGKWIAN A, KWON J K, KANG B C. Identifying candidate genes for Phytophthora capsici resistance in pepper (Capsicum annuum) via genotyping-by-sequencing-based QTL mapping and genome-wide association study[J]. Scientific Reports, 2019, 9(1): 9962. DOI:10.1038/s41598-019-46342-1 |
| [67] |
GUO G, WANG S, LIU J, PAN B, DIAO W, GE W, GAO C, SNYDER J C. Rapid identification of QTLs underlying resistance to cucumber mosaic virus in pepper (Capsicum frutescens)[J]. Theoretical and Applied Genetics, 2016, 130(1): 41-52. DOI:10.1007/s00122-016-2790-3 |
| [68] |
RO N, HAILE M, HUR O, KO H C, YI J Y, WOO H J, CHOI Y M, RHEE J, LEE Y J, KIM D A, DO J W, KIM G W, KWON J K, KANG B C. Genome-wide association study of resistance to anthracnose in pepper (Capsicum chinense) germplasm[J]. BMC Plant Biology, 2023, 23(1): 389. DOI:10.1186/s12870-023-04388-4 |
| [69] |
VAN HAPEREN P, VOORRIPS R E, VAN KAAUWEN M, VAN EEKELEN H, DE VOS R C H, VAN LOON J J A, VOSMAN B. Fine mapping of a thrips resistance QTL in Capsicum and the role of diterpene glycosides in the underlying mechanism[J]. Theoretical and Applied Genetics, 2021, 134(5): 1557-1573. DOI:10.1007/s00122-021-03790-6 |
(责任编辑 张辉玲)
 2024, Vol. 51
2024, Vol. 51