文章信息
基金项目
- 广东省国际科技合作项目(2021A0505030047);广东省农业农村厅科技专项(2020KJ136-02);广东省农业科学院农业优势产业学科团队建设项目(202104TD); 财政部和农业农村部:国家现代农业产业技术体系项目(CARS-13)
作者简介
-
王润风(1995—),女,博士,助理研究员,研究方向为花生栽培育种,E-mail:wangrunfeng@gdaas.cn
王润风,博士,助理研究员。2015年6月毕业于浙江大学农学系,获农学学士学位。2020年12月毕业于浙江大学农业与生物技术学院作物所,获农学博士学位。2021年6月入职广东省农业科学院作物研究所花生研究室,主要从事花生栽培育种研究。发表SCI论文5篇,其中以第一作者发表SCI论文2篇; 李少雄,三级研究员,1993年7月毕业于华南农业大学植物遗传育种专业,国家花生产业技术体系广州综合试验站站长,现从事花生遗传育种工作。参加工作以来,承担国家和省科技项目多项,先后培育出9个花生品种通过国家品种审(鉴)定,13个品种通过省级品种审定,10个品种获植物新品种保护权,这些品种在产量、品质和抗病性方面均有显著的提高,且多个品种被列为广东省和其他华南各省(区)主导品种和最受欢迎的花生品种,并在我国南方花生产区大面积推广应用。先后获省(部)级科学技术奖励10项,广东省农业技术推广奖6项。获授权国家发明专利4项,植物新品种保护权10个。在国内外学术刊物发表学术论文50多篇.
通讯作者
- 李少雄(1970—),男,研究员,研究方向为花生遗传育种,E-mail:lishaoxiong@gdaas.cn.
文章历史
- 收稿日期:2021-10-29
2. 云浮市农业科学及技术推广中心,广东 云浮 527300
2. Yunfu Agricultural Sciences and Technology Extension Center, Yunfu 527300, China
【研究意义】花生作为我国的主要油料作物之一,年种植面积多达466.7万hm2,占世界花生面积的20%,居第2位。在国内主要油料作物中,花生的栽培面积仅次于油菜,占油料作物面积的25%[1]。我国花生产区大体可分为北方产区和南方产区,不同产区在气候条件、土壤类型、栽培制度和花生品种方面存在很大的差异。南方花生产区年播种面积达186万hm2,占全国的40% 左右,在我国花生生产中占有重要地位[1]。因此,对南方产区花生育成品种进行遗传多样性分析,了解现有品种的遗传基础,对于培育适宜南方产区的花生品种至关重要。【前人研究进展】近几十年来,随着科技水平的提高,我国花生生产发展迅速,其中高产花生品种的培育及推广发挥了重要作用。然而,我国花生育种在品质、抗病及抗逆性上与国际先进水平仍有较大差距,近几年在产量上有较大突破的品种也较少。造成这一现象的原因一方面在于我国花生主要以常规杂交育种为主,育种周期长,对产量、抗病、抗逆、品质等复杂农艺性状的遗传改良慢,且育成品种遗传基础相对单一;另一方面则在于当前我国花生基础研究相对滞后,特别是分子生物学研究上与其他主要作物的育种研究相比严重滞后。近年来,主要农作物分子育种研究进展迅速,并在遗传连锁图谱构建、重要农艺性状基因定位与克隆、标记辅助选择等方面的研究取得较大进展。但在花生育种研究方面,花生重要农艺性状相关基因的定位克隆以及标记辅助选择等相关的研究仍相对贫乏,致使目前花生遗传育种存在很大的盲目性和不可预测性,造成育种效率低、周期长,对病虫害或逆境因子的抵抗力脆弱,产量潜力不大。适宜的分子标记是鉴定花生品种及辅助选择育种的重要前提,SSR标记在花生栽培种间存在相对丰富的多态性[2-5],成为当前检测栽培种分子多态性的主要标记之一[6-7],并已成功应用于我国多个省份育成的花生品种的遗传多态性分析及遗传图谱构建[8-10],但目前对于南方产区花生品种的遗传多样性分析少有报道。【本研究切入点】本研究通过100对SSR引物分析了54份中国南方花生区试品种的遗传多样性,并进行聚类分析,同时通过品种系谱分析其中21份南方花生区试品种的亲本来源。【拟解决的关键问题】本研究利用SSR标记对54份我国南方区试花生品种进行遗传多样性分析,旨在揭示我国南方花生品种遗传多样性,明确品种间的亲缘关系,为今后花生品种的遗传改良提供分子依据。
1 材料与方法 1.1 试验材料1.1.1 植物材料 供试材料为1996年以来参加南方区域试验的花生品种54份,由广东省农业科学院作物研究所提供,详见表 1。选取饱满有活力的花生种子播种于穴盘中,待种子发芽后取花生幼苗期叶片(10~14 d)存于-80℃备用。
1.1.2 生化试剂 供试SSR引物序列共100对(表 2),引物由上海英骏生物科技有限公司合成,Taq酶及其他常规试剂分别购自大连宝生物和北京鼎国生物技术有限责任公司。
1.2 试验方法
1.2.1 花生叶片DNA提取 花生叶片DNA提取方法参照卢圣栋[11]的方法并略作简化改进,提取的DNA于-20 ℃保存备用。
1.2.2 PCR扩增 PCR反应在PE9700热循环仪(美国ABI公司)上进行。PCR反应体系(20 μL):1 × PCR buffer(50 mmol/L KCl、10 mmol/L Tris-HCl,pH 8.3,1.5 mmol/L MgCl2,0.1% 明胶),1 U Taq DNA聚合酶,50~100 ng模板DNA,0.15 μmol/L引物,0.2 mmol/L dNTP。反应程序为94 ℃ 5 min → 94 ℃ 1 min + 55 ℃ 30 s + 72 ℃ 1 min(35个循环)→ 72 ℃ 5 min。扩增产物在6.0% 非变性聚丙烯酰胺凝胶中电泳(1×TBE,75 V,2.5 h),0.1% AgNO3染色10~15 min,之后于BIORAD凝胶成像系统下观察记录、照相。本实验方法经3次重复操作,结果稳定。
1.2.3 数据分析 统计SSR电泳图谱上有差异且易于识别的多态性条带,在相同迁移率的位置上,有带记为“1”,无带记为“0”,组成1和0的原始矩阵。采用NTSYS-pc 2.10软件进行聚类分析。对原始矩阵用SimQual程序求Dice相似系数,并获得相似系数矩阵。用SHAN程序中的UPGMA(Unweighted pair-group method using arithmeticaverages)方法进行聚类分析,并通过Treeplot模块生成聚类图。根据供试品种的育种单位,采用Weir[12]的方法按下列公式计算标记位点的多态性信息量(Polymorphism information content,PIC):
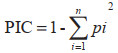
|
式中,n为该位点的等位基因数,pi为第i个等位基因在群体中的频率。
2 结果与分析 2.1 扩增产物的多态性100对SSR引物在54个中国南方区试花生品种中进行扩增,其中有47对引物检测出多态性、占总引物的47%。47对多态性引物在54个品种中共检测出等位基因138个,每对引物检测出2~7个、平均2.93个。每对多态性引物在供试品种中的多态性信息量(PIC)为0.067~0.764、平均为0.336(图 1、表 3)。进一步分析福建、广东、广西三省花生区试品种的SSR多态性表明,供试引物分别有37、39和34对在福建、广东、广西区试品种中检查出多态性,共检测出的等位基因数为85、91、76个,平均为2.26、2.33、2.24个,多态性信息量为0.315、0.388、0.326。

|
| 图 1 SSR标记Ah1TC3G05在14个花生区试品种的PCR电泳结果 Fig. 1 PCR electrophoretogram of SSR marker Ah1TC3G05 in 14 peanut varieties for regional tests |

|
SSR多态性分析结果表明,南方区试花生品种总体上遗传多样性较低、品种间遗传差异小。另外,福建、广东、广西省内育种单位育成品种间的遗传多态性相近,存在遗传基础狭窄的缺点。
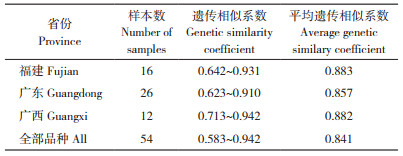
2.2 类型间的聚类分析根据47对多态性引物扩增的条带,利用NTSYS2.10分析软件计算供试花生品种间的遗传相似系数,并进行遗传聚类分析。由表 4可知,54份供试花生品种间的遗传相似系数介于0.642~0.960之间,平均为0.841,其中汕头市农业科学研究所育成花生品种间的遗传相似系数介于0.692~0.960之间,平均为0.883;广东省农业科学院作物研究所育成花生品种间的遗传相似系数变化范围为0.673~0.960,平均为0.857。t测验显示,不同育种单位育成的花生品种间遗传相似系数不存在显著差异,表明南方区试品种的遗传背景相似。
|
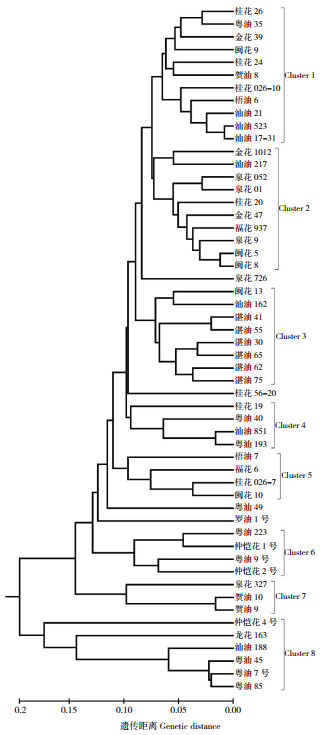
将47对多态性引物在供试品种中的扩增带型数字化后聚类分析,结果(图 2)显示,47对引物能够完全将54个品种区分开。在遗传距离为0.2处,54个品种中有50个被聚为7个类群。其中,第1类群包括5个广西品种(桂花26、桂花24、贺油8号、桂花026-10、梧油6号)、4个广东品种(粤油35、汕油21、汕油523、汕油71-31)和2个福建品种(金花39、闽花9号);第2类群包括8个福建品种(金花1012、泉花052、泉花01、金花47、福花937、泉花9、闽花5、闽花8)、1个广东品种(汕油217)和1个广西品种(桂花20);第3类群包括7个广东品种(汕油162、湛油41、湛油55、湛油30、湛油65、湛油62、湛油75)和1个福建品种(闽花13);第4类群包括3个广东品种(粤油40、汕油851、粤油193)和1个广西品种(桂花19);第5类群包括2个广西品种(梧油7号、桂花026-7)和2个福建品种(福花6、闽花10);第6类群包括4个广东品种(粤油223、仲恺花1号、粤油9号、仲恺花2号);第7类群包括2个广西品种(贺油10号、贺油9号)和1个福建品种(泉花327);第8类群包括5个广东品种(仲恺花4号、汕油188、粤油45、粤油7号、粤油85)和1个福建品种(龙花163)。
|
| 图 2 54个花生栽培品种的SSR聚类树状图 Fig. 2 Clustering dendrogram of 54 cultivated peanut varieties based on SSR markers |
统计结果显示,在第2类群的10个品种聚集了8个福建省品种;第3类群的8个品种聚集了7个广东省品种,其中6个为湛油系列;广东省品种还聚集在第4、第6和第8类群中。根据上述结果可知,尽管福建、广东、广西3个省份的花生品种在聚类时存在交叉现象,但同一省份的品种相对聚类在一起,亲缘关系相对较近,遗传基础相似。
2.3 系谱分析根据中国花生品种及其系谱数据库[13]、《中国花生品种及其系谱》[1]中的花生系谱相关信息,确认其亲本来源,绘制了其中21个南方区试花生品种的系谱图。从系谱图(图 3)可以发现,福建、广东、广西3个省份的花生品种亲本来源是相互交叉的。系谱分析结果发现,在已确认亲本来源的21个花生品种中,有20个花生品种的祖先亲本为伏花生和狮头企,其中以伏花生和狮头企为主培育出的粤油551、粤油551-116、汕油27及在此基础上培育出的汕油71、汕油523是南方培育花生栽培品种常用的骨干亲本,大量重复利用这些品种作为亲本可能是导致南方栽培花生品种遗传多样性低的原因之一。
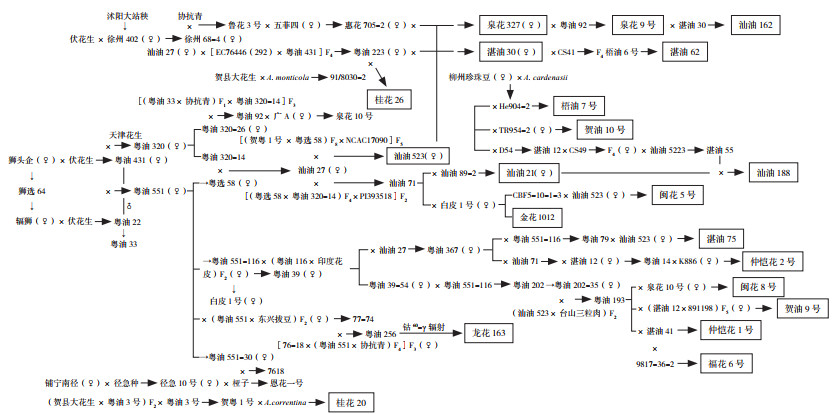
|
| 图 3 南方21个花生栽培品种系谱图 Fig. 3 Pedigree chart of 21 cultivated peanut varieties in southern China |
3 讨论
随着现代农业种业的发展,遗传丰富的农家品种被育种家们选育出的优质抗病品种取代,作物品种趋于单调,显著降低了作物的遗传多样性。作物遗传多样性的降低意味着作物基因资源的单一化,导致作物对环境的适应性降低,增加了作物在生产上的风险,不利于农业的可持续发展。为此,明确作物品种间的亲缘关系并对作物品种进行遗传多样性的鉴定及分析至关重要。
目前,SSR标记已广泛应用于作物遗传多样性的鉴定分析。李清等[14]通过30对SSR引物分析了96份广东省大豆种质资源,鉴定了其资源多样性,并成功构建了96份大豆资源的DNA身份证。本研究中同样发现47对SSR引物可以对54个南方区试花生品种进行精确的特异性鉴定,且鉴定结果与品种来源存在一定相关性,表明今后在进行品种鉴定、种质资源筛选等研究时可以考虑将SSR标记作为花生栽培品种分类的辅助手段。
本研究采用100对SSR引物扩增54个花生栽培品种,结果有47% 的引物能够在栽培品种间检测出多态性,平均检测出的等位基因数为2.93个、平均PIC值仅0.336,表明南方区试花生品种间遗传差异小、遗传基础薄弱。洪彦彬等[4]利用110对SSR引物检测了28个花生地方品种,平均检测出的等位基因数为3.76个、平均PIC值为0.43。由此可见,南方区试花生品种间的遗传多态性低于地方品种。花生品种的遗传相似系数及系谱分析结果同样表明,南方区试花生品种的遗传背景相似。
我国南方花生育成品种的遗传多态性低与育种亲本的选择有很大关系。虽然我国目前收集的花生种质资源已超过8 000份,但对花生种质资源的利用率不足,多数育成品种为狮头企和伏花生的衍生品种[1, 15]。结合广东、广西、福建3省的花生育种工作及品种推广情况发现,福建省的花生品种主要以引进的广东品种为基础亲本通过系统选育、杂交等方式选育得到[1, 16-17];广西审定花生品种主要以广西的中间材料和广东的育成品种为直接亲本通过系统选种、杂交等方式选育得到,其中花生栽培种和野生种种间杂交育种是广西育成花生新品种的重要途径[1, 18];广东省主要通过系统选育、杂交育种、辐射诱变等方法先后选育出系列高产抗病的花生品种[1, 19]。其中,以狮头企和伏花生为主选育出的粤油551和粤油116(即粤油551-116)及后续选育出的汕油523、汕油71和粤油223在南方各省大面积推广应用,并成为南方花生产区的骨干亲本[1, 19]。这些情况与系谱分析的结果基本一致,也在一定程度上解释了聚类分析的结果。综上可知,南方花生现有骨干亲本遗传基础狭窄,进而导致南方花生育成品种遗传多态性低。
花生育成品种遗传基础的狭窄引起了花生科研工作者的重视。早在1985年,于善新等[20]就山东省花生种植情况指出了品种单一化带来的弊端,并提出花生品种应当多样化。陈静等[21]和高斌等[22]通过SSR标记对北方花生育成品种进行分析发现,我国北方花生骨干亲本利用情况相似度较高、遗传基础狭窄。遗传基础的狭窄给花生生产带来了风险,严重限制了花生育种工作的发展。
对国内外花生种质资源的鉴定及遗传多样性的研究,为花生品种的遗传改良提供了分子依据。任小平等[23]通过SSR标记发现ICRISAT花生微核心种质资源具有丰富的遗传多样性。姜慧芳等[24]通过SSR标记鉴定表明中国花生种质资源与ICRISAT资源存在较大差异,且我国花生种质资源的遗传多样性比ICRISAT资源丰富。因此,今后在品种选育过程中,应充分利用国内外的花生种质资源,培育新遗传背景下的骨干亲本,拓宽花生育成品种的遗传基础,增加花生品种的遗传多样性。
4 结论本研究对来自广东、广西、福建3个省份的54份花生品种进行了SSR分子标记鉴定及聚类分析,结果表明3个省份的花生品种遗传多样性较低。系谱分析发现,广东、广西、福建3个省份的花生品种使用的骨干亲本多为狮头企衍生品种。南方花生现有骨干亲本遗传基础狭窄,进而导致南方花生育成品种遗传多态性低,限制了花生育种工作的发展。在未来的花生品种选育过程中,需重视花生分子育种的基础研究,挖掘并鉴定适宜的分子标记,同时合理利用国内外花生资源,培育新遗传背景下的花生骨干亲本,才能为花生育种工作带来新的突破。
| [1] |
禹山林. 中国花生品种及其系谱[M]. 上海: 上海科学技术出版社, 1999. YU S L. Chinese peanut varieties and their genealogy[M]. Shanghai: Shanghai Science and Technology Press, 1999. |
| [2] |
王玉龙, 黄冰艳, 王思雨, 杜培, 齐飞艳, 房元瑾, 孙子淇, 郑峥, 董文召, 张新友. 四倍体野生种花生A. monticola全基因组SSR的开发与特征分析[J]. 中国农业科学, 2019, 52(15): 2567-2585. DOI:10.3864/j.issn.0578-1752.2019.15.002 WANG Y L, HUANG B Y, WANG S Y, DU P, QI F Y, FANG Y J, SUN Z Q, ZHENG Z, DONG W Z, ZHANG X Y. Development and characterization of whole genome SSR in tetraploid wild peanut(Arachis monticola)[J]. Scientia Agricultura Sinica, 2019, 52(15): 2567-2585. DOI:10.3864/j.issn.0578-1752.2019.15.002 |
| [3] |
徐志军, 赵胜, 徐磊, 胡小文, 安东升, 刘洋. 基于RNA-seq数据的栽培种花生SSR位点鉴定和标记开发[J]. 中国农业科学, 2020, 53(4): 695-706. DOI:10.3864/j.issn.0578-1752.2020.04.003 XU Z J, ZHAO S, XU L, HU X W, AN D S, LIU Y. Discovery of microsatellite markers from RNA-seq data in cultivated peanut (Arachis hypogaea)[J]. Scientia Agricultura Sinica, 2020, 53(4): 695-706. DOI:10.3864/j.issn.0578-1752.2020.04.003 |
| [4] |
洪彦彬, 梁炫强, 陈小平, 林坤耀, 周桂元, 李少雄, 刘海燕. 花生栽培种(Arachis hypogaea L.)类型间遗传差异的SSR分析[J]. 分子植物育种, 2008, 6(1): 71-78. DOI:10.3969/j.issn.1672-416X.2008.01.011 HONG Y B, LIANG X Q, CHEN X P, LIN K Y, ZHOU G Y, LI S X, LIU H Y. Genetic differences in peanut cultivated types(Arachis hypogaea L.)revealed by SSR polymorphism[J]. Molecular Plant Breeding, 2008, 6(1): 71-78. DOI:10.3969/j.issn.1672-416X.2008.01.011 |
| [5] |
刘洪, 徐振江, 饶得花, 鲁清, 李少雄, 刘海燕, 陈小平, 梁炫强, 洪彦彬. 基于形态学性状和SSR标记的花生品种遗传多样性分析和特异性鉴定[J]. 作物学报, 2019, 45(1): 26-36. DOI:10.3724/SP.J.1006.2019.84060 LIU H, XU Z J, RAO D H, LU Q, LI S X, LIU H Y, CHEN X P, LIANG X Q, HONG Y B. Genetic diversity analysis and distinctness identification of peanut cultivars based on morphological traits and SSR markers[J]. Acta Agronomica Sinica, 2019, 45(1): 26-36. DOI:10.3724/SP.J.1006.2019.84060 |
| [6] |
LU Q, HONG Y B, LI S X, LIU H, LI H F, ZHANG J N, LAN H F, LIU H Y, LI X Y, WEN S J. Genome-wide identification of microsatellite markers from cultivated peanut(Arachis hypogaea L.)[J]. BMC Genomics, 2019, 20(1): 799-807. DOI:10.1186/s12864-019-6148-5 |
| [7] |
MA J J, ZHAO Y H, CHEN H, FU C, ZHU L, ZHOU X M, XIA H, HOU L, LI G H, ZHUANG W J, WANG X J, ZHAO C Z. Genome-wide development of polymorphic microsatellite markers and their application in peanut breeding program[J]. Electronic Journal of Biotechnology, 2020, 44: 25-32. DOI:10.1016/j.ejbt.2020.01.004 |
| [8] |
白冬梅, 薛云云, 张鑫, 王鹏冬, 黄莉, 田跃霞, 姜慧芳. 基于SSR标记分析山西地方花生种质遗传多样性[J]. 中国油料作物学报, 2020, 42(5): 753-759. DOI:10.19802/j.issn.1007-9084.2020067 BAI D M, XUE Y Y, ZHANG X, WANG P D, HUANG L, TIAN Y X, JIANG H F. Genetic diversity of peanut local germplasm resources in Shanxi province based on SSR markers[J]. Chinese Journal of Oil Crop Sciences, 2020, 42(5): 753-759. DOI:10.19802/j.issn.1007-9084.2020067 |
| [9] |
侯睿, 张小军, 岳福良, 张小红, 李爽, 刘行, 张相琼, 李文均. 基于SSR的四川花生遗传多样性分析[J]. 中国农学通报, 2019, 35(25): 43-53. DOI:10.11924/j.issn.1000-6850.casb18030136 HOU R, ZHANG X J, YUE F L, ZHANG X H, LI S, LIU X, ZHANG X Q, LI W J. Genetic diversity analysis of peanut in Sichuan based on SSR markers[J]. Chinese Agricultural Science Bulletin, 2019, 35(25): 43-53. DOI:10.11924/j.issn.1000-6850.casb18030136 |
| [10] |
郭敏杰, 殷君华, 邓丽, 苗建利, 李阳, 芦振华, 房元瑾, 李绍伟, 任丽. 开农系列高油酸花生遗传多样性分析[J]. 花生学报, 2020, 49(4): 14-22. DOI:10.14001/j.issn.1002-4093.2020.04.003 GUO M J, YIN J H, DENG L, MIAO J L, LI Y, LU Z H, FANG Y J, LI S W, REN L. Genetic diversity analysis on Kainong series cultivars of high oleic acid peanut(Arachis hypogaea L.)[J]. Journal of Peanut Science, 2020, 49(4): 14-22. DOI:10.14001/j.issn.1002-4093.2020.04.003 |
| [11] |
卢圣栋. 现代分子生物学实验技术[M]. 北京: 中国协和医科大学出版社, 1999: 101-136. LU S D. Experimental technologies in modern molecular biology[M]. Beijing: Peking Union Medical College Press, 1999: 101-136. |
| [12] |
WEIR B S. Genetic data analysis: Methods for discrete genetic data[M]. Sunderland: Sinauer Associates, 1990.
|
| [13] |
国家花生数据中心. 中国花生品种及其系谱数据库[EB/OL]. (2017-01-31)[2021-10-19]. http://www.peanutdata.cn/variety/. National Peanut Data Center. Database of Chinese Peanut varieties and their genealogy[EB/OL]. (2017-01-31)[2021-10-19]. http://www.peanutdata.cn/variety/. |
| [14] |
李清, 罗永坚, 吴柔贤, 贾俊婷, 张文虎, 宋松泉, 刘军. 广东省大豆种质资源遗传多样性分析及DNA分子身份证构建[J]. 广东农业科学, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 LI Q, LUO Y J, WU R X, JIA J T, ZHANG W H, SONG S Q, LIU J. Analysis on genetic diversity and construction of DNA molecular identity card of soybean germplasm resources in Guangdong province[J]. Guangdong Agricultural Sciences, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 |
| [15] |
周小静, 任小平, 黄莉, 罗怀勇, 陈玉宁, 刘念, 陈伟刚, 廖伯寿, 雷永, 姜慧芳. 花生种质资源研究进展与展望[J]. 植物遗传资源学报, 2020, 21(1): 33-39. DOI:10.13430/j.cnki.jpgr.20191028002 ZHOU X J, REN X P, HUANG L, LUO H Y, CHEN Y N, LIU N, CHEN W G, LIAO B S, LEI Y, JIANG H F. Research progress and prospect for peanut germplasm resources[J]. Journal of Plant Genetic Resources, 2020, 21(1): 33-39. DOI:10.13430/j.cnki.jpgr.20191028002 |
| [16] |
陈永水, 陈剑洪, 郭陞垚, 肖宇, 陈茹艳, 王金线. 福建省审(认)定花生品种系谱及主要性状遗传改良分析[J]. 中国农学通报, 2014, 30(18): 136-144. CHEN Y S, CHEN J H, GUO S Y, XIAO Y, CHEN R Y, WANG J X. Analysis of pedigree and main traits improvement of peanut cultivars registered in Fujian[J]. Chinese Agricultural Science Bulletin, 2014, 30(18): 136-144. |
| [17] |
黄金堂, 陈海玲, 郑国栋, 李清华, 邱国清, 李淑萍, 谢志琼. 福建花生品种系谱及其性状演变分析[J]. 中国农学通报, 2012, 28(27): 31-36. DOI:10.3969/j.issn.1000-6850.2012.27.007 HUANG J T, CHEN H L, ZHENG G D, LI Q H, QIU G Q, LI S P, XIE Z Q. Analysis of pedigree and evolution of traits for peanut varieties registered in Fujian[J]. Chinese Agricultural Science Bulletin, 2012, 28(27): 31-36. DOI:10.3969/j.issn.1000-6850.2012.27.007 |
| [18] |
林秀芳, 叶万余, 吴春玲, 刘海东, 徐小媛, 陈灿. 广西审定花生品种系谱及农艺性状演变[J]. 中国油料作物学报, 2020, 42(5): 760-766. DOI:10.19802/j.issn.1007-9084.2019202 LIN X F, YE W Y, WU C L, LIU H D, XU X Y, CHEN C. Evolution of agronomic traits and pedigree for authorized peanut cultivars by Guangxi[J]. Chinese Journal of Oil Crop Sciences, 2020, 42(5): 760-766. DOI:10.19802/j.issn.1007-9084.2019202 |
| [19] |
黎穗临. 狮头企亲缘花生品种系谱分析[J]. 花生科技, 2000(4): 5-10. DOI:10.14001/j.issn.1002-4093.2000.04.002 LI S L. Pedigree analysis of peanut varieties related to Shitouqi[J]. Peanut Science and Technology, 2000(4): 5-10. DOI:10.14001/j.issn.1002-4093.2000.04.002 |
| [20] |
于善新, 孙中瑞. 从山东省当前种植的花生品种看品种单一化的弊端[J]. 中国油料, 1985(1): 6-9. YU S X, SUN Z R. The disadvantages of peanut variety simplification in Shandong province[J]. Chinese Oil, 1985(1): 6-9. |
| [21] |
陈静, 胡晓辉, 苗华荣, 石运庆, 禹山林. SSR标记分析国家北方花生区试品种的遗传多样性[J]. 植物遗传资源学报, 2009, 10(3): 360-366. DOI:10.13430/j.cnki.jpgr.2009.03.004 CHEN J, HU X H, MIAO H R, SHI Y Q, YU S L. Analysis of genetic diversity of peanut(Arachis hypogaea L.)varieties under the region tests of north China based on SSR marker[J]. Journal of Plant Genetic Resources, 2009, 10(3): 360-366. DOI:10.13430/j.cnki.jpgr.2009.03.004 |
| [22] |
高斌, 李洪珍, 崔顺立, 郭丽果, 陈焕英, 穆国俊, 杨鑫雷, 刘立峰. 北方花生育成品种(系)分子标记鉴定及系谱分析[J]. 植物遗传资源学报, 2019, 20(6): 1472-1485. DOI:10.13430/j.cnki.jpgr.20181218003 GAO B, LI H Z, CUI S L, GUO L G, CHEN H Y, MU G J, YANG X L, LIU L F. Molecular marker identification and pedigree analysis for peanut cultivars(lines)in northern China[J]. Journal of Plant Genetic Resources, 2019, 20(6): 1472-1485. DOI:10.13430/j.cnki.jpgr.20181218003 |
| [23] |
任小平, 张晓杰, 廖伯寿, 雷永, 黄家权, 陈玉宁, 姜慧芳. ICRISAT花生微核心种质资源SSR标记遗传多样性分析[J]. 中国农业科学, 2010, 43(14): 2848-2858. DOI:10.3864/j.issn.0578-1752.2010.14.003 REN X P, ZHANG X J, LIAO B S, LEI Y, HUANG J Q, CHEN Y N, JIANG H F. Analysis of genetic diversity in ICRISAT mini core collection of peanut(Arachis hypogaea L.)by SSR markers[J]. Scientia Agricultura Sinica, 2010, 43(14): 2848-2858. DOI:10.3864/j.issn.0578-1752.2010.14.003 |
| [24] |
姜慧芳, 任小平, 张晓杰, 黄家权, 雷永, 晏立英, 廖伯寿, HARI D U, CORLEY C H. 中国花生小核心种质与ICRISAT微核心种质的SSR遗传多样性比较[J]. 作物学报, 2010, 36(7): 1084-1091. DOI:10.3724/SP.J.1006.2010.01084 JIANG H F, REN X P, ZHANG X J, HUANG J Q, LEI Y, YAN L Y, LIAO B S, UPADHYAYA H D, HOLBROOK C C. Comparison of genetic diversity between peanut mini core collections from China and ICRISAT by SSR markers[J]. Acta Agronomica Sinica, 2010, 36(7): 1084-1091. DOI:10.3724/SP.J.1006.2010.01084 |
(责任编辑 张辉玲)
 2021, Vol. 48
2021, Vol. 48