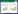文章信息
基金项目
- 财政部和农业农村部:国家现代农业产业技术体系项目(CARS-13);广州市科技计划项目(201904010190);广东省农业科学院农业优势产业学科团队建设项目(202104TD);广东省农业科学院中青年学科带头人培养项目(R2020PY-JX004)
作者简介
- 邓权清(1992—),男,博士,研究方向为花生油脂合成生理,E-mail:dqq648841569@163.com; 陈小平,博士,三级研究员,广东省农业科学院“十四五”农业优势产业杰出团队学科带头人,现任广东省农业科学院作物研究所所长。主要从事花生基因组学与遗传育种研究,与印度国际半干旱热带作物研究所(ICRISAT)、美国佐治亚大学等研究机构合作开展花生野生种和栽培种全基因组测序工作。迄今已在《美国国家科学院院刊》(PNAS)、Molecular Plant和Plant Biotechnology Journal等国内外学术刊物发表论文70余篇,其中SCI收录40多篇。先后承担国家自然科学基金、广东省重点研发计划、广东省自然科学基金、广东省国际合作项目等国家和省级以上项目20余项。通过国家和省审(鉴)定或登记的花生新品种13个,获植物新品种权14个,获国家发明专利6项,获省(部)级科技奖励3项.
通讯作者
- 陈小平(1976—),男,博士,研究员,研究方向为花生基因组学,E-mail:chenxiaoping@gdaas.cn.
文章历史
- 收稿日期:2021-10-29
【研究意义】花生(Arachis hypogaea L.)是一种世界性的油料和蛋白质作物,也是我国四大油料作物之一,其单产和总产长期居油料作物之首[1]。近年来,我国花生食品加工发展迅速,除了传统食用植物油和副食品外,还开发出极具竞争优势的花生营养健康产品[2]。油酸是单不饱和脂肪酸,有较高的氧化稳定性,能有效预防心血管疾病[2-3]。高油酸花生(油酸含量>50%)突出的保健作用与经济效益,使得选育高油酸花生品种成为国内花生品质改良育种的目标之一[3]。与其他植物油相比,高油酸花生油具有货架期长、不饱和脂肪酸含量高(如油酸与亚油酸含量占油脂80%)、无豆腥和不含植物芥酸等特点,越发受到花生榨油企业与消费者青睐[4]。脂质作为花生油脂的主要成分,其代谢物的转化、合成与降解过程能够动态地影响着花生油脂的品质[5]。然而,由于脂质种类多(其中主要成分为甘油三酯混合物),分子结构复杂,现阶段鲜有对花生脂质准确定性定量的研究[6-7]。因此,探索一种能甄别品种与鉴评品质的检测方法,不仅能提高花生育种效率,还能为食品安全提供有效分析手段。
【前人研究进展】花生栽培种属于异源四倍体豆科作物,与其他模式作物相比,花生的基础研究相对薄弱[8]。花生品种鉴定主要通过形态特征和表型观察,该法可靠性差、难以批量化操作[9]。随着分子标记辅助选择育种技术在作物育种中普遍应用,花生种质也开发出大量的分子标记,如SSR、AFLP、RAPD和RFLP等[10-13],但由于这些分子标记特异性与重复性欠佳,导致分析结果可能有所差异。此外,植物油脂的质量鉴定主要通过检测脂肪酸的含量与成分,即脂肪酸分析水平[7]。近年来,油脂研究人员对脂质分子结构与组成的研究愈发深入,直接推动植物油脂检测提升至脂质分子层面[6-7]。
【本研究切入点】脂质组学是通过采用超高效液相色谱- 串联质谱(Ultra performance liquid chromatography tandem mass spectrometry,UPLC-MS/MS)技术对所有内源性高丰度的脂质分子进行定性定量分析,从而更直观地了解生物体内各种脂质代谢与合成的机理[6]。而应用近红外光谱分析技术(Near infrared spectroscopy,NIRS)能够准确快速、批量化与标准化地测定这些花生品质指标[4]。本研究所利用得广泛靶向脂质代谢组技术综合传统靶向与非靶向代谢组检测方法,通过检测平台的自建数据库MWDB(Metware database)及代谢物信息公共数据库,与多元统计分析相结合的手段研究样品间的脂质组差异[14-16]。同时,将脂质与蛋白质含量作为考察花生品质的主要指标[2]。
【拟解决的关键问题】本研究通过利用广泛靶向脂质代谢组分析技术获得高油酸花生与普通花生的核心差异脂质代谢物,结合近红外标准化测定的品质指标,运用多元统计分析,进而聚类生成可视化的特征图谱,最终实现从表型与脂质分子层面鉴评花生品种。
1 材料与方法 1.1 试验材料供试材料选取两个高、低油酸花生品种,高油酸品种开农1715(KN1715)、低油酸品种冀花7号JH7H。试验材料于2020年秋季(7—11月)种植在广东省农业科学院白云试验基地(113' 44"E,23' 39"N)。田间试验采用随机区组设计,每小区种植36株(6行×6列),行株间距20 cm,3次重复。收获时,每个花生品种随机选取15株进行表型数据收集与广泛靶向脂质代谢组学检测。
1.2 试验方法1.2.1 农艺性状调查 参照鲁清等[17]的方法,农艺性状检测指标包括主茎长、第一侧枝长、单株果数、单株饱果数、百果重、百仁重,并计算单株饱果率、单株秕果率与出仁率。
1.2.2 品质性状分析 花生收获后,清洗成熟的花生荚果,去除表面泥土,40 ℃烘干脱水后,使用近红外谷物分析仪DA7250(Perten,瑞典)对种子进行品质检测,其中脂质指标包括脂肪(Fat)、油酸(Oleic acid,CHO)、亚油酸(Linoleic acid,CLA)、油酸/ 亚油酸含量比值(CHO/ CLA)、棕榈酸(Palmitic acid)、硬脂酸(Stearic acid)、花生酸(Arachidic acid)、山嵛酸(Docosanoic acid)、二十四烷酸(Lignoceric acid),蛋白指标包括蛋白质(Protein,Pr)、氨基酸(Amino acid,AA)、苏氨酸(Threonine,Thr)、缬氨酸(Valine,Val)、蛋氨酸(Methionine,Met)、异亮氨酸(Isoleucine,Ile)、亮氨酸(Leucine,Leu)、苯丙氨酸(Phenylalanine,Phe)、赖氨酸(Lysine,Lys)、组氨酸(Histidine,His)、精氨酸(Arginine,Arg)、脯氨酸(Proline,Pro)[4]。
1.2.3 广泛靶向脂质代谢组学分析 将烘干脱水后的花生种子(各品种3个重复)用研磨仪研磨(30 Hz、1.5 min)至粉末状;准确称取50 mg粉末,用1 mL脂质提取液提取;涡旋2 min,超声5 min,加入500 μL水;40 ℃,12 000 r/min离心,吸取上清液500 μL于1.5 mL离心管中,真空冷冻干燥(VIRTIS,美国)浓缩,复溶后0.22 μm微孔滤膜过滤,保存于进样瓶中。后续的UPLC-MS/MS检测与分析委托迈维(武汉)生物技术有限公司进行。
1.3 数据处理与分析花生农艺与品质性状数据采用Excel软件进行统计,并利用GraphPad Prism软件绘图。品种间采用成对T检验,使用SPSS 21版软件进行。广泛靶向脂质代谢组学原始数据的多元分析,如热图、火山图、排序图(Patterns hunter),主成分分析(Principal component analysis,PCA),偏最小二乘- 判别分析PLS-DA(Partial least squares-discriminant analysis,PLS-DA)和得分图通过MetaboAnalyst软件(http://www.metaboanalyst.ca)与图图云平台(https://www.cloudtutu.com/)进行[18]。
2 结果与分析 2.1 高、低油酸花生品种农艺性状比较比较分析结果(图 1)表明,高、低油酸花生品种的农艺性状差异显著,其中高油酸品种KN1715的主茎、第一侧枝长度比低油酸品种JH7H分别高11.72% 和33.74%(图 1A、B),差异显著;除单株秕果率外,KN1715的单株果数、单株饱果率、百果重、百仁重与出仁率比JH7H分别低46.23%、5.02%、5.35%、11.68% 和17.17%(图 1C~H),差异显著。

|
| * 表示差异显著,** 表示差异极显著 *represents significant difference, **represents extremely significant difference 图 1 高、低油酸花生的农艺性状 Fig. 1 Agronomic traits of high and low oleic acid peanuts |
2.2 高、低油酸花生品质性状比较
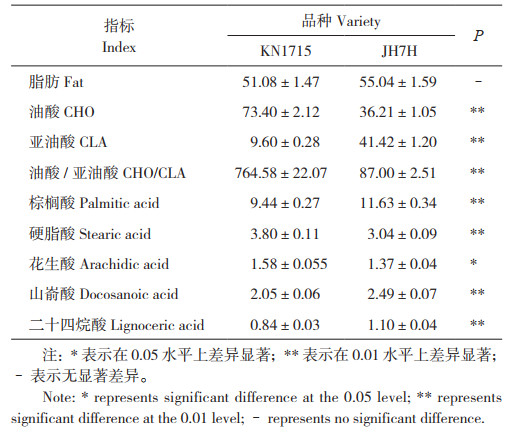
由表 1可知,脂质指标中,KN1715的油酸/ 亚油酸含量比值为JH7H的8.79倍,油酸、硬脂酸、花生酸含量比JH7H分别高102.71%、25.00%、15.33%,但亚油酸、棕榈酸、山嵛酸、二十四烷酸含量分别低76.83%、18.83%、17.67%、23.64%,差异均达显著水平;蛋白指标中,与JH7H相比,KN1715的蛋白质、氨基酸、缬氨酸、蛋氨酸、异亮氨酸、亮氨酸、苯丙氨酸、组氨酸、精氨酸与脯氨酸含量分别高16.35%、16.89%、16.47%、23.81%、17.54%、17.19%、18.63%、19.03%、19.81%、13.85%,差异显著(表 2)。
|
|
2.3 高、低油酸花生脂质组学特征
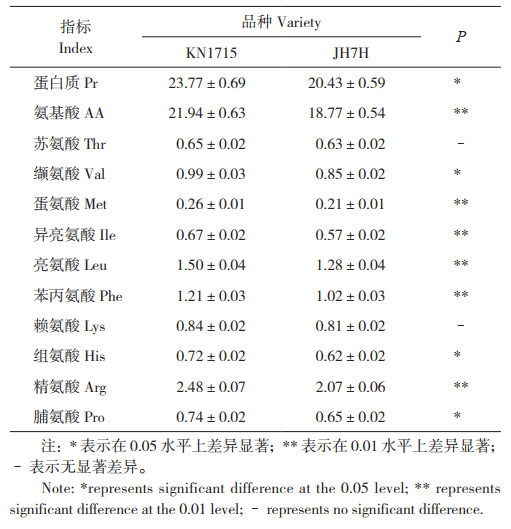
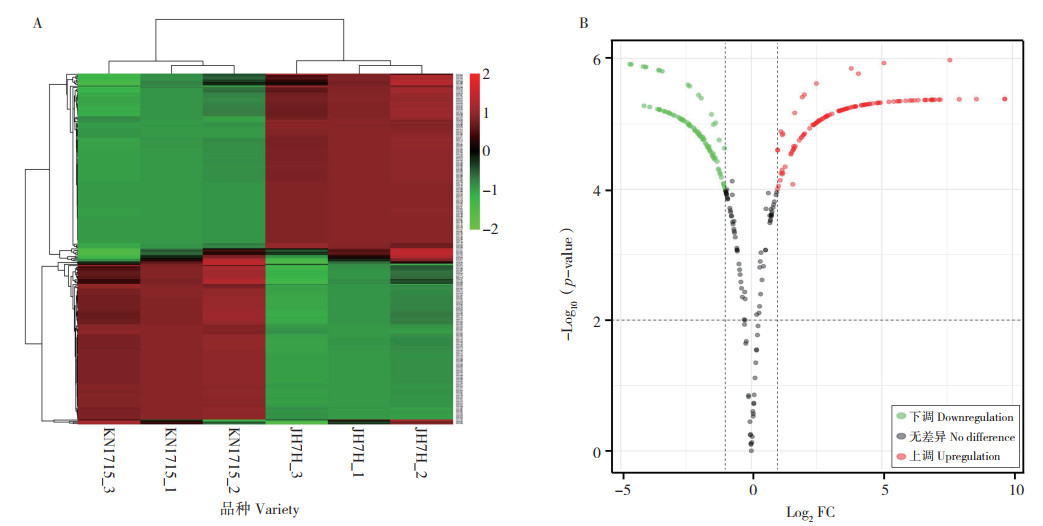
基于KN1715与JH7H的广泛靶向脂质代谢组学数据进行可视化数据分析,从而显示出两品种的总样本特征(图 2)。热图结果表明,在两个品种的种子中均鉴定出16类295种脂质代谢物,且两品种间脂质代谢物含量差异明显(图 2A)。此外,各品种的3个生物重复均能分别聚类,说明得到的数据重复性好、可靠性强。火山图根据差异倍数(Fold change,FC ≥ 2,P ≤ 0.05)进一步筛选差异代谢物,结果表明,KN1715中含量比JH7H高的脂质代谢物有90种,比JH7H低的有114种,无差异的有91种(图 2B)。
|
| A:KN1715与JH7H的差异脂质代谢物聚类分析热图;B:差异脂质代谢物火山图;红、绿与黑色分别表示正、负与无相关性,颜色越深相关性越强 A: Heat map of cluster analysis of differential lipid metabolites between KN1715 and JH7H; B: Volcano map of differential lipid metabolites; Red, green and black represent positive, negative and no correlation, respectively, the darker the color the stronger the correlation 图 2 高、低油酸花生的脂质组学总样本特征 Fig. 2 Total sample characteristics of lipidomics of high and low oleic acid peanuts |
初步可视化结果(图 2)表明,高、低油酸花生品种中存在核心差异脂质代谢物。为了进一步分析KN1715与JH7H的脂质代谢物变化规律,以显著差异的测试数据为自变量进行PCA分析和偏最小PLS-DA分析,以进一步展现脂质代谢测试数据的一致性,同时确定两个品种分离的关键代谢物。同时,以PLS-DA变量投影重要度(Variable importance in projection,VIP)衡量各测定指标对各组样本分类判别的影响强度和解释能力,从而辅助关键指标的筛选(通常以VIP值>1.0作为筛选标准)。PCA分析结果(图 3A)表明,所有测定指标降维为2个主成分,分别为PC1和PC2方差百分比为99.4% 和0.6%,其中PC1可以很好地表征所有测定指标99.4% 的信息;PLS-DA分析(图 3B)则降维出2个变量,即变量1(99.4%)和变量2(0.6%),其中变量1也可以很好地表征所有测定指标99.40% 的信息。从图 3C、D可以看出,PCA与PLS-DA分析结果均可以将KN1715、JH7H的脂质代谢物完全分离为两组,根据PLS-DA分析的变量1生成VIP得分图进一步分析,以量化测定参数对两个品种分离的贡献。从图 3E可以看出,两个品种中存在3类(即甘油二酯DG 3.10%、甘油三酯TG 93.80%、磷脂酰胆碱PC 3.10%)31种脂质代谢物的VIP值> 1.0,说明这31种脂质代谢物是形成两品种分离的核心差异脂质代谢物。

|
| A、C:PCA分析;B、D:PLS-DA分析;E:PLS-DA分析变量1的VIP得分 A, C: PCA analysis; B, D: PLS-DA analysis; E: Variable importance in projection(VIP)to the Component 1 of PLS-DA 图 3 脂质代谢物的PCA与PLS-DA分析及PLS-DA分析变量1的VIP得分 Fig. 3 PCA and PLS-DA analysis of lipid metabolites and variable importance in projection(VIP)to the component 1 of PLS-DA |
2.4 脂质组核心差异代谢物与品质指标的特征谱及其相关性
通过31种脂质组核心差异代谢物与标准化测定的品质指标进行聚类分析,可以得到鉴定花生品种的可视化脂质组特征图谱(图 4)。图 4结果显示,脂质代谢物与品质指标在KN1715与JH7H两个高、低油酸品种间具有明显的差异,在KN1715中,有19种脂质分子含量显著上调,其余12种则相反,这也可作为区分高油酸花生与普通花生的特异性脂质生物标志物。
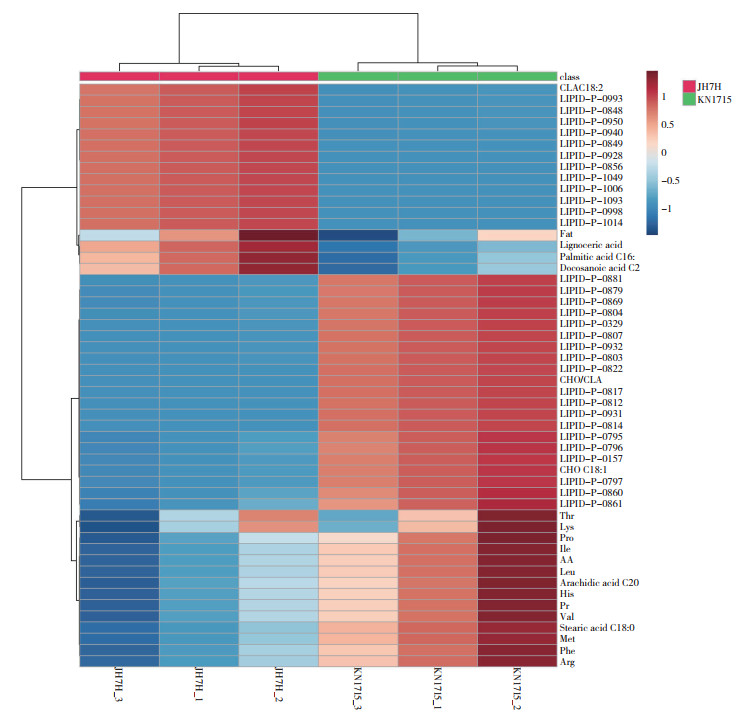
|
| 图 4 脂质组核心差异代谢物与品质指标的特征谱 Fig. 4 Characteristics mapping for core differential lipidomics metabolites and the quality indexes |
对脂质组核心差异代谢物与品质指标的相关性进行分析,既可进一步探索代谢物与表型的关系,也佐证脂质组核心差异代谢物。图 5A结果表明,高油酸花生品种代谢物与品质指标的相关性热图,红、蓝与白分别表示正、负、无相关性,其颜色越深相关性越强。由于KN1715与JH7H分别为高、低油酸品种,故重点关注与油酸相关的指标。由图 5可知,油酸与部LIPID-P-0795、LIPID-P-0796、LIPID-P-0157、LIPID-P-0797、LIPID-P-0881、LIPID-P-0879、LIPID-P-0869、LIPID-P-0804、LIPID-P-0822、CHO/CLA、LIPID-P-0817、LIPID-P-0931、LIPID-P-0814、LIPID-P-0812、LIPID-P-0329、LIPID-P-0807、LIPID-P-0932、LIPID-P-0803、硬脂酸C18:0、花生酸、Met、Phe、Arg、Pro、Ile、AA、Leu、His、Pr、Val)之间存在显著正相关,与Fat、Thr、Lys及两各品种均无显著相关性外,与其他指标之间则存在显著负相关。进而对所有与油酸密切相关的指标进行分类排序,根据相关系数>0.5进行选择,与油酸相关性由强到弱排序的结果见图 5B。
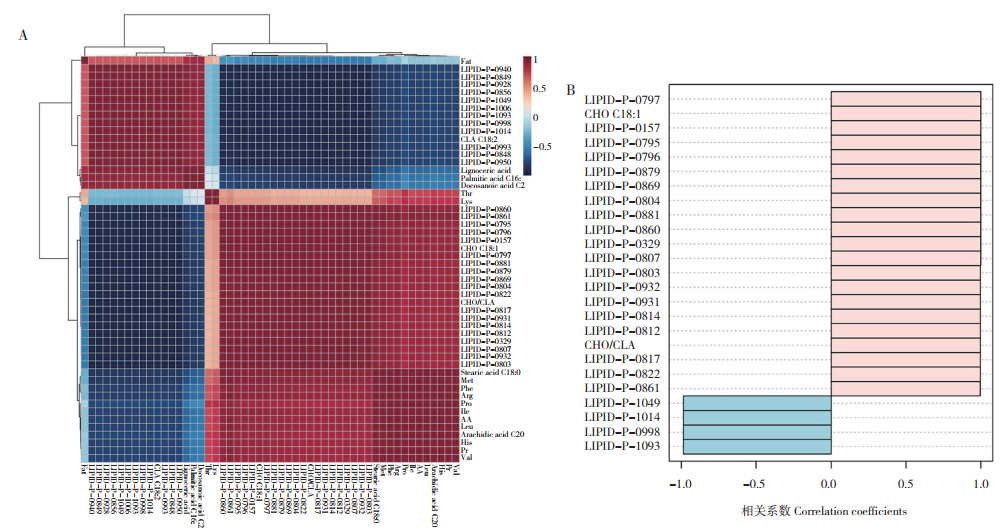
|
| A:红、蓝与白分别表示正、负、无相关性,颜色越深相关性越强;B:相关性越强与油酸越靠近,其直方柱状越长 A: Red, blue and white represent positive, negative and no correlation respectively, the darker the color the stronger the correlation; B: The stronger the correlation and the closer the oleic acid, the longer the histogram 图 5 脂质组核心差异代谢物与品质指标的相关性热图(A)及与油酸相关的指标排序图(B) Fig. 5 Heat map(A)for core differential lipidomics metabolites and the quality indicators, and the top indexes correlated with oleic acid(B) |
3 讨论
本研究首次利用近红外光谱技术与广泛靶向脂质组学,基于高、低油酸花生的品质指标与种类繁多的脂质分子的原始数据,通过多元统计分析,最终获得品种鉴定与品质评价兼备的表型与脂质分子特征图谱,以及筛选高油酸花生特异性脂质生物标志物。目前,近红外光谱技术作为一种无损、快速、低成本的检测技术,已被广泛应用于植物品种鉴定与选育[19]、作物品质评价[20]和食用植物油脂与油料品质检测[21]。本研究结果显示,油酸与亚油酸含量成反比,这与禹山林等[22]最先利用近红外技术测定花生的油脂与蛋白质含量的结果类似。本研究中,基于近红外光谱检测到的物质均是脂肪酸水平的脂质代谢物。而花生油作为广东省主要的食用油之一,其不断增加的市场需求,导致不良商家使用不健康或较便宜的油进行掺假,而掺假物与纯花生油脂的光谱非常相似,因此,研制能识别和量化相应掺假物的通用、可靠的分析方法是今后研究的重要方向[23]。
脂质组学主要的研究对象是较高丰度的内源性脂质代谢物,而且其普遍采用的研究手段是UPLC-MS/MS技术,使得该技术天然具备高稳定与高精度属性。因此,广泛靶向脂质组学成功应用于不同领域,如长链脂肪酸的生物合成与棉纤维发育[24]、植物细胞表面脂质盐受体[25]。本研究使用UPLC-MS/MS广泛靶向脂质组学对2个油酸差异显著的花生品种进行分析,经过多元统计分析最终得到3类31种核心差异脂质分子,即甘油三酯(93.80%)、甘油二酯(3.10%)和磷脂酰胆碱(3.10%)。甘油三酯作为植物油脂的主要成分,在植物脂质代谢途径中起到关键作用[26],而甘油三酯的功能决定于其骨架结构上的脂肪酸种类[7],这也是本研究中31种核心差异脂质代谢物的来源。此外,近年来多组学关联分析已被广泛应用于作物遗传育种与生理生化等机理研究[27-28],本研究利用多元统计分析方法对花生表型组与脂质组数据进行集成分析,并初步获得与油酸含量相关的表型与脂质组指标,这些结果将为揭示花生脂质合成与代谢机制提供研究思路与理论依据。
4 结论本研究分析了成熟期高、低油酸花生KN1715与JH7H的表型性状(农艺与品质性状)与脂质组的差异。就表型性状而言,KN1715的主茎、第一侧枝长度、油酸、硬脂酸、花生酸、蛋白质、氨基酸、缬氨酸、蛋氨酸、异亮氨酸、亮氨酸、苯丙氨酸、组氨酸、精氨酸与脯氨酸含量显著高于JH7H;就脂质组而言,两品种间存在较大的差异,共筛选到16类295种脂质分子,进一步通过对差异代谢物多元统计分析,最终筛选到3类31种核心差异脂质分子,其在花生品种鉴定起到关键作用。另外,本研究集成近红外与广泛靶向脂质组学数据,获得KN1715与JH7H的可视化脂质组特征图谱,建立从品质表型与脂质分子层面进行花生品种鉴定与品质评价的新型分析方法。
| [1] |
李海东, 李文金, 康涛, 任志红, 陈建生, 张利民. 花生主要光合性状对产量形成的重要性差异分析[J]. 花生学报, 2016, 45(4): 61-65. DOI:10.14001/j.issn.1002-4093.2016.04.011 LI H D, LI W J, KANG L, REN Z H, CHEN J S, ZHANG L M. Theimportance difference analysis of main photosynthetic parameters of peanut on yield formation[J]. Journal of Peanut Science, 2016, 45(4): 61-65. DOI:10.14001/j.issn.1002-4093.2016.04.011 |
| [2] |
刘浩, 鲁清, 李海芬, 李少雄, 陈小平, 梁炫强, 洪彦彬. 花生硬脂酰-ACP酸脱饱和基因FAB2表达的分子机制[J]. 作物学报, 2019, 45(11): 1638-1648. DOI: 10.3724/SP.J.1006.2019.94003. LIU H, LU Q, LI H F, CHEN X P, LIANG X Q, HONG Y B. Molecular mechanism of stearoyl-ACP desaturase gene FAB2 expression in peanut[J]. Acta Agronomica Sinica, 2019, 45(11): 1638-1648. DOI: 10.3724/SP.J.1006.2019.94003. |
| [3] |
LIU H, LI H F, GU J Z, DENG L, REN L, HONG Y B, LU Q, CHEN X P, LIANG X Q. Identification of the candidate proteins related to oleic acid accumulation during peanut(Arachis hypogaea L.)seed development through comparative proteome analysis[J]. International Journal of Molecilar Sciences, 2018, 19(4): 1235. DOI:10.3390/ijms19041235 |
| [4] |
刘浩, 洪彦彬, 鲁清, 李海芬, 陈小平, 梁炫强, 李少雄. 南方花生区试品种主要品质性状与产量变化趋势分析[J]. 广东农业科学, 2018, 45(7): 8-15. DOI:10.16768/j.issn.1004-874X.2018.07.002 LIU H, HONG Y B, LU Q, LI H F, CHEN X P, LIANG X Q, LI S X. Trend vibration analysis of major quality traits and yield in peanut varieties under national regional trail in Southern China[J]. Guangdong Agricultural Sciences, 2018, 45(7): 8-15. DOI:10.16768/j.issn.1004-874X.2018.07.002 |
| [5] |
LIU H, HO NG, Y B, LU Q, LI H F, GU J Z, REN L, DENG L., ZHOU B J, CHEN X P, LIANG X Q. Integrated analysis of comparative lipidomics and proteomics reveals the dynamic changes of lipid molecular species in high-oleic acid peanut seed[J]. Journal of Agricultural and Food Chemistry, 2020, 68: 426-438. DOI:10.1021/acs.jafc.9b04179 |
| [6] |
赵新楠, 王秀嫔, 李培武, 印南日, 万立昊, 王晓, 张良晓. 基于超高效液相色谱-高分辨质谱法的油料作物脂质组学分析[J]. 分析测试学报, 2020, 39(6): 697-704. DOI:10.3969/j.issn.1004-4957.2020.06.001 ZHAO X N, WANG X P, LI P W, YIN N R, WAN L H, WANG X, ZHANG L X. Lipidomics analysis of oil crops based on ultra-high performance liquid chromatography-high resolution mass spectrometry[J]. Journal of Instrumental Analysis, 2020, 39(6): 697-704. DOI:10.3969/j.issn.1004-4957.2020.06.001 |
| [7] |
杨悠悠, 刘佟, 苗雨田, 王浩, 杨永坛. 8种常见食用油中甘油三酯组成的分析[J]. 中国粮油学报, 2017, 32(5): 122-126. YANG Y Y, LIU D, MIAO Y T, WANG H, YANG Y T. Composition analysis of triacylglycerols in eight common edible oils[J]. Journal of the Chinese Cereals and Oils Association, 2017, 32(5): 122-126. |
| [8] |
LIU H, HU D X, DU P X, WANG L, LIANG X Q, LI H F, LU Q, LI S X, LIU H, CHEN X P, VARSHNEY R K, HONG Y B. Single-cell RNA-seq describes the transcriptome landscape and identifies critical transcription factors in the leaf blade of the allotetraploid peanut (Arachis hypogaea L.)[J]. Plant Biotechnology Journal, 2021, 13656. DOI:10.1111/pbi.13656 |
| [9] |
蔺儒侠, 郭凤丹, 王兴军, 夏晗, 侯蕾. 花生分子育种研究进展[J]. 作物杂志, 2021(5): 1-5. DOI:10.16035/j.issn.1001-7283.2021.05.001 LIN R X, GUO F D, WANG X J, XIA H, HOU L. Advances in peanut molecular breeding[J]. Crops, 2021(5): 1-5. DOI:10.16035/j.issn.1001-7283.2021.05.001 |
| [10] |
ZHAO C Z, QIU J J, GAURAV A, WANG J S, REN X Z, XIA H, GUO B Z, MA C, WAN S B, BERTIOLI D J, VARSHNEY R K, PANDEY M K, WANG X J. Genome-wide discovery of microsatellite markers from diploid progenitor species, Arachis duranensis and A. ipaensis, and their application in cultivated peanut(A. hypogaea)[J]. Frontiers in Plant Science, 2017, 8: 1029. DOI:10.3389/fpls.2017.01209 |
| [11] |
MILLA S R, ISLEIB T G, STALKER H T. Taxonomic relationships among Arachis sect. Arachis species as revealed by AFLP markers[J]. Genome, 2005, 48(1): 1-11. DOI:10.1139/g04-089 |
| [12] |
CRESTER S, TSAI S M, VALLS J F M, GIMENES M A, LOPES C R. Genetic characterization of brazilian annual Arachis species from aections Arachis and Heteranthae using RAPD Markers[J]. Genetic Resources and Crop Evolution, 2005, 52(8): 1079-1086. DOI:10.1007/s10722-004-6098-9 |
| [13] |
HALWARD T, STALKER H T, KOCHERT G. Development of an RFLP linkage map in diploid peanut species[J]. Theoretical and Applied Genetics, 1993, 87(3): 379-384. DOI:10.1007/BF01184927 |
| [14] |
钱瑞, 任锡毅, 刘永翔, 蒋选利. 基于广泛靶向代谢组学的竹黄活性成分分析[J]. 菌物学报, 2019, 38(5): 631-645. DOI:10.13346/j.mycosystema.180321 QIAN R, REN X Y, LIU Y X, JIANG X L. Widely-targeted metabolomics analysis of Shiraia bambusicola bioactive components[J]. Mycosystema, 2019, 38(5): 631-645. DOI:10.13346/j.mycosystema.180321 |
| [15] |
方贤胜, 吴涛, 肖良俊. 基于广泛靶向代谢组学的浅黄色和紫色核桃内种皮成分差异分析[J]. 食品科学, 2021, 42(12): 215-221. DOI:10.7506/spkx1002-6630-20191108-099 FANG X S, WU T, XIAO L J. Broadly targeted metabolomics analysis of differential metabolites between light yellow and purple Walnut endocarps[J]. Food Science, 2021, 42(12): 215-221. DOI:10.7506/spkx1002-6630-20191108-099 |
| [16] |
鲁忠富, 李艳伟, 汪颖, 吴晓花, 吴新义, 王尖, 汪宝根, 李国景. 基于广泛靶向代谢组学的瓠瓜果实鲜味差异代谢物分析[J]. 中国蔬菜, 2021(6): 34-41. DOI:10.19928/j.cnki.1000-6346.2021.1025 LU Z F, LI Y W, WANG Y, WU XH, WU X Y, WANG J, WANG B G, LI G J. Analysis of fruit umami differential metabolites of bottle gourd based on widely targeted metabolomics[J]. China Vegetables, 2021(6): 34-41. DOI:10.19928/j.cnki.1000-6346.2021.1025 |
| [17] |
鲁清, 刘浩, 李海芬, 陈小平, 洪彦彬, 刘海燕, 李少雄, 周桂元, 梁炫强. 花生不同株型主要农艺性状的相关分析及其对单株产量的影响[J]. 热带作物学报, 2019, 40(6): 1115-1121. DOI:10.3969/j.issn.1000-2561.2019.06.012 LU Q, LIU H, LI H F, CHEN X P, HONG Y B, LIU HY, LI S X, ZHOU G Y, LIANG X Q. Correlation analysis of main agronomic traits of different plant types and path analysis of yield per plant in peanut (Arachis hypogaea L.)[J]. Chinese Journal of Tropical Crops, 2019, 40(6): 1115-1121. DOI:10.3969/j.issn.1000-2561.2019.06.012 |
| [18] |
DENG Q Q, YU T B, ZENG Z, ASHRAF U, SHI Q H, HUANG S H, LIAN T X, CHEN J W, MUZAFFAR W, SHEN W K. Silicon application modulates the growth, rhizosphere soil characteristics, and bacterial community structure in sugarcane[J]. Frontiers in Plant Science, 2021, 12: 710139. DOI:10.3389/fpls.2021.710139 |
| [19] |
黄艳华, 杜娟, 刘斯佳, 李洪超, 张蕴薇. 近红外光谱在植物种及品种鉴定中的应用[J]. 中国农学通报, 2014, 30(6): 46-51. HUANG Y H, DU J, LIU S J, LI H C, ZHANG Y W. The application of near infrared spectroscopy in identification of plant species and cultivars[J]. Chinese Agricultural Science Bulletin, 2014, 30(6): 46-51. |
| [20] |
李丹丹, 曹军, 张冬妍. 近红外光谱技术在坚果检测中的应用研究[J]. 食品研究与开发, 2021, 42(4): 198-202. DOI:10.12161/j.issn.1005-6521.2021.04.034 LI D D, CAO J, ZHANG D Y. Studies on detection of nuts by near infrared spectroscopy technology[J]. Food Research and Development, 2021, 42(4): 198-202. DOI:10.12161/j.issn.1005-6521.2021.04.034 |
| [21] |
李雪, 王督, 白艺珍, 喻理, 岳晓凤, 张文, 张良晓, 李培武. 我国油料产品品质的近红外光谱快速检测技术研究进展[J]. 分析测试学报, 2020, 39(10): 1189-1195. DOI:10.3969/j.issn.1004-4957.2020.10.002 LI X, WANG D, BAI Y Z, YU L, YUE X F, ZHANG W, ZHANG L X, LI P W. Research progress of near infrared spectroscopy in rapid determination on quality of oilseed products in China[J]. Journal of Instrumental Analysis, 2020, 39(10): 1189-1195. DOI:10.3969/j.issn.1004-4957.2020.10.002 |
| [22] |
禹山林, 朱雨杰, 闵平, 杨庆利, 曹玉良, 王传堂, 刘旭, 周学秋. 傅立叶近红外漫反射非破坏性测定花生种子主要脂肪酸含量[J]. 花生学报, 2010, 39(1): 11-14, 48. DOI:10.14001/j.issn.1002-4093.2010.01.008 YU S L, ZHU Y J, MIN P, YANG Q L, CAO Y L, WANG C T, LIU X, ZHOU X Q. Nondestructive measurement of main fatty acid composition in peanut seeds with near infrared reflectance spectroscopy[J]. Journal of Peanut Science, 2010, 39(1): 11-14, 48. DOI:10.14001/j.issn.1002-4093.2010.01.008 |
| [23] |
RAFAEL C C, RIBEIRO D S M, SANTOS J L M, PASCOA R. Comparison of near infrared spectroscopy and raman spectroscopy for the identification and quantification through MCR-ALS and PLS of peanut oil adulterants[J]. Talanta, 2021, 230: 122373. DOI:10.1016/j.talanta.2021.122373 |
| [24] |
HU W J, CHEN L, QIU X Y, WEI J, LI F G, SHEN G X. AKR2A participates in the regulation of cotton fifibre development by modulating biosynthesis of very long-chain fatty acids[J]. Plant Biotechnology Journal, 2019, 13221. DOI:10.1111/pbi.13221 |
| [25] |
JIANG Z, ZHOU X, TAO M, YUAN F, LIU L L, HU Z L, PEI Z M. Plant cell-surface GIPC sphingolipids sense salt to trigger Ca2+ influx[J]. Nature, 2019, 572: 341-346. DOI:10.1038/s41586-019-1449-z |
| [26] |
ROBINSON C V, ROHACS T, HANSEN S B. Tools for understanding nanoscale lipid regulation of ion channels[J]. Trends in Biochemical Sciences, 2019, 44(9): 795-806. DOI:10.1016/j.tibs.2019.04.001 |
| [27] |
李洁, 姚晓华. 多组学关联分析作物耐逆境胁迫研究进展[J]. 广东农业科学, 2019, 46(8): 22-28. DOI:10.16768/j.issn.1004-874X.2019.08.004 LI J, YAO X H. Research progress on multi-omics association analysis of crop resistance to adversity stress[J]. Guangdong Agricultural Sciences, 2019, 46(8): 22-28. DOI:10.16768/j.issn.1004-874X.2019.08.004 |
| [28] |
李涛, 孙保娟, 李植良, 黎振兴, 罗少波, 徐小万, 衡周, 宫超, 游倩. 茄子对青枯病的抗性机制研究现状与展望[J]. 广东农业科学, 2021, 48(9): 40-50. DOI:10.16768/j.issn.1004-874X.2021.09.005 LI T, SUN B J, LI Z L, LI Z X, LUO S B, XU X W, HENG Z, GONG C, YOU Q. Research status and prospects of resistance mechanism of eggplant to bacterial wilt[J]. Guangdong Agricultural Sciences, 2021, 48(9): 40-50. DOI:10.16768/j.issn.1004-874X.2021.09.005 |
(责任编辑 邹移光)
 2021, Vol. 48
2021, Vol. 48