文章信息
基金项目
- 国家自然科学基金(61675003);广东省自然科学基金(2018A030310153);广州市科技计划项目(201707010346)
作者简介
- 陈楚汉(1997—),男,在读硕士生,研究方向为近红外光谱研究,E-mail: 597748426@qq.com.
通讯作者
- 代芬(1978—),女,博士,副教授,研究方向为农业信息与技术研究,E-mail: sunflower@scau.edu.cn.
文章历史
- 收稿日期:2020-12-05
2. 广东省蚕业技术推广中心,广东 广州 510640;
3. 华南农业大学动物科学学院,广东 广州 510642
2. Guangdong Sericulture Technology Promotion Center, Guangzhou 510640, China;
3. College of Animal Science, South China Agricultural University, Guangzhou 510642, China
【研究意义】蚕茧雌雄鉴别是蚕茧杂交育种的重要环节。从熟蚕上蔟到蚕蛹化蛾共约14 d,蚕种场一般在第8天进行削茧鉴蛹辨别雌雄,削茧鉴蛹时间只有4~5 d,在短时间内完成该环节任务需要大量人工,劳动成本高。因此,研究蚕蛹性别自动鉴定具有重要的实践意义。【前人研究进展】目前关于蚕蛹性别自动鉴定的方法有荧光蚕茧辨性(Fluorescent cocoon sex identification, FCSI)、磁共振成像(Magnetic resonance imaging, MRI)、X射线成像技术等[1],但这些方法成本高、速度慢,不适于在实际生产中推广。龚攀等[2]用871A和7532两个品种的蚕蛹图像进行了识别试验,每一品种取50个样本进行训练、50个样本进行测试,BP网络训练收敛,准确率分别为98% 和96%。然而计算机视觉的方法需要削开茧壳,并将蚕蛹腹部置于相机镜头位置,检测速度太慢而无法满足实际生产需求。近红外光谱分析技术在蚕茧雌雄检测的应用上有良好效果[3-5],基于近红外漫透射光谱信息的蚕茧无损雌雄检测[6-7],不需要削茧取蛹进行判别即可实现实时检测和无损检测。
【本研究切入点】由于近红外光谱分析技术具有成本低、速度快、无损样品等优点,因此适用于农产品检测领域[8-10]。近红外光谱数据是一维数据,传统机器学习方法分析一维数据有良好效果[11-12],例如支持向量机(Support vector machine,SVM)、梯度提升决策树(Gradient boosting decision tree,GBDT)等方法。但是传统机器学习方法需要特定的光谱预处理方法,例如主成分分析(Principal component analysis,PCA),光谱修正、基线校正、平滑和归一化等方法。卷积神经网络(Convolutional neural networks,CNN)具有非常强的数据拟合能力,可学习到泛化能力更强的特征表达[13],一般应用在计算机视觉领域,近年来也有许多人将CNN应用于近红外光谱分析[14-15],在不使用预处理方法的情况下,CNN也能取得良好的效果[16]。迁移学习即一种学习对另一种学习的影响,它广泛存在于知识、技能、态度和行为规范的学习中。在计算机视觉、自然语言识别和人类行为识别等不同领域[17-18],迁移学习都取得了良好效果。
【拟解决的关键问题】目前基于近红外光谱的蚕茧检测技术存在以下问题:第一,各地茧场的环境不同,各地采集蚕茧的近红外光谱也有差异;第二,蚕茧品种不同,各个品种采集蚕茧的近红外光谱也有差异;第三,不同型号光谱仪采集蚕茧的近红外光谱也有差异。因此要建立一个较好的分类模型,理论上要采用不同光谱仪采集大量不同品种的样本光谱并且对样本进行标记,显然这在实际中是不可能完成的。为了解决以上问题,本研究使用大量数据建立一个良好的源域模型,将目标域数据集分成30%~70%的训练集,间隔为10%,用目标域数据集的训练集分别微调源域模型和建立SVM和RF两种模型,并对比分析目标域测试集准确率。为了验证卷积神经网络提取特征的能力,对源域模型中间层输出进行可视化分析,将目标域数据集输入到源域模型中,获取中间层输出,用中间层输出分别建立SVM和RF两种模型,并对比分析测试集准确率。
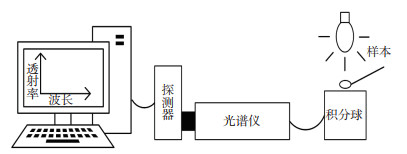
1 材料与方法 1.1 设备介绍样本漫透射光谱的采集采用自行研制的种茧自动分选样机完成,蚕茧雌雄快速无损检测系统如图 1所示,检测步骤如下:
|
| 图 1 蚕茧雌雄快速无损检测系统 Fig. 1 Fast and nondestructive system of sex detection of silkworm |
(1)自动分选样机将蚕茧逐个送到蚕茧检测工位。
(2)光源(100 W卤灯泡)从上向下照射蚕茧,积分球在蚕茧下面采集蚕茧的漫透射光,通过600μm光纤连接光谱仪。
(3)光谱仪通过USB线将样机采集的光谱数据传输给电脑端,电脑端保存数据。
海洋光谱仪部件采用NirQuest512型便携式光纤光谱仪,OTO光谱仪部件采用SW2540型便携式光纤光谱仪。检测范围:903~1 599 nm,光谱仪设置积分时间为200 ms,平均次数为4以提高数据的稳定性,平滑宽度为4以匹配系统的分辨率。
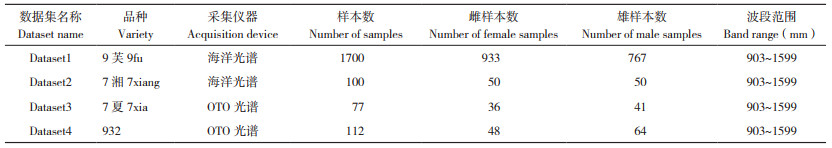
1.2 试验样本供试的家蚕种茧样本来自于广东省蚕业推广中心。按照试验设计要求将所有样本分成4个数据集(表 1)。根据蚕蛹的腹部第8、9腹节外部特征,削茧取蛹进行雌雄辨别,对难以判别的则解剖蚕蛹的腹部,以是否有卵来判别雌雄。
1.3 数据预处理
本研究采集的是蚕茧的近红外漫透射光谱,其中近红外光谱的透射率计算公式如下:

|
式中,R为漫透射光谱,Iraw为原始光谱,Ireference为本研究采集的是蚕茧的近红外漫透射光谱,参照光谱(在采光口不放任何样本的情况下采集其中近红外光谱的透射率计算公式如下:的光谱),Iblack为暗电流光谱(遮挡采光口后采集的光谱)。每次采集环境亮度发生明显改变都需要重新采集1次参考光谱和暗电流光谱。
CNN不需要任何预处理方法。使用SVM和RF时,为了减少噪声对模型产生的影响,对原始光谱进行标准归一化处理和卷积平滑滤波(Savitzky-Golay smoothing,SG)处理。标准归一化公式如下:
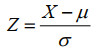
|
式中,X为原始光谱,μ为原始光谱均值,σ为原始光谱的方差。
近红外光谱波长k处经滤波后的平均值为:
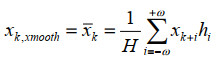
|
式中,hi为平滑系数,ω为平滑窗口,H为归一化因子,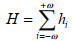
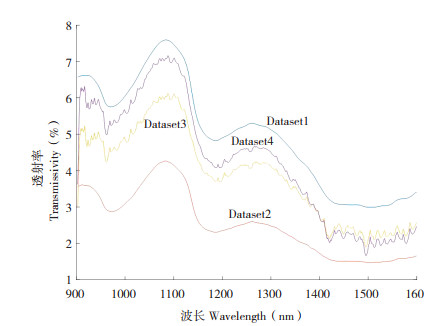
在同种条件下,样本的近红外光谱的谱峰波长一致,而因为设备或样本品种不同,谱峰波长会略有差别。本研究针对迁移学习的问题着重对样本的平均近红外光谱进行研究,图 2为4个数据集的近红外平均光谱。Dataset1的平均近红外光谱有5个谱峰,峰值波长分别为918、970、1 084、1 186、1 269 nm;Dataset2的平均近红外光谱有5个谱峰,峰值波长分别为913、973、1 084、1 186、1 262 nm;Dataset3的平均近红外光谱有5个谱峰,峰值波长分别为917、962、1 088、1 186、1 267 nm;Dataset4的平均近红外光谱有5个谱峰,峰值波长分别为917、962、1 088、1 186、1 267 nm。
|
| 图 2 4个数据集的平均光谱 Fig. 2 Mean spectra of 4 data sets |
Dataset1和Dataset2的近红外光谱均由海洋光谱仪采集,它们的峰值波长非常接近,样本品种分别为9芙和7湘,9芙的平均光谱透射率高于7湘的。Dataset3和Dataset4的近红外光谱均由OTO光谱仪采集,它们的峰值波长一致,而它们的样本品种分别为7夏和932,在波长为1 397 nm之前,932的平均光谱透射率高于7夏的,在1 397 nm之后7夏的平均光谱透射率则高于932的。上述结果表明海洋光谱仪和OTO光谱仪采集的不同品种样本间近红外光谱峰值十分接近,但不同仪器信噪比不同,不同品种的透射率也不同。
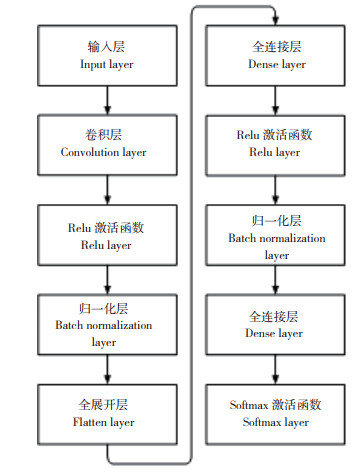
1.5 试验设计1.5.1 卷积神经网络设计 本研究设计出一个基于keras框架的卷积神经网络,CNN结构如图 3所示。该网络有1个卷积层和2个全连接层。CNN的卷积层有16个filters,kernel size大小为50,strides为25,激活函数为relu。第1个全连接层神经元个数为20,激活函数为relu,最后一层全连接层为分类层,激活函数为softmax。
|
| 图 3 卷积神经网络结构 Fig. 3 Structure of convolutional neural network |
通常卷积神经网络中的池化层或者汇合层(pooling)起到特征降维作用和一定程度的防过拟合作用,并不是卷积神经网络必需的操作,近期有研究表明构建只含卷积操作的网络可以达到甚至超过传统卷积神经网络(卷积层、池化层的交替)的分类精度。在本试验中,输入的数据通过卷积之后,特征数量较少,因此降采样降维并不必要,直接省去池化层。批量归一化层(Batch normalization,BN)不仅极大提升了训练速度,收敛过程大大加快,还能增加分类准确率[19]。relu激活函数可以防止过拟合;压缩数据,去除冗余;强化特征,减小误差;加快收敛速度。Categorical_crossentropy损失函数,当误差大时,权重更新快;当误差小时,权重更新慢。除了最后一层分类层的激活函数是softmax,其他层激活函数都是relu。
模型训练使用随机梯度下降(Stochastic gradient descent,SGD)最优化函数,正则化系数设置为0.001,采用衰减的学习率,当epoch为0~39时,学习率为0.001;当epoch为40~59时,学习率为0.0001;当epoch ≥ 60时,学习率为0.00001。
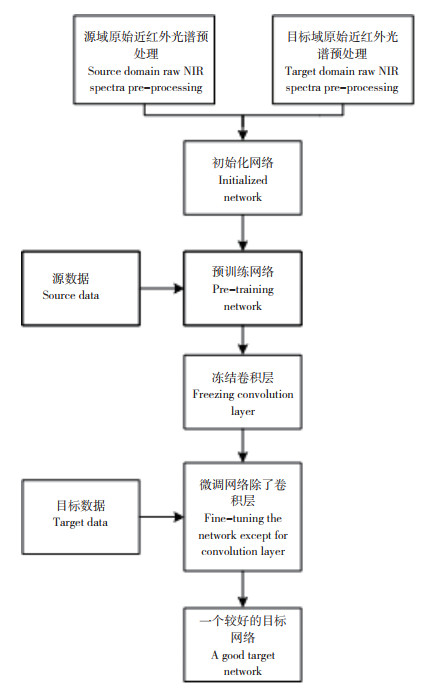
1.5.2 迁移学习 迁移学习是接受预训练模型(由其他人在大数据集上训练过的网络的权重和参数)并用自己的数据集对模型进行“微调”的过程[20]。在自然图像上,神经网络学习的第一层特征不是特定于某个数据集或任务,而是通用的[21]。因此本研究设计的迁移学习模型,将预训练网络的第一层卷积层冻结,再用目标数据集对网络进行训练。深度迁移学习预测模型如图 4所示,试验步骤如下所示:
|
| 图 4 深度迁移学习预测模型流程 Fig. 4 Flowchart of prediction model of deep transfer learning |
(1)根据试验内容准备好预训练数据集和目标数据集。
(2)将预训练数据集和目标数据集分别分成60% 训练集和40% 测试集。
(3)使用源域的训练集进行训练,对测试集数据准确率进行评估。
(4)若预训练网络表现好,则保存网络,否则重新训练。
(5)冻结预训练网络的卷积层。
(6)用目标数据集的训练集对冻结后的预训练网络进行“微调”。
(7)获得经过迁移学习后的目标网络,并对目标数据集的测试集进行准确率评估。
1.5.3 试验结果计算与试验环境 所有试验重复50次得出结果,然后再分别求平均值和标准差,得出的结果是平均值加上或减去3倍的标准差,置信度为99.73%,测试集的准确率(A)为:
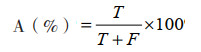
|
式中,T为测试集分类正确的数量,F为测试集分类错误的数量。
所有试验均是在计算机上(Intel Core i5-4200,2.8 GHz CPU和12 GB内存)用anaconda(python版本3.7.1,tensorflow版本1.14.0)进行。
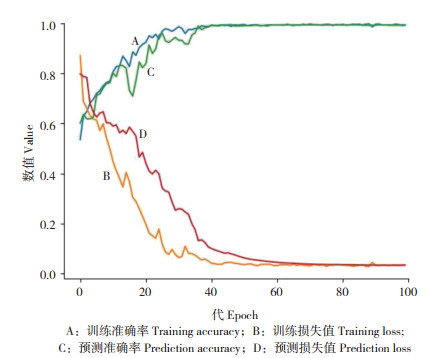
2 结果与分析 2.1 源域模型训练Dataset1分为60%的训练集和40%的测试集,用训练集训练源域模型,重复50次训练,训练集准确率为99.41(±0.30)%, 测试集准确率为99.56(±0.22)%。图 5为源域数据集训练集和测试集的精度和损失图,训练迭代次数达到60次后,模型达到最优。为了选取较好的源域模型,重复试验,选取最优的模型作为迁移学习源域模型。
|
| 图 5 源域模型精度和损失结果 Fig. 5 Result of accuracy and loss of source domain model |
2.2 源域模型中间输出可视化
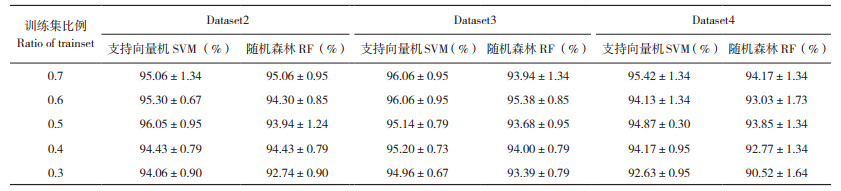
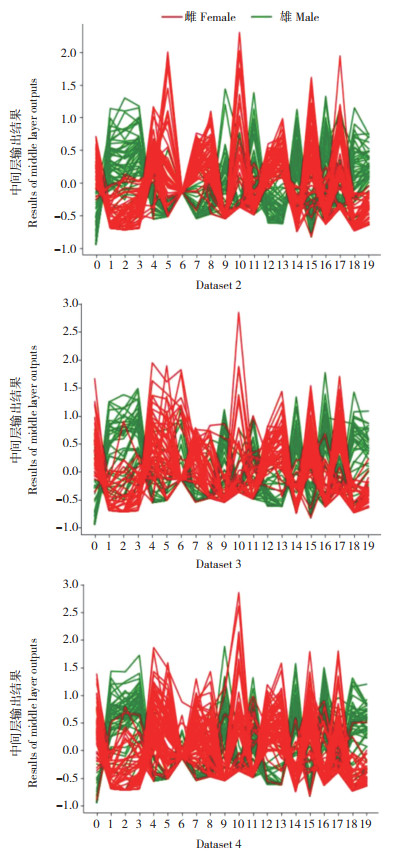
将目标域数据集Dataset2、Dataset3和Dataset4输入到源域模型中,获取中间层输出(CNN倒数第2层的输出,输出大小为20),中间层输出如图 6所示, 其中红色为雌样本,绿色为雄样本。从图 6可以看出,中间层输出具有明显的可分性。为了验证卷积神经网络提取特征的能力,用中间层输出分别建立SVM和RF两种模型,由于中间层输出已经具有可分性,不需要对数据进行预处理,训练集占目标数据集的30%~70%,得出测试集结果如表 2所示。中间层输出建立的模型准确率都在90% 以上,其中SVM的效果优于RF。结果表明,源域模型的卷积层具有很好的特征提取能力。
|
| 图 6 3个目标域数据集的源域模型中间层输出 Fig. 6 Middle layer outputs of source domain model of three target domain data sets |
2.3 相同设备不同品种间的迁移学习
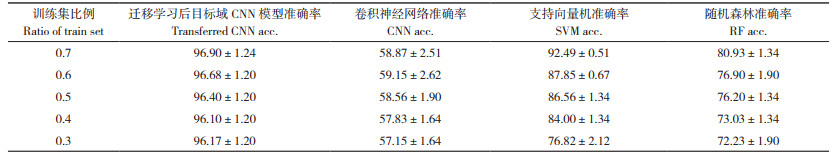
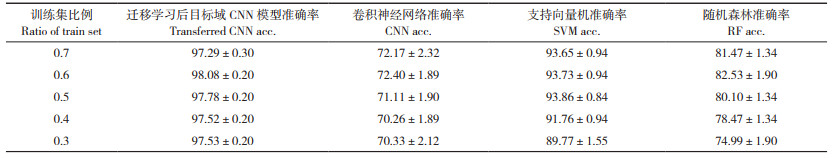
冻结源域模型的卷积层,把目标数据集Dataset2的30%~70% 作为训练集,用训练集微调源域CNN网络,得到迁移学习后目标域CNN模型。同时,用训练集重新训练独立的CNN、SVM和RF3种模型,然后比较迁移学习后目标域CNN模型和重新训练的独立模型的测试集准确率。由表 3可知,在训练集占总数据集比例为0.7时,使用独立CNN建模测试集准确率只有58.87%,而SVM和RF的测试集准确率分别为92.49% 和80.93%,表明数据量较少的情况下使用传统机器学习的方法优于CNN。在训练集占总数据集比例为0.3时,迁移学习后目标域CNN模型测试集准确率为96.17%,而SVM和RF准确率分别为76.82% 和72.23%,表明在数据量较少的情况下,迁移学习后目标域CNN模型要优于传统机器学习模型。本研究结果证明了在不同品种情况下迁移学习的可行性。
2.4 不同设备不同品种间的迁移学习
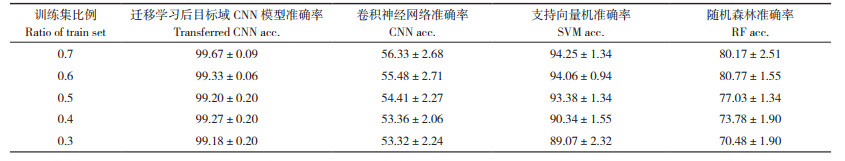
冻结源域模型的卷积层,把目标数据集Dataset3和Dataset4的30%~70% 作为训练集,用训练集微调源域CNN网络,得到迁移学习后目标域CNN模型。同时,用训练集重新训练独立的CNN、SVM和RF 3种模型,然后比较迁移学习后目标域CNN模型和重新训练的独立模型的测试集准确率。由表 4、表 5可知,迁移学习后CNN模型准确率都在97% 以上,而重新训练的独立CNN准确率只有50% 以上,这是由于训练数据太少,造成独立的CNN拟合不足。传统机器学习相对CNN网络对样本数量的要求要少一些,但是传统机器学习方法需要对输入数据进行预处理。由表 4、表 5可知,SVM的准确率为90% 以上,RF准确率在73%~81% 之间,两种方法都比迁移学习后目标域CNN模型准确率低,造成这一结果原因之一是样本数量少,而且没有使用输入数据预处理。在训练数据较少的情况下,使用迁移学习可以解决训练样本不足的问题。上述结果证明了在不同品种、不同光谱仪的情况下迁移学习的可行性。
3 讨论
实现快速家蚕雌雄鉴别有重要的实践意义,一方面家蚕雌雄鉴别是品种选育的重要环节,另一方面可以合理利用和分配雌雄蛹资源。目前,家蚕品种选育朝着多元化的方向发展[22],也就是说家蚕品种也呈多样性,这就增加了家蚕性别自定鉴别的难度。偏最小二乘法判别分析(Partial least squares discrimination analysis,Pls-da),SVM和RF等传统机器学习方法常用于近红外光谱分析中,且具有良好效果,然而由于近红外光谱数据存在噪声或者数据分布不均等问题,使用传统近机器学习方法需要使用特定的光谱预处理方法。刘燕德等[23]使用近红外光谱对叶片可溶性糖进行分析,使用了不同的数据预处理方法对模型进行优化。由于蚕茧品种和光谱仪器型号的多样性,蚕茧的近红外光谱数据也具有多样性,因此构建一个泛化能力较强的分类模型十分困难。在基于近红外光谱分析的家蚕自动鉴别中,一般采用监督学习的方式,需要专业人员对数据进行标注,而且对数据标注工作量大。利用近红外光谱技术对同一品种的蚕蛹进行准确的性别鉴别是一种有效的方法,然而由于采集条件或品种变化导致模型效果变差。针对不同品种间光谱数据的差异而造成模型效果变差的问题,Xie等[24]等使用半监督学习的方法,通过预标记的方式,选取较为可靠的新品种数据更新训练原始模型,提高了模型在不同品种之间的泛化能力。但使用传统机器学习方法需要对数据进行预处理,不同的光谱数据的预处理方法可能不一样。卷积神经网络具有良好的提取特征能力,使用卷积神经网络能够提取出不同光谱仪采集的不同品种蚕茧的近红外光谱数据的共同特征。本研究首次使用不同光谱仪的家蚕近红外光谱数据进行分析,同时通过迁移学习,使用少量数据“微调”预训练网络,可以得到一个效果较好的目标域模型,从而减少了重新搭建一个蚕茧雌雄分类模型的成本。
目前设计一个能提取所有数据分布特征的卷积神经网络较为困难。后续研究我们将使用更多的不同品种和不同光谱仪型号的家蚕近红外光谱数据进行试验,同时在网络中加入深度可分离卷积等结构对模型进行优化,以期在实际生产中得到应用。
4 结论本研究分析了基于近红外光谱的家蚕种茧雌雄鉴别模型在多设备和多品种间的迁移,得出以下结论:
(1)用1 700个9芙样本(NirQuest512光谱仪)构建的CNN源域模型,家蚕种茧雌雄分辨准确率达到99% 以上。结果表明使用大量数据构建的CNN模型具有很好的雌雄分辨能力。
(2)为了验证卷积层提取雌雄特征的能力,将目标域数据集Dataset2、Dataset3和Dataset4输入到源域模型中,获取中间层输出,并将中间层输出分成不同比例的训练集和测试集,然后用中间层输出的训练集作为输入训练SVM和RF模型,不同训练集比例建模比较,SVM和RF模型的测试集准确率分别为92% 和90% 以上。结合上述结果,通过可视化分析表明卷积层能很好地提取雌雄特征。
(3)用不同训练集比例的数据分别建立迁移学习CNN、独立CNN、SVM和RF模型,通过试验比较发现,在数据量较少的情况下,迁移学习后CNN模型效果最优,传统机器学习方法次之,深度CNN模型最差。结果表明,通过迁移学习能够快速建立一个针对不同品种蚕茧或不同光谱仪的雌雄鉴别模型。
| [1] |
蔡健荣, 刘兵, 李永平, 孙力. X射线成像技术判别蚕茧性别[J]. 农业工程学报, 2012, 28(8): 275-279. DOI:10.3969/j.issn.1002-6819.2012.08.044 CAI J R, LIU B, LI Y P, SUN L. Sexual discrimination of silkworm cocoon based on X-ray imaging[J]. Transactions of the Chinese Society of Agricultural Engineering, 2012, 28(8): 275-279. DOI:10.3969/j.issn.1002-6819.2012.08.044 |
| [2] |
龚攀, 李光林. 基于计算机视觉的蚕蛹性别识别应用研究[J]. 农机化研究, 2014(1): 206-209. DOI:10.3969/j.issn.1003-188X.2014.01.050 GONG P, LI G L. The recognition application research of the tupa gender based on computer vision[J]. Agricultural Mechanization Research, 2014(1): 206-209. DOI:10.3969/j.issn.1003-188X.2014.01.050 |
| [3] |
ZHU Z Q, YUAN H F, SONG C F, LI X Y, FANG D W, GUO Z Y, ZHU X Y, LIU W, YAN G D. High-speed sex identification and sorting of living silkworm pupae using near-infrared spectroscopy combined with chemometrics[J]. Sensors and Actuators B: Chemical, 2018, 268: 299-309. DOI:10.1016/j.snb.2018.04.093 |
| [4] |
TAO D, WANG Z R, LI G L, QIU G Y. Accurate identification of the sex and species of silkworm pupae using near infrared spectroscopy[J]. Journal of Applied Spectroscopy, 2018, 85(5): 949-952. DOI:10.1007/s10812-018-0744-z |
| [5] |
TAO D, QIU G, LI G. A novel model for sex discrimination of silkworm pupae from different species[J]. IEEE Access, 2019(7): 165328-165335. DOI:10.1109/ACCESS.2019.2953040 |
| [6] |
代芬, 吴玲, 叶观艳, 钟杨生, 洪添胜, 黄晓燕. 基于近红外漫透射光谱信息的蚕茧雌雄检测[J]. 农业机械学报, 2015, 46(12): 280-284. DOI:10.6041/j.issn.1000-1298.2015.12.038 DAI F, WU L, YE G Y, ZHONG Y S, HONG T S, HUANG X Y. Non-destructive detection of male and female silkworm cocoon based on transmission near infrared spectroscopy[J]. Transactions of The Chinese Society of Agricultural Machinery, 2015, 46(12): 280-284. DOI:10.6041/j.issn.1000-1298.2015.12.038 |
| [7] |
代芬, 车欣欣, 彭斯冉, 杨晓帆, 钟杨生, 李震, 吕石磊. 近红外漫透射光谱快速无损鉴别家蚕种茧茧壳内蚕蛹雌雄[J]. 华南农业大学学报, 2018, 39(2): 103-109. DOI:10.7671/j.issn.1001-411X.2018.02.016 DAI F, CHE X X, PENG S R, YANG X F, ZHONG Y S, LI Z, LYU S L. Fast and nondestructive gender detection of Bombyx mori chrysalis in the cocoon based on near infrared transmission spectroscopy[J]. Journal of South China Agricultural University, 2018, 39(2): 103-109. DOI:10.7671/j.issn.1001-411X.2018.02.016 |
| [8] |
ZHANG C, WU W Y, ZHOU L, CHENG H, YE X Q, HE Y. Developing deep learning based regression approaches for determination of chemical compositions in dry black goji berries(Lycium ruthenicum Murr.) using near-infrared hyperspectral imaging[J]. Food Chemistry, 2020, 319: 126536. DOI:10.1016/j.foodchem.2020.126536 |
| [9] |
ZHOU D R, YU Y, HU R W, LI Z M. Discrimination of Tetrastigma hemsleyanum according to geographical origin by near-infrared spectroscopy combined with a deep learning approach[J]. Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2020, 238: 118380. DOI:10.1016/j.saa.2020.118380 |
| [10] |
吴全金, 周喆, 孙威江. 近红外光谱技术在茶叶品质调控中的应用[J]. 广东农业科学, 2019, 46(1): 91-100. DOI:10.16768/j.issn.1004-874X.2019.01.014 WU Q J, ZHOU Z, SUN W J. Review on the application of near-infrared spectroscopy technology in tea quality management[J]. Guangdong Agricultural Sciences, 2019, 46(1): 91-100. DOI:10.16768/j.issn.1004-874X.2019.01.014 |
| [11] |
NOLASCO PEREZ I M, BADARO A T, BARBON JR S, AC BARBON A P, RODRIGUES POLLONIO M A, BARBIN D F. Classification of chicken parts using a portable near-infrared(NIR)spectrophotometer and machine learning[J]. Applied Spectroscopy, 2018, 72(12): 1774-1780. DOI:10.1177/0003702818788878 |
| [12] |
GAO J, NUYTTENS D, LOOTENS P, HE Y, PIETERS J G. Recognising weeds in a maize crop using a random forest machine-learning algorithm and near-infrared snapshot mosaic hyperspectral imagery[J]. Biosystems Engineering, 2018, 170: 39-50. DOI:10.1016/j.biosystemseng.2018.03.006 |
| [13] |
WANG C, MAHADEVAN S. Heterogeneous domain adaptation using manifold alignment//IJCAI proceedings of the 22nd international joint conference on artificial intelligence[C]. 2011: 1541-1546.
|
| [14] |
CHEN X, CHAI Q, LIN N, LIU X H, WANG W. 1D convolutional neural network for the discrimination of aristolochic acids and their analogues based on near-infrared spectroscopy[J]. Analytical Methods, 2019, 11(40): 5118-5125. DOI:10.1039/C9AY01531K |
| [15] |
MALEK S, MELGANI F, BAZI Y. One‐dimensional convolutional neural networks for spectroscopic signal regression[J]. Journal of Chemometrics, 2018, 32(5): e2977. DOI:10.1002/cem.2977 |
| [16] |
AULIA M N, KHODRA M L, KOESOEMA A P. Predicting macronutrient of baby food using near-infrared spectroscopy and deep learning approach[J]. IOP Conference Series Materials Science and Engineering, 2020, 803: 012019. DOI:10.1088/1757-899X/803/1/012019 |
| [17] |
NG W, MINASNY B, MONTAZEROLGHAEM M, PADARIAN J, FERGUSON R, BAILEY S, MCBRATNEY B A. Convolutional neural network for simultaneous prediction of several soil properties using visible/near-infrared, mid-infrared, and their combined spectra[J]. Geoderma, 2019, 352: 251-267. DOI:10.1016/j.geoderma.2019.06.016 |
| [18] |
FERGUSON M K, RONAY A K, LEE Y T T, LAW K H. Detection and segmentation of manufacturing defects with convolutional neural networks and transfer learning[J]. Smart and Sustainable Manufacturing Systems, 2018, 2: 1-15. DOI:10.1520/SSMS20180033 |
| [19] |
IOFFE S, SZEGEDY C. Batch normalization: Accelerating deep network training by reducing internal covariate shift[Z/OL]. (2015-02-11)[2015-03-02]. https://arxiv.org/abs/1502.03167v2.
|
| [20] |
SHIN H C, ROTH H R, GAO M, LU L, XU Z, NOGUES I, YAO J, MOLLURA D, SUMMERS M R. Deep convolutional neural networks for computer-aided detection: CNN architectures, dataset characteristics and transfer learning[J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1285-1298. DOI:10.1109/TMI.2016.2528162 |
| [21] |
YOSINSKI J, CLUNE J, BENGIO Y, LIPSON H. How transferable are features in deep neural networks?[J]. Advances in Neural Information Processing Systems, 2014, 27: 3320-3328. |
| [22] |
邹宇晓, 王思远, 廖森泰. 广东省农业科学院蚕桑资源多元化利用研究进展[J]. 广东农业科学, 2020, 47(11): 153-161. DOI:10.16768/j.issn.1004-874X.2020.11.017 ZOU Y X, WANG S Y, LIAO S T. Research progress in diversified utilization of sericultural resources from Guangdong Academy of Agricultural Sciences[J]. Guangdong Agricultural Sciences, 2020, 47(11): 153-161. DOI:10.16768/j.issn.1004-874X.2020.11.017 |
| [23] |
刘燕德, 肖怀春, 韩如冰, 孙旭东, 朱丹宁, 曾体伟, 李泽敏. 柑桔叶片可溶性糖近红外检测非线性模型研究[J]. 广东农业科学, 2016, 43(11): 43-49. DOI:10.16768/j.issn.1004-874X.2016.11.007 LIU Y D, XIAO H C, HAN R B, SUN X D, ZHU D N, ZENG T W, LI Z M. Study on NIR detection non-linear model of soluble sugar in citrus leaves[J]. Guangdong Agricultural Sciences, 2016, 43(11): 43-49. DOI:10.16768/j.issn.1004-874X.2016.11.007 |
| [24] |
XIE L, YANG Z, TAO D, LI G L, YANG X D, SONG J, LIU X W. The model updating based on near infrared spectroscopy for the sex identification of silkworm pupae from different varieties by a semi-supervised learning with pre-labeling method[J]. Spectroscopy Letters, 2019, 52(10): 642-652. DOI:10.1080/00387010.2019.1681463 |
 2021, Vol. 48
2021, Vol. 48