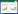文章信息
基金项目
- 国家重点研发计划项目(2019YFD1002205)
作者简介
- 郑雪文(1991—),女,在读硕士生,研究方向为果树生理及分子生物学,E-mail:272880361@qq.com.
通讯作者
- 杨转英(1975—),女,博士,副教授,研究方向为果树生理及分子生物学,E-mail:332933829@qq.com.
文章历史
- 收稿日期:2021-12-06
2. 海南大学园艺学院,海南 海口 570228
2. College of Horticulture, Hainan University, Haikou 570228, China
【研究意义】菠萝(Ananas comosus L. Merr)是凤梨科最重要的经济作物,在许多热带和亚热带国家广泛种植[1],是世界最著名的热带水果之一[2]。根据FAO数据,2019年全球菠萝年产量约为2 600万t。菠萝果实中含有人体必需的维生素C、多种菠萝蛋白酶和有机酸等,具有丰富的营养价值且风味独特,深受广大消费者的喜爱[3]。种植2~3年的菠萝自然结果率一般在60%~80% 之间,开花时间具有不确定性。人工诱导开花不仅能促使菠萝提早开花、增强开花时间的一致性,还能显著提高结果率[4]。乙烯是迄今为止已发现的能直接启动菠萝生殖生长的唯一一种植物激素,国内外学者对外源刺激诱导菠萝成花的研究发现,乙烯利诱导开花的效果除受施用浓度、外界环境和植株生长状况影响外,不同品种菠萝对乙烯利诱花的效果也存在较大差异,这也导致一些对乙烯利不敏感的优良品种在市场上难以推广[5-8]。鉴于此,揭示菠萝成花机理对生产上诱导菠萝开花具有重要的指导作用。【前人研究进展】自然界中植物的花形态各异,花器官通常由萼片、花瓣、雄蕊和心皮组成,以上组成部分在花的生长发育中均具有独特作用[9]。根据ABC模型,花器官的4个组成部分一般由3类基因调控,后来又发现了D类基因SINGSTICK(STK)和E类基因SEPALLATA1-4(SEP1-4)[10-11],构成ABCDE模型。目前研究验证在拟南芥中调控花发育的相关基因有APETALA1(AP1)和APETALA2(AP2)、APETALA3/PISTILLATA(AP3/ PI)、LEAFY(LFY)、FRUITFULL(FUL)、AGAMOUS(AG)等,除AP2外均属于MADSbox家族成员[9, 12-13]。MADS-box转录因子广泛存在于植物中,是参与花器官形成、花期调控、果实发育等的关键基因。MADS-box基因分为Ⅰ型和Ⅱ型两类,Ⅱ型基因比Ⅰ型多3个保守域,可分为MIKC* 和MIKCC两个亚家族。AP3是MADS-box家族MIKCC亚族的一个分支,属于ABCDE模型中的B类功能基因,在拟南芥中主要参与植物花发育中花瓣与雄蕊的形成[11, 14-17]。目前,在多种植物花发育过程中发现AP3起着重要作用,如藏红花CsAP3可调节柱头发育[18]。白菜和油菜的B.AP3.a基因决定花瓣和雄蕊的发育,而B.AP3.b基因只参与雄蕊的发育[19];在番茄中,TAP3参与雄蕊和花瓣的发育,TM6则主要调控雄蕊的发育[20]。【本研究切入点】目前在菠萝中关于AP3基因及其功能的研究尚没有相关报道。【拟解决的关键问题】本研究从乙烯诱导后的巴厘品种菠萝中克隆出一个新的AP3同源全长基因,命名为AcMADS41,通过生物信息学技术对该基因结构、保守结构域、物种进化树等进行系统分析,并利用RNA-sep分析其在菠萝花芽发育不同阶段和不同花器官中的表达差异,为进一步研究AcMADS41在菠萝成花过程中的功能奠定基础。
1 材料与方法 1.1 试验材料供试菠萝植株品种为巴厘(Ananas comosus L. cv. Comte de Paris),选取200株健康生长植株,使用30 mL浓度为200 mg/L的乙烯利灌心处理[4],以清水为对照,在处理后0、4、8、16、32、48 d分别对花芽进行取样,花器官分化后对不同花器官分别采样单独保存,用液氮速冻,保存于-80℃。
RNA提取试剂盒Quick RNA isolation Kit购于北京华越洋生物科技有限公司,逆转录试剂盒Thermo Scientific RevertAid First Strand cDNA Synthesis Kit购于Thermo Fisher Scientific,Trans TaqⓇ DNA polymerase High Fidelity试剂盒、pEASY-T1载体和DH5α大肠杆菌转化菌株购于北京全式金生物技术有限公司。试验所用仪器包括Eppendorf离心机、Germany超微量分光光度计、Thermo Fisher Scientific PCR仪。
1.2 总RNA的提取及反转录根据多糖多酚植物RNA提取试剂盒说明书操作,从菠萝花芽与花器官中提取总RNA,并用1% 琼脂糖凝胶电泳检测RNA完整性。通过超微量紫外分光光度计检测RNA浓度和质量后,用Thermo Scientific RevertAid First Strand cDNA Synthesis Kit进行反转录,得到cDNA[5, 21]。
1.3 目的基因的克隆通过前期转录组测序结果分析得到AcMADS41的CDS序列,使用Primer Premier设计目的基因的引物(AcMADS-F、AcMADS-R,表 1),以菠萝花芽的混合cDNA为模板。用Trans TaqⓇ DNA Polymerase High Fidelity试剂盒克隆目的基因的片段,PCR反应程序为:94℃预变性3 min,94℃变性30 s、55℃退火30 s、72℃延伸30 s,38个循环,72℃后延伸7 min。扩增结果用1% 琼脂糖凝胶电泳进行检测。将得到的目的基因进行切胶回收,得到纯化产物与pEASY-T1载体连接并转化大肠杆菌感受态中,阳性检测正确后送测序。
1.4 AcMADS41的生物信息学分析
1.4.1 AcMADS41的理化性质分析 从菠萝基因组数据中搜索得到AcMADS41的全长序列,通过拟南芥数据库(https://www.arabidopsis.org/index.jsp)得到拟南芥AtAP3的CDS序列和DNA全长序列,将CDS序列和DNA全长序列用Bioxm 2.6软件进行比对,比对结果通过Exon-Intron Graphic Maker在线网站进行修饰。用Expasy-Translate分析并翻译AcMADS41的CDS序列,使用ExpasyProtParam(https://web.expasy.org/protparam/)分析该基因氨基酸序列的等电点(PI)、分子量(MW)、氨基酸组成、蛋白不稳定性指数和脂肪族指数等。使用SWISS-MODEL预测蛋白的三级结构模型(https://swissmodel.expasy.org/)[3]。
1.4.2 AcMADS41基因结构、保守域和同源性分析 通过NCBI数据库(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析AcMADS41蛋白结构域。在NCBI和PlantTFDB数据库进行blastP得到AcMADS41不同物种的同源蛋白序列并下载,通过clustalX对同源蛋白序列进行多序列比对,用Mega6.0通过邻位连接法(NJ)构建系统进化树,Bootstrap重复次数为1 000次。选取其中7个物种的同源蛋白序列和AcMADS41蛋白序列用DNAMAN进行多序列比对。
1.4.3 菠萝AcMADS41启动子顺式作用元件分析 通过菠萝基因组数据库(http://pineapple.angiosperms.org/pineapple/html/index.html)搜索得到AcMADS41基因的启动子序列,在PlantCARE在线网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)上进行分析[22],得到的数据再通过Tbtools软件进一步处理。
1.5 菠萝AcMADS41基因的表达分析通过菠萝基因组数据库搜索得到菠萝花、果、叶和根组织部位的基因组数据进行分析。从转录组(论文待发表)数据中分析AcMADS41在乙烯利处理后0、4、8、16、32、48 d菠萝花芽中的表达情况,并分析其在不同花器官中的表达。使用实时荧光定量PCR(qRT-PCR)方法对菠萝AcMADS41在花器官不同组成部分的表达进行分析。使用Primer Premier设计目的基因的引物,以Acactin作为内参基因(表 2)。试验所用试剂为Thermo Fisher Scientific的PowerUp TM SYBR TM Green预混液,仪器为LightCycler 480 Ⅱ实时荧光定量PCR仪。qRT-PCR反应终体系为10 µL,qRTPCR反应条件:50 ℃ 2 min,95 ℃ 2 min,95 ℃ 15 s、57 ℃ 15 s、72 ℃ 30 s,共40个循环。每个样品3次重复,利用2-ΔΔCt法计算AcMADS41相对表达量。
1.6 菠萝AcMADS41蛋白互作分析
以拟南芥作为参考,使用String在线网站(https://cn.string-db.org/)对AcMADS41蛋白与其他蛋白间的互作进行预测。
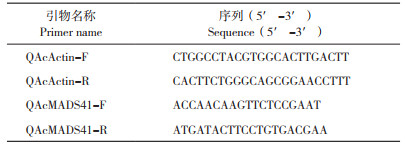
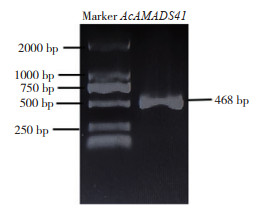
2 结果与分析 2.1 AcMADS41基因的全长克隆从菠萝花芽与花器官中提取总RNA,通过1% 琼脂糖凝胶电泳和超微量紫外分光光度计检测质量和浓度,电泳条带清晰完整,浓度为500~ 1 000 ng/μL,OD260/280比值在2.0~2.2之间,可以进行后续试验。以反转录得到的cDNA为模板,通过PCR技术扩增AcMADS41基因的全长cDNA序列,电泳检测其长度为468 bp(图 1)。连接pEASY-T1载体并转化大肠杆菌感受态(DH5α),测序结果显示序列正确。
|
| 图 1 AcMADS41基因的全长克隆电泳结果 Fig. 1 Full-length clone electrophoresis result of AcMADS41 gene |
2.2 AcMADS41的生物信息学分析
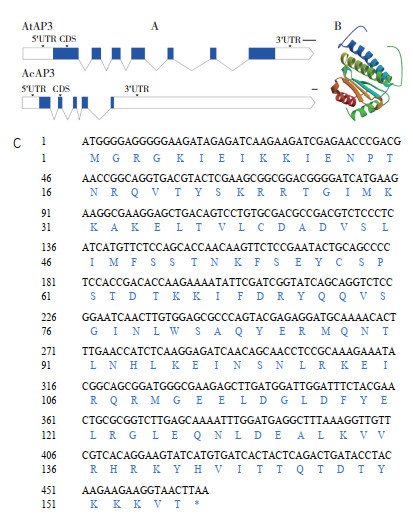
2.2.1 AcMADS41的理化性质分析 AcMADS41 基因组DNA全长序列为5 098 bp,编码区为458 bp,编码155个氨基酸(图 2C)。将拟南芥AP3(AtAP3)和菠萝AP3(AcMADS41)AcMADS41基因组全长序列比对后发现该基因内含子、外显子与拟南芥的差异显著(图 2A)。AtAP3基因含有5'UTR、3'UTR、7个外显子和6个内含子,而AcMADS41基因含有5'UTR、3'UTR、5个外显子和4个内含子,后者3'UTR的长度远大于前者3'UTR的长度。根据Expasy-ProtParam预测分析结果,AcMADS41蛋白的分子量(MW)为18 242.90,等电点(PI)为9.56,赖氨酸(Lys)含量最高、占总氨基酸数的10.2%,带负电荷的残基总数(Asp + Glu)为20,带正电荷的残基总数(Arg + Lys)为29。该蛋白的不稳定性指数(Ⅱ)为31.21,表明蛋白质稳定;脂肪指数为75.42,亲水性的平均值(GRAVY)为-0.849,表明该蛋白为亲水蛋白。以6byy.2.A为模板[23],通过SWISS-MODEL预测AcAP3蛋白的三级结构模型发现,该模型为同型二聚体,序列相似性为0.43,全球模型质量估计(GMQE)为0.23,QMEAN为-0.54(图 2B),表明预测模型的质量较高。
|
| A:菠萝和拟南芥AP3基因的DNA结构;B:菠萝AcMADS41蛋白的三级结构模型预测;C:菠萝AcMADS41 cDNA序列及编码氨基酸序列 A: DNA structure of AP3 genes in pineapple and Arabidopsis thaliana; B: Prediction of the tertiary structure model of pineapple AcMADS41 protein; C: cDNA sequence and coding amino acid sequence of AcMADS41 in pineapple 图 2 AcMADS41的理化性质预测分析 Fig. 2 Prediction and analysis of physicochemical property of AcMADS41 |
2.2.2 AcMADS41蛋白结构域预测和同源性比对 通过NCBI数据库预测AcAP3蛋白结构域,结果(图 3A)显示,该蛋白在2~80 bp处含有高度保守的MADS域(MADS-box),在83~153 bp处含有K域(K-box),表明该蛋白为MIKC型MADS-box基因中MIKCc型蛋白的一员。
通过NCBI数据库和PlantTFDB数据库得到多个与AcMADS41同源的其他蛋白序列,并选取海枣(Phoenix dactylifera)、油棕(Elaeis guineensis)、石刁柏(Asparagus officinalis)、鹤顶兰(Phaius tancarvilleae)、拟南芥(Arabidopsis thaliana)、短柄草(Brachypodium distachyon)和本氏烟草(Nicotiana benthamiana)等7个物种的AP3同源蛋白与AcMADS41蛋白进行多序列比对,结果(图 3B)发现其序列长度相对较短,但在同等长度部分与其他序列之间的相似性较高,均含有MADS-box和K-box结构域。

|
| 图 3 AcMADS41蛋白的保守域预测分析 Fig. 3 Prediction and analysis of AcMADS41 protein domain |
利用MEGA6.0软件对AcMADS41氨基酸序列与其他22个物种中的同源蛋白进行系统进化树分析,结果(图 4)发现Ac进化树主要分为单子叶植物和双子叶植物两大类。菠萝AcMADS41氨基酸序列与海枣和油棕被聚类到同一条分支上,可见其与海枣、油棕等热带植物中AP3同源蛋白的亲缘关系较近。
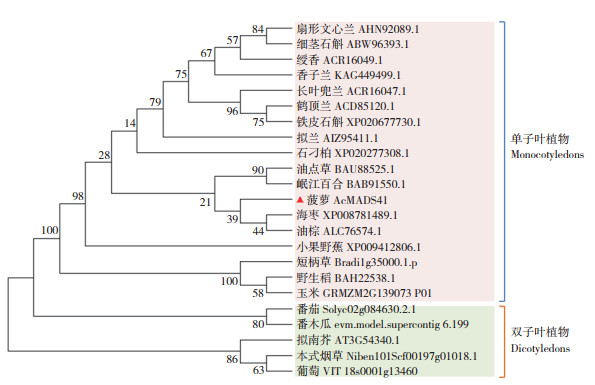
|
| 图 4 AcMADS41的系统进化分析 Fig. 4 Phylogenetic analysis of AcMADS41 |
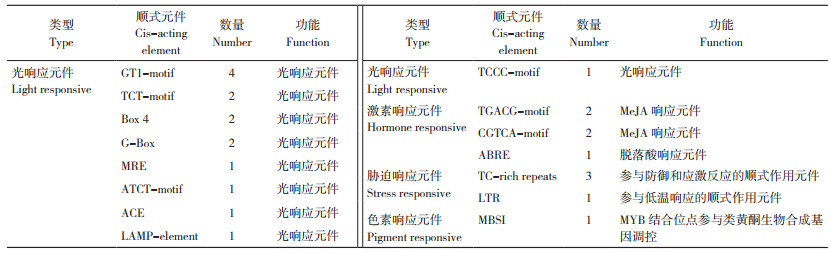
2.2.3 菠萝AcMADS41启动子顺式作用元件预测分析 在菠萝基因组数据库搜索得到一个2 468 bp的AcMADS41启动子。经PlantCARE预测启动子顺式作用元件结果(图 5)显示,AcMADS41启动子有19种、97个顺式作用元件,共97个,除TATA-box和CAAT-box等启动子基本元件外,还有光响应元件、激素响应元件、胁迫响应元件等(表 3),其中光响应元件的数量最多。
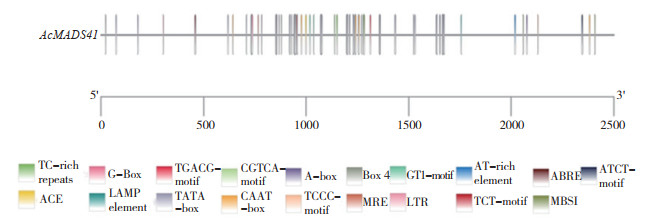
|
| 图 5 菠萝AcMADS41基因启动子顺式作用元件预测分析 Fig. 5 Prediction and analysis of cis-acting elements of AcMADS41 gene promoter in pineapple |
|
2.3 菠萝AcMADS41基因的表达分析
从菠萝基因组数据中搜索得到菠萝花、叶、果、根4个部位的AcMADS41基因的表达数据并分析,结果(图 6 A)发现该基因只在菠萝花组织中高度表达,表明其可能与菠萝花发育有关。从转录组数据中分析菠萝AcMADS41在花芽发育过程中的表达情况,发现该基因在乙烯利处理后32 d才开始变化,在处理后48 d高度表达(图 6 B),推测该基因主要作用于菠萝花器官发育阶段。
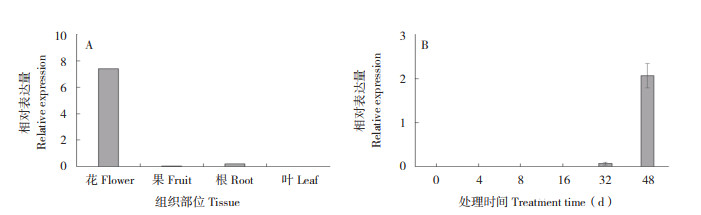
|
| 图 6 菠萝AcMADS41基因表达分析 Fig. 6 Expression analysis of ACMADS41 in pineapple |
利用qRT-PCR对AcMADS41在不同组织部位和花器官中的相对表达量进行分析,以热图形式展示定量结果。由图 7可以看出,AcMADS41在花器官- 花柱中的表达量最高,且明显高于其他花器官组成部分,其次在花瓣和雄蕊中也有部分表达量,推测AcMADS41的作用主要是参与菠萝花柱的发育。
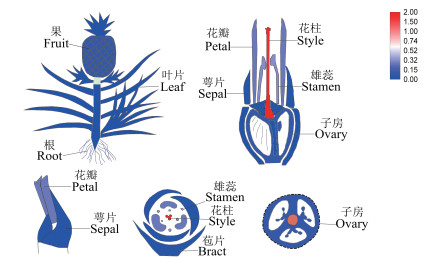
|
| 图 7 菠萝AcMADS41的组织特异性表达 Fig. 7 Specific expression of AcMADS41 in pineapple tissues |
2.4 AcMADS41蛋白互作预测分析
使用String在线网站对AcMADS41蛋白与其他蛋白间的互作进行预测,以拟南芥作为参考,预测结果(图 8)显示该蛋白可能与LFY、FYPP3、UFO、PI等10个蛋白进行互作。UFO与LFY功能联系综合得分相对较高,互作的可能性更大,推测该蛋白参与了花的发育。AcMADS41还可能与PI和SUP共表达。PI与AP3的同源性最高,在拟南芥中PI与AP3形成异源二聚体,共同参与花瓣和雄蕊的发育[24]。推测菠萝中的AcMADS41蛋白也有可能与PI蛋白形成异源二聚体共同参与花的发育。
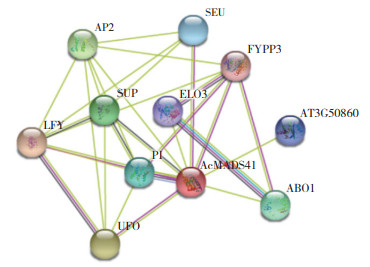
|
| 图 8 菠萝AcMADS41蛋白与其他蛋白的功能预测 Fig. 8 Function prediction of AcMADS41 protein and other proteins in pineapple |
3 讨论
花和果实发育的机理研究一直是植物界研究的重要方向,MADS-box基因家族作为调控花果发育的一类关键基因近年来也备受关注。AP3是MADS-box家族MIKCC亚族的一个分支,含有典型的MADS-box和K-box结构域,属于ABCDE开花模型中的B类功能基因[17],在拟南芥[12]、萝卜[25]、番茄[26]、梅花[27]、莲[28]等多种植物中已被证实参与调控花瓣和雄蕊的发育。但近年发现AP3基因不仅限于花瓣和雄蕊中表达。在兰花中,高阶SP复合体(OAP3-1/OAGL6-1/ OAGL6-1/OPI)调控萼片和花瓣的发育;OAP3和OAGL6基因的协同作用可能调控花被和唇瓣的颜色分化[29-30]。在藏红花中,CsAP3基因参与花被和雄蕊的发育,是调节柱头发育的重要基因[18, 31]。在葡萄中,VvAP3在花瓣、心皮和雄蕊中均为高表达[32]。本研究从菠萝花芽中克隆出一个AP3同源基因AcMADS41,通过保守域分析证实其含有典型MADS-box和K-box结构域。通过与拟南芥DNA结构和7个不同物种的AP3同源序列进行多序列分析,发现其氨基酸序列长度较短,但在DNA结构中3'UTR序列长度较长。
通过系统进化树分析发现该基因与油棕和海枣的亲缘关系较近,菠萝、油棕和海枣均为单子叶热带植物。油棕中AP3同源蛋白EgDEF1通常在细胞内形成DEF/GLO异二聚体复合物,共同参与花器官的表达,目前已证实EgGLO2在雄花和雌花中均有表达,可见EgDEF1也可能参与雄花和雌花的形成[33];石刁柏AoDEF基因则在雄蕊和内部花被中表达[34]。
根据基因组数据分析AcMADS41基因在菠萝根、果、花和叶4个部位的相对表达量,发现AcMADS41在花中的表达量最高,在其他3个部位几乎不表达,更加验证了“AcMADS41参与菠萝花的发育”这一推断。B类功能基因一般参与花瓣和雄蕊的形成,AcMADS41是AP3-Like基因,分析其在菠萝花芽发育不同阶段的转录组数据,发现其在成花后期表达量最高、在前期几乎不表达,推测它可能参与花器官的形成。本研究发现AcMADS41在花器官- 花柱中的表达量最高,其次在花瓣和雄蕊中也有表达,推测AcMADS41主要参与了菠萝花柱的发育,这与藏红花中的CsAP3基因功能[18, 31]相似。
通过与其他蛋白质的互作关系预测,发现AcMADS41与LFY、FYPP3、UFO、PI等10个蛋白可能进行互作,其中与UFO、LFY蛋白互作的可能性更大。这些蛋白大多与成花相关,推测AcMADS41蛋白在菠萝成花过程中可起到除参与花器官形成外的其他作用,这为进一步研究菠萝成花分子机制奠定了理论基础。
4 结论本研究在菠萝中克隆得到1个新的AP3全长基因AcMADS41,通过对AcMADS41进行一系列生物信息学预测分析,发现AcMADS41含有MIKC型MADS-box家族特有的MADS-BOX和K-BOX结构域,其蛋白质较稳定。经系统进化树分析发现菠萝AcMADS41与海枣、油棕等热带单子叶植物中AP3同源蛋白的亲缘关系最近。AcMADS41启动子中有19种顺式作用元件,除TATA-box和CAAT-box等启动子基本元件外,光响应元件的数量最多。经基因组与转录组数据分析和组织特异性表达分析发现,AcMADS41在花柱中相对表达量最高,推测AcMADS41在菠萝花柱发育过程中起着重要作用。
| [1] |
RIOS V M, GOMEZ HERRERA M D, SUGITA N H, ALAYON LUACES P. Water status response of pineapple using destructive and non-destructive indicators and their relations in two contrasting seasons[J]. Journal of the Saudi Society of Agricultural Sciences, 2020, 19(8): 538-547. DOI:10.1016/j.jssas.2020.10.002 |
| [2] |
HADIDI M, AMOLI P I, JELYANI A Z, HASIRI Z, ROUHAFZA A, IBARZ A, KHAKSAR F B, TABRIZI S T. Polysaccharides from pineapple core as a canning by-product: Extraction optimization, chemical structure, antioxidant and functional properties[J]. International Journal of Biological Macromolecules, 2020, 163: 2357-2364. DOI:10.1016/j.ijbiomac.2020.09.092 |
| [3] |
荆永琳, 耿宏叶, 徐立, 李志英. 菠萝叶刺形成相关基因AcSPL14克隆与载体构建[J]. 分子植物育种, 2019, 17(2): 487-493. DOI:10.13271/j.mpb.017.000487 JING Y L, GENG H Y, XU L, LI Z Y. Cloning and vector construction of pineapple leaf thorn related gene AcSPL14[J]. Molecular Plant Breeding, 2019, 17(2): 487-493. DOI:10.13271/j.mpb.017.000487 |
| [4] |
林朝宗. 人工控制菠萝开花结果技术初探[J]. 福建热作科技, 1987(3): 16-17. LIN C Z. Preliminary study on artificial control of flowering and fruiting of pineapple[J]. Fujian Science & Technology of Tropical Crops, 1987(3): 16-17. |
| [5] |
张红娜, 刘胜辉, 孙伟生, 李运合, 孙光明, 林文秋, 张秀梅, 吴青松. 49菠萝对乙烯利诱花的敏感性差异研究[J]. 热带作物学报, 2018, 39(6): 1087-1094. DOI:10.3969/j.issn.1000-2561.2018.06.008 ZHANG H N, LIU S H, SUN W S, LI Y H, SUN G M, LIN W Q, ZHANG X M, WU Q S. Ethylene induced sensitivity of difference pineapple varieties[J]. Chinese Journal of Tropical Crops, 2018, 39(6): 1087-1094. DOI:10.3969/j.issn.1000-2561.2018.06.008 |
| [6] |
张治礼, 范鸿雁, 华敏, 何凡. 菠萝开花诱导及其生理与分子基础[J]. 热带作物学报, 2012, 33(5): 950-955. DOI:10.3969/j.issn.1000-2561.2012.05.032 ZHANG Z L, FAN H Y, HUA M, HE F. Induction of pineapple flowering and underlying physiological and molecular bases[J]. Chinese Journal of Tropical Crops, 2012, 33(5): 950-955. DOI:10.3969/j.issn.1000-2561.2012.05.032 |
| [7] |
刘胜辉, 吴青松, 张秀梅, 孙伟生, 孙光明. 活性炭与分子筛吸附乙烯诱导巴厘菠萝开花的研究[J]. 广东农业科学, 2014, 41(10): 38-41. DOI:10.16768/j.issn.1004-874X.2014.10.034 LIU S H, WU Q S, ZHANG X M, SUN W S, SUN G M. Effects of active carbon and zeolite molecular sieve with saturated ethylene on flower forcing of Yellow Mauritius pineapple[J]. Guangdong Agricultural Sciences, 2014, 41(10): 38-41. DOI:10.16768/j.issn.1004-874X.2014.10.034 |
| [8] |
刘胜辉, 臧小平, 孙光明, 叶春海. 乙烯利对珍珠菠萝综合催花效应研究[J]. 广东农业科学, 2009, 8(8): 78-80. DOI:10.16768/j.issn.1004-874X.2009.08.047 LIU S H, ZANG X P, SUN G M, YE C H. Integrated effects of ethrel on pineapple's(Zhenzhu cv.)flower induction[J]. Guangdong Agricultural Sciences, 2009, 8: 78-80. DOI:10.16768/j.issn.1004-874X.2009.08.047 |
| [9] |
IRISH V. The ABC model of floral development[J]. Current Biology, 2017, 27(17): 887-890. DOI:10.1016/j.cub.2017.03.045 |
| [10] |
JIANG Y C, WANG M M, ZHANG R, XIE J H, DUAN X S, SHAN H Y, XU G X, KONG H Z. Identification of the target genes of AqAPETALA3-3(AqAP3-3)in Aquilegia coerulea(Ranunculaceae) helps understand the molecular bases of the conserved and non-conserved features of petals[J]. New Phytologist, 2020, 227(4): 1235-1248. DOI:10.1111/nph.16601 |
| [11] |
WON S Y, JUNG J A, KIM J S. Genome-wide analysis of the MADS-Box gene family in Chrysanthemum[J]. Computational Biolog y and Chemist r y, 2021, 90: 107424. DOI:10.1016/j.compbiolchem.2020.107424 |
| [12] |
戚晓利, 卢孟柱. 拟南芥APETALA1基因在花发育中的网络调控及其生物学功能[J]. 中国农学通报, 2011, 27(8): 103-107. QI X L, LU M Z. Regulation network and biological roles of APETALA1 of Arabidopsis thaliana in f lower development[J]. Chinese Agricultural Science Bulletin, 2011, 27(8): 103-107. |
| [13] |
ZHANG Z B, JIN Y J, WAN H H, CHENG L, FENG Z G. Genome-wide identification and expression analysis of the MADS-box transcription factor family in Camellia sinensis[J]. Journal of Applied Genetics, 2021, 62(2): 249-264. DOI:10.1007/s13353-021-00621-8 |
| [14] |
ABRAHAM-JUAREZ M J, SCHRAGER-LAVELLE A, MAN J, WHIPPLE C, HANDAKUMBURA P, BABBITT C, BARTLETT M. Evolutionary variation in MADS-box dimerization affects floral development and protein abundance in maize[J]. The Plant Cell, 2020, 32(11): 3408-3424. DOI:10.1105/tpc.20.00300 |
| [15] |
ZHU P P, DONG T T, XU T, KANG H S. Identification, characterisation and expression analysis of MADS-box genes in sweetpotato wild relative Ipomoea trifida[J]. Acta Physiologiae Plantarum, 2020, 42(11): 1-13. DOI:10.1007/s11738-020-03153-6 |
| [16] |
GHORBANI M M, BAGHERI H, GHOLAMI M. Genome-wide study of flowering-related MADS-box genes family in Cardamine hirsuta[J]. Biotech, 2020, 10(12): 1-14. DOI:10.1007/s13205-020-02521-w |
| [17] |
范丙友, 高水平, 侯小改, 胥华伟, 史国安, 孔祥生. Col生态型拟南芥AP3基因启动子克隆及植物表达载体构建[J]. 基因组学与应用生物学, 2011, 30(1): 21-26. DOI:10.3969/gab.030.000021 FAN B Y, GAO S P, HOU X G, XU H W, SHI G A, KONG X S. Molecular cloning of promoter of AP3 gene from Arabidopsis thaliana ecotype(Col)and construction of the plant expression vector[J]. Plant Gene and Trait, 2011, 30(1): 21-26. DOI:10.3969/gab.030.000021 |
| [18] |
WAFAI A H, HUSAINI A M, QADRI R A. Temporal expression of floral proteins interacting with CArG1 region of CsAP3 gene in Crocus sativus L[J]. Gene Reports, 2019, 16: 100446. DOI:10.1016/j.genrep.2019.100446 |
| [19] |
ZHANG Y F, WANG X F, ZHANG W X, YU F, TIAN J H, LI D R, GUO A G. Functional analysis of the two brassica AP3 genes involved in apetalous and stamen carpelloid phenotypes[J]. PLOS ONE, 2011, 6(6): e20930. DOI:10.1371/journal.pone.0020930 |
| [20] |
DE MARTINO G, PAN I, EMMANUEL E. Functional analyses of two tomato APETALA3 genes demonstrate diversification in their roles in regulating floral development[J]. The Plant Cell, 2006, 18(8): 1833-1845. DOI:10.1105/tpc.106.042978 |
| [21] |
刘开, 余炳伟, 李丹丹, 陈娜, 曹必好. 茄子SmWRKY65基因克隆及其对青枯病的抗性分析[J]. 广东农业科学, 2021, 48(3): 42-52. DOI:10.16768/j.issn.1004-874X.2021.03.006 LIU K, YU B W, LI D D, CHEN N, CAO B H. Cloning of SmWRKY65 gene and its resistance to bacterial wilt in eggplant[J]. Guangdong Agricultural Sciences, 2021, 48(3): 42-52. DOI:10.16768/j.issn.1004-874X.2021.03.006 |
| [22] |
周梦蝶, 王泽玉, 王辰雨, 杜娟, 宋剑灵, 刘华, 陈曙, 解新明, 钟天秀. 御谷RAV1基因及启动子的克隆与生物信息学分析[J/OL]. 分子植物育种, 2021: 1-20[2021-03-16]. http://kns.cnki.net/kcms/detail/46.1068.S.20210303.0957.004.html. ZHOU M D, WANG Z Y, WANG C Y, DU J, SONG J L, LIU H, CHEN S, XIE X M, ZHONG T X. Cloning and bioinformatics analysis of the RAV1 gene and promoter in pennisetum glaucum[JJ/OL]. Molecular Plant Breeding, 2021: 1-20[2021-03-16]. http://kns.cnki.net/kcms/detail/46.1068.S.20210303.0957.004.html. |
| [23] |
STEINEGGER M, MEIER M, MIRDITA M, VOHRINGER H, HAUNSBERGER S J, SODING J. HH-suite3 for fast remote homology detection and deep protein annotation[J]. BMC Bioinformatics, 2019, 20(1). DOI:10.1186/s12859-019-3019-7 |
| [24] |
WUEST S E, O'MAOILEIDIGH D S, RAE L, KWASNIEWSKA K, RAGANELLI A, HANCZARY K, LOHAN A J, LOFTUS B, LORTUS B, GRACIET E, WELLMER F. Molecular basis for the specification of floral organs by APETALA3 and PISTILLATA[J]. Proceedings of the National Academy of Sciences, 2012, 109(33): 13452-13457. DOI:10.1073/pnas.1207075109 |
| [25] |
LI C, WANG Y, XU L, NIE S S, CHEN Y L, LIANG D Y, SUN X C, KARANJA B K, LUO X B, LIU L W. Genome-wide characterization of the MADS-Box gene family in radish(Raphanus sativus L.) and assessment of its roles in flowering and floral organogenesis[J]. Frontiers in Plant Science, 2016, 26(7): 1390. DOI:10.3389/fpls.2016.01390 |
| [26] |
WANG Y S, ZHANG J L, HU Z L, GUO X H, TIAN S B, CHEN G P. Genome-wide analysis of the MADS-Box transcription factor family in Solanum lycopersicum[J]. International Journal of Molecular Sciences, 2019, 20(12): 2961. DOI:10.3390/ijms20122961 |
| [27] |
XU Z D, ZHANG Q X, SUN L D, DU D L, CHENG T G, PAN H T, YANG W R, WANG J. Genome-wide identification, characterisation and expression analysis of the MADS-box gene family in Prunus mume[J]. Molecular Genetics and Genomics, 2014, 289(5): 903-920. DOI:10.1007/s00438-014-0863-z |
| [28] |
LIN Z Y, CAO D G, DAMARIS R N, YANG P F. Genome-wide identification of MADS-box gene family in sacred lotus(Nelumbo nucifera)identifies a SEPALLATA homolog gene involved in floral development[J]. BMC Plant Biology, 2020, 20(1): 1-15. DOI:10.1186/s12870-020-02712-w |
| [29] |
HSU H F, CHEN W H, SHEN Y H, HSU W H, MAO W T, YANG C H. Multifunctional evolution of B and AGL6 MADS box genes in orchids[J]. Nature Communications, 2021, 12(1): 1-12. DOI:10.1038/s41467-021-21229-w |
| [30] |
LI B J, ZHENG B Q, WANG J Y, TSAI W C, LU H C, ZOU L H, WAN X, ZHANG D Y, QIAO H J, LIU Z J, WANG Y. New insight into the molecular mechanism of colour differentiation among floral segments in orchids[J]. Communications Biology, 2020, 3(1): 6253-6255. DOI:10.1038/s42003-020-0821-8 |
| [31] |
WAFAI A H, BUKHARI S, MOKHDOMI T A, AMIN A, WANI Z, HUSSAINI A, MIR J I, QADRI R A. Comparative expression analysis of senescence gene CsNAP and B-class floral development gene CsAP3 during different stages of flower development in Saffron(Crocus sativus L.)[J]. Physiology and Molecular Biology of Plants, 2015, 21(3): 459-463. DOI:10.1007/s12298-015-0307-1 |
| [32] |
GRIMPLET J, MARTINEZ-ZAPATER J M, CARMONA M J. Structural and functional annotation of the MADS-box transcription factor family in grapevine[J]. BMC Genomics, 2016, 17(1): 1-23. DOI:10.1186/s12864-016-2398-7 |
| [33] |
ADAM H, JOUANNIC S, MORCILLO F, RICHAUD F, DUVAL Y, TREGEAR J W. MADS Box genes in oil palm(Elaeis guineensis): Patterns in the evolution of the SQUAMOSA, DEFICIENS, GLOBOSA, AGAMOUS, and SEPALLATA subfamilies[J]. Journal of Molecular Evolution, 2006, 62(1): 15-31. DOI:10.1007/s00239-005-0333-7 |
| [34] |
PARK J H, ISHIKAWA Y, YOSHIDA R, KANNO A, KAMEYA T. Expression of AODEF, a B-functional MADS-box gene, in stamens and inner tepals of the dioecious species Asparagus officinalis L[J]. Plant Molecular Biology, 2003, 51(6): 867-875. DOI:10.1023/a:1023097202885 |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49