文章信息
基金项目
- 国家自然科学基金(31871716);广东省农业科学院院长基金(202023);广州市科技计划项目(202102021009);广东省农业科学院创新基金(202118)
作者简介
-
贾俊婷,发育生物学博士,助理研究员。毕业于中国科学院华南植物研究所,主要从事农作物种质资源深度鉴评及创新利用。先后主持广东省基础与应用基础研究粤穗联合基金项目、广州市基础与应用基础研究项目和广东省农业科学院院长基金等科研项目,在《BMC Plant Biology》 《Journal of Experimental Botany》《BMC Genomics》《Plant Biotechnology Journal》《Frontiers in Plant Science》等国内外学术期刊上发表论文、著作13篇,其中第一作者4篇。申请国家发明专利4项,授权1项。
贾俊婷(1988—),女,博士,助理研究员,研究方向为优异基因挖掘及功能,E-mail:jiajunting123@126.com; 刘军,博士,研究员,现任广东省农业科学院农业生物基因研究中心首席专家,广东省农作物种质资源保存与利用重点实验室主任,长期致力于农作物种质资源收集保存以及相关种子生物学研究。先后主持国家自然科学基金、广东省科技计划项目、广东省自然科学基金项目等30余项。以笫一作者或通信作者在《Plant Journal》《Rice》 《Plant and Cell Physiology》《BMC Plant Biology》《植物学报》《中国农业科学》等国内外期刊发表高水平论文40多篇;编写和参与编写著作3部;以笫一发明人获得授权发明专利10项;已研发产品6个;获省市各级科技成果奖励6项.
通讯作者
- 刘军(1969—),男,博士,研究员,研究方向为农作物种质资源保存与利用,E-mail:liujun@gdaas.cn.
文章历史
- 收稿日期:2022-09-05
【研究意义】非核苷酸序列的改变而产生的遗传变异称之为表观遗传,常见的有组蛋白修饰、染色质重塑和DNA甲基化等[1]。水稻作为我国重要的粮食作物,对其组蛋白进行深入研究具有重大意义。目前我们对水稻的表观调控机制仍知之甚少。本研究以水稻组蛋白去甲基化酶基因OsJMJ719为研究对象,对其进行基因克隆,预测其蛋白质分子结构,并进一步探究该基因与其他不同物种相似蛋白的亲缘关系,为进一步解析OsJMJ719基因的功能奠定基础。【前人研究进展】染色质的基本结构单位是核小体,核小体由146 bp DNA和碱性核心组蛋白组成。核心组蛋白特定位点的赖氨酸和精氨酸残基上容易发生甲基化、乙酰化、磷酸化、泛素化等共价修饰[2],从而提供表观遗传信息,调控染色质的功能,如基因的活性、染色质的结构、表观遗传记忆等。组蛋白上的各种共价修饰都是动态的、可逆的酶促反应过程。组蛋白甲基转移酶和去甲基化酶协同调控组蛋白甲基化。组蛋白甲基化修饰通常发生在组蛋白H3和H4 N末端的精氨酸和赖氨酸残基上[3]。组蛋白赖氨酸去甲基化酶(histone lysine demethylase,KDM)根据其结构特征分为两大类,一类是以FAD为辅因子的LSD1家族蛋白(lysine specific demethylase 1);另一类是以亚铁离子(Fe2+)和α-酮戊二酸(α-KG)为辅因子的JmjC家族蛋白(JHDM, JmjC-domain-containing histone demethylase),在组蛋白的去甲基化调控过程中起主导作用[4]。相关研究表明,水稻中的JmjC结构蛋白在调控基因表达、控制水稻发育方面具有一定功能,其编码基因有20个,依次为OsJMJ701、OsJMJ702……OsJMJ719、OsJMJ720。根据进化关系JmjC蛋白可分为5个亚家族,分别为JHDM1/KDM5家族、JHDM3/KDM4家族、JHDM2/KDM3家族、JMJD6家族和JmjC domain-only家族蛋白[5]。近年研究表明,组蛋白去甲基化酶在植物的生长发育、种子萌发和逆境反应等方面都起着重要作用[6-9]。OsJMJ703专一性作用于水稻组蛋白H3K4me2/3,T-DNA插入突变体与野生型相比,株高降低、二次枝梗减少且粒型发生变化[10];OsJMJ703也参与干旱胁迫,敲除突变体表现为对干旱的耐受性增强[11]。OsJMJ705是一个特异的水稻组蛋白H3K27me2/3去甲基化酶,正调控白叶枯病菌抗性基因的表达,还参与水稻生物胁迫应答和生殖过程,表现为突变体植株矮小、结实率下降[12]。在体外OsJMJ706可以导致H3K9me2/3的去甲基化,其通过调控花发育有关基因,影响小穗发育,导致内、外稃数目改变及雄、雌蕊数改变[13]。OsJMJ714作用于H3K9me1/2/3的去甲基化,调控水稻中多种植物激素如生长素代谢相关基因的表达,造成水稻根系的发育过程紊乱[14]。【本研究切入点】组蛋白H3末端的甲基化修饰可以抑制或者沉默植物生长发育基因及逆境响应基因的表达,本研究以一个组蛋白去甲基化酶基因为研究对象,研究其启动子结构、亚细胞定位及在响应非生物胁迫过程中的表达模式。【拟解决的关键问题】本研究从水稻品种中花11中克隆出OsJMJ719(LOC_Os02g01940)基因;通过生物信息学技术对该基因结构、蛋白结构、物种进化树等进行系统分析;构建其亚细胞定位载体,对其蛋白进行亚细胞定位进行观察;并利用实时荧光定量PCR分析其在水稻组织表达模式、不同逆境条件下的表达模式,从而为进一步研究OsJMJ719的生物学功能奠定基础。
1 材料与方法 1.1 试验材料供试水稻品种为粳稻(Oryza sativa ssp. Japonica)ZH11;小叶烟草为本实验室保存,用于亚细胞定位观察,在温度22~23 ℃,光周期为16 h/8 h条件下培养1个月后备用。基因克隆载体pMD18-T simple购于TaKaRa公司;亚细胞定位GFP载体pCAMBIA1302购买于武汉艾迪晶生物科技有限公司、大肠杆菌E. coli DH5α和农杆菌GV1301为本实验室保存。RNA提取试剂盒购自GenStar公司,反转录试剂盒Prime-ScriptTMRT reagent Kit购自TaKaRa公司。水稻中花11两周大的幼苗,分别使用50 μmol/L ABA、300 mmol/L NaCl、20% PEG、4 ℃低温、25 μmol/L乙烯前体ACC处理1 h和3 h,取地上部分,设3个生物学重复,用液氮速冻后置于-80 ℃冰箱备用。
1.2 试验方法1.2.1 水稻OsJMJ719基因的克隆OsJMJ719 (NCBI GenBank登录号:XM_015767632.2)预测为Oryza sativa Japonica Group lysine-specific demethylase,根据其保守序列,用Primer 5.0设计引物,引物序列如下:正向5'-GCCGCAATTTGA TTTGATTTCCTCCTT-3',反向5'-GCCATCTGAC TGACTCCACAATACAAT-3'。采用TRIzol法提取植物总RNA,反转录,使用高保真酶PrimeSTAR HS DNA Polymerase扩增得到全长产物,RCR反应体系如下:PrimeSTAR Max Primix(2×)25μL,Primer 1 10 pmol,Primer 2 10 pmol,cDNA 200 ng,添加ddH2O至总体积为50 μL。反应条件:98 ℃变性3 min;98 ℃ 10 s、55 ℃ 15 s、72℃ 10 s,33个循环。取20 μL扩增产物用1% 琼脂糖凝胶电泳进行鉴定,回收并纯化PCR产物连接载体测序。
1.2.2 生物信息学分析 利用国家水稻数据中心网站(https://www.ricedata.cn/index.htm)获得OsJMJ719的CDS序列、启动子序列和氨基酸序列;在PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)网站对基因启动子区域进行顺式作用元件分析;使用MEGA6.0对OsJMJ719蛋白序列和其他物种的蛋白序列进行系统进化树分析;利用软件SWISS-MODEL(https://swissmodel.expasy.org/)对蛋白三维结构进行预测;利用网址Netphos 3.1 ESPASY对OsJMJ719的亲疏水性进行预测;利用在线网站SignalP6.0(https://services.healthtech.dtu.dk/service.php?SignalP)进行信号肽预测;利用TMHMM2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行跨膜结构预测;采用Netphos 3.1 Server(http://www.cbs.dtu.dk/services/NetPhos/)网站对OsJMJ719的磷酸化水平进行预测;使用String在线网站(https://string-db.org)对OsJMJ719蛋白与其他蛋白间的互作进行预测。
1.2.3 表达模式分析 以ZH11为材料,利用qRT-PCR的方法分析OsJMJ719基因在转录水平的表达,具体取材方法如下:不同组织部位(根、茎、叶、剑叶、穗、种子),不同老化阶段(人工老化11 d和14 d的种子)和不同胁迫处理的材料(50 μmol/L ABA;300 mmol/L NaCl;20% PEG;4 ℃低温;25 μmol/L乙烯前体ACC处理1 h和3 h),分析OsJMJ719基因的表达模式。设计Real-time PCR引物,对OsJMJ719基因进行表达模式分析,引物序列如下:正向5'-GCTGGTACCCGCATCTAACA-3',反向5'-ACCATCGGCAAACTCCTTGT-3'。定量PCR按照TaKaRa公司编号为RR820Q的SYBR ® Premix Ex Taq ™ II(Tli RNaseH Plus)试剂盒说明书进行操作,使用LightCycler480(Roche, Germeny)定量PCR仪进行qRT-PCR,反应条件如下:95 ℃ 30 s;95 ℃ 5 s、68 ℃ 30 s,45个循环。qRT-PCR反应结束后,使用2–ΔΔCt方法对所得数据进行相对定量分析。
水稻人工老化处理:将种子用纱网袋装好,置于15 ℃、相对湿度(R.H) 85% 的密封的干燥器中预处理2 d后,转移到已经平衡好的42 ℃、相对湿度85% 干燥器中处理5~15 d, 然后分别置于25 ℃、32% 干燥3 d,密封贮藏于-20 ℃。以未经老化处理且储存在种质资源长期保存库(-20 ℃,相对湿度小于50%)的材料为对照。
1.2.4 亚细胞定位分析 析设计分别带BglII和SpeI酶切位点的引物:正向5'-gaagatctATGCCGC CCAAGAGGAAGCG-3',反向5'-ggactagtACTAAC ACTTGGGGCAGACGC-3',以克隆引物测序正确的质粒做克隆模板,克隆OsJMJ719基因的CDS(coding sequence, CDS)区域并连接到pMD18-T simple载体上,转化DH5α,通过测序方法进一步鉴定序列是否正确,确保无移码现象发生;配制酶切反应液,同时酶切pCAMBIA1302及pMD18-Tsimple-OsJMJ719质粒,37 ℃酶切4 h后,对酶切产物进行电泳检测,回收及纯化酶切目的产物;目标片段OsJMJ719连接到表达载体pCAMBIA1302,体系为10 µL,其中OsJMJ719的酶切产物1 µL,酶切后载体pCAMBIA1302 2 µL,双蒸水2 µL,Solution I 5 µL,在16 ℃ 30 min时,4 ℃放置过夜;转化DH 5α大肠杆菌感受态细胞,筛选阳性克隆,质粒提取酶切鉴定,获得pCAMBIA1302-OsJMJ719-GFP重组质粒。将重组质粒分别转化水稻和烟草进行表达和亚细胞定位观察。
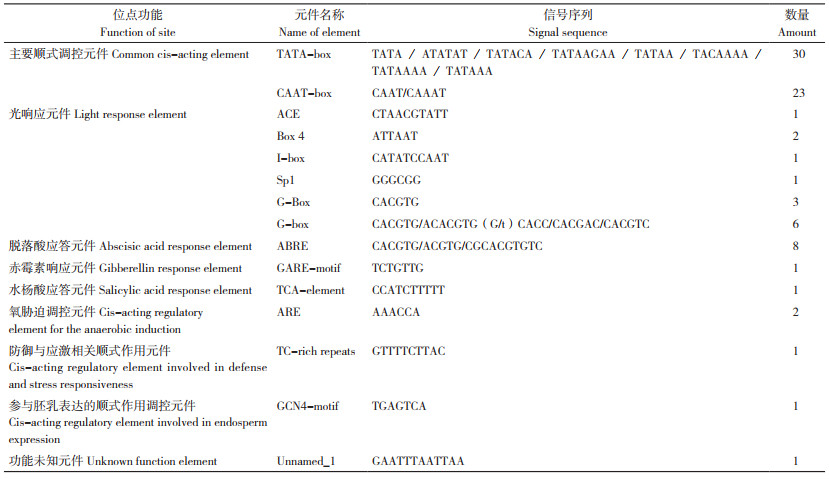
2 结果与分析 2.1 OsJMJ719基因克隆及启动子作用元件分析从粳稻品种中花11中提取总RNA,经过反转录得到cDNA,用于基因克隆。在国家水稻数据中心网站搜索OsJMJ719(LOC_Os02g01940)的基因序列,用Primer 5.0设计引物,设计克隆引物,以cDNA为模板对OsJMJ719的CDS(coding sequence, CDS)进行PCR扩增,测序得到CDS全长为2 994 bp(图 1),该基因编码998个氨基酸。
|
| 图 1 水稻OsJMJ719基因的克隆 Fig. 1 Cloning of OsJMJ719 gene in rice |
为了解OsJMJ719受到的潜在调控因素,对其启动子顺式作用元件进行预测分析。选取OsJMJ719基因起始密码子ATG前2 000 bp的序列作为启动子序列,在PlantCARE网站分析其顺式作用元件(表 1)。结果表明,在OsJMJ719基因启动子序列中,存在很多种类的顺式作用元件,作用于植物生长调控的多个方面:第一类为启动子主要顺式作用元件,包括CAAT-box(23个)、TATA-box(30个);第二类为参与光响应元件和光响应调控元件,包括ACE(1个)、I-box(1个)、Sp1(1个)、Box4(2个)、G-Box(3个)和G-box(6个);第三类为植物激素响应元件,包括赤霉素响应元件GARE-motif(1个)、水杨酸响应元件TCA-element(1个)和脱落酸响应元件ABRE(8个);第四类为环境胁迫调控相关元件,包括ARE(2个)、TC-rich repeats(1个);第五类为参与胚乳表达的调控元件GCN4-motif(1个);第六类为Unnamed_1元件(1个),预测其为蛋白结合位点元件。分析表明,OsJMJ719基因启动子具有光响应相关元件14个、植物激素响应元件10个、环境胁迫调控相关元件3个,推测OsJMJ719可能参与植物生长发育及逆境、激素等外界因素的响应。
2.2 系统进化树构建与分析
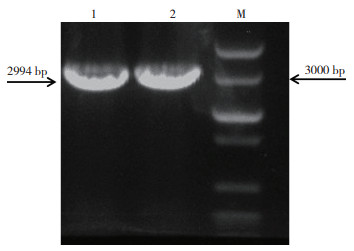
将OsJMJ719的蛋白序列提交到NCBI数据库,获得登录号BFA07558.1;利用BLASTP工具搜索不同物种中OsJMJ719的同源序列,用邻接法构建系统进化树,聚类后分析发现,OsJMJ719蛋白首先与水稻(Oryza sativa Japonica)、沼生菰(Zizania palustris)、粗山羊草(Aegilops tauschii)、小麦(Triticum aestivum)、大麦(Hordeum vulgare)的JmjC基因蛋白序列有较高的同源性,表明水稻与沼生菰、粗山羊草、小麦、大麦亲缘关系较近,其次是黑麦草和二穗短柄草(图 2)。
|
| 图 2 OsJMJ719蛋白和其他物种相似蛋白的同源性比对 Fig. 2 Homology comparison of OsJMJ719 proteins and homologous proteins from other species |
2.3 水稻OsJMJ719的蛋白结构及亲水性分析
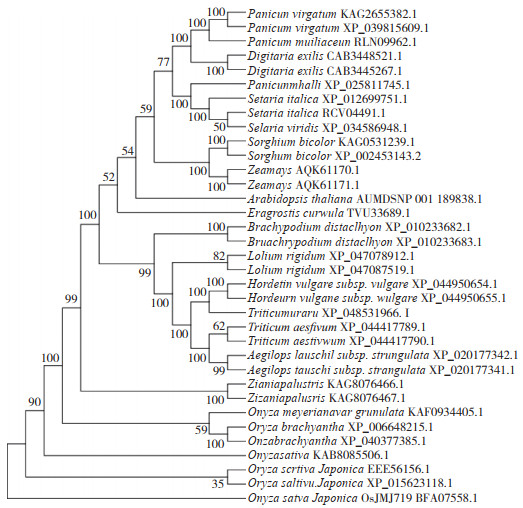
通过SWISS-MODEL对OsJMJ719蛋白的三维结构进行预测,发现该蛋白的三级结构以无规卷曲和α-螺旋为主,其中构成α螺旋的氨基酸313个、占31.39%,形成延伸链的氨基酸110个、占11.03%,构成无规则卷曲的氨基酸537个、占53.86%,β转角的氨基酸37个、占3.71%(图 3A)。利用网址ESPASY对OsJMJ719的亲疏水性进行预测(图 3B)。图 3B中,横轴代表氨基酸序列,纵轴代表亲水性平均系数,以0为分界线,数值为正数疏水性越强,数值为负数亲水性越强。该段基因编码的蛋白质亲水平均系数为-0.751,推测该蛋白为亲水性蛋白质。
|
| 图 3 OsJMJ719蛋白的三维结构(A)和亲水性预测(B) Fig. 3 Tertiary structure(A)and hydrophilic prediction(B)of OsJMJ719 protein |
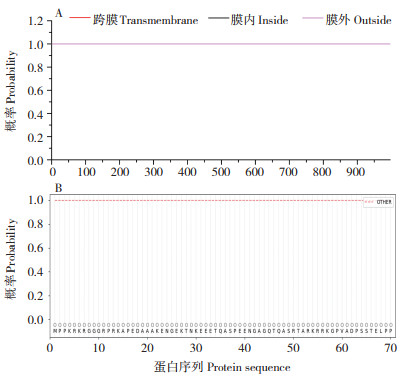
2.4 水稻OsJMJ719的跨膜结构及信号肽预测
使用工具TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)进行OsJMJ719蛋白跨膜结构预测,结果表明OsJMJ719蛋白为膜外蛋白,无跨膜区域,也无膜内区域(图 4A)。使用在线网站SignalP 6.0对OsJMJ719蛋白是否存在信号肽进行预测,发现OsJMJ719蛋白存在信号肽的可能性为0.15%,因此OsJMJ719蛋白不存在信号肽的可能性为99.85%(图 4B)
|
| 图 4 OsJMJ719蛋白跨膜结构(A)和信号肽预测(B) Fig. 4 Protein transmembrane structure(A)and signal peptide prediction(B)of OsJMJ719 |
2.5 水稻OsJMJ719蛋白磷酸化水平分析
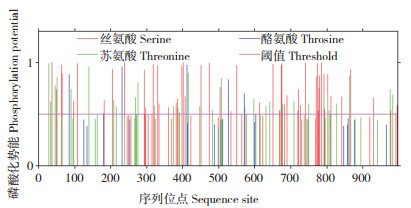
磷酸化作为一种常见的翻译后修饰,在能量代谢、信号传导、神经活动等维持机体细胞的生理过程中发挥作用,是一种重要的调节蛋白质功能的机制。采用Netphos 3.1 Server网站对OsJMJ719的磷酸化水平进行预测(图 5),发现OsJMJ719含有82个蛋白质磷酸化位点,其中丝氨酸的预测位点有47个,最高得分位点是第64位,评分0.998,序列为VADPSSTEL;苏氨酸的预测位点有29个,最高得分位点是第28位,评分0.985,序列为NGEKTNKEE;酪氨酸的预测位点有6个,最高得分位点是第413位,评分0.967,序列为DNYIYCPTA。表明OsJMJ719拥有较多的磷酸化位点。
|
| 图 5 OsJMJ719蛋白磷酸化位点分析 Fig. 5 Phosphorylation site analysis of OsJMJ719 |
2.6 OsJMJ719蛋白互作预测
使用String在线网站对OsJMJ719蛋白与其他蛋白间的互作进行预测,以拟南芥为参考,预测结果(图 6)显示该蛋白可能与SWIB等6个蛋白进行互作,以未鉴定蛋白(uncharacterized protein)最多。OsJMJ719与SWIB功能联系综合得分相对较高,互作的可能性更大。OsJMJ719还可能通过调控绿色未鉴定蛋白,从而间接调控S-腺苷甲硫氨酸脱羧酶(S-adenosylmethionine decarboxylase, SAMDC)。

|
| 图 6 OsJMJ719蛋白与其他蛋白间的互作预测 Fig. 6 Prediction of interactions between OsJMJ719 protein and other proteins |
2.7 水稻OsJMJ719蛋白亚细胞定位分析
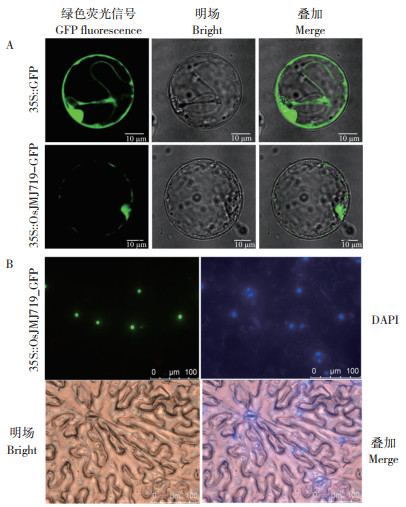
为进一步观察OsJMJ719的亚细胞定位,将35S启动子所驱动的OsJMJ719与GFP进行融合,通过原生质体瞬时转化法将构建的35S: : OsJMJ719-GFP转化到原生质体中进行蛋白表达。利用激光共聚焦显微镜可以观察到空载对照35S: : GFP在细胞质、细胞核以及细胞膜中均有表达;OsJMJ719-GFP的荧光可以在原生质体的细胞核中观察到细胞质和细胞膜中有极微弱的荧光, 因此推测OsJMJ719蛋白主要定位于细胞核中(图 7A)。同时使用农杆菌转化法将35S: : OsJMJ719-GFP瞬时转化烟草,转染2~3 d后,进行激光共聚焦显微镜观察。结果显示,在瞬时表达的烟草下表皮细胞中,GFP信号和DAPI信号完全重合,说明OsJMJ719在烟草瞬时表达系统中定位于烟草下表皮的细胞核中(图 7B)。
|
|
A:OsJMJ719在水稻原生质体中的亚细胞定位; B:OsJMJ719在烟草中的亚细胞定位 A: Subcellular localization of OsJMJ719 in rice protoplasts; B: Subcellular localization of OsJMJ719 in tobacco leaves 图 7 OsJMJ719蛋白的亚细胞定位分析 Fig. 7 Subcellular localization of OsJMJ719 protein |
2.8 基因表达模式分析
通过对OsJMJ719启动子作用元件的分析,发现启动子中有10个植物激素相应元件和3个环境胁迫调控元件,为此利用ABA、ACC(乙烯前体)、低温、高盐和干旱胁迫处理对OsJMJ719基因进行组织表达和老化的定量分析,发现OsJMJ719在水稻各组织中呈现组成型表达;成熟的种子中高丰度表达,在叶鞘和穗中的表达量较少;且在种子老化过程中表达量显著下调。在ABA、低温、高盐、干旱和ACC胁迫处理下,中花11幼叶中OsJMJ719基因的表达模式分析结果如图 10所示,低温处理后,水稻OsJMJ719的表达水平开始持续升高,在处理3 h时的表达水平约为处理前表达水平的6倍;在300 mmol/L NaCl胁迫处理下,叶片中OsJMJ719基因在3 h的表达量上调显著,约为起始量的15.5倍;在20% PEG的处理下,OsJMJ719基因的表达量陆续上升,在处理1 h时的表达量约为0 h的4倍,在处理3 h时的表达量约为0 h的10倍;在50 μmol/L ABA的处理下,处理1 h时OsJMJ719基因的表达量与0 h无差异,在处理3 h时上调显著,约为0 h的8倍;25 μmol/L乙烯前体ACC处理1 h和3 h的情况下,OsJMJ719基因的表达量表现为先上升后下降的趋势(图 8)。

|
| S:茎;L:叶;LS:叶鞘;SP:穗;R:根;Se:成熟种子;11 d: 人工老化11 d的种子;14 d:人工老化14 d的种子 S: Stem; L: Leaf; LS: Leaf sheath; SP: Spike; R: Root; Se: Mature seed; 11 d: Artificially aged seeds for 11 days; 14 d: Artificially aged seeds for 14 days 图 8 OsJMJ719基因的组织表达及不同胁迫处理条件下的相对表达量分析 Fig. 8 Relative expression level analysis of OsJMJ719 gene in different tissues and under different abiotic stresses |
3 讨论
组蛋白修饰作为一种重要的表观遗传修饰机制,在植物开花时间调控、果实发育以及逆境胁迫响应中起重要作用[15]。JmjC结构蛋白作为KDM家族中重要的一员,可以通过参与组蛋白修饰和DNA甲基化之间的相互作用,维持体内组蛋白甲基化的稳态,在表观遗传过程、基因表达和植物发育中发挥重要作用[16]。
启动子对基因时空表达的调控具有关键作用, 对基因的启动子序列进行分析可为研究基因表达模式和潜在功能提供一定的参考价值。刘圣杰等[17]对一个水稻乙二醛酶基因OsGLYI11.2的启动子片段进行了克隆和顺式作用元件分析,发现该基因的启动子在水稻的生长发育及逆境胁迫响应中发挥作用。因此,OsJMJ719基因启动子具有多种植物生长激素、非生物胁迫、光响应调控元件以及逆境相关元件,暗示逆境胁迫、激素及光信号等在一定程度上调节OsJMJ719基因的表达,并且推测OsJMJ719很可能在应对逆境过程中起着重要作用。除此之外,OsJMJ719基因启动子有调控胚乳表达的元件,说明该基因介导胚乳的表达。在今后研究转录调节的分析机制时,可以通过分析启动子结构及组成和相应的生物学功能,结合植株的表型特性,解析该基因启动子对植株发育和应对逆境中的作用。
使用String在线网站对OsJMJ719蛋白与其他蛋白间的互作进行预测,发现该蛋白可能与SWIB互作的可能性更大。SWIB是ATP依赖的染色体重塑复合物(ATP-dependent chromatin remodeling complex)中SWI/SNF亚家族的成员[18]。SWI/SNF染色体重塑复合物介导了的组蛋白与DNA之间的结合,参与了植物发育和逆境胁迫调节[19]。二者在水稻体内是否发生互作,是否参与染色体重塑复合体的形成,需要我们更加深入的研究。互作蛋白分析结果显示OsJMJ719蛋白可以间接与SAMDC互作。作为多胺合成的关键限速酶,SAMDC主要参与植物防御干旱胁迫的调控过程[20],因此推测OsJMJ719可能参与植物的干旱胁迫反应。
在对不同物种的JMJC蛋白进行分析,可直观地得到JMJC蛋白均含典型的JmjC结构域,且在进化上十分保守,不同物种之间的JmjC结构域相似度较高。同一个基因家族中,拥有相同保守元件的基因功能有可能相似[21]。随着不断深入研究,越来越多含JmjC结构域蛋白的功能逐渐被挖掘。JMJ17可以通过调控ABA响应基因促进种子萌发和幼苗生长,也可以参与植物的脱水胁迫响应[22-23]。H3K9去甲基化酶JMJ27参与调控干旱胁迫反应[7]。水稻JMJ706可以影响水稻花器官发育和基因MADS-box的表达[13],JMJ703可以影响根系菌群[24]。本研究克隆到的OsJMJ719基因编码的蛋白在序列上与拟南芥IBM1/JMJ25较为接近,属于KDM3/JHDM2家族;IBM1/JMJ25是一个H3K9me2和H3K9me1的去甲基化酶,ibm1突变体植株表现出不同类型的发育缺陷,如叶片变小变窄、花器官发育不正常、花粉败育和生殖能力降低等表型;JMJ25亚细胞定位也位于细胞核内[25-27]。通过组织定量表达分析,发现OsJMJ719在成熟种子中表达量较高,且在老化过程中持续下调表达,推测可能在种子萌发、贮藏过程中起重要作用。本研究结果表明OsJMJ719受ABA、NaCl、PEG和乙烯的诱导表达,OsJMJ719启动子区域存在与逆境响应相关的元件,暗示OsJMJ719可能参与水稻的非生物胁迫应对过程。本研究后续工作将通过构建OsJMJ719的crispr敲除和过表达载体,创制突变体和获得过表达株系,从而验证OsJMJ719在种子贮藏和非生物胁迫中的功能。
4 结论本研究从水稻中花11总RNA中反转录得到cDNA,由此克隆得到1个JMJC家族成员OsJMJ719基因,该基因ORF全长2 994 bp,编码997个氨基酸;OsJMJ719蛋白是不稳定的亲水蛋白,与沼生菰、粗山羊草、小麦同源性较高,无信号肽及跨膜区域,拥有较多的磷酸化位点。蛋白质三级结构分析表明,OsJMJ719蛋白结构以无规则卷曲和α-螺旋为主;亚细胞定位结果显示该蛋白定位于细胞核。在OsJMJ719基因的启动子序列中,存在14个参与光响应元件和光响应调控元件、10个植物激素响应元件、3个环境胁迫调控元件,且OsJMJ719基因受ABA、高盐、干旱和ACC胁迫诱导均上调表达,推测其可能参与水稻响应多种非生物胁迫,在逆境响应中发挥重要作用。
| [1] |
BIRD A. Perceptions of epigenetics[J]. Nature, 2007, 447(7143): 396-8. DOI:10.1038/nature05913 |
| [2] |
LUGER K, MADER A W, RICHMOND R K, SARGENT D F, RICHMOND T J. Crystal structure of the nucleosome core particle at 2.8 A resolution[J]. Nature, 1997, 389(6648): 251-60. DOI:10.1038/38444.PMID:9305837 |
| [3] |
ZHAO W, NEYT P, VAN LIJSEBETTENS M, SHEN W H, BERR A. Interactive and noninteractive roles of histone H2B monoubiquitination and H3K36 methylation in the regulation of active gene transcription and control of plant growth and development[J]. New Phytologist, 2019, 221(2): 1101-1116. DOI:10.1111/nph.15418 |
| [4] |
ACCARI S L, FISGER P R. Emerging roles of JmjC domain-containing proteins[J]. International Review of Cell and Molecular Biology, 2015, 319: 165-220. DOI:10.1016/bs.ircmb.2015.07.003 |
| [5] |
LU F L, LI G L, CUI X, LIU C Y, WANG X J, CAO X F. Comparative analysis of JmjC domain-containing proteins reveals the potential histone demethylases in Arabidopsis and rice[J]. Journal of Integrative Plant Biology, 2008, 50(7): 886-896. DOI:10.1111/j.1744-7909 |
| [6] |
MOSAMMAPARAST N, YANG S. Reversal of histone methylation: biochemical and molecular mechanisms of histone demethylases[J]. Annual Review of Biochemistry, 2010, 79: 155-179. DOI:10.1146/annurev.biochem.78.070907.103946 |
| [7] |
WANG Q L, LIU P, JING H, ZHOU X F, ZHAO B, LI Y, JIN J B. JMJ27-mediated histone H3K9 demethylation positively regulates drought-stress responses in Arabidopsis[J]. New Phytologist, 2021, 232(1): 221-236. DOI:10.1111/nph.17593 |
| [8] |
HUNG F Y, CHEN J H, FENG Y R, LAI Y C, YANG S G, WU K Q. Arabidopsis JMJ29 is involved in trichome development by regulating the core trichome initiation gene GLABRA3[J]. The Plant Journal, 2020, 103(5): 1735-1743. DOI:10.1111/tpj.14858 |
| [9] |
CHEN H H, TONG J H, FU W, LIANG Z W, RUAN J X, YU Y G, SONG X, YUAN L B, XIAO L T, LIU J, CUI Y H, HUANG S Z, LI C L. The H3K27me3 demethylase RELATIVE OF EARLY FLOWERING6 suppresses seed dormancy by inducing abscisic acid catabolism[J]. Plant Physiology, 2020, 184(4): 1969-1978. DOI:10.1104/pp.20.01255 |
| [10] |
CUI X K, JIN P, CUI X, GU L F, LU Z K, XUE Y M, WEI L Y, QI J F, SONG X W, LUO M, AN G, CAO X F. Control of transposon activity by a histone H3K4 demethylase in rice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(5): 1953-1960. DOI:10.1073/pnas.1217020110 |
| [11] |
SONG T, ZHANG Q, WANG H, HAN J B, XU Z Q, YAN S N, ZHU Z G. OsJMJ703, a rice histone demethylase gene, plays key roles in plant development and responds to drought stress[J]. Plant Physiology and Biochemistry, 2018, 132: 183-188. DOI:10.1016/j.plaphy.2018.09.007 |
| [12] |
LI T T, CHEN X S, ZHONG X C, ZHAO Y, LIU X Y, ZHOU S L, CHENG S F, ZHOU D X. Jumonji C domain protein JMJ705-mediated removal of histone H3 lysine 27 trimethylation is involved in defense-related gene activation in rice[J]. The Plant Cell, 2013, 25(11): 4725-4736. DOI:10.1105/tpc.113.118802 |
| [13] |
SUN Q W, ZHOU D X. Rice jmjC domain-containing gene JMJ706 encodes H3K9 demethylase required for floral organ development[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(36): 13679-13684. DOI:10.1073/pnas.0805901105 |
| [14] |
秦巧. OsJMJ714调控水稻根系发育与生长素含量的功能研究[D]. 北京: 中国农业科学院, 2013. QIN Q. The function of OsJMJ714 in regulation of root development and auxin content in rice[D]. Beijing: Chinese Academy of Agricultural Sciences, 2013. |
| [15] |
卢晶霞, 杨松光, 吴克强. 番茄组蛋白去乙酰化酶HD2家族生物信息学及表达模式分析[J]. 广东农业科学, 2014, 41(12): 138-143. DOI:10.16768/j.issn.1004-874X.2014.12.004 LU J X, YANG S G, WU K Q. Bioinformatics and expression pattern analysis of histone deacetylase HD2 subfamily in tomato (Solanum lycopersicum)[J]. Guangdong Agricultural Sciences, 2014, 41(12): 138-143. DOI:10.16768/j.issn.1004-874X.2014.12.004 |
| [16] |
THINNES C C, ENGLAND K S, KAWAMURA A, CHOWDHURY R, SCHOFIELD C J, HOPKINSON R J. Targeting histone lysine demethylases-progress, challenges, and the future[J]. Biochimice Et Biophysica Acta Gene Regulatogy Mechanisms, 2014, 1839(12): 1416-1432. DOI:10.1016/j.bbagrm.2014.05.009 |
| [17] |
刘圣杰, 赖剑云, 刘文华, 刘勤坚, 陈沛, 张文虎, 朱庆锋, 肖应辉, 刘军. 水稻OsGLYI11.2基因启动子的克隆及表达分析[J/OL]. 分子植物育种, 2021: 1-14. http://kns.cnki.net/kcms/detail/46.1068.S.20210707.1337.006.html. LIU S J, LAI J Y, LIU W H, LIU Q J, CHEN P, ZHANG W H, ZHU Q F, XIAO Y H, LIU J. Cloning and expression analysis of OsGLYI11.2 gene promoter from Oryza sativa[J/OL]. Molecular Plant Breeding, 2021: 1-14. http://kns.cnki.net/kcms/detail/46.1068.S.20210707.1337.006.html. |
| [18] |
ZHOU C, MIKI B, WU K. CHB2, a member of the SWI3 gene family, is a global regulator in Arabidopsis[J]. Plant Molecular Biology, 2003, 52(6): 1125-1134. DOI:10.1023/b:plan.0000004305.60407.8b |
| [19] |
SAMOWSKA E, GRATKOWSKA D M, SACHAROWSKI S P, CWIEK P, TOHGE T, FERNIE A R, SIEDLECKI J A, KONCZ C, SAMOWSKI T J. The role of SWI/SNF chromatin remodeling complexes in hormone crosstalk[J]. Trends in Plant Science, 2016, 21(7): 594-608. DOI:10.1016/j.tplants.2016.01.017 |
| [20] |
唐文武, 吴秀兰. 沙糖橘S-腺苷甲硫氨酸脱羧酶基因克隆及表达分析[J]. 南方农业学报, 2020, 51(6): 1369-1376. DOI:10.3969/j.issn.2095-1191.2 TANG W W, WU X L. Cloning and expression analysis of S-adenosylmethionine decarboxylase gene (CrSAMDC) in Shatangju[J]. Journal of Southern Agriculture, 2020, 51(6): 1369-1376. DOI:10.3969/j.issn.2095-1191.2 |
| [21] |
NAKANO T, SUZUKI K, FUJIMURA T, SHINSHI H. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiology, 2006, 140(2): 411-432. DOI:10.1104/pp.105.073783 |
| [22] |
WANG T J, HUANG S, ZHANG A, GUO P, LIU Y, XU C, CONG W, LIU B, XU Z Y. JMJ17-WRKY40 and HY5-ABI5 modules regulate the expression of ABA-responsive genes in Arabidopsis[J]. New Phytologist, 2021, 230(2): 567-584. DOI:10.1111/nph.17177.Epub2021Feb12.PMID:33423315 |
| [23] |
HUANG S Z, ZHANG A, JIN J B, ZHAO B, WANG T J, WU Y, WANG S, LIU Y T, WANG J, GUO P, AHMAD R, LIU B, XU Z Y. Arabidopsis histone H3K4 demethylase JMJ17 functions in dehydration stress response[J]. New Phytologist, 2019, 223(3): 1372-1387. DOI:10.1111/nph.15874 |
| [24] |
LYU Z Y, DAI R, XU H R, LIU Y X, BAI B, MENG Y, LI H Y, CAO X F, BAI Y, SONG X W, ZHANG J Y. The rice histone methylation regulates hub species of the root microbiota[J]. Journal of Genetics and Genomics, 2021, 48(9): 836-843. DOI:10.1016/j.jgg.2021.06.005 |
| [25] |
INAGAKI S, MIURA-KAMIO A, NAKAMURA Y, LU F, CUI X, CAO X, KIMURA H, SAZE H, KAKUTANI T. Autocatalytic differentiation of epigenetic modifications within the Arabidopsis genome[J]. The EMBO Journal, 2010, 29(20): 3496-3506. DOI:10.1038/emboj.2010.227 |
| [26] |
LIU X L, ZHAO J H, GUO H S. IBM1-dependent H3K9 demethylation enables self-silencing of an exogenous silencer for the non-cell autonomous silencing of an endogenous target gene[J]. Journal of Genetics and Genomics, 2019, 46(3): 149-153. DOI:10.1016/j.jgg.2019.02.006 |
| [27] |
FAN D, DAI Y, WANG X C, WANG Z J, HE H, YANG H C, CAO Y, DENG X W, MA L G. IBM1, a JmjC domain-containing histone demethylase, is involved in the regulation of RNA-directed DNA methylation through the epigenetic control of RDR2 and DCL3 expression in Arabidopsis[J]. Nucleic Acids Research, 2012, 40(18): 8905-8916. DOI:10.1093/nar/gks647 |
(责任编辑 邹移光)
 2022, Vol. 49
2022, Vol. 49