文章信息
基金项目
- 广东省科技计划项目(2020B121201008,2020B020220008);广州市科技计划项目(202102021008);广东省农业科学院院长基金(202021)
作者简介
-
李清,博士,助理研究员,毕业于中国农业大学,2018年进入广东省农业科学院农业生物基因研究中心工作至今,主要从事水稻种子活力调控机制研究与种质资源鉴评。先后主持广州市科技计划项目和广东省农业科学院院长基金等科研项目,参与国家自然科学基金、广东省自然科学基金、广东省重点研发专项等多个项目。在《Plant Journal》《Plant Physiology》《Journal of Experimental Botany》等期刊发表科技论文多篇。
李清(1989—),女,博士,助理研究员,研究方向为农作物种质资源基因挖掘,E-mail:411066120@qq.com; 刘军,博士,三级研究员,硕士研究生导师,广东省农业科学院农业生物基因研究中心学科带头人,广东省农作物种质资源保存与利用重点实验室负责人,兼任广东省植物生理学会理事。长期从事作物种子生物学、种质资源保存与利用研究,先后主持国家自然科学基金、广东省科技攻关项目、广东省自然科学基金项目和中央补助地方科技基础条件专项资金项目等30余项。以第一作者或通信作者在《Plant Journal》《BMC Plant Biology》《Frontiers in Plant Sciences》《植物学报》《中国农业科学》等国内外期刊发表高水平论文30多篇,编写或参与编写著作3部;以第一发明人获得授权发明专利10项;已研发产品4个。获省市各级科技成果奖励5项,包括广东省科技进步奖一等奖、广东省农业技术推广奖一等奖、广州市科技进步奖二等奖等。主持完成的“水稻种子寿命与活力控制理论与技术研究应用”成果经专家评价总体达国际先进水平,部分国际领先,该成果已在多家种业应用,取得显著的经济和社会效益.
通讯作者
- 刘军(1969—),男,博士,研究员,研究方向为农作物种质资源保存与利用,E-mail:liujun@gdaas.cn.
文章历史
- 收稿日期:2022-09-30
【研究意义】遗传密码是连接核酸和蛋白质的纽带,在生物体遗传信息的传递中起重要作用,其中mRNA共有64个密码子,编码20个氨基酸和3个终止信号,除Met和Trp仅由1个密码子编码外,大多数氨基酸由多个同义密码子编码[1]。然而,蛋白质合成过程中不同密码子的使用概率并不相同。一个物种或者一个基因更倾向于使用一个或多个特定的同义密码子,这被称为同义密码子使用偏好性(Synonymous Codon Usage Bias,SCUB)[2]。密码子使用偏好性的特征在原核生物和真核生物中普遍存在。大量研究表明,密码子使用偏好模式可能由于基因在基因组进化过程中受到多种因素影响引起,包括自然选择、组成突变模式、翻译选择、基因长度、tRNA丰度和mRNA二级结构等[3-5]。因此,密码子使用偏好性的研究不仅可以揭示一个物种或相关物种基因之间的起源、进化和突变方式,而且密码子的优化可以提高外源序列在转基因研究中的表达效率,进而提高转基因位点的准确性[6]。【前人研究进展】叶绿体是调节植物光合作用的主要细胞器,具有感知外界环境胁迫信号的能力[7]。由于叶绿体基因组的小尺寸和大拷贝数,其功能受到广泛关注[8]。与核基因转化相比,叶绿体基因组具有外源基因表达效率高、定点整合无位置效应现象、遗传稳定、不随花粉漂移等优点,现已被广泛应用于分子进化、系统发育和遗传表达等领域[9-10]。随着高通量测序技术的快速发展,目前已有许多植物的叶绿体基因组完成测序,并且完成了密码子特征的分析,包括木薯(Manihot esculenta L.)[11]、菠萝(Ananas comosus L.)[12]、稷(Panicum miliaceum L.)[13]、芝麻(Sesamum indicum L.)[14]等。
【本研究切入点】显齿蛇葡萄(Ampelopsis grossedentata),是葡萄科(Vitaceae)蛇葡萄属(Ampelopsis)中的一种藤本植物,其嫩茎叶富含二氢杨梅素等黄酮类化合物,具有抗氧化、抗肿瘤、护肝、降血糖和调血脂等多种功效,开发利用前景广阔[15]。目前,关于显齿蛇葡萄的研究主要集中在抗氧化活性、二氢杨梅素测定、药理作用等方面,有关质量标准、指纹图谱等方面也有所报道[16],然而关于显齿蛇葡萄叶绿体基因组密码子偏好性方面的还未见相关研究报道。【拟解决的关键问题】本研究分析显齿蛇葡萄叶绿体基因组的密码子使用偏好、变异来源和筛选最优密码子,并利用多元统计分析方法探讨显齿蛇葡萄叶绿体基因组的密码子使用模式以及分析其影响因素,不仅对提高显齿蛇葡萄叶绿体基因的外源表达水平具有指导意义,同时也为显齿蛇葡萄的后续基因组研究及其遗传改良育种提供理论基础。
1 材料与方法 1.1 试验材料从NCBI数据库下载显齿蛇葡萄完整叶绿体基因组(GenBank登录号:MT267294),序列全长为162 147 bp,为了提高分析密码子偏好性的准确性,首先从82个CDS中剔除长度小于300 bp的序列,其次选择以ATG为起始密码子,TAA、TAG或TGA为终止密码子的CDS序列,最终筛选出59条序列进行后续数据分析[17]。
1.2 试验方法1.2.1 密码子碱基组成分析 使用CodonW 1.4.2软件分析59个显齿蛇葡萄叶绿体基因组CDS的同义密码子相对使用度(Relative Synonymous Codon Usage, RSCU)、有效密码子数(Effective Number of Codon, ENC)、密码子适应指数(Codon Adaptation Index,CAI)、氨基酸长度(Length of Amino Acid, Laa)、第3位碱基上A、T、C、G含量以及最优密码子使用频率(Frequency of Optimal Codons, FOP),使用在线软件工具EMBOSS(http://www.bioinformatics.nl/emboss-explorer)分析各基因密码子上第1、2、3位碱基的GC含量(GC1、GC2、GC3),并计算各基因的总GC含量(GCall),并利用SPSS计算GC1、GC2、GC3、Laa、ENC的相关性。
1.2.2 中性绘图分析 中性绘图是一种可初步判别影响密码子偏好性因素的方法,以GC12(GC1和GC2的均值)为纵坐标,GC3为横坐标绘制散点图,图中每一个点代表一个基因的位置,并对GC12和GC3进行相关性分析,推断影响密码子使用偏好性的因素。若GC12和GC3呈显著相关,则说明密码子3个位点的碱基具有相同变异模式,密码子使用偏好性主要受突变影响,若相关性不显著,则说明3个位点的碱基变异模式较大,主要受自然选择影响[18]。
1.2.3 ENC-plot分析 为探究密码子使用偏好性与基因碱基组成之间的关系,以GC3为横坐标、ENC为纵坐标绘制散点图,以基因与ENC标准曲线的距离衡量影响密码子偏好性的主要因素,ENC的期望值ENCexp=2+GC3+29/ ﹝GC32+(1-GC3)2﹞[19]。
1.2.4 PR2-plot分析 通过偏倚分析密码子第3位碱基A、T和C、G之间突变是否平衡,计算密码子第3位碱基上A、T、C、G的含量,以A3/(A3+T3)为纵坐标,G3/(G3+C3)为横坐标进行偏倚分析,在平面图上显示各密码子第3位碱基的组成状态[20]。
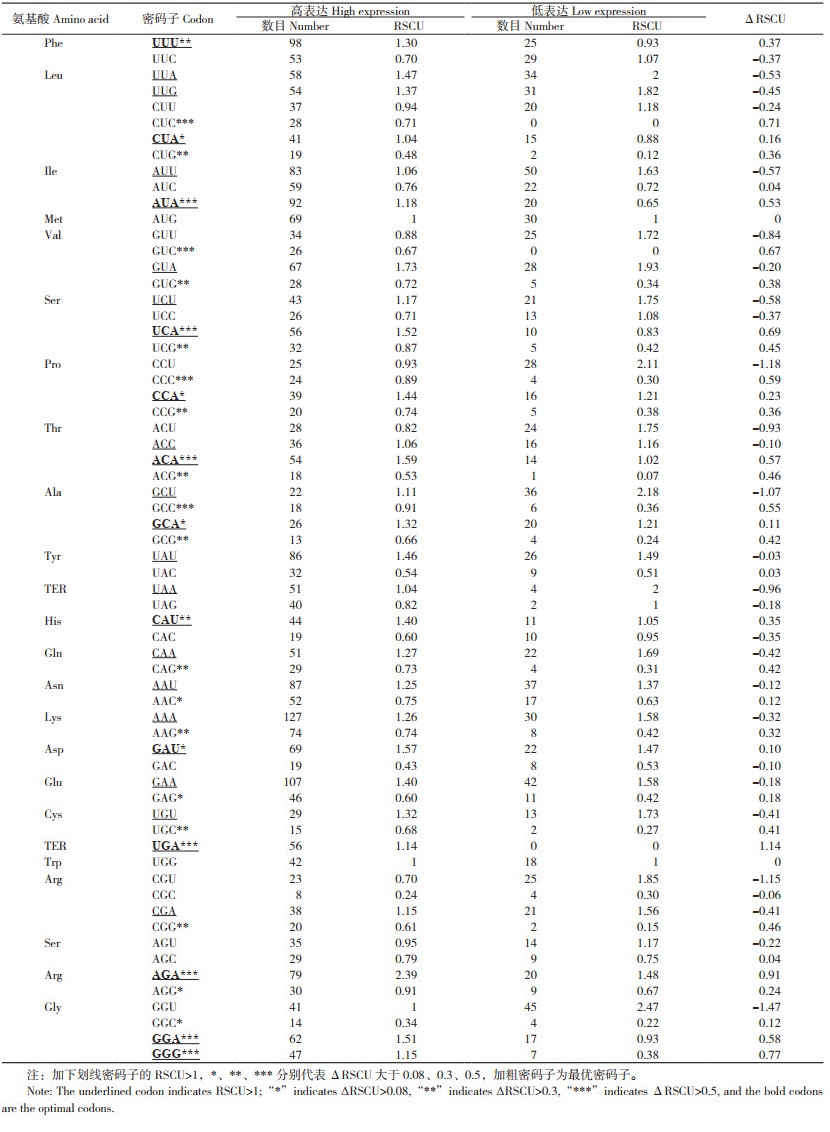
1.2.5 最优密码子分析 将59个基因按照CAI值从高到低排列,分别选择两端10% 基因数(共12个)作为高、低表达库并计算。RSCU值≥ 1的为高频密码子,ΔRSCU(RSCU 高表达-RSCU 低表达)值> 0.08为高表达密码子;若该密码子ΔRSCU ≥ 0.08且RSCU ≥ 1,则被确定为最优密码子[21]。
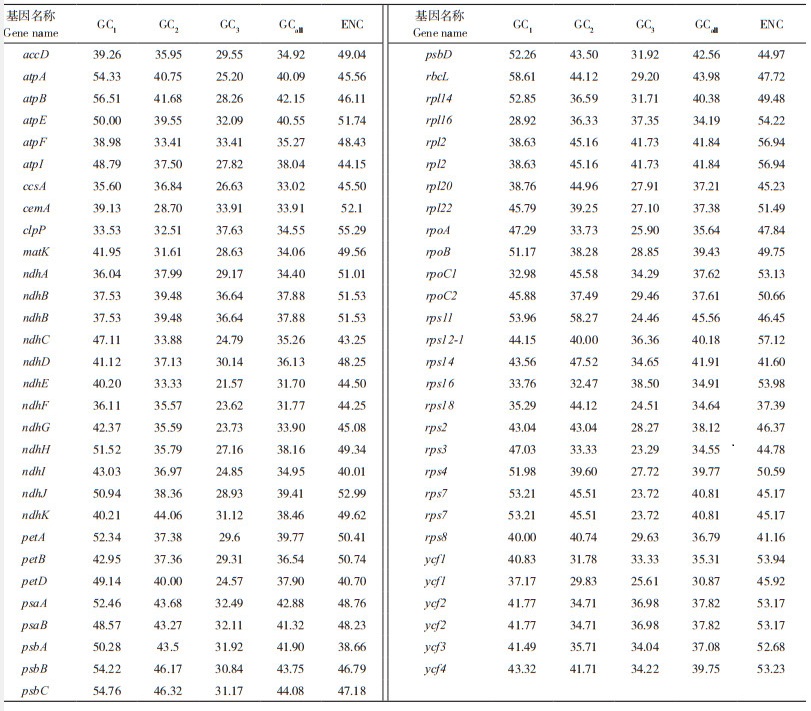
2 结果与分析 2.1 显齿蛇葡萄叶绿体基因组密码子的组成特征通过利用Codon W 1.4.2软件分析59条显齿蛇葡萄叶绿体中的编码蛋白基因,由表 1可知,有核糖体基因11个(rps11、rps12-1、rps14、rps16、rps18、rps2、rps3、rps4、rps7、rps7、rps8),光合作用基因9个(petA、petB、petD、psaA、psaB、psbA、psbB、psbC、psbD),烟碱脱氢酶基因12个(ndhA、ndhB、ndhB、ndhC、ndhD、ndhE、ndhF、ndhG、ndhH、ndhI、ndhJ、ndhK),ATP基因6个(accD、atpA、atpB、atpE、atpF、atpI),其他基因21个;平均GC含量为37.98%,其中密码子的3位碱基的平均GC含量分别为GC1(44.88%) > GC2(38.13%) > GC3(29.45%),显齿蛇葡萄叶绿体基因密码子末尾碱基偏好以A/T(U)结尾。表 1显示,ENC取值范围为37.39 ~ 57.12,平均值为48.48,可见显齿蛇葡萄叶绿体基因组密码子偏好性较弱。
|
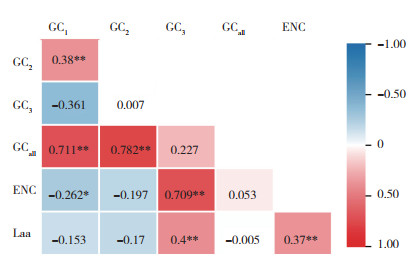
基因密码子各参数之间的相关性分析结果(图 1)表明,GC1与GC2、GC为极显著相关,与GC3和ENC为显著负相关,GC2与GC3为极显著相关,说明叶绿体基因组中,第1位碱基与第2位碱基组成具有较大的相似性,但均与第3位碱基具有较大差异;GC3与ENC和Laa为极显著相关,说明第3位碱基组成的变化对密码子的使用偏好性影响较大,且基因长度影响第3位碱基的组成,ENC与Laa均达到极显著相关,表明序列长度对密码子使用偏好性造成较大影响。
|
| *、** 分别表示显著相关和极显著相关 * and ** represent significant correlation and extremely significant correlation, respectively 图 1 显齿蛇葡萄叶绿体基因组密码子各参数间的相关分析 Fig. 1 Correlation analysis between the indexes of chloroplast genome codon in Ampelopsis grossedentata |
使用CodonW计算59条编码蛋白序列的相对同义密码子使用度RSCU,结果(图 2)显示,29个密码子的RSCU > 1、为高频密码子,其中12个以U结尾、14个以A结尾、2个以G结尾和1个以C结尾,A/U结尾的密码子占89.65%,表明显齿蛇葡萄叶绿体基因组密码子更偏向A/U结尾,以G/C结尾的密码子偏少。
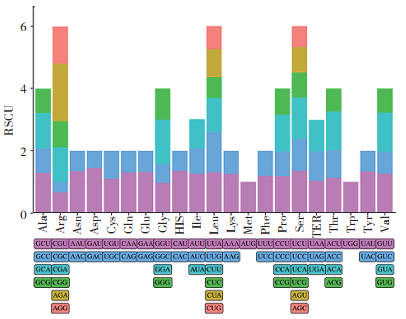
|
| 图 2 显齿蛇葡萄叶绿体基因组的RSCU分析 Fig. 2 RSCU analysis of chloroplast genome in Ampelopsis grossedentata |
2.2 中性绘图分析
中性绘图分析结果(图 3)显示,GC12的平均值为32.63%~56.12%,GC3的取值范围为21.57%~41.73%。图中各基因均位于对角线上方,且标准曲线斜率为-0.2401,表明第3位密码子碱基与第1、2位碱基组成不同。GC12和GC3的相关系数为0.0608,相关性不显著,表明显齿蛇葡萄叶绿体基因密码子3个位点的碱基组成存在较大差异,且显齿蛇葡萄叶绿体基因组的密码子偏好性主要受自然选择因素影响,然而有3个基因(clpP、rps16、rpl16)位于对角线下方,说明这3个基因受突变影响造成。
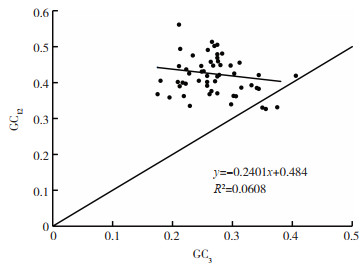
|
| 图 3 中性绘图分析结果 Fig. 3 Analysis result of neutral plot |
2.3 ENC-plot分析
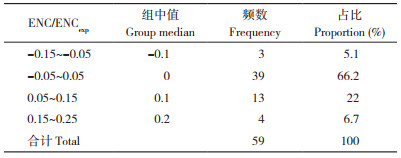
以GC3为横坐标、ENC为纵坐标绘制散点图(图 4),并与ENC的期望值ENCexp进行比较,ENC < 35表示密码子偏性较强,而ENC > 35则表示密码子偏性较弱[19]。ENCexp取值范围为42.95~58.43,说明密码子使用偏好性较弱。计算ENC/ENCexp比值(表 2)发现,比值在-0.05~0.05间有39个,占基因总数的66.2%,说明基因落在标准曲线附近,表明突变对密码子偏好性的形成起重要作用。
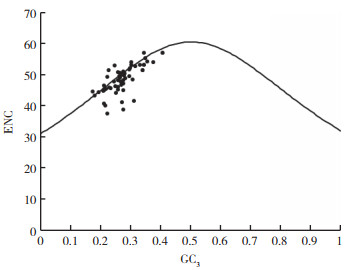
|
| 图 4 ENC-plot分析结果 Fig. 4 Analysis result of ENC-plot |
2.4 PR2-plot分析
偏倚分析可探究突变和自然选择对密码子使用偏好的影响,PR2-plot分析结果(图 5)表明,基因位点在4个平面中分布并不均匀,从使用频率上看T > A、G > C,其基因密码子主要分布在右下方区域(23个),表明显齿蛇葡萄叶绿体基因组密码子第3位碱基T、G的使用频率高于A、C,说明显齿蛇葡萄叶绿体基因组密码子使用偏好性主要由自然选择因素决定,但同时也受到自身内部碱基突变因素的影响。
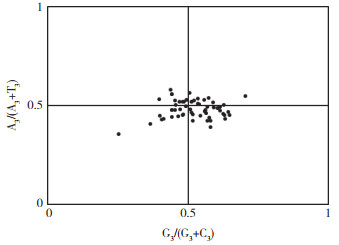
|
| 图 5 PR2-plot分析结果 Fig. 5 Analysis result of PR2-plot |
2.5 显齿蛇葡萄叶绿体基因组最优密码子鉴定
以RSCU > 1为标准共筛选出28个高频密码子(表 3);通过CAI值对编码蛋白基因降序排序,取高低两端前10% 序列(分别各取8条)进行最优密码子计算,以ΔRSCU > 0.08为标准确定了31个高表达密码子,其中12个以A/T结尾,19个以C/G结尾;以ΔRSCU > 0.08、且RSCU > 1为标准共确定13个最优密码子(UUU、CUA、AUA、UCA、CCA、ACA、GCA、CAU、GAU、UGA、AGA、GGA、GGG),其中12个以A/U结尾、1个以G结尾。
3 讨论
同义密码子使用偏好是生物群中一种不可避免的现象,生物获得特定的密码子使用模式以适应起源、进化、自然选择和突变压力等多种因素[22]。密码子第3个碱基的同义突变不能改变氨基酸的类型,但被认为是决定氨基酸类型的重要特征,因此GC3经常被用作密码子偏向的重要指标[23-24]。本研究对密码子碱基组成的分析表明,显齿蛇葡萄叶绿体基因组的CDS序列GC3含量为29.45%,更倾向于使用A/T密码子,这与王义华[25]分析葡萄属叶绿体基因组的研究结果一致。RSCU值分析结果也证实了这一点,显齿蛇葡萄的叶绿体基因组中存在A/T密码子使用偏向,这与大多数高等植物的模式一致[26]。当密码子的使用受到自然选择的影响时,GC3值往往分布在一个较小的范围内,GC12和GC3之间没有显著的相关性[27]。中性绘图结果显示,GC12和GC3之间的相关性较弱,前2个碱基的组成与密码子的第3个碱基的组成不同,而各参数相关性分析也证实了这一点,说明所分析的显齿蛇葡萄叶绿体基因组的密码子使用模式主要受自然选择的影响。这一结果与许多物种叶绿体基因组的密码子使用情况一致,如肖蒲桃(Syzygium acuminatissimum)[28]、朝文心兰(Oncidium gower ramsey)[29]、木薯[11]等。此外,结合ENC-Plot和PR2-Plot分析的结果推测,显齿蛇葡萄叶绿体基因组的密码子使用偏好性受多种因素影响,包括突变压力、碱基组成和基因长度,其中主要的影响因素是自然选择,重要影响因素是突变。
密码子偏好性可以通过调节基因翻译的准确性和效率影响基因表达,基因表达水平越高,密码子偏好性越强[30-31]。本研究挖掘到显齿蛇葡萄叶绿体基因组共31个高频密码子,其中13个被确定为最优密码子。筛选到的最优密码子可以用于设计叶绿体基因表达载体以提高叶绿体基因组中基因的表达水平,也可以利用已知密码子的使用偏好来推测和预测未知基因的表达和功能[32-33],可为今后从遗传水平上进行显齿蛇葡萄育种改良提供参考。
4 结论本研究从显齿蛇葡萄叶绿体基因组中共筛选出59条蛋白编码序列,基于显齿蛇葡萄的叶绿体基因组数据进行密码子使用偏好分析,结果表明,显齿蛇葡萄叶绿体基因组的GC含量为37.98%,说明AT含量略高;共筛选出13个最优密码子,分别为UUU、CUA、AUA、UCA、CCA、ACA、GCA、CAU、GAU、UGA、AGA、GGA和GGG,除GGG外大多数密码子以A或U结尾。显齿野葡萄叶绿体基因组的密码子使用偏好受核苷酸组成、自然选择、突变压力和基因表达水平的影响,最主要影响因素为自然选择。本研究结果为显齿蛇葡萄的密码子使用模式研究提供了新的视角,为显齿蛇葡萄的分子育种奠定了基础。
| [1] |
WEI L, HE J, JIA X, QI Q, LIANG Z S, ZHENG H, PING Y, LIU S Y, SUN J C. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution[J]. BMC Evolutionary Biology, 2014, 14(1): 262. DOI:10.1186/s12862-014-0262-4 |
| [2] |
TANG D, WEI F, CAI Z. Analysis of codon usage bias and evolution in the chloroplast genome of Mesona chinensis Benth[J]. Development Genes and Evolution, 2020, 231(1/2): 1-9. DOI:10.1007/s00427-020-00670-9 |
| [3] |
POP C, ROUSKIN S, INGOLIA N T, HAN L, PHIZICKY E M, WEISSMAN J S, KOLLER D. Causal signals between codon bias, mRNA structure, and the efficiency of translation and elongation[J]. Molecular Systems Biology, 2014, 10(12): 770. DOI:10.15252/msb.20145524 |
| [4] |
QUAX T E F, CLAASSENS N J, S LL D, OOST J V D. Codon bias as a means to fine-tune gene expression[J]. Molecular Cell, 2015, 59(2): 149-161. DOI:10.1016/j.molcel.2015.05.035 |
| [5] |
TULLER T, WALDMAN Y Y, KUPIEC M, RUPPIN E. Translation efficiency is determined by both codon bias and folding energy[J]. Proceedings of the National Academy of Sciences, 2010, 107(8): 3645-3650. DOI:10.1073/pnas.0909910107 |
| [6] |
SHENG J, SHE X, LIU X, WANG J, HU Z LI. Comparative analysis of codon usage patterns in chloroplast genomes of five Miscanthus species and related species[J]. Peer J, 2021, 9: e12173. DOI:10.7717/peerj.12173 |
| [7] |
LV R, LI Z, LI M, DOGRA V, LV S, LIU R Y, LEE K P, KIM C. Uncoupled expression of nuclear and plastid photosynthesis-associated genes contributes to cell death in a lesion mimic mutant[J]. The Plant Cell, 2019, 31(1): 210-230. DOI:10.1105/tpc.18.00813 |
| [8] |
XU C, CAI X, CHEN Q, ZHOU H, CAI Y, BEN A. Factors affecting synonymous codon usage bias in chloroplast genome of Oncidium Gower Ramsey[J]. Evolutionary Bioinformatics, 2011, 7: 271-278. DOI:10.4137/EBO.S8092 |
| [9] |
KWAK S-Y, LEW T T S, SWEENEY C J, KOMAN V B, WONG M H, BOHMERT-TATAREV K, SNELL K D, SEO J S, CHU NAM-HAI, MICHAEL S. Chloroplast-selective gene delivery and expression in planta using chitosan-complexed single-walled carbon nanotube carriers[J]. Nature Nanotechnology, 2019, 14(5): 447-455. DOI:10.1038/s41565-019-0375-4 |
| [10] |
RUF S, FORNER J, HASSE C, KROOP X, SEEGER S, SCHOLLBACH L, SCHADACH A, BOCK R. High-efficiency generation of fertile transplastomic Arabidopsis plants[J]. Nature Plants, 2019, 5(3): 282-289. DOI:10.1038/s41477-019-0359-2 |
| [11] |
赵英, 付海天, 金刚, 徐钏, 宋恩亮, 罗燕春, 莫永龙. 木薯叶绿体基因组密码子偏好性分析[J]. 江西农业学报, 2022, 34(4): 46-52. DOI:10.19386/j.cnki.jxnyxb.2022.04.008 ZHAO Y, FU H T, JIN G, XU C, SONG E L, LUO Y C, MO Y L. Analysis of codon usage bias in chloroplast genome of cassava[J[J]. Acta Agriculturae Jiangxi, 2022, 34(4): 46-52. DOI:10.19386/j.cnki.jxnyxb.2022.04.008 |
| [12] |
杨祥燕, 蔡元保, 谭秦亮, 覃旭, 黄显雅, 吴密. 菠萝叶绿体基因组密码子偏好性分析[J]. 热带作物学报, 2022, 43(3): 439-446. DOI:10.3969/j.issn.1000-2561.2022.03.001 YANG X Y, CAI Y B, TAN Q L, QIN X, HUANG X Y, WU M. Analysis of codon usage bias in the chloroplast genome of Ananas comosus[J]. Chinese Journal of Tropical Crops, 2022, 43(3): 439-446. DOI:10.3969/j.issn.1000-2561.2022.03.001 |
| [13] |
LI G, ZHANG L, XUE P. Codon usage pattern and genetic diversity in chloroplast genomes of Panicum species[J]. Gene, 2021, 15(802): 145866. DOI:10.1016/j.gene.2021.145866 |
| [14] |
MEBEASELASSIE A, ZHU C Y. Genome-wide analysis of codon usage in sesame (Sesamum indicum L.)[J]. Heliyon, 2022, 8(1): e08687. |
| [15] |
常敬芳. 显齿蛇葡萄茎主要活性成分含量测定及其指纹图谱研究[D]. 南宁: 广西中医药大学, 2018. CHANG J F. Determination of the main active components in the stem of Ampelopsis grossedentata stems and the fingerprint study[D]. Nanning: Guangxi University of Chinese Medicine, 2018. |
| [16] |
张朝阳, 马世龙, 秦邦, 向班贵, 胡百顺, 陈永波, 刘瑶, 陈娥. 野生藤茶资源的鉴别及指纹图谱评价[J]. 山东农业大学学报(自然科学版), 2022, 53(2): 188-196. DOI:10.3969/j.issn.1000-2324.2022.02.003 ZHANG C Y, MA S L, QIN B, XIANG B G, HU B S, CHEN Y B, LIU Y, CHEN E. Identification and evaluation of fingerprint for the wild Ampelopsis grossedentata resources[J]. Journal of Shandong Agricultural University(Natural Science Edition), 2022, 53(2): 188-196. DOI:10.3969/j.issn.1000-2324.2022.02.003 |
| [17] |
PETROV D A, HARTL D L. Patterns of nucleotide substitution in Drosophila and mammalian genomes[J]. Proceedings of the National Academy of Sciences, 1999, 96(4): 1475-1479. DOI:10.1073/pnas.96.4.1475 |
| [18] |
WRIGHT F. Comparative analysis of flower-meristem-identity gene APETALA2 (AP2) codon in different plant species[J]. Gene, 1990, 87(1): 23-29. DOI:10.1016/0378-1119(90)90491-9 |
| [19] |
WRIGHT F. The 'effective number of codons' used in a gene[J]. Gene, 1990, 87(1): 23-29. DOI:10.1016/0378-1119(90)90491-9 |
| [20] |
SUEOKA N. Near Homogeneity of PR2-Bias fingerprints in the human genome and their implications in phylogenetic analyses[J]. Journal of Molecular Evolution, 2001, 53(4/5): 469-476. |
| [21] |
SHARP P M, LI W H. The codon adaptation index-a measure of directional synonymous codon usage bias, and its potential applications[J]. Nucleic Acids Research, 1987, 15(3): 1281-1295. DOI:10.1093/nar/15.3.1281 |
| [22] |
QI AN W, ZHANG J. Codon usage bias and nuclear mRNA concentration: Correlation vs. Causation[J]. Proceedings of the National Academy of Sciences, 2021, 118: e2104714118. DOI:10.1073/pnas.2104714118 |
| [23] |
GU W J, ZHOU T, MA J, SUN X, LU Z. The relationship between synonymous codon usage and protein structure in Escherichia coli and Homo sapiens[J]. Biosystems, 2004, 73: 89-97. DOI:10.1016/j.biosystems.2003.10.001 |
| [24] |
INGVARSSON P K. Gene Expression and protein length influence codon usage and rates of sequence evolution in populus tremula[J]. Molecular Biology and Evolution, 2006, 24(3): 836-844. DOI:10.1093/molbev/msl212 |
| [25] |
王义华. 葡萄属叶绿体基因组分析及其系统发育研究[D]. 武汉: 华中农业大学, 2018. WANG Y H. Chloroplast genome sequences of vitis: structural organization and phylogenetic analysis[D]. Wuhan: Huazhong Agricultural University, 2018. |
| [26] |
MINGZHAO S, FANG L, JINPING H. Analysis on codon usage of chloroplast genome of Gossypium hirsutum.[J]. Scientia Agricultura Sinica, 2011, 44(2): 245-253. DOI:10.1371/journal.pone.0242624 |
| [27] |
KAWABE A, MIYASHITA N T. Patterns of codon usage bias in three dicot and four monocot plant species[J]. Genes & Genetic Systems, 2003, 78(5): 343-352. DOI:10.1266/ggs.78.343 |
| [28] |
邓演文, 刘晓洲, 卓定龙, 曾凤. 肖蒲桃叶绿体基因组结构特征及系统发育关系[J]. 广东农业科学, 2021, 48(6): 38-46. DOI:10.16768/j.issn.1004-874X.2021.06.006 DENG Y W, LIU X Z, ZHUO D L, ZENG F. Structural characteristics and phylogenetic relationship of chloroplast genome of Syzygium acuminatissimum[J]. Guangdong Agricultural Sciences, 2021, 48(6): 38-46. DOI:10.16768/j.issn.1004-874X.2021.06.006 |
| [29] |
李冬梅, 吕复兵, 朱根发, 操君喜, 孙映波, 刘海林, 王真. 文心兰叶绿体基因组密码子使用的相关分析[J]. 广东农业科学, 2012, 39(10): 61-65. DOI:10.3969/j.issn.1004-874X.2012.10.019 LI D M, LYU F B, ZHU G F, CAO J X, SUN Y B, LIU H L, WANG Z. Analysis on codon usage of chloroplast genome of Oncidium 'Gower Ramsey'[J]. Guangdong Agricultural Sciences, 2012, 39(10): 61-65. DOI:10.3969/j.issn.1004-874X.2012.10.019 |
| [30] |
HERSHBERG R, PETROV D A. Selection on codon bias[J]. Annual Review of Genetics, 2008, 42: 287-299. DOI:10.1146/annurev.genet.42.110807.091442 |
| [31] |
ZHOU Z, DANG Y, ZHOU M, LI L, YU C H, FU J J, CHEN S, LIU Y. Codon usage is an important determinant of gene expression levels largely through its effects on transcription[J]. Proceedings of the National Academy of Sciences, 2016, 113(41): 6117-6125. DOI:10.1073/pnas.1606724113 |
| [32] |
XING Z B, CAO L, ZHOU M, XIU L S. Analysis on codon usage of chloroplast genome of Eleutherococcus senticosus[J]. China Journal of Chinese Materia Medica, 2013, 38(5): 661-5. DOI:10.7501/j.issn.0253-2670.2013.05.020 |
| [33] |
WU X M. The analysis method and progress in the study of codon bias[J]. Hereditas, 2007, 29(4): 420. DOI:10.1360/yc-007-0420 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49