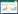文章信息
基金项目
- 眉山职业技术学院科研基金(22KY03)
作者简介
- 何帅(1995—),男,在读硕士生,研究方向为预防兽医学,E-mail:heshuai_china@yeah.net.
通讯作者
- 柴俊(1973—),女,硕士,高级实验师,研究方向为动物微生物学与免疫学,E-mail:1620728462@qq.com.
文章历史
- 收稿日期:2022-08-26
2. 云南农业职业技术学院,云南 昆明 650031;
3. 云南农业大学,云南 昆明 650201
2. Yunnan Vocational College of Agriculture, Kunming 650031, China;
3. Yunnan Agricultural University, Kunming 650201, China
【研究意义】猪圆环病毒2型(PCV2)被认为是猪圆环病毒相关疾病的主要病原体,主要侵害机体的免疫系统,造成免疫抑制,每年对世界各地的养猪业造成巨大的经济损失[1-2]。PCV2在猪群中普遍存在,与大多数单链RNA病毒一样具有快速的核苷酸更新率,增加了PCV2的遗传变异[3-4]。因此,对PCV2流行毒株的研究将有利于预测病毒的遗传演变趋势和评估现有疫苗的保护效果。【前人研究进展】我国于1998年首次发现PCV2,2001—2003年在我国不同地区进行了一次全面的PCV2分子流行病学调查[5],目前研究报道PCV2有6个基因亚型,即PCV2a、PCV2b、PCV2c、PCV2d、PCV2e和PCV2h[6],Franzo等[7]提出了一个新的分类方案,基于3个标准:最大基因型内p-距离为13%(根据ORF2基因计算),相应内部节点处的bootstrap支持度高于70%,具有至少15个可用序列。根据该方案可定义8种基因型(PCV-2a~PCV-2h),其中6种是以前提出的。PCV2的核苷酸高取代率,使得PCV2流行毒株的基因亚型持续发生演变,让流行形势变得更为复杂。PCV2病毒粒子直径14~17 nm,呈20面体对称结构,无囊膜,含有共价闭合的单股环状负链DNA,其基因组大小为1 767 bp或1 768 bp,ORF1(945 nt)是复制酶(Rep)蛋白,编码病毒DNA复制相关的蛋白质,ORF2(702 nt或705 nt)是衣壳(Cap)蛋白、病毒主要抗原决定簇[8-9]。猪圆环病毒家族成员包括PCV1、PCV2、PCV3和首次在我国湖南发现的PCV4[10]。1999年Naya等[11]首次在患呼吸系统疾病的牛的肺组织或流产牛胎儿的肺组织中发现圆环病毒PCR产物,与分离自猪的PCV2型基因组几乎完全相同,核苷酸序列同源性达99%,该报道证明了猪圆环病毒与牛呼吸道合胞病毒、牛病毒性腹泻病毒混合感染牛。2014年我国Zhai等[12-13]对奶牛、水牛和黄牛样品采用PCR分析,结果表明PCV2感染仅存在于水牛(16%,8/50)中,此外,在同一只水牛样品中,通过PCR鉴定出不同PCV2亚型DNA。【本研究切入点】目前云南省尚无PCV2在非自然宿主(牛)群体中流行情况的研究报道,本研究对云南地区PCV2在猪、牛群体间的感染情况进行调查,可以有效弥补这一空白。【拟解决的关键问题】调查云南地区PCV2在猪、牛群体间的流行情况,丰富PCV2在非自然宿主(牛)群体中流行病学资料,分析影响PCV2的流行因素,对PCV2云南株序列进行同源性分析,对ORF2蛋白进行氨基酸突变位点分析,并构建PCV2遗传进化树,为后续PCV2的防控和疫苗免疫奠定了基础。
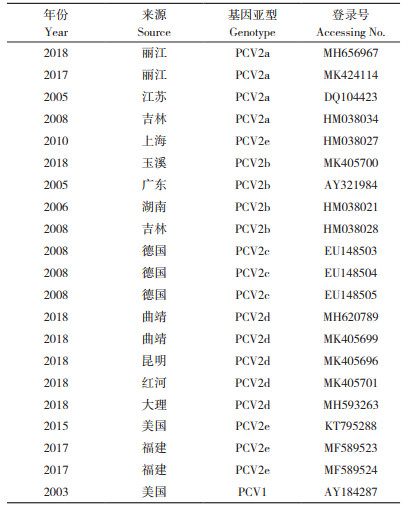
1 材料与方法 1.1 试验材料2019年9月至2020年12月调查PCV2在云南省猪群、牛群中的流行情况,从云南省普洱、昆明、曲靖、楚雄、红河、玉溪、大理、临沧、怒江、德宏、文山采集了511份疑似感染PCV2的猪组织、血液等样品,从大理、怒江、曲靖、普洱采集了272份荷斯坦奶牛的粪便、血液样品,共计783份样品(表 1),-80 ℃保存备用。
|
试验主要试剂包括2×EasyTaq® PCR SuperMix (+dye)、EasyPure® Plasmid MiniPrep Kit、EasyPure® Quick Gel Extraction Kit,均购自北京全式金生物科技有限公司;TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0、pMD™ 19-T Vector Cloning Kit、E.coli DH5α Competent Cells、DL2000 DNA marker,均购自宝生物工程(大连)有限公司。
1.2 试验方法1.2.1 引物设计与合成 根据GenBank已有的PCV2序列,设计合成PCV2-549检测引物1对(F:5'CGCTGCCACATCGAGAAAGC3',R:5'ATCAGACCCCGTTGGAATGG3'),PCV2-1767全基因反向扩增引物1对(F:5'CTCTCTATCGGAGGATTACT3',R:5' ATTACTTCCTTGGTATTTTG3')。引物由北京擎科生物科技有限公司昆明分公司合成。
1.2.2 DNA提取 将样品迅速转移至液氮预冷的研钵中,用研杵研磨组织,其间不断加入液氮,直至样品研磨成粉末状。抗凝血直接取200 μL于1.5 mL离心管中,提取DNA。样品DNA提取按照TaKaRa MiniBEST Universal Genomic DNA Extraction Kit Ver.5.0试剂盒使用说明书进行提取。
1.2.3 PCR扩增及测序 PCR反应液配制:12.5 μL 2×Easy Taq SuperMix,PCV2-549上下游引物各1 μL,1 μL DNA模板,9.5 μL ddH2O。PCR反应程序:94 ℃预变性4 min;94 ℃变性30 s、58 ℃退火30 s、72 ℃延伸50 s,35个循环;72 ℃延伸8 min,4 ℃保存。取PCR产物进行1% 琼脂糖凝胶电泳检测,凝胶成像系统拍照分析。
按照EasyPure Quick Gel Extraction Kit说明书纯化回收目的片段,将目的片段与pMD19-T载体连接,转化到DH5α感受态细胞中,在含氨苄西林的LB固体培养基培养12 h,挑取单一白色菌落,在LB液体培养基中进行增菌培养,按照EasyPure HiPure Plasmid MiniPrep Kit说明书提取质粒,并由昆明擎科生物科技有限公司测序。
1.2.4 PCV2序列分析 使用DNA Star 7.0软件MegAlign程序,采用ClustalW算法将本研究得到的6条云南PCV2流行毒株与GenBank数据库中选取的20条PCV2(a/b/d)参考毒株(表 2)进行PCV2全基因同源性分析,并以PCV1序列(AY184287)为遗传进化树树根,利用MEGA软件(7.0版)ClustalW算法,采用邻近连接法(NJ)参数模型构建PCV2系统发育进化树。
1.2.5 ORF2氨基酸序列分析 利用MegAlign软件Clustal W法对本研究得到的PCV2毒株与2株疫苗株SH(HMO38027)、LG(HMO38034)和10株GenBank数据库中选取的PCV2(a/b/d)参考毒株进行PCV2衣壳蛋白氨基酸突变位点分析。
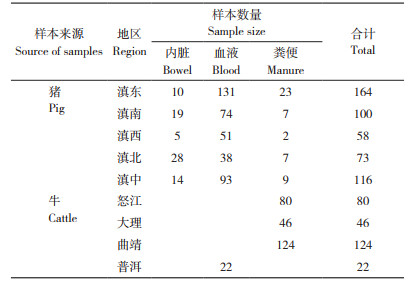
2 结果与分析 2.1 云南省PCV2的PCR检测结果通过PCR检测(图 1)发现,511份猪样品PCV2阳性率为48.73%(249/511),272份荷斯坦奶牛样品PCV2核酸检测结果均为阴性。猪的组织样品PCV2阳性率为57.89%(44/76),血液样品PCV2阳性率为51.42%(199/387),粪便样品PCV2阳性率为12.5%(6/48);滇中样品的PCV2阳性率为48.28%(56/116),滇东样品的PCV2阳性率为50.61%(83/164),滇南样品的PCV2阳性率为52.00%(52/100),滇西样品的PCV2阳性率为44.83%(26/58),滇北样品的PCV2阳性率为43.84%(32/73)。
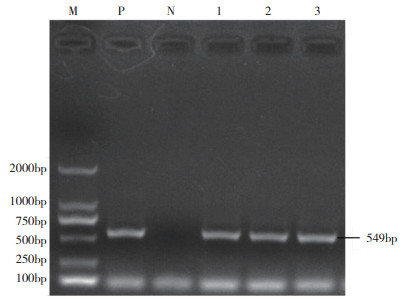
|
| M:DL2000 DNA marker;P:阳性对照;N:阴性对照;1~3:样品 M: DL-2000 DNA marker; P: Positive control; N: Negative control; 1-3: Samples 图 1 PCV2的PCR检测电泳结果 Fig. 1 PCR electrophoresis result of PCV2 |
从阳性样品中成功扩增出6条PCV2全基因序列,其中5株全基因序列为1 767bp(图 2),1株全基因序列为1 768 bp,将其序列上传至GenBank数据库,得到如下登录号:LinCang-202006(MW217511)、JinNing-202006(MW653451)、LuQuan-202004(MW653452)、WenShan-202004(MW653453)、DeHong-202005(MW653450)、WenShan-202009(MW653449)。
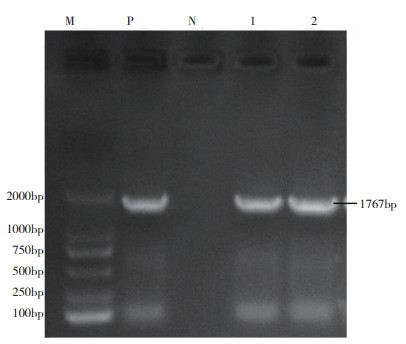
|
| M:DL2000 DNA marker;P:阳性对照;N:阴性对照;1~2:阳性样品 M: DL2000 DNA marker; P: Positive control; N: Negative control; 1-2: Positive samples 图 2 PCV2全基因扩增电泳结果 Fig. 2 Whole gene amplification electrophoresis result of PCV2 |
2.2 PCV2全基因组序列比对分析
本研究得到的6条PCV2全基因组序列同源性在94.6%~99.9% 之间(图 3),其中DeHong-202005毒株与WenShan-202009毒株同源性最高(99.9%),DeHong-202005毒株与WenShan-202004毒株、WenShan-202004毒株与WenShan-202009毒株同源性最低(94.6%);与疫苗株LG(HM038034)和SH(HM038027)同源性在95.0%~97.9% 之间,与GenBank数据库中20条来自国内外不同地区的PCV2(a/b/c/d/e)参考株同源性在91.4%~99.8% 之间。

|
| 图 3 云南PCV2流行毒株同源性分析 Fig. 3 Homology analysis of PCV2 epidemic strains in Yunnan |
以PCV1序列(AY184287)为系统发育进化树树根,构建Neighbor-Joining Tree系统发育树(图 4)。本研究得到的6条PCV2毒株经系统发育分析,结果显示为有2株PCV2a(WenShan-202004、LuQuan-202004)、2株PCV2b(JinNing-202006、LinCang-202006)、2株PCV2d(DeHong-202005、WenShan-202009)。据系统发育树可知,2株PCV2a毒株与PCV2a参考株分离形成一个独立进化分支,与疫苗株LG(HM038034)亲缘关系较远;2株PCV2b毒株与参考毒株MK405700形成独立进化支,与疫苗株SH(HM038027)亲缘关系较远;2株PCV2d毒株与KunMing(MK405696)亲缘关系较近,形成独立进化分支,云南PCV2d流行毒株自2019年开始与之前鉴定的PCV2d流行毒株分离形成一个独立进化分支。
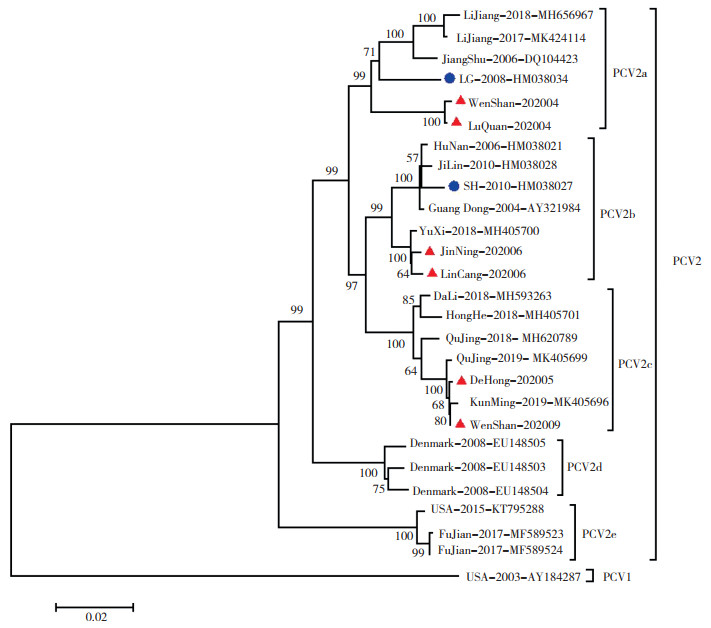
|
| ●:PCV2疫苗株;▲:新鉴定云南PCV2流行株 ●: PCV2 vaccine strain; ▲ : Newly identified PCV2 epidemic strain in Yunnan 图 4 云南PCV2流行毒株全基因序列系统发育树 Fig. 4 Phylogenetic tree based on the whole gene sequence of PCV2 epidemic strain in Yunnan |
2.3 PCV2 ORF2氨基酸序列分析
WenShan-202004、LuQuan-202004、JinNing-202006、LinCang-202006毒株ORF2均含233个氨基酸序列,DeHong-202005、WenShan-202009毒株ORF2均含234个氨基酸序列,DeHong-202005毒株和WenShan-202009毒株与PCV2(a/b)参考毒株比较,在第234位多1个赖氨酸(K) 残基(图 5)。WenShan-202004、LuQuan-202004具有PCV2a的典型氨基酸序列,位于第86~91位氨基酸的TNKISI序列,WenShan-202004、LuQuan-202004与LG (HM038034)疫苗株相比、在21(L21S)、47 (T41A)、80(V80L)、130(V130F)、133(V133S)、185(L185M)、187(L187I) 等氨基酸位点发生突变,WenShan-202004还在3 (Y3H)、219(V219I) 等氨基酸位点发生突变。JinNing-202006、LinCang-202006两株具有PCV2b的典型氨基酸序列,分别是位于86~91的SNPRSV与位于191、206、210位点的GIE氨基酸序列;JinNing-202006、LinCang-202006与SH (HM038027)疫苗株相比,在59(R59K)、63 (K63R)、190(A190T)、228(A228D)、230(T230P) 等氨基酸位点发生突变,JinNing-202006在219氨基酸位点(V219A)发生突变,LinCang-202006在203氨基酸位点(E203G) 发生突变。DeHong-202005、WenShan-202009两株具有位于86~91位点的SNPLTV和190、191、206、210、234位点的TGIDK典型氨基酸序列,这些位点均属于PCV2d的典型氨基酸序列。

|
| 图 5 云南PCV2流行毒株ORF2突变位点分析 Fig. 5 Analysis of mutation sites of ORF2 from PCV2 epidemic strains in Yunnan |
3 讨论
近年来,我国多个省份相继检测到猪群感染PCV2的高阳性率[14-15],PCV2感染引发的相关疾病造成生猪生产性能下降甚至死亡,给生猪养殖业带来严重的经济损失。据Lyu等[16]报道,云南省2016—2019年PCV2阳性率为60.93%,高于本次调查结果,说明PCV2在云南省的流行程度有减弱趋势,近年的PCV2防控措施取得了一定成果。据宋春莲等[17]调查发现,PCV2在滇中的阳性率为48.62%,在滇南的阳性率为29.03%,在滇西的阳性率为47.37%,在滇东的阳性率为27.27%,而本次云南省PCV2猪群流行情况调查结果显示,PCV2在滇南地区的阳性率最高、为52.00%,这可能是滇南地区靠近越南、缅甸、泰国等国家,边境线防疫难度大,生物安全风险系数较高所致。
Zhai等[12-13]在2014年采集50份水牛组织样品,其中有8份呈PCV2阳性,而来自奶牛的180份血液样品和来自黄牛的50份组织样品均呈PCV2阴性,且PCV2毒株在水牛中具有遗传多样性,可分为3种不同的基因型(PCV2b、PCV2d和PCV2e)。本研究的272份荷斯坦奶牛样品呈PCV2阴性,与Zhai等的调查研究报告一致,但Zhai等确定PCV2在水牛群体中流行,这可能与水牛的生活习惯有关。一般来说,我国大多数奶牛和黄牛都是在牧场圈养,而水牛主要在我国南方的农村地区放牧,周边的水系经常被猪的粪便污染,这可能是导致水牛感染PCV2的原因。
1996—2000年初,PCV2a是临床感染猪中最普遍的基因型,之后PCV2b占主导地位(“基因型转变”) [18-19],PCV2d最早在我国报道[20],第二次“基因型转变”(从PCV2b到PCV-2d) 似乎正在全球发生[21],这可能是由全球使用PCV2疫苗推动的。Lyu等[16]在2020年公开的15株PCV2云南毒株,其中有2株PCV2a亚型、1株PCV2b亚型、12株PCV2d亚型,PCV2d成为主要流行基因型。本研究获得的6株PCV2分别属于PCV2a、PCV2b和PCV2d这3个基因亚型,每种基因亚型各2株,表明PCV2感染在云南猪群中的遗传多样性不断增加,3种基因型在猪群中共同存在,可见近年来云南PCV2的流行情况较为复杂。
不同PCV2基因型的ORF2编码衣壳蛋白可以根据Cap蛋白中的典型序列来区分,PCV2a存在典型的氨基酸序列TNKISI,PCV2b存在典型的氨基酸序列TNKISI、PNPRSV和A/TGIE,PCV2d存在典型的氨基酸序列SNPLTV和TGID[22]。本研究的6株PCV2毒株,Cap蛋白具有其基因亚型的典型氨基酸序列,其中2株(MW653449、MW653450,PCV2d亚型)ORF2长度为705 bp,编码234个氨基酸,其余4个PCV2毒株的ORF2长度均为702 bp,编码233个氨基酸。本研究的PCV2d毒株均在Cap蛋白的C末端终止密码子之前增加了一个赖氨酸(K),DeHong-202005/WenShan-202009毒株与参考毒株相比在第169位氨基酸位点(S169R/T)发生特有突变,该演化可能会对毒株的毒力和免疫逃避产生影响。
4 结论本研究获得6株PCV2毒株,PCV2a、PCV2b和PCV2d基因亚型各2株,这说明PCV2感染在云南地区猪群中呈多样性,防控难度加大;PCR检测结果证明PCV2在云南地区猪群中普遍存在,荷斯坦牛奶尚未感染PCV2;PCV2云南株遗传进化分析结果证明,PCV2(a/b)衣壳蛋白氨基酸序列朝着PCV2d基因亚型不断进化,DeHong-202005/WenShan-202009毒株与参考毒株相比在第169位氨基酸位点(S169R/T)发生特有突变,PCV2在亲缘关系上受地域影响较小,受时间因素影响较大。本研究结果明确了云南省PCV2感染现状及流行毒株的分子流行病学特点、遗传演化情况,可为云南地区今后更有效地预防控制PCV2感染及疫苗选择提供参考。
| [1] |
OPRIESSNIG T, MENG X J, HALBUR P G. Porcine circovirus type 2 associated disease: update on current terminology, clinical manifestations, pathogenesis, diagnosis, and intervention strategies[J]. Journal of Veterinary Diagnostic Investigation, 2007, 19(6): 591-615. DOI:10.1177/104063870701900601 |
| [2] |
曹宗喜, 郑心力, 张艳, 叶保国, 林哲敏, 王峰. PCV2海南株全基因组的克隆与序列分析[J]. 广东农业科学, 2015, 42(8): 118-123. DOI:10.16768/j.issn.1004-874X.2015.08.00 CAO Z X, ZHENG X L, ZHANG Y, YE B G, LIN Z M, WANG F. Cloning and sequence analysis of the whole genome of PCV2 Hainan strain[J]. Guangdong Agricultural Sciences, 2015, 42(8): 118-123. DOI:10.16768/j.issn.1004-874X.2015.08.00 |
| [3] |
FIRTH C, CHARLESTON MA, DUFFY S, SHAPIRO B, HOLMES E C. Insights into the evolutionary history of an emerging livestock pathogen: porcine circovirus 2[J]. Journal of Virology, 2009, 83(24): 12813-12821. DOI:10.1128/JVI.01719-09 |
| [4] |
WANG H, GU J, XING G, QIU X, AN S, WANG Y, ZHANG C, LIU C, GONG W, TU C, SU S, ZHOU J. Genetic diversity of porcine circovirus type 2 in China between 1999-2017[J]. Transboundary and Emerging Diseases, 2019, 66(1): 599-605. DOI:10.1111/tbed.13040 |
| [5] |
ZHAI S L, CHEN S N, XU Z H, TANG M H, WANG F G, LI XJ, SUN B B, DENG S F, HU J, LYU D H, WEN X H, YUAN J, LUO M L, WEI W K. Porcine circovirus type 2 in China: an update on and insights to its prevalence and control[J]. Virology Journal, 2014, 11: 88. DOI:10.1186/1743-422X-11-88 |
| [6] |
WIEDERKEHR D D, SYDLER T, BUERGI E, HAESSIG M, ZIMMERMANN D, POSPISCHIL A, BRUGNERA E, SIDLER X. A new emerging genotype subgroup within PCV-2b dominates the PMWS epizooty in Switzerland[J]. Veterinary Microbiology, 2009, 136(1/2): 27-35. DOI:10.1016/j.vetmic.2008.10.028 |
| [7] |
FRANZO G, SEGAL S J. Porcine circovirus 2 (PCV-2) genotype update and proposal of a new genotyping methodology[J]. PLoS ONE, 2018, 13(12): e0208585. DOI:10.1371/journal.pone.0208585 |
| [8] |
NAWAGITGUL P, MOROZOV I, BOLIN S R, HARMS P A, SORDEN S D, PAUL P S. Open reading frame 2 of porcine circovirus type 2 encodes a major capsid protein[J]. Journal of General Virology, 2000, 81(9): 2281-2287. DOI:10.1099/0022-1317-81-9-2281 |
| [9] |
LIU J, CHEN I, DU Q, CHUA H, KWANG J. The ORF3 protein of porcine circovirus type 2 is involved in viral pathogenesis in vivo[J]. Journal of Virology, 2006, 80(10): 5065-5073. DOI:10.1128/JVI.80.10.5065-5073.2006 |
| [10] |
ZHANG H H, HU W Q, LI JY, LIU T N, ZHOU J Y, OPRIESSNIG T, XIAO C T. Novel circovirus species identified in farmed pigs designated as Porcine circovirus 4, Hunan province, China[J]. Transboundary and Emerging Diseases, 2020, 67(3): 1057-1061. DOI:10.1111/tbed.13446 |
| [11] |
NAYAR G P, HAMEL A L, LIN L, SACHVIE C, GRUDESKI E, SPEARMAN G. Evidence for circovirus in cattle with respiratory disease and from aborted bovine fetuses[J]. Canadian Veterinary Journal, 1999, 40(4): 277-278. |
| [12] |
ZHAI S L, CHEN R A, CHEN S N, WEN X H, LYU D H, WU D C, YUAN J, HUANG Z, ZHOU X R, LUO M L, HE D S, WEI W K. First molecular detection of porcine circovirus type 2 in bovids in China[J]. Virus Genes, 2014, 49(3): 507-511. DOI:10.1007/s11262-014-1117-1 |
| [13] |
ZHAI S L, HE D S, QI W B, CHEN S N, DENG S F, HU J, LI X P, LI L, CHEN R A, LUO M L, WEI W K. Complete genome characterization and phylogenetic analysis of three distinct buffalo-origin PCV2 isolates from China[J]. Infection, Genetics and Evolution, 2014, 28: 278-282. DOI:10.1016/j.meegid.2014.10.005 |
| [14] |
李玲, 李国新, 周艳君, 童武, 王礞礞, 童光志. 2008 ~2011年中国部分地区猪圆环病毒2型的分子流行病学调查[J]. 中国动物传染病学报, 2012, 20(2): 1-10. LI L, LI G X, ZHOU Y J, TONG W, WANG M M, TONG G Z. Molecular epidemiological investigation of porcine circovirus type 2 in some areas of China from 2008 to 2011[J]. Chinese Journal of Animal Infectious Diseases, 2012, 20(2): 1-10. |
| [15] |
李娇, 王文秀, 谢金文, 李峰, 沈志强. 我国猪圆环病毒病的流行现状分析[J]. 养猪, 2016(6): 97-100. DOI:10.13257/j.j.cnki.21-1104/s.2016.06.032 LI J, WANG W X, XIE J W, LI F, SHEN Z Q. Analysis of the prevalence of porcine circovirus disease in China[J]. Pig Farming, 2016(6): 97-100. DOI:10.13257/j.j.cnki.21-1104/s.2016.06.032 |
| [16] |
LYU N, ZHU L, LI W, LI Z, QIAN Q, ZHANG T, LIU L, HONG J, ZHENG X, WANG Y, ZHANG Y, CHAI J. Molecular epidemiology and genetic variation analyses of porcine circovirus type 2 isolated from Yunnan Province in China from 2016-2019[J]. Veterinary Research, 2020, 16(1): 96. DOI:10.1186/s12917-020-02304-8 |
| [17] |
宋春莲, 曾敬元, 陶杨, 张雪, 谢相悦, 刘永波, 吴常月, 李鑫, 凌万旭, 何锦, 张桂生, 兰春林, 王艳芬, 李鲜, 舒相华. 2012年—2016年云南省部分地区规模猪场4种常见病毒感染情况的调查[J]. 动物医学进展, 2018, 39(1): 117-120. DOI:10.16437/j.cnki.1007-5038.2018.01.021 SONG C L, ZENG J Y, TAO Y, ZHANG X, XIE X Y, LIU Y B, WU C Y, LI X, LING W X, HE J, ZHANG G S, LAN C L, WANG Y F, LI X, SHU X H. Survey of four common viral infections in large-scale pig farms in some areas of Yunnan Province from 2012 to 2016[J]. Advances in Animal Medicine, 2018, 39(1): 117-120. DOI:10.16437/j.cnki.1007-5038.2018.01.021 |
| [18] |
FRANZO G, SEGAL S J. Porcine circovirus 2 (PCV-2) genotype update and proposal of a new genotyping methodology[J]. PLoS ONE, 2018, 13(12): e0208585. DOI:10.1371/journal.pone.0208585 |
| [19] |
WIEDERKEHR D, SYDLER T, BUERGI E, HAESSIG M, ZIMMERMANN D, POSPISCHIL A, BRUGNERA E, SIDLER X. A new emerging genotype subgroup within PCV-2b dominates the PMWS epizooty in Switzerland[J]. Veterinary Microbiology, 2009, 136(1/2): 27-35. DOI:10.1016/j.vetmic.2008.10.028 |
| [20] |
GUO L J, LU Y H, WEI Y W, HUANG L P, LIU C M. Porcine circovirus type 2 (PCV2): genetic variation and newly emerging genotypes in China[J]. Virology Journal, 2010, 7: 273. DOI:10.1186/1743-422X-7-273.PMC2967541 |
| [21] |
FRANZO G, CORTEY M, SEGAL S J, HUGHES J, DRIGO M. Phylodynamic analysis of porcine circovirus type 2 reveals global waves of emerging genotypes and the circulation of recombinant forms[J]. Molecular Phylogenetics and Evolution, 2016, 100: 269-280. DOI:10.1016/j.ympev.2016.04.028 |
| [22] |
CHEUNG A K. Homologous recombination within the capsid gene of porcine circovirus type 2 subgroup viruses via natural co-infection[J]. Archives of Virology, 2009, 154(3): 531-534. DOI:10.1007/s00705-009-0329-5 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49