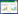文章信息
基金项目
- 黑龙江省青年科学基金(QC2018027);黑龙江省博士后资助经费(LBH-Z17203)
作者简介
- 孙莉莉(1983—),女,博士,副研究员,研究方向为分子遗传育种,E-mail:sunlilineau@163.com.
通讯作者
- 牟蕴慧(1965—),女,研究员,研究方向为寒地核果类果树育种与栽培技术,E-mail:yunhuimu@163.com.
文章历史
- 收稿日期:2022-01-05
2. 哈尔滨体育学院运动人体科学学院,黑龙江 哈尔滨 150008
2. College of Sport Human Sciences, Harbin Sport University, Harbin 150008, China
【研究意义】李(Prunus salicina L.)属蔷薇科(Rosaceae)李属(Prunus)植物,李属分类复杂且较难区分,世界上共有30~40种[1],国际上将李亚属分为杏组(Sect. Armeniaca)、真李组(Sect. Euprunus)和樱李组(Sect. Prunocerasus)[2]。真李组包括中国李(Prunus salicina Lindl.)、杏李(Prunus simonii Carr.)、乌苏里李(Prunus ussuriensis Kov. & kost.)、欧洲李(Prunus domestica L.)、樱桃李(Prunus cerasifera Ehrh.)及黑刺李(Prunus spinosa L.)等6种,其中商品李为二倍体的中国李(又称日本李)和六倍体的欧洲李[3]。我国李属植物分布广、适应性强,在浙江、福建、广东、云南、四川、陕西、东北等地均有驯化栽培的品种[3],其资源种类及数量均居世界第1位[4]。黑龙江省位于我国最北区域,李的抗寒种质资源丰富,与其他作物相比,李的经济效益比较显著[5]。由于优质、大果、耐贮运材料稀少,加之传统育种方法耗时长、成效低,亟需借助分子生物学手段加快育种进程。高质量遗传图谱的构建是分子标记辅助育种的基础。【前人研究进展】随着分子生物学的快速发展,DNA分子标记技术已经成为一种重要研究方法广泛应用于分子鉴定[6]、杂交种纯度分子检测[7]、种质资源多样性[8-9]、分子遗传图谱构建[10]、遗传多样性分析[11-13]及指纹图谱构建[14-15]等多个领域。DNA分子标记是遗传图谱构建的主要手段,孙文英等[16]利用AFLP分子标记技术构建梨(Pyrus L.)的遗传连锁图谱。艾小艳等[17]概述了构建桃(Prunus persica L.)遗传图谱的多种分子标记方法,包括基于第一、第二代分子标记的RFLP(Restriction Fragment Length Polymorphism,限制性内切酶片段长度多态性)、SSR(Simple Sequence Repeat,简单重复序列)、AFLP(Amplified Fragment Length Polymorphism,扩增片段长度多态性)和RAPD(Random Amplified Polymorphic DNA,随机引物扩增多态性DNA)技术以及基于第三代分子标记的SNP(Single Nucleotide Polymorphism,单核苷酸多态性)技术。另外,Linge等[18]也利用SNP技术构建桃的高密度遗传图谱。章秋平等[19]利用SSR和SRAP(Sequence-related Amplified Polymorphism,相关序列扩增多态性)标记构建了两张杏(Prunus armeniaca L.)遗传连锁图谱。Dang等[20]通过SRAP、AFLP和ISSR(Inter-simple Sequence Repeat,内部简单重复序列)标记构建芒果(Mangifera indica L.)遗传连锁图谱。由此可见,通过分子标记技术构建遗传连锁图谱是切实可行的方法之一。分子遗传连锁图谱主要用于性状定位、分子标记辅助育种及功能基因克隆研究等,同时在遗传学、遗传分子育种及功能基因组学等方面发挥重要作用[21-23]。此外,对于遗传背景复杂的木本植物而言,“双假测交”理论的提出为多年生果树遗传图谱的研究提供了理论依据[24]。【本研究切入点】目前,国内外有关李的研究主要集中在种质资源调查[3]、遗传多样性[25-26]、果实性状分析[27]等方面,而关于李遗传连锁图谱构建方面的研究鲜见报道[28]。东北地区独特的地理环境和丰富的李种质资源,有利于开展优质、抗寒新品种的选育工作。如前所述,分子标记辅助育种技术可以加快育种进程,而遗传连锁图谱又是分子辅助育种的基础。【拟解决的关键问题】本研究以东北地区品种吉林6号和龙园秋李作为亲本,以其F 1后代作为作图群体,通过ISSR和SRAP分子标记构建李的遗传连锁图谱,为后续李的分子辅助育种提供理论依据,推进优质、抗寒李品种的选育。
1 材料与方法 1.1 试验材料供试材料来源于黑龙江省农业科学院园艺分院,以吉林6号和龙园秋李为亲本材料,其中母本为吉林6号,树势矮小,果实较小,平均单果质量40 g,果实成熟时为红色,离核,8月中旬果实成熟;父本为龙园秋李(九三杏梅×台湾李),抗寒、丰产、树姿直立,耐红点病;果实扁圆形,平均单果质量75 g,最大果质量110 g;成熟时全面紫红色,半离核,较耐贮运,9月初果实成熟,是北方寒地果实最大品种。
以F1子代实生苗为作图群体,选取80个F1子代进行样本采集。杂交群体于2009年杂交,2010年播种,2012年定植于黑龙江省农业科学院园艺分院核果试验园,长势较好,稀有开花结果,没有进行剔选。同时对F1子代进行连续两年的数据调查,调查内容包括部分生长性状和果实性状,其中叶片长、宽和果实横径、纵径经数据分析呈正态分布(数据未显示),用于QTL定位分析。
1.2 试验方法1.2.1 DNA提取 分别采集吉林6号和龙园秋李亲本及F1子代植株的幼嫩叶片,于-80℃冰箱中保存备用。根据天根植物DNA提取试剂盒(DP305)的相关步骤进行DNA的提取,提取后采用1.0% 琼脂糖凝胶电泳进行DNA质量的检测,电泳缓冲液为1×TAE,观察并拍照,用于后续引物筛选及遗传图谱的构建。
1.2.2 F1子代真实性鉴定 根据本课题组前期工作积累,选取特异性条带清晰的SSR引物UDP96-005(F′GTAACGCTCGCTACCACAAA、R′CCTGCATATCACCACCCAG)来鉴定子代的真实性,比较F1群体与双亲的基因型,去除后代中与双亲基因型不一致的植株。
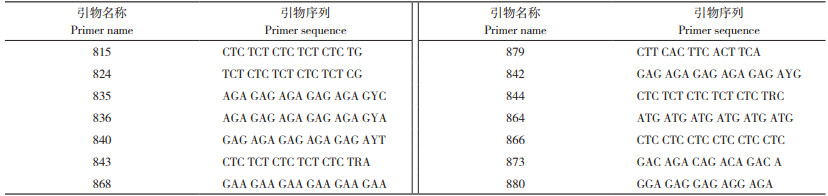
1.2.3 ISSR及SRAP引物筛选 试验所用的ISSR引物从UBC800-UBC900中随机选取44条,SRAP引物采用已公布的引物序列[29],由me1-me10及em1-em20组合成200对引物进行筛选,引物均由上海生工生物工程技术服务有限公司合成。
对亲本材料和2个杂交后代基因组DNA进行ISSR和SRAP引物筛选,试验选取PCR扩增条带清晰、重复性好且多态性较丰富的14条ISSR引物(表 1)和16对SRAP引物(表 2)用于后续研究。
1.2.4 PCR扩增 ISSR-PCR反应体系为20 μL:10 × PCR Buffer 2μL,引物(50 pmol /μL)2 μL,dNTP(each 10 mmol/L)0.4 μL,模板(DNA)1 μL,Taq Plus DNA Polymerase(2 U/μL)0.5 μL,加ddH2O 14.1 μL。PCR反应程序:95℃预变性5 min;94℃变性1 min,52~62℃退火1 min,72℃延伸2 min,40个循环;72℃延伸10 min;4℃保存。1.5% 琼脂糖凝胶,1×TAE,150 V,100 mA,30 min电泳观察。
SRAP-PCR反应体系为15 μL:Taq PCR Master Mix,7.5 μL,正向引物(50 pmol /μL)1 μL,反向引物(50 pmol /μL)1 μL,模板(DNA)1μL,加ddH2O 4.5 μL。PCR反应程序:94℃预变性5 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,30个循环;72℃延伸8 min;4℃保存。1.5% 琼脂糖凝胶,1×TAE,150 V,100 mA,30 min电泳观察。
1.3 数据分析根据Join Map 4.0软件中CP作图模型(hk×hk、nn×np、lm×ll、ef×eg、ab×cd等5种分离类型)的要求,将PCR所得凝胶结果在Excel中按照模型要求进行数据录入,将作图群体个体数和每个个体的基因型横向录入,将引物位点名称和分离类型纵向录入,其中hk×hk(双亲均为杂合的基因型,h对k为显性)、nn×np(母本为纯隐性基因型nn、父本为杂合基因型np,p对n为显性)、lm×ll(母本为杂合基因型lm、父本为纯隐性基因型ll,m对l为显性),选用LOD值为0.5~10.0的连锁群,采用Kosambi函数计算图谱距离。本试验引物编号为引物名+ 条带分子量,用于构建李遗传图谱。
2 结果与分析 2.1 DNA提取采用植物DNA提取试剂盒,分别对2个亲本及80个F1子代进行DNA提取,提取后采用1% 琼脂糖凝胶电泳检测DNA质量。部分电泳结果见图1,图 1显示基因组DNA条带完整无降解。
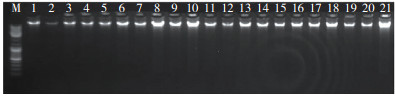
|
| Marker;1~21:F1部分后代 Marker; 1-21: Some F1 progenies 图 1 F1后代DNA电泳结果 Fig. 1 Electrophoresis result of DNA from F1 progenies |
2.2 F1子代真实性鉴定
根据双亲基因型和子代群体的基因型,挑选出与双亲基因型不符的子代,通过筛选有72个个体为真实性子代,用于后期群体验证。F1子代真实性部分鉴定结果见图 2,根据亲本条带分布对子代条带进行对比分析,如图 2子代2和子代10箭头所示,出现亲本中没有的条带,被鉴定为非真实性子代,排除群体之外。
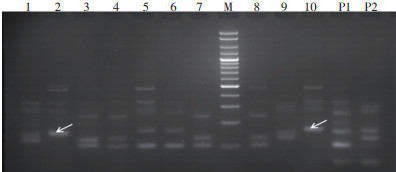
|
| Marker;P1:母本;P2:父本;1~10:F1部分后代 Marker; P1: Female parent; P2: Male parent; 1-10: Some F1 progenies 图 2 部分F1子代真实性鉴定结果 Fig. 2 Authenticity identification results of some F1 progenies |
2.3 分子标记的多态性分析
首先将44条ISSR引物、200对SRAP引物在亲本和2个真实性后代中进行多态性筛选,根据PCR凝胶结果分析得知,44条ISSR引物中有14条引物条带稳定且清晰,引物比例为31.8%,ISSR和SRAP获得的标记数量如表 3所示,ISSR引物产生等位基因数为74个,每条引物均产5.3个;200对SRAP引物中有16对具有多态性,引物比例为8.0%,SRAP引物产生等位基因数为46个,每对引物均产2.9个。本研究将筛选出的14条ISSR引物和16对SRAP引物进行亲本和72个F1后代的PCR扩增,以构建遗传图谱。引物在F1群体中产生标记位点120个,经χ2检验后,偏分离位点有9个,频率为7.5%。
2.4 标记分离分析
利用ISSR和SRAP分子标记对李F1后代进行亲本和群体检测,发现多态性标记在作图群体中有不同的分离类型,根据Join Map 4.0软件中的CP作图模型,将亲本及F 1后代的基因型划分为hk×hk、nn×np、lm×ll、ef×eg和ab×cd等5种类型,每种分离类型在ISSR和SRAP标记中的数量如表 3所示,nn×np类型总数相对较高。hk×hk型分离比例为1 ︰ 2 ︰ 1(图 3A),nn×np型(图 3B)、lm×ll型(图 3C)分离比例为1 ︰ 1,ef×eg型如图 3D所示,ab×cd型如图 3E所示。

|
| A: ISSR868-2500;B: ISSR842-600;C: ISSR844-1000;D: me4em15-900;E: ISSR824-2000。1~14:F1 图 3 ISSR及SRAP引物检测到亲本和F1后代中的杂合位点分离类型 Fig. 3 Segregation pattern of heterozygous loci in parents and F1 progenies detected by ISSR and SRAP primers |
2.5 分子遗传连锁图谱的构建
将获得数据导入Join Map 4.0软件的CP模型中,将LOD=5.0设置为标记连锁群,同时将偏分离的9个标记去掉,对120个标记位点进行分析,成功构建了李遗传连锁图谱,该图谱包含38个标记,其中ISSR标记31个、SRAP标记7个,分布在16个遗传连锁群内,如图 4所示。该遗传图谱在李基因组中覆盖528.5 cM,标记间平均图距13.9 cM,连锁群LG1最长(71.1 cM),LG4最短(9.6 cM)。分布在连锁群上的标记数最多为4个,最少为2个。多态性标记间最大遗传距离为52.8 cM,位于LG5的ISSR842-2250与me1em20-700之间;最小遗传距离为1.4 cM,位于LG4的ISSR880-500与ISSR880-600之间。
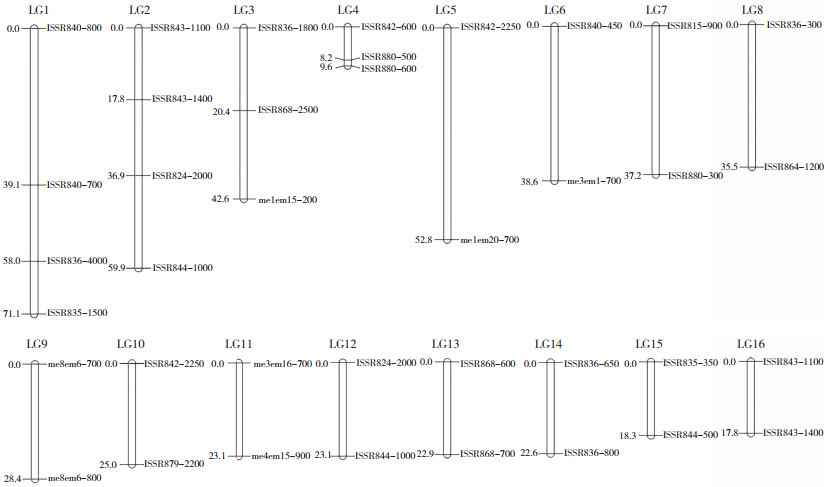
|
| 图 4 李遗传连锁图谱 Fig. 4 Genetic linkage map of Prunus salicina L. |
3 讨论
遗传研究和分子育种方法需要基本的基因组资源,如分子标记和连锁图谱。为开发李遗传研究资源,本研究利用分子标记方法构建李遗传连锁图谱。李为多年生木本植物,遗传背景复杂,且为异花授粉植物,很难获得自交群体[30],而“双假测交”理论可以很好地解决这一难题,为李的遗传图谱构建和分子辅助育种提供了理论依据。同时,对遗传图谱构建来说,用于作图的亲本和群体选择尤为重要,遗传图谱作图亲本的选择应以遗传背景差异较大、亲缘关系较远为好。本研究选择吉林6号和龙园秋李为作图亲本,依据前期遗传多样性分析结果显示,两者亲缘关系较远[31],符合遗传图谱亲本选择的要求。另外,关于作图群体数量的阐述,章秋平等[23]研究表明,核果类果树遗传图谱群体大小范围为48~297株子代不等,本研究通过F1子代真实性鉴定,72株植株为真实性子代,群体数量适宜进行遗传图谱构建的研究。
分子标记方法具有引物通用性好、操作简单易行、多态性好且分布均匀等优点,被广泛应用于许多林木的遗传连锁图谱构建中[32-33]。本研究利用ISSR和SRAP分子标记,通过多态性筛选选出14条ISSR引物和16对SRAP引物来构建李的遗传连锁图谱。通过查阅文献得知,关于分子标记引物个数和标记方法的选择,不同研究存在较大差异。战晴晴等[34]利用28条ISSR和14对SSR多态性引物构建了北柴胡(Bupleurum chinense DC.)遗传连锁图谱,包含72个ISSR和8个SSR位点;王晓敏等[35]利用14对SRAP引物组合、9条ISSR引物及10对TRAP引物构建了金针菇(Flammulina filiformis)分子遗传连锁图谱,获得185个标记位点,其中SRAP标记76个、ISSR标记33个、TRAP标记74个、不亲和性因子位点2个。本研究中,共产生120个清晰稳定的标记位点,其中ISSR标记74个、SRAP标记46个,最终有38个标记,其中31个ISSR标记和7个SRAP标记。由此可见,不同标记的组合选择可以最大程度地覆盖基因组,有利于增加图谱密度和提高图谱质量。
有研究表明,李是一种自交不亲和的二倍体种(2n=2X=16)[36],Juan等[37]对日本李进行SNP连锁分析,获得8个连锁群,同时将不同性状进行QTL定位,其中果实成熟时间定位在LG4上,果皮颜色定位在LG3和LG4上。Basilio等[38]利用GBS(Gnotyping-by-Sequencing)方法获得日本李高质量SNP标记1 441个,分布在8个连锁群内。本研究获得的连锁群数为16个,与上述研究结果不一致,可能原因是本研究采用的分子标记方法较少,获得的标记数较少,在基因组中分布较分散导致的,可以通过增加后代和分子标记的数量来缩小差距、提高连锁图谱的密度和分辨率。本研究结果丰富了李遗传连锁图谱的分子标记类型和标记位点,能更好地为李的基因定位和分子遗传育种提供理论依据。
4 结论本研究构建了李遗传连锁图谱,以东北地区吉林6号和龙园秋李为作图亲本,利用SSR分子标记方法筛选出72个真实性子代。分别利用14条ISSR引物获得74个等位基因,16对SRAP引物产生46个等位基因。对PCR扩增结果进行条带分析,获得亲本及F1后代的5种基因型,分别是hk×hk、nn×np、lm×ll、ef×eg和ab×cd,其中nn×np类型数目相对较高。引物在F1群体中产生标记位点120个,经χ2检验后,偏分离频率为7.5%,利用Join Map 4.0软件的CP作图模型进行Kosambi函数计算,构建含有16个连锁群的连锁图谱,包含38个分子标记位点,覆盖基因组长度为528.5 cM。李遗传连锁图谱的成功构建可以推进东北地区李优质、抗寒新品种的选育和生产实践。
| [1] |
TOPP B L, RUSSELL D M, NEUMULLER M, DALBO M A, LIU W. Fruit breeding, handbook of plant breeding[M]. BADENES M L, BYRNE D H, Eds. . New York: Springer Science + Business Media, 2012: 571-621.
|
| [2] |
REALES A, SARGENT D J, TOBUTT K R, RIVERA D. Phylogeneticsof Eurasian plums, Prunus Lsection Prunus (Rosaceae), according to coding and non-coding chloroplast DNA sequences[J]. Tree Genetics & Genomes, 2010, 6: 37-45. DOI:10.1007/s11295-009-0226-9 |
| [3] |
魏潇, 章秋平, 刘威生. 中国李种质资源研究进展[J]. 园艺学报, 2020, 47(6): 1203-1212. DOI:10.16420/j.issn.0513-353x.2019-0677 WEI X, ZHANG Q P, LIU W S. Research progress on plum germplasm resources in China[J]. Acta Horticulturae Sinica, 2020, 47(6): 1203-1212. DOI:10.16420/j.issn.0513-353x.2019-0677 |
| [4] |
张加延. 我国李杏种质资源调查研究的突破性进展[J]. 园艺与种苗, 2011(2): 7-10, 37. DOI:10.3969/j.issn.2095-0896.2011.02.002 ZHANG J Y. Disruptive progress of plum and apricot germplasm resources investigation in China[J]. Horticulture & Seed, 2011(2): 7-10, 37. DOI:10.3969/j.issn.2095-0896.2011.02.002 |
| [5] |
牟蕴慧, 甄灿福, 刘海军, 金庆明. 黑龙江省李、杏生产现状及产业发展建议[J]. 中国园艺文摘, 2013(6): 44-47. MU Y H, ZHEN C F, LIU H J, JIN Q M. Production status and industrial development suggestions of plum and apricot in Heilongjiang Province[J]. Chinese Horticulture Abstracts, 2013(6): 44-47. |
| [6] |
石乃星, 文国松, 赵明富. 黄精属植物DNA分子鉴定技术应用研究进展[J]. 植物遗传资源学报, 2021, 22(5): 1209-1218. DOI:10.13430/j.cnki.jpgr.20210315005 SHI N X, WEN G S, ZHAO M F. Application of DNA molecular identification technology in Polygonatum Mill[J]. Journal of Plant Genetic Resources, 2021, 22(5): 1209-1218. DOI:10.13430/j.cnki.jpgr.20210315005 |
| [7] |
陈梦洁, 吕长平, 秧拯民, 石杨, 帅佳琪. 基于SSR标记的牡丹杂交子代真实性鉴定[J/OL]. 分子植物育种, 2021. https://kns.cnki.net/kcms/detail/46.1068.S.20210406.1628.012.html. CHEN M J, LYU C P, YANG Z M, SHI Y, SHUAI J Q. Authenticity identification of hybrid progenies of Paeonia suffruticosa based on SSR Markers[J/OL]. Molecular Plant Breeding, 2021. https://kns.cnki.net/kcms/detail/46.1068.S.20210406.1628.012.html. |
| [8] |
李晋华, 王少铭, 罗莉斯, 冷家归, 侯颖辉, 李德文. 基于SRAP标记的生姜种质资源遗传多样性分析[J/OL]. 分子植物育种, 2021. https://kns.cnki.net/kcms/detail/46.1068.S.20211203.0004.002.html. LI J H, WANG S M, LUO L S, LENG J G, HOU Y H, LI D W. Genetic diversity of ginger germplasm resources based on SRAP Markers [J/OL]. Molecular Plant Breeding, 2021. https://kns.cnki.net/kcms/detail/46.1068.S.20211203.0004.002.html. |
| [9] |
李清, 罗永坚, 吴柔贤, 贾俊婷, 张文虎, 宋松泉, 刘军. 广东省大豆种质资源遗传多样性分析及DNA分子身份证构建[J]. 广东农业科学, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 LI Q, LUO Y J, WU R X, JIA J T, ZHANG W H, SONG S Q, LIU J. Analysis on genetic diversity and construction of DNA molecular identity card of soybean germplasm resources in Guangdong Province[J]. Guangdong Agricultural Sciences, 2020, 47(12): 221-228. DOI:10.16768/j.issn.1004-874X.2020.12.023 |
| [10] |
胡会刚, 叶自行, 胡桂兵, 董晨. 应用RAPD标记构建柑桔分子遗传图谱[J]. 广东农业科学, 2013, 40(18): 123-126. DOI:10.16768/j.issn.1004-874X.2013.18.060 HU H G, YE Z X, HU G B, DONG C.. Construction of citrus genetic linkage map based on RAPD markers[J]. Guangdong Agricultural Sciences, 2013, 40(18): 123-126. DOI:10.16768/j.issn.1004-874X.2013.18.060 |
| [11] |
刘荣, 龚德勇, 刘清国, 黄海, 范建新. 基于SRAP分子标记的13份贵州芒果种质资源遗传多样性分析[J]. 热带作物学报, 2019, 40(1): 87-91. DOI:10.3969/j.issn.1000-2561.2019.01.013 LIU R, GONG D Y, LIU Q G, HUANG H, FAN J X. Genetic diversity analysis of thirteen Mango germplasm resources based on SRAP Molecular Markers[J]. Chinese Journal of Tropical Crops, 2019, 40(1): 87-91. DOI:10.3969/j.issn.1000-2561.2019.01.013 |
| [12] |
宋芸, 张鑫瑞, 李政, 孙哲, 李澳旋, 杜晓蓉, 乔永刚. 基于SSR分子标记的柴胡遗传多样性与遗传结构分析[J/OL]. 药学学报,
2021. https://kns.cnki.net/kcms/detail/11.2163.R.20211209.0137.009.html. SONG Y, ZHANG X R, LI Z, SUN Z, LI A X, DU X R, QIAO Y G. Genetic diversity and genetic structure analysis of Bupleurum hinense DC. based on S SR molecular marker[J/OL]. Acta Pharmaceutica Sinica, 2021. https://kns.cnki.net/kcms/detail/11.2163.R.20211209.0137.009.html. |
| [13] |
李东霞, 石鹏, 杨伟波, 符海泉, 李虹, 刘立云. 利用SSR分子标记分析海南与国内外花生种质资源的遗传多样性[J]. 广东农业科学, 2015, 42(20): 118-124. DOI:10.16768/j.issn.1004-874X.2015.20.021 LI D X, SHI P, YANG W B, FU H Q, LI H, LIU L Y. Analysis of genetic diversity in peanut (Arachis hypogaea L.)germplasm resources from Hainan and other regions by using SSR markers[J]. Guangdong Agricultural Sciences, 2015, 42(20): 118-124. DOI:10.16768/j.issn.1004-874X.2015.20.021 |
| [14] |
董胜君, 王新鑫, 张皓凯, 陈建华, 刘权钢, 徐豪. 基于SSR标记的东北杏遗传多样性分析及指纹图谱构建[J]. 沈阳农业大学学报, 2021, 52(6): 668-676. DOI:10.3969/j.issn.1000-1700.2021.06.004 DONG S J, WANG X X, ZHANG H K, CHEN J H, LIU Q G, XU H. Genetic diversity analysis and DNA fingerprint construction of Armeniaca mandshurica based on SSR Markers[J]. Journal of Shenyang Agricultural University, 2021, 52(6): 668-676. DOI:10.3969/j.issn.1000-1700.2021.06.004 |
| [15] |
石秀兰, 陈平, 于得水, 韩瑞宏, 刘萍. 狼尾草属优质牧草SRAP遗传多样性分析与指纹图谱构建[J]. 广东农业科学, 2018, 45(10): 55-60. DOI:10.16768/j.issn.1004-874X.2018.10.009 SHI X L, CHEN P, YU D S, HAN R H, LIU P. Analysis of genetic diversity and construction of fingerprint of Pennisetum by SRAP[J]. Guangdong Agricultural Sciences, 2018, 45(10): 55-60. DOI:10.16768/j.issn.1004-874X.2018.10.009 |
| [16] |
孙文英, 张玉星, 李秀根, 王龙. 梨AFLP分子连锁图谱的构建与分析[J]. 华北农学报, 2009, 24(3): 179-183. SUN W Y, ZHANG Y X, LI X G, WANG L. Construction and analysisof AFLP Molecular Linkage Map in pear[J]. Acta Agriculturae Boreali-Sinica, 2009, 24(3): 179-183. |
| [17] |
艾小艳, 何华平, 龚林忠, 王富荣, 王会良, 刘勇. 桃分子遗传图谱构建及红肉性状基因定位概述[J]. 湖北农业科学, 2016, 55(4): 817-819, 854. DOI:10.14088/j.cnki.issn0439-8114.2016.04.001 AI X Y, HE H P, GONG L Z, WANG F R, WANG H L, LIU Y. Construction of Molecular Genetic Map and gene mapping of blood-fresh traits in peach ﹝ Prunus persica (L) Batsch ﹞[J]. Hubei Agricultural Sciences, 2016, 55(4): 817-819, 854. DOI:10.14088/j.cnki.issn0439-8114.2016.04.001 |
| [18] |
LINGE C D S, ANTANAVICIUTE L, ABDELGHAFAR A, ARÚS P, BASSI D, ROSSINI L, FICKLIN S, GASIC K. High-density multipopulation consensus genetic linkage map for peach[J]. PLoS ONE, 2018, 13(11): e0207724. DOI:10.1371/journal.pone.0207724 |
| [19] |
章秋平, 刘威生, 刘宁, 张玉萍, 刘硕, 魏潇, 刘有春. 杏杂合位点共显性标记的分离方式及连锁图谱构建[J]. 园艺学报, 2011, 38(10): 1983-1990. DOI:10.16420/j.issn.0513-353x.2011.10.020 ZHANG Q P, LIU W S, LIU N, ZHANG Y P, LIU S, WEI X, LIU Y C. Segregation type of heterozygous loci in F1 and construction of Molecular Markers Linkage Map in Apricot[J]. Acta Horticulturae Sinica, 2011, 38(10): 1983-1990. DOI:10.16420/j.issn.0513-353x.2011.10.020 |
| [20] |
DANG Z G, CHEN Y Y. Construction of a G enetic Linkage Map of mango based on SRAP, AFLP and ISSR Markers[J]. Agricultural Biotechnology, 2017, 6(6): 9-12, 16. DOI:10.19759/j.cnki.2164-4993.2017.06.003 |
| [21] |
LIN H, GUO Y S, YANG X X, KONDO S, ZHAO Y H, LIU Z D, LI K, GUO X W. QTL identification and candidate gene identification for monoterpene content in grape (Vitis vinifera L.) berries[J]. Vitis, 2020(59): 19-28. DOI:10.5073/vitis.2020.59.19-28 |
| [22] |
杨晓旭. 葡萄果实香气物质的QTL定位及关联分析研究[D]. 沈阳: 沈阳农业大学, 2017. YANG X X. QTL Mapping and association study for aromatic compounds in grape (Vitis vinifera L. )[D]. Shenyang: Shenyang Agricultural University, 2017. |
| [23] |
章秋平, 王力荣, 李疆, 曹珂, 陈昌文. 核果类果树遗传连锁图谱的研究进展[J]. 果树学报, 2009, 26(4): 532-538. DOI:10.13925/gsxb.2009.04.026 ZHANG Q P, WANG L R, LI J, CAO K, CHEN C W.. Research advances on genetic linkage mapping construction of stone fruit trees[J]. Journal of Fruit Science, 2009, 26(4): 532-538. DOI:10.13925/gsxb.2009.04.026 |
| [24] |
WEEDEN N F, HEMMATT M, LAWSON D M, LODHI M, BELL R L, MANGANARIS A G, REISCHS B I, BROWN S K, YE G N. Development and application of molecular marker linkage map in woody fruit crops[J]. Euphytica, 1994, 77-71-75. DOI:10.1007/BF02551464 |
| [25] |
WEI X, SHEN F, ZHANG Q P, LIU N, ZHANG Y P, XU M, LIU S, ZHANG Y J, MA X X, LIU W S. Genetic diversity analysis of Chinese plum (Prunus salicina L) based on whole-genome resequencing[J]. Tree Genetics & Genomes, 2021, 17(3): 26-36. DOI:10.1007/S11295-021-01506-X |
| [26] |
魏潇, 章秋平, 刘宁, 张玉萍, 徐铭, 刘硕, 张玉君, 马小雪, 刘威生. 不同来源中国李(Prunus salicina L) 的多样性与近缘种关系[J]. 中国农业科学, 2019, 52(3): 568-578. DOI:10.3864/j.issn.0578-1752.2019.03.017 WEI X, ZHANG Q P, LIU N, ZHANG Y P, XU M, LIU S, ZHANG Y J, MA X X, LIU W S. Genetic diversity of the Prunus salicina L. from different sources and their related species[J]. Scientia Agricultura Sinica, 2019, 52(3): 568-578. DOI:10.3864/j.issn.0578-1752.2019.03.017 |
| [27] |
孙海龙, 鲁晓峰, 邵静, 徐树广. 不同李种质果实性状分析及综合评价[J]. 中国果树, 2021(12): 53-59. DOI:10.16626/j.cnki.issn1000-8047.2021.12.010 SUN H L, LU X F, SHAO J, XU S G. Analysis and comprehensive evaluation of fruit characters of different plum germplasm[J]. China Fruits, 2021(12): 53-59. DOI:10.16626/j.cnki.issn1000-8047.2021.12.010 |
| [28] |
CARRASCO B, GONZÁLEZ M, GEBAUER M, GONZÁLEZ R G, MALDONADO J, SILVA H. Construction of a highly saturated linkage map in Japanese plum (Prunus salicina L.) using GBS for SNP marker
calling[J]. PLoS ONE, 13(12): e0208032. DOI:10.1371/journal.pone.0208032 |
| [29] |
LI G, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: Its application to mapping and gene tagging in brassica[J]. Theoretical and Applied Genetics, 2001, 103(2): 455-461. DOI:10.1007/s001220100570 |
| [30] |
万怡震, 王跃进, 张今今, 孙马, 杨克强, 徐炎. 多年生果树植物分子遗传作图[J]. 园艺学报, 2002, 29(S): 629-634. WAN Y Z, WANG Y J, ZHANG J J, SUN M, YANG K Q, XU Y. Molecular Genetic Mapping in fruit crops[J]. Acta Horticulturae Sinica, 2002, 29(S): 629-634. |
| [31] |
孙莉莉, 牟蕴慧, 甄灿福, 叶万军. 寒地李和杏遗传多样性SSR分析[J]. 北方园艺, 2017(13): 50-55. DOI:10.11937/bfyy.20170286 SUN L L, MU Y H, ZHEN C F, YE W J. Genetic diversity of SSR analysis of plum and apricot in cold region[J]. Northern Horticulture, 2017(13): 50-55. DOI:10.11937/bfyy.20170286 |
| [32] |
GULSEN O, UZUN A, CANAN I, SEDAY U, CANIHOS E. A new citrus linkage map based on SRAP, SSR, ISSR, POGP, RGA and RAPD markers[J]. Euphytica, 2010, 173(2): 265-277. DOI:10.1007/s10681-010-0146-7 |
| [33] |
VENKATESWARLU M, URS S R, NATH B S, SHASHIDHAR H E, MAHESWARAN M, VEERAIAH T M, SABITHA M G. A first genetic linkage map of mulberry (Morus spp.)using RAPD, ISSR, and SSR markers and pseudotestcross mapping strategy[J]. Tree Genetics & Genomes, 2006, 3(1): 15-24. DOI:10.1007/s11295-006-0048-y |
| [34] |
战晴晴, 隋春, 魏建和, 范圣此, 张杰. 利用ISSR和SSR分子标记构建北柴胡遗传图谱[J]. 药学学报, 2010, 45(4): 517-523. DOI:10.16438/j.0513-4870.2010.04.001 ZHAN Q Q, SUI C, WEI J H, FAN S C, ZHANG J. Construction of genetic linkage map of Bupleurum chinense DCusing ISSR and SSR markers[J]. Acta Pharmaceutica Sinica, 2010, 45(4): 517-523. DOI:10.16438/j.0513-4870.2010.04.001 |
| [35] |
王晓敏, 吕瑞娜, 李长田. 应用SRAP、ISSR和TRAP标记构建金针菇分子遗传连锁图谱[J]. 分子植物育种, 2020, 18(13): 4377-4383. DOI:10.13271/j.mpb.018.004377 WANG X M, LU R N, LI C T. Construction of Flammulina filiformis Molecular Genetic Linkage Map with SRAP, ISSR and TRAP Markers[J]. Molecular Plant Breeding, 2020, 18(13): 4377-4383. DOI:10.13271/j.mpb.018.004377 |
| [36] |
JANICK J, MOORE J N. Fruit breeding, tree and tropical fruits[M]. 2nd ed. New York: John Wiley & Sons Inc, 1995: 559-607.
|
| [37] |
JUAN A S, IGOR P, PAULINA S, PATRICIO Z, CLAUDIA S, MALLIKARJUNA A, DIANNE V, DAVID R, PEDRO M G, RODRIGO I. Genotyping by sequencing for SNP-based linkage analysis and identification of QTLs linked to fruit quality traits in Japanese Plum (Prunus salicina Lindl.)[J]. Frontiers in Plant Science, 2017, 8: 476. DOI:10.3389/fpls.2017.00476 |
| [38] |
BA SI L IO C, MÁ XIMO G, MARLENE G, ROL ANDO G G, JONATHAN M, HERMAN S. Construction of a highly saturated linkage map in Japanese plum (Prunus salicina L.)using GBS for SNP
marker calling[J]. PLoS ONE, 2018, 13(12): e0208032. DOI:10.1371/journal.pone.0208032 |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49