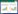文章信息
基金项目
- 国家重点研发计划项目(2019YFD10005002-0303);农业农村部农垦局物种资源保护项目(18220019)
作者简介
- 官鑫(1997—),女,在读硕士生,研究方向为橡胶树抗病资源鉴定发掘,E-mail:2922057051@qq.com.
通讯作者
- 涂敏(1980—),女,博士,副研究员,研究方向为橡胶树抗病育种,E-mail:tm_tumin@163.com.
文章历史
- 收稿日期:2022-03-08
2. 云南农业大学植物保护学院,云南 昆明 650201
2. Plant Protection College, Yunnan Agricultural University, Kunming 650201, China
【研究意义】白粉病是橡胶树生产上最重要的叶部病害,其病原菌鉴定为Erysiphe quercicola[1-2],为专性寄生菌。该病自1918年在印尼瓜哇被发现,目前已遍及亚非各国植胶园,主要为害橡胶树嫩叶(古铜期及变色期叶片)、嫩芽、嫩梢和花序,条件适合时其分生孢子可全年重复侵染,降低橡胶树光合作用,导致橡胶树生长迟缓、胶乳产量降低,最高可造成橡胶树减产45%[3-4]。因此,通过对橡胶树叶片上白粉病菌活菌量的快速检测,能及时开展橡胶树白粉病预测预报及防控,将其对生产的损失降至最低。【前人研究进展】qPCR是利用荧光信号积累实时监测PCR进程中每个反应循环扩增产物量的变化,最后通过标准曲线对未知模板进行定量分析的方法[5]。qPCR技术能够定量检测病菌[6-7],但不能区分病菌的死活,易出现假阳性结果。研究表明,细胞死亡后DNA仍然能够保留数天时间。PMA是一种光敏感、与DNA高亲和力结合的染料,其光解后能穿过死菌的细胞膜与死菌DNA结合形成稳定的共价键,从而抑制死菌DNA扩增。活菌因其具有完整的细胞膜,可将PMA排除在细胞膜外[8]。因此,利用PMA对死活细胞的选择性,结合qPCR技术,可以实现准确定量检测活菌的目的。目前该技术已广泛应用于食源性致病菌[9-10]、医学病原微生物[11]、水环境病原微生物[12]等,对植物病原菌检测也有越来越多的报道,已在玉米细菌性枯萎病菌(Pantoea stewartii subsp. stewartii)[13]、西瓜细菌性果斑病菌(Acidovorax citruui)[14]、青枯菌(Ralstonia solanacearum)[15-17]、猕猴桃溃疡病菌(Pseudomonas syringae pv. actinidiae)[18]、黄瓜白粉病菌(Podosphaera xanthii)[19]、梨火疫病菌(Erwinia amylovora)[20]、十字花科黑斑病菌(Pseudomonas syringae pv. maculicola)[21]等建立了活菌PMA-qPCR定量的检测方法。【本研究切入点】鉴于目前PMA-qPCR技术在橡胶树白粉病活菌检测方面尚无研究报道,根据橡胶树白粉菌的ITS保守区序列设计特异性引物,构建PMA-qPCR分子检测体系。【拟解决的关键问题】本研究拟建立PMA-qPCR对橡胶树白粉病活菌的快速检测体系,为该病害预测预报及防控提供理论依据和技术支持。
1 材料与方法 1.1 试验材料橡胶白粉菌(Erysiphe quercicola)、橡胶灵芝菌(Ganoderma pseudoferreum)和禾谷镰孢菌(Fusarium graminearum)已纯化3代以上,由中国热带农业科学院橡胶研究所种质资源课题组分离保存;大肠杆菌(Escherichia coli)菌株由该所阳江华老师赠予。白粉菌接种材料为橡胶树无性系热研73397古铜期组培苗。试验于2021年6月至2022年2月在中国热带农业科学院橡胶研究所进行。
1.2 活/ 死分生孢子悬浮液制备从接种显症的热研73397橡胶树白粉病病叶上刮取约1 g白色粉状物,置于装有30 mL ddH2O的50 mL离心管中,涡旋振荡,摇匀,用血球计数板计数,制备浓度为1×105个/mL的新鲜活孢子悬浮液。取15 mL孢子悬浮液于新的离心管,100 ℃水浴30 min,获得灭活孢子悬浮液。
1.3 引物设计及特异性检测用Primer Premier 5.0软件根据本课题组鉴定的橡胶白粉菌的rDNA-ITS保守区设计引物,其序列为Eq-F:5’ TGCCTGTTCGAGCGTCAT 3’;Eq-R:5’ GCAAGTGGGTTGTTCTGG 3’,扩增产物片段大小为177 bp。引物由铂尚生物技术(上海)有限公司合成。以橡胶白粉菌、橡胶灵芝菌和禾谷镰刀菌及大肠杆菌所提取的总DNA作为模板,以双蒸水作为空白对照,验证引物对Eq-F/Eq-R对橡胶白粉菌qPCR扩增的特异性。qPCR反应体系与程序参考谢学文等[22]的方法。
1.4 重组质粒制备以提取的橡胶白粉病菌DNA为模板进行PCR扩增,PCR反应体系及反应程序参考谢学文等[22]的方法,其中反应程序中退火温度为60 ℃。对PCR产物进行1% 琼脂糖凝胶电泳回收目的条带,PCR产物经纯化后与pMD19-T于16 ℃连接,转至DH5α感受态细胞中,37 ℃过夜培养,挑取白色单克隆,转于含有Amp的LB液体培养基中,37 ℃下200 r/min振荡培养12 h。通过PCR验证和测序〔铂尚生物技术(上海)有限公司〕检测插入目的片段的正确性。将测序正确的菌液提取重组质粒,超微量分光光度计测定质粒DNA浓度和纯度,根据摩尔定律,计算单位体积质粒所含的DNA拷贝数浓度,质粒拷贝数=〔质粒浓度(ng/μL)×6.02×1023]/〔(载体长度bp+ 片段长度bp)×660 g/mol〕。
1.5 qPCR标准曲线建立及重复性检验将定量后的高拷贝数重组质粒按10倍梯度稀释为3.0×109、3.0×108、3.0×107、3.0×106、3.0×105、3.0×104、3.0×103、3.0×102、3.0×101 copies/μL,在实时荧光定量仪上进行qPCR扩增,反应体系参照1.3,重复3次。反应结束后获得线性和可靠的标准曲线(相关系数R2>0.99)。选择3.0×102、3.0×104、3.0×106 copies/μL的质粒进行组间和组内重复实验5次,以确定qPCR过程中标准曲线的准确性。
1.6 PMA-qPCR对活/ 死分生孢子悬浮液检测将1.2中的活/ 死分生孢子悬浮液按表 1的成分加入1.5 mL灭菌的离心管中混合,用铝箔纸将各样品离心管管盖以下部分包起来以避光,分别加入20 µL PMA工作液,使其终浓度为20 µg/mL,置于避光摇床中振荡孵育5 min(150 r/min)。之后置于冰上,在距离样品15 cm处利用100 W蓝光灯照射15 min,期间不断转动离心管使其均匀照射。光照结束后于离心机10 000 r/min离心10 min,弃上清液,采用真菌DNA提取试剂盒提取DNA。以未加PMA处理的样品为对照,参照1.3反应体系对样品进行检测。
1.7 PMA-qPCR灵敏度检测
将1.2定量后的活孢子悬浮液按10倍梯度稀释为1.0×105、1.0×104、1.0×103、1.0×102、1.0×101、1.0×100个/mL,按1.6中PMA处理后,提取DNA。使用特异引物Eq-F/Eq-R进行qPCR扩增,反应体系参照1.3,获取检测样品Ct值,对PMA-qPCR灵敏度进行分析。
1.8 PMA-qPCR检测丙环唑处理后活菌变化选取10株古铜期橡胶树热研73397芽接苗接种白粉病菌,待植株发病后,使用40 mg/L丙环唑喷施病叶,采集喷施后0、15、30、60、90、120 min 6个时期的叶片,每个时期3次重复。随后将样品用液氮研磨成粉末,分别迅速称取2管100 mg粉末于1.5 mL灭菌离心管中,一管直接提取基因组DNA,另一管内加入1 mL灭菌的PBS,振荡15 s。用铝箔纸将各样品离心管管盖以下部分包裹避光,按1.6中PMA处理后提取DNA。以未加PMA处理的样品为对照,参照1.3反应体系对样品进行检测。
试验数据使用Excel 2010记录,利用IBM SPSS Statistics进行单因素ANOVA检验分析差异显著性,采用OriginPro 2021软件作图。
2 结果与分析 2.1 引物特异性验证利用设计的引物Eq-F/Eq-R对橡胶白粉菌、橡胶灵芝菌、禾谷镰刀菌及大肠杆菌的DNA进行qPCR扩增,结果表明橡胶白粉菌扩增的循环数(Ct值)为20.57,而其他菌株及空白对照均无扩增曲线(图 1A);反应产物熔解曲线显示,当Tm值在87.50 ℃时呈单一熔解峰,无引物二聚体和非特异性扩增(图 1B),表明本试验设计的引物能区分橡胶白粉菌和其他病原菌,特异性良好,能够用于后续PMA-qPCR方法的建立。

|
| 1:橡胶白粉菌;2:橡胶灵芝菌;3:禾谷镰刀菌;4:大肠杆菌;N:双蒸水 1:E. quercicola; 2: G. pseudoferreum; 3: F. graminearum; 4: E. coli; N: Double distilled water 图 1 引物Eq-F/Eq-R对橡胶白粉菌的qPCR特异性扩增曲线(A)和熔解曲线(B) Fig. 1 qPCR specific amplification curve (A) and melting curve (B) of primer Eq-F/Eq-R against powdery mildew of rubber tree |
2.2 质粒标准品和标准曲线的制备
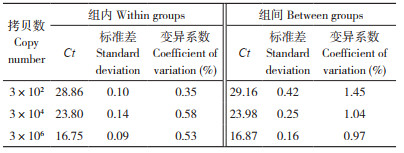
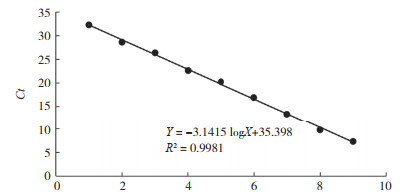
从白粉病菌基因组DNA扩增的PCR产物片段大小为177 bp(图 2)。测序结果表明重组质粒pMD-19T-Eq构建成功。提取的重组质粒浓度为94.6 ng/μL,根据1.4中公式计算的拷贝数约为3.0×1010 copies/μL。通过梯度PCR确定最佳退火温度为60 ℃。以标准质粒10倍梯度稀释拷贝浓度为3.0×101~3.0×108 copies/μL,根据qPCR检测扩增结果绘制标准曲线(图 3),扩增反应循环数Ct值与质粒DNA拷贝数的对数值之间有良好的线性关系:Y=-3.1415×logX+35.398,相关系数R2=0.9981,扩增效率Eff=107.97%。式中,Y为Ct值,X为基因拷贝浓度。
|
| 图 2 橡胶白粉菌ITS保守区PCR扩增 Fig. 2 PCR amplification of ITS conserved region of Erysiphe quercicola |
|
| 质粒标准品浓度对数值 Plasmid concentration logarithmic value 图 3 橡胶白粉菌标准曲线 Fig. 3 Standard curve of Erysiphe quercicola |
2.3 qPCR重复性检测
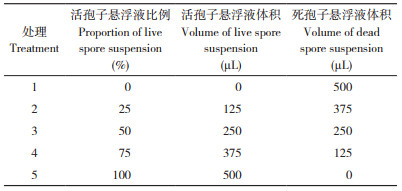
选择3×102、3×104、3×106 copies/μL 3个梯度的质粒进行组间和组内的重复试验,以确定qPCR过程中标准曲线的准确性。结果(表 2)显示,组内变异系数为0.35%~0.58%,组间变异系数为0.97%~1.45%,表明qPCR的重复性良好,可用于后续试验。
2.4 PMA-qPCR对活/ 死分生孢子悬浮液的检测
以qPCR为对照,对不同比例的活/ 死分生孢子悬浮液进行PMA-qPCR检测,验证该体系对混合样品扩增的准确性。结果(图 4)显示,当悬浮液全部为死孢子时,qPCR的Ct值为24.50,而PMA-qPCR的Ct值为31.91,差异显著。当悬浮液活孢子比例为25%、50%、75% 时,随着活孢子悬浮液比例的增加,PMA-qPCR的Ct值逐渐减小,分别为29.00、26.92和26.00;而qPCR的Ct值无明显变化,分别为24.65、24.16和23.36,且qPCR样品检测的Ct值均小于PMA-qPCR。当样品悬浮液中活孢子比例达到100% 时,PMA-qPCR的Ct值与qPCR的Ct值无显著差异,说明加入PMA对活孢子的扩增几乎无影响。同时,悬浮液中随着活孢子比例的增大,PMA-qPCR的Ct值相应减少,说明死孢子DNA被PMA抑制不能扩增,从而降低了qPCR检测中由于悬浮液死孢子扩增造成的假阳性。

|
| *表示差异显著 P* Represent significant difference 图 4 不同比例活/ 死孢子悬浮液qPCR与PMA-qPCR结果 Fig. 4 PMA-qPCR results of different proportions of live/dead spore suspensions |
2.5 PMA-qPCR灵敏度检测
将活孢子悬浮液1×105个/mL按10倍浓度梯度进行稀释,经PMA处理后提取DNA进行PMA-qPCR反应。随着稀释倍数增加,反应体系中活孢子数量减少,Ct值上升。根据标准曲线得到定量结果见表 3,PMA-qPCR最低可检测拷贝数为4.77×102 copies/μL。

|
2.6 PMA-qPCR检测丙环唑处理后活菌比例
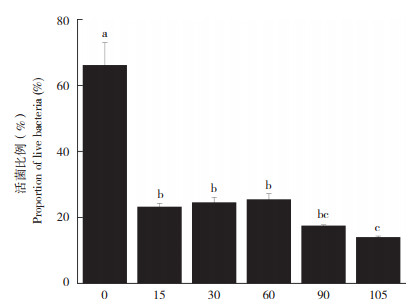
橡胶叶片喷施丙环唑后,白粉菌活菌比例随着处理时间的延长整体呈减少趋势(图 5)。其中,处理后15 min,活菌比例从66.13% 下降至23.18%(P < 0.05);处理后90 min,活菌比例下降至17.43%(P < 0.05);处理后105 min活菌比例为13.97%(P < 0.05)。表明PMA-qPCR能准确定量检测出经丙环唑处理后橡胶树白粉病病叶中的活菌数。
|
| 柱上小写英文字母不同者表示差异显著 Different lowercase letters above the column indicate significant differences 图 5 丙环唑处理后橡胶树白粉病病叶活菌比例 Fig. 5 Proportion of live bacteria in powdery mildew of rubber tree leaves treated with propiconazole |
3 讨论
自Nogva等[23]发现叠氮溴化乙锭(Ethidium monoazide,EMA)能与PCR技术结合区分活细菌与死细菌以来,该方法已在细菌[24]、病毒[25]等病原微生物中得到应用。但EMA会部分渗透入活细胞中,造成活菌中基因组DNA的损失[26-27]。研究发现PMA能弥补EMA的这一不足,提高检测的准确性。目前PMA-qPCR技术在定量检测植物病原细菌中应用较为广泛[28-29],而在植物病原真菌中的应用较少[30]。本研究首次报道了橡胶树白粉病PMA-qPCR检测体系的建立及其应用。
本研究利用PMA-qPCR方法对不同活/ 死孢子悬浮液比例的检测发现,当样品全部为死孢子悬浮液时,也会有少量扩增,Tian等[31]、Elizaquível等[32]在对西瓜细菌性果斑病菌和食源性细菌检测中也发现了同样的现象。研究认为,若样品中活菌和死菌比例过低时,PMA-qPCR检测会比实际样品中的活菌数高。前人研究发现扩增片段长度越大,对死菌DNA扩增的抑制效果越好[33],但靶基因片段过长会减弱扩增的荧光信号,通常qPCR扩增产物的大小在50~200 bp时有较高的扩增效率[34]。本研究设计的特异性引物扩增长度为177 bp,对目标菌特异性检测良好,建立的标准曲线具有较好的线性关系,但当样品均为死孢子悬浮液时仍有一定扩增量,可能与本试验中死孢子悬浮液是通过加热致死获得、导致其细胞膜未能完全损伤有关,造成了PMA与死孢子悬浮液DNA没有充分结合,从而影响最终的扩增效果,该推测还需进一步验证。
橡胶树白粉病分子检测方法较少,目前主要有环介导等温扩增(Loop-mediated isothermal amplification,LAMP)[35]和巢式PCR技术[36]两种。本研究建立的PMA-qPCR体系对活孢子悬浮液检测灵敏度为4.77×102 copies/μL,对橡胶树白粉病早期诊断具有重要作用。目前研究橡胶树白粉病的生物和化学防治效果主要采用孢子萌发试验和盆栽试验。如黄有航等[37]采用上述两种方法得出枯草芽胞杆菌S43对橡胶树白粉病的防效为86.12%;罗婵娟等[38]利用盆栽实验测定苯并噻二唑(BTH)对橡胶树白粉病的防效为57.8%。但上述两种方法均无法检测处理后橡胶树白粉病活菌数。而本研究建立的PMA-qPCR体系能快速定量检测出橡胶树白粉病活菌数。在丙环唑处理15 min后活菌比例由66.13% 下降至23.18%,较处理前有显著下降,表明本体系可以应用到橡胶树白粉病防治效果检测中。
4 结论本研究建立的橡胶树白粉病PMA-qPCR体系,具有重复性好和灵敏度高的特点,能快速、准确地检测样品中分生孢子悬浮液活菌量,弥补了常规分子检测方法不能区分病原菌死活状态的不足。该体系还能在丙环唑处理橡胶树白粉病病叶15 min后,快速定量检测到橡胶树白粉病病叶中的活菌比例较未处理前显著下降,可及时有效地检测药剂对橡胶树白粉病的防效。该PMA-qPCR体系的建立为橡胶树白粉病的早期预测预报和防效检测提供了精确的技术手段。
| [1] |
WU H, PAN Y W, RONG D, HE Q G, MAMY J N R, LIU W B, ZHENG F C, MIAO W G. Molecular identification of the powdery mildew fungus infecting rubber trees in China[J]. Forest Pathology, 2019, 49(5): 1-8. DOI:10.1111/efp.12519 |
| [2] |
TAM L T T, DUNG P N, LIEM N V. First report of powdery mildew caused by Erysiphe quercicola on mandarin in Vietnam[J]. Plant Disease, 2016, 100(6): 1777. DOI:10.1094/PDIS-11-15-1381-PDN |
| [3] |
LIYANAGE K K, KHAN S, BROOKS S, MORTIMER P E, KARUNARATHNA S C, XU J C, HYDE K D. Taxonomic revision and phylogenetic analyses of rubber powdery mildew fungi[J]. Microbial Pathogenesis, 2017, 105: 185-195. DOI:10.1016/j.micpath.2017.01.054 |
| [4] |
LIYANAGE K K, KHAN S, MORTIMER P E, HYDE K D, BROOKS S, MING Z. Powdery mildew disease of rubber tree[J]. Forest Pathology, 2016, 46(2): 90-103. DOI:10.1111/efp.12271 |
| [5] |
黄海泉. 实时荧光定量PCR技术在植物检疫中应用的研究进展[J]. 湖北农业科学, 2012, 51(1): 5-8. DOI:10.14088/j.cnki.issn0439-8114.2012.01.016 HUANG H Q. Research progress of application of real time fluorescent quantitative PCR technology in plant quarantine[J]. Hubei Agricultural Sciences, 2012, 51(1): 5-8. DOI:10.14088/j.cnki.issn0439-8114.2012.01.016 |
| [6] |
张纪利, 齐是, 栾新博, 金亚波, 黄崇峻, 黎平, 韦建玉, 颜健. 植烟土壤中枯萎病菌和黑胫病菌的快速检测方法[J]. 广东农业科学, 2022, 49(2): 101-108. DOI:10.16768/j.issn.1004-874X.2022.02.012 ZHANG J L, QI S, LUAN X B, JIN Y B, HUANG C J, LI P, WEI J Y, YAN J. Method for rapid detection of Fusarium oxysporum and Phytophthora parasitica var. nicotianae in tabacco planting soil[J]. Guangdong Agricultural Sciences, 2022, 49(2): 101-108. DOI:10.16768/j.issn.1004-874X.2022.02.012 |
| [7] |
陈霞, 崔一平, 彭埃天, 宋晓兵, 程保平, 凌金锋. 仁化县贡柑主要病害的调查与检测[J]. 广东农业科学, 2021, 48(2): 92-99. DOI:10.16768/j.issn.1004-874X.2021.02.012 CHEN X, CUI Y P, PENG A T, SONG X B, CHENG B P, LING J F. Investigation and detection of main diseases on gonggan in Renhua county[J]. Guangdong Agricultural Sciences, 2021, 48(2): 92-99. DOI:10.16768/j.issn.1004-874X.2021.02.012 |
| [8] |
ANDREAS N, CHING Y C, ANNE K C. Comparison of propidium monoazide with ethidium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells[J]. Journal of Microbiological Methods, 2006, 67(2): 310-320. DOI:10.1016/j.mimet.2006.04.015 |
| [9] |
LI D, TONG T Z, ZENG S Y, LIN Y W, WU S X, HE M. Quantification of viable bacteria in wastewater treatment plants by using propidium monoazide combined with quantitative PCR (PMA-qPCR)[J]. Journal of Environmental Sciences, 2014, 26(2): 299-306. DOI:10.1016/S1001-0742(13)60425-8 |
| [10] |
GAO S J, SUN C Z, HONG H L, GOONERATNE R, MUTUKUMIRA A, WU X Y. Rapid detection of viable Cronobacter sakazakii in powdered infant formula using improved propidium monoazide (PMAxx) and quantitative recombinase polymerase amplification (qRPA) assay[J]. Food Control, 2021, 124: 1-8. DOI:10.1016/j.foodcont.2021.107899 |
| [11] |
任洪林, 张士军, 胡盼, 卢士英, 柳增善, 王海波, 李岩松, 柳溪林, 张英. 一种基于PMA-qPCR技术的布鲁氏菌活菌计数方法: CN110343744A[P]. 2019-10-18. REN H L, ZHANG S J, HU P, LU S Y, LIU Z S, WANG H B. LI Y S, LIU X L, ZHANG Y. A live Brucella count method based on PMA-qPCR technology: CN110343744A[P]. 2019-10-18. |
| [12] |
KIBBEE R J, RMECI B. Development of a sensitive and false-positive free PMA-qPCR viability assay to quantify VBNC Escherichia coli and evaluate disinfection performance in wastewater effluent[J]. Journal of Microbiological Methods, 2017, 132: 139-147. DOI:10.1016/j.mimet.2016.12.004 |
| [13] |
冯建军, 刘杰, 王飞, 李秋枫, 刘新娇, 李一农, 章桂明. PMA结合实时荧光PCR进行玉米细菌性枯萎病菌细胞活性检测初步研究[J]. 植物检疫, 2014, 28(2): 27-32. FENG J J, LIU J, WANG F, LI Q F, LIU X J, LI Y N, ZHANG G M. Preliminary study on differentiation of live and dead cell of Pantoea stewartii subsp. stewartii using propidium monoazide combined with real-time fluorescent PCR[J]. Plant Quarantine, 2014, 28(2): 27-32. |
| [14] |
赵璟源, 胡佳续, 张莹, 刘鹏, 唐芳, 廖芳. 西瓜细菌性果斑病病菌活细胞PMA-PCR检测方法的建立[J]. 中国植保导刊, 2015, 35(9): 12-15. ZHA0 J Y, HU J X, ZHANG Y, LIU P, TANG F, LIAO F. Establishment of detection method for viable cells of Acidovorax avenae subsp. citrulli with PMA-PCR[J]. China Plant Protection, 2015, 35(9): 12-15. |
| [15] |
曹梦琪, 王旭东, 王俊, 盛晟, 吴福安. 基于PMA-qPCR检测青枯菌5号生理小种活菌的方法[J]. 蚕业科学, 2015, 41(6): 1004-1010. DOI:10.13441/j.cnki.cykx.2015.06.005 CAO M Q, WANG X D, WANG J, SHENG S, WU F A. Detection of living cells of Ralstonia solanacearum race 5 by PMA-qPCR Method[J]. Acta Sericologica Sinica, 2015, 41(6): 1004-1010. DOI:10.13441/j.cnki.cykx.2015.06.005 |
| [16] |
高达芳, 刘霞, 杨艳丽. 马铃薯青枯病病菌PMA-PCR活菌检测体系优化与初步应用[J]. 中国植保导刊, 2017, 37(10): 5-13. GAO D F, LIU X, YANG Y L. Optimization and preliminary application of PMA-PCR system for detection of viable Ralstonia solanacearum in potato[J]. China Plant Protection, 2017, 37(10): 5-13. |
| [17] |
王帅, 徐进, 许景升, 张昊, 冯洁. PMA-qPCR定量检测青枯菌活菌方法的建立[J]. 植物保护, 2018, 44(6): 122-128. DOI:10.16688/j.zwbh.2017483 WANG S, XU J, XU J S, ZHANG H, FENG J. Development of a PMA-qPCR method for quantitative detection of Ralstonia solanacearum[J]. Plant Protection, 2018, 44(6): 122-128. DOI:10.16688/j.zwbh.2017483 |
| [18] |
肖妍, 刘芸宏, 高贵田, 曹凡, 马燕萍, 赵武奇, 雷玉山. PMA-qPCR方法检测陕西猕猴桃溃疡菌优势病原菌活菌的研究[J]. 食品与机械, 2019, 35(4): 48-53. DOI:10.13652/j.issn.1003-5788.2019.04.009 XIAO Y, LIU Y H, GAO G T, CAO F, MA Y P, ZHAO W Q, LEI Y S. PMA-qPCR for detecting live bacteria of Shaanxi kiwifruit cancer dominant pathogen[J]. Food & Machinery, 2019, 35(4): 48-53. DOI:10.13652/j.issn.1003-5788.2019.04.009 |
| [19] |
李金婷, 张佐然, 梁雅静, 孔维文, 李云龙, 何晓青. 黄瓜白粉病的PMA-qPCR方法建立及应用[J]. 福建农林大学学报(自然科学版), 2019, 45(5): 550-555. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2019.05.002 LI J T, ZHANG Z R, LIANG Y J, KONG W W, LI Y L, HE X Q. Establishment and application of a PMA-qPCR method for cucumber powdery mildew[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2019, 45(5): 550-555. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2019.05.002 |
| [20] |
袁英哲, 韩剑, 王岩, 罗明, 包慧芳, 张春竹, 黄伟. 梨火疫病菌活菌快速定量检测方法的建立[J]. 果树学报, 2020, 37(9): 1425-1433. DOI:10.13925/j.cnki.gsxb.20200056 YUAN Y Z, HAN J, WANG Y, LUO M, BAO H F, ZHANG C Z, HUANG W. Establishment of rapid quantitative detection of viable Erwinia amylovora[J]. Journal of Fruit Science, 2020, 37(9): 1425-1433. DOI:10.13925/j.cnki.gsxb.20200056 |
| [21] |
于璇, 王卫芳, 李献锋, 冯黎霞, 乾义柯, 张承军, 魏霜. PMA-qPCR检测十字花科黑斑病菌活菌方法的建立[J]. 植物检疫, 2021, 35(4): 49-54. DOI:10.19662/j.cnki.issn1005-2755.2021.04.009 YU X, WANG W F, LI X F, FENG L X, QIAN Y K, ZHANG C J, WEI S. PMA-qPCR assay for the detection of viable Pseudomonas syringae pv. maculicola[J]. Plant Quarantine, 2021, 35(4): 49-54. DOI:10.19662/j.cnki.issn1005-2755.2021.04.009 |
| [22] |
谢学文, 陈利达, 曹金强, 韩道杰, 石延霞, 柴阿丽, 李磊, 李宝聚. 茄匍柄霉菌实时荧光定量PCR检测方法的建立及应用[J]. 植物病理学报, 2021, 51(4): 618-625. DOI:10.13926/j.cnki.apps.000717 XIE X W, CHEN L D, CAO J Q, HAN D J, SHI Y X, CAI A L, LI L, LI B J. DNA detection and quantification of Stemphylium solani in heat-treated infected leaf tissues in vary moisture conditions using real-time qPCR[J]. Acta Phytopathologica Sinica, 2021, 51(4): 618-625. DOI:10.13926/j.cnki.apps.000717 |
| [23] |
NOGVA H K, DROMTORP S M, NISSEN H, RUDI K. Ethidium monoazide for DNA-based differentiation of viable and dead bacteria by 5'-nuclease PCR[J]. BioTechniques, 2003, 34(4): 804-812. DOI:10.2144/03344rr02 |
| [24] |
FUKUZAWA S, SHIHO H Y, FUJITA T. Selective detection of DNA from viable Mycobacterium tuberculosis complex strains using EMA-PCR method[J]. Japanese Journal of Infectious Diseases, 2018, 72(1): 19-22. DOI:10.7883/yoken.JJID.2018.111 |
| [25] |
YU Q Y, LI S S, JIAN J, SHI L, LIU Y, QIN Y, YU Q. Study on application of EMA-PCR rapid identification of infectious and non-infectious adenovirus[J]. Journal of Sichuan University, 2017, 48(1): 132-135. DOI:10.13464/j.scuxbyxb.2017.01.028 |
| [26] |
LEE J L, LEVIN R E. A comparative study of the ability of EMA and PMA to distinguish viable from heat killed mixed bacterial flora from fish fillets[J]. Journal of Microbiological Methods, 2009, 76(1): 93-96. DOI:10.1016/j.mimet.2008.08.008 |
| [27] |
赵红波, 熊丽娜, 莫自耀. 荧光定量PCR结合EMA/PMA染色法快速检测军团菌活菌[J]. 中国卫生检验杂志, 2013, 23(7): 1661-1664. ZHAO H B, XIONG L N, MO Z Y. Quick detection of viable Legionella by real-time PCR combined with EMA/PMA[J]. Chinese Journal of Health Laboratory Technology, 2013, 23(7): 1661-1664. |
| [28] |
WANG H, TURECHEK W W. Detection of viable Xanthomonas fragariae Cells in strawberry using Propidium Monoazide and Long-Amplicon Quantitative PCR[J]. Plant Disease, 2019, 104(4): 1105-1112. DOI:10.1094/PDIS-10-19-2248-RE |
| [29] |
SOTO M L, TEIXIDO N, USALL J, VINAS I, CRESPO S A, TORRES R. Development of PMA real-time PCR method to quantify viable cells of Pantoea agglomerans CPA-2, an antagonist to control the major postharvest diseases on oranges[J]. International Journal of Food Microbiology, 2014, 180: 49-55. DOI:10.1016/j.ijfoodmicro.2014.04.011 |
| [30] |
VILANOVA L, USALL J, TEIXIDO N, TORRES R. Assessment of viable conidia of Monilinia fructicola in flower and stone fruit combining propidium monoazide (PMA) and qPCR[J]. Plant Pathology, 2017, 66(8): 1276-1287. DOI:10.1111/ppa.12676 |
| [31] |
TIAN Q, FENG J J, HU J, ZHAO W J. Selective detection of viable seed-borne Acidovorax citrulli by real-time PCR with propidium monoazide[J]. Scientific Reports, 2016, 6(1): 35457. DOI:10.1038/srep35457 |
| [32] |
ELIZAQUIVEL P, SANCHEZ G, AZNAR R. Quantitative detection of viable foodborne E. coli O157:H7, Listeria monocytogenes and Salmonella in fresh-cut vegetables combining propidium monoazide and real-time PCR[J]. Food Control, 2012, 25(2): 704-708. DOI:10.1016/j.foodcont.2011.12.003 |
| [33] |
LI B, CHEN J Q. Development of a sensitive and specific qPCR assay in conjunction with propidium monoazide for enhanced detection of live Salmonella spp. in food[J]. BMC Microbiology, 2013, 13: 259-273. DOI:10.1186/1471-2180-13-273 |
| [34] |
MAKKAR H P S, MCSWEENEY C S. Methods in gut microbial ecology for ruminants[M]. Dordrecht: Springer Netherlands, 2005. DOI:10.1007/1-4020-3791-0
|
| [35] |
史梦玉. 基于LAMP检测技术的橡胶树白粉病早期测报技术初探[D]. 海口: 海南大学, 2018. SHI M Y. Technical study on early prediction and forecast of powdery mildew of rubber tree based on LAMP detection[D]. Haikou: Hainan University, 2018. |
| [36] |
毛宇宁, 梁鹏, 刘文波, 林春花, 缪卫国, 郑服丛. 橡胶树白粉病菌分子检测技术的建立[J]. 植物保护, 2016, 42(4): 119-123. DOI:10.3969/j.issn.0529-1542.2016.04.019 MAO Y N, LIANG P, LIU W P, LIN C H, MIAO W G, ZHENG F C. PCR and nested-PCR for the molecular detection of Oidium heveae Steinm[J]. Plant Protection, 2016, 42(4): 119-123. DOI:10.3969/j.issn.0529-1542.2016.04.019 |
| [37] |
黄有航, 谭志琼, 王寅寒, 张荣意. 一株橡胶树白粉病生防菌的鉴定及在橡胶树叶片定殖规律研究[J]. 植物保护, 2013, 39(2): 77-81. DOI:10.3969/j.issn.0529-1542.2013.02.015 HUANG Y H, TAN Z Q, WANG Y H, ZHANG R Y. Identification of a bacterial isolate for biocontrol of the rubber tree powdery mildew and its colonization in rubber tree leaves[J]. Plant Protection, 2013, 39(2): 77-81. DOI:10.3969/j.issn.0529-1542.2013.02.015 |
| [38] |
罗婵娟, 范志伟, 沈奕德, 程汉亭, 刘丽珍. BTH诱导橡胶树对白粉病的抗性效果和相关酶活性测定[J]. 热带作物学报, 2011, 32(3): 475-479. DOI:10.3969/j.issn.1000-2561.2011.03.022 LUO C J, FAN Z W, SHEN Y D, CHENG H T, LIU L Z. Effects of rubber tree resistance induced by BTH to Oidium heveae and assay of resistance-related enzymes[J]. Chinese Journal of Tropical Crops, 2011, 32(3): 475-479. DOI:10.3969/j.issn.1000-2561.2011.03.022 |
(责任编辑 杨贤智)
 2022, Vol. 49
2022, Vol. 49