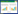文章信息
基金项目
- 广东省基础与应用基础研究基金- 粤穗联合基金(2020A1515111138,2021A1515111214);广东省农业科学院中青年学科带头人项目(R2020PY-JG003);广东省农业科学院优秀博士项目(R2021YJ-YB2004)
作者简介
- 钱玉磊(1997—), 男, 在读硕士生, 研究方向为蔬菜分子生物学, E-mail: qylei2022@163.com.
通讯作者
- 吴智明(1981—), 男, 博士, 教授, 研究方向为蔬菜分子生物学, E-mail: wuzm2012@zhku.edu.cn; 江彪(1983—), 男, 博士, 研究员, 研究方向为蔬菜遗传育种与分子生物学, E-mail: jiangbiao@gdaas.cn.
文章历史
- 收稿日期:2022-05-15
2. 广东省农业科学院蔬菜研究所/广东省蔬菜新技术研究重点实验室,广东 广州 510640
2. Vegetable Research Institute, Guangdong Academy of Agricultural Sciences/Guangdong Key Laboratory for New Technology Research on Vegetables, Guangzhou 510640, China
【研究意义】冬瓜(Benincasa hispida Cogn.)是葫芦科冬瓜属一年生蔓生草本植物,富含多种维生素和膳食纤维,是典型的高钾低钠蔬菜,兼具药用和保健功效[1-2]。冬瓜耐储运、货架期长,在调节蔬菜淡季、保证周年供应中发挥着重要作用[3-4]。抗氧化(Oxidation Resistance, OXR)基因广泛存在于真核细胞中,是一类能有效降低细胞中多余活性氧(Reactive Oxygen Species, ROS)的基因[5]。研究表明,OXR基因在植物抵抗逆境胁迫过程中发挥着重要作用[6-7],因此开展冬瓜BhiOXR基因的克隆及其在非生物胁迫下的表达特征分析,对该基因的功能研究具有重要意义。【前人研究进展】OXR基因最早在动物中发现,其C端含有TLDc〔TBC(Tre2/Bub2/Cdc16)、LysM(Lysine motif)、Domain catalytic〕保守结构域,具有重要的抗氧化作用[5]。自2019年在模式植物拟南芥中鉴定出6个OXR基因家族成员以来[6],相继在向日葵[7]、十字花科植物[8]和小麦[9]等多种植物的全基因组范围内鉴定出OXR家族成员。拟南芥AtOXR2是一种线粒体蛋白,能够减轻酵母oxr1突变体氧化应激的敏感性,也可通过影响体内的活性氧水平提高过表达AtOXR2拟南芥的光合作用效率和对环境胁迫的耐受性,促进植株生长,从而提高植株整体的生物量和种子产量[6]。此外,研究发现,过表达AtOXR2也可通过影响转基因拟南芥体内水杨酸代谢来增强植株的抗病性[10];拟南芥和玉米中异源过表达向日葵HaOXR2,可增强植株非生物氧化应激胁迫的耐受性[7]。李寒等[9]研究发现,小麦TaOXR1.6A和TaOXR2.5D在一定程度上响应氧化胁迫,可通过调控水杨酸途径来增强植株的免疫力。【本研究切入点】2019年11月,本课题组发表了第一张冬瓜高质量基因组图谱[11],这为基于全基因组鉴定、克隆和分析冬瓜BhiOXR基因奠定了基础,已有研究表明OXR基因在响应逆境胁迫过程中发挥重要作用,然而截至目前仍未有冬瓜BhiOXR基因在非生物胁迫响应方面的相关研究报道。【拟解决的关键问题】本研究以冬瓜高代自交系B227(参考基因组测序所用材料)的幼苗为试验材料,克隆冬瓜6个BhiOXR基因,并分析其响应4种非生物胁迫处理(低温、高温、盐和干旱)的表达特征,以期为深入研究冬瓜BhiOXR基因的功能奠定基础。
1 材料与方法 1.1 试验材料及处理本研究所用材料为冬瓜高代自交系B227,由广东省农业科学院蔬菜研究所提供,于2020年7月播种在培养钵中,置于广东省农业科学院蔬菜研究所温室进行培养。待幼苗长至两叶一心期,分别将长势一致的冬瓜幼苗置于4℃和40℃培养箱(16 h光照、8 h黑暗)进行低温和高温处理,利用150 mmol/L NaCl溶液模拟盐胁迫、10%PEG6000溶液模拟干旱胁迫,每个处理5次重复。分别取未处理植株、不同胁迫处理后4、8、12和24 h植株的适量叶片,放置于液氮中速冻,带回实验室后置于-80℃超低温冰箱中保存备用。
1.2 冬瓜BhiOXR基因家族成员鉴定及克隆以拟南芥6个AtOXR基因家族成员的蛋白质序列为种子文件,通过hmmsearch和BLASTP搜索获得冬瓜BhiOXR基因,并利用MEGA11对冬瓜BhiOXR基因进行系统进化分析。分别利用EasyPure®RNA Kit试剂盒(TransGen公司)和FastKing gDNA Dispelling RT SuperMix试剂盒(TIANGEN公司)提取冬瓜叶片总RNA和反转录成cDNA。
根据冬瓜基因组数据库中BhiOXR基因的CDS序列,在ORF两端设计全长引物(表 1),利用RT-PCR技术扩增BhiOXR基因的CDS全长。反应体系为:25μL Primer STAR Max DNA Polymerase,上、下引物(10μmol/L)各1μL, 1μL cDNA(10 ng/μL), 22μL ddH2O。反应程序为:98℃预变性3 min; 98℃变性10 s、56℃退火30 s、72℃延伸30 s; 72℃终延伸5 min, 35个循环。PCR产物采用1%琼脂糖凝胶电泳检测,并将目的片段回收测序。
1.3 冬瓜BhiOXR基因的生物信息学分析
根据基因的位置信息,利用MG2C(http://mg2c.iask.in/mg2c_v2.1/)将其定位到相应的染色体上。提取每个BhiOXR基因起始密码子前2 500 bp序列,在Plant Care网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)获得顺式作用元件信息;提取冬瓜BhiOXR基因蛋白质序列,提交植物蛋白亚细胞定位预测网站(Plant-mPLoc, http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)预测其编码的蛋白质在细胞中的具体位置[12];利用在线SOPMA工具(https://npsa-prabi.ibcp.fr/cgibin/npsa_automat.pl?page=npsa_sopma.html)预测BhiOXR蛋白含有的二级结构[13];利用SWISS-MODEL在线软件预测冬瓜BhiOXR基因的蛋白三级结构[14]。
1.4 冬瓜BhiOXR基因在非生物胁迫下的表达分析在NCBI Primer-Blast上设计6个BhiOXR基因和内参基因BhiUBQ的特异性qRT-PCR引物(表 2),引物由广州生工生物工程有限公司合成。提取不同非生物胁迫处理样品的总RNA、反转录成cDNA(方法参考材料与方法1.2), qRT-PCR使用TransStart®Green qPCR SuperMix试剂盒(TransGen公司)在CFX ConnectTM实时荧光定量仪器〔BIO-RAD(美国)公司〕上进行反应。反应体系为:5μL 2×TransStart®Green qPCR SuperMix,上、下向引物(10μmol/L)各0.5μL, 2μL cDNA(10 ng/μL), 2μL ddH2O。反应程序为:94℃预变性30 s; 94℃变性5 s、55℃退火15 s、72℃延伸10 s, 40个循环,每个反应3个技术重复。
以冬瓜BhiUBQ作为内参对照,每个BhiOXR基因处理0 h的Ct值为基准,采用2-∆∆Ct方法计算冬瓜BhiOXR基因在4种非生物胁迫处理后4、8、12、24 h后的相对表达量,并采用LSD检验方法分析差异显著性。
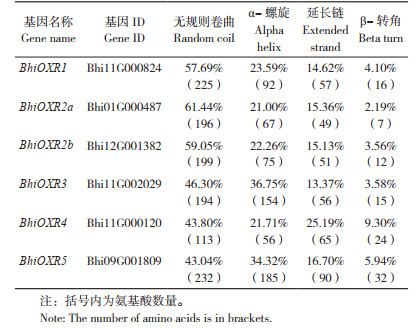
2 结果与分析 2.1 冬瓜BhiOXR基因的鉴定及克隆以拟南芥AtOXR基因为种子序列,在冬瓜基因组数据库中共鉴定得到6个冬瓜BhiOXR基因。系统进化分析可将6个基因分为5个亚族(OXR1~OXR5),其中OXR2亚族有2个成员、其余亚族仅有1个成员。因此,将冬瓜6个BhiOXR基因分别命名为BhiOXR1、BhiOXR2a、BhiOXR2b、BhiOXR3、BhiOXR4、BhiOXR5。根据BhiOXR基因的CDS序列设计全长引物,以冬瓜自交系B227的叶片cDNA为模板,利用RT-PCR进行扩增,成功克隆获得BhiOXR2a、BhiOXR3、BhiOXR4和BhiOXR5等4个基因的全长CDS(图 1),且克隆产物经测序与冬瓜基因组数据库CDS序列一致。经尝试采用不同DNA聚合酶和扩增条件仍无法克隆BhiOXR1和BhiOXR2b的全长CDS后,利用新的反向引物克隆获得截短CDS片段(图 1)。
|
| 图 1 6个冬瓜BhiOXR基因的扩增结果 Fig. 1 Amplification results of six BhiOXR genes in wax gourd |
2.2 冬瓜BhiOXR基因的生物信息学分析
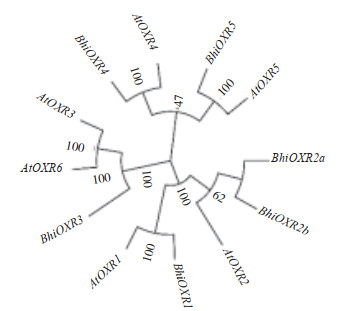
对克隆获得的BhiOXR基因进行同源性分析发现,各个亚族内BhiOXR基因和AtOXR基因同源性较高(图 2)。6个冬瓜BhiOXR基因的CDS序列长度变化范围为777~1 620 bp,编码的氨基酸数量最多有539个(BhiOXR5),最少仅258个(BhiOXR4)。染色体定位结果(表 3)显示,冬瓜BhiOXR基因不均匀分布于4条染色体,其中1、9、12号染色体各有1个成员,11号染色体有3个成员。亚细胞定位预测表明BhiOXR1、BhiOXR2a和BhiOXR2b的编码产物位于细胞核,BhiOXR3和BhiOXR5的编码产物位于叶绿体,而BhiOXR4的编码产物存在于多个细胞器中。
|
| 图 2 冬瓜BhiOXR基因与拟南芥AtOXR基因的同源性分析 Fig. 2 Homology analysis of BhiOXR genes from wax gourd and AtOXR gene from Arabidopsis |
 |
蛋白质二级结构预测结果(表 4)表明,BhiOXR所编码蛋白质的二级结构主要由4种结构组成:无规则卷曲(构成无规则卷曲的氨基酸个数占总氨基酸个数的57.69%~43.04%)、α-螺旋(34.32%~21.00%)、延伸链(13.37%~25.19%)和
β-转角(2.19%~9.30%)。蛋白质三级结构预测结果(图 3)显示,6个BhiOXR基因之间的三级结构具有高度相似性,暗示其可能具有相似的生物学功能。
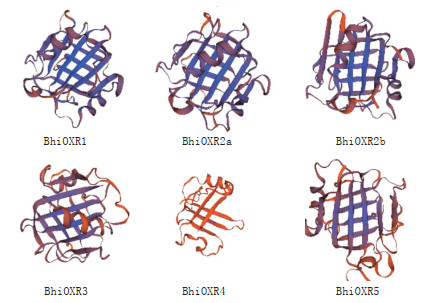
|
| 图 3 冬瓜BhiOXR蛋白三级结构预测 Fig. 3 Prediction of tertiary structure of BhiOXR proteins in wax gourd |
顺式作用元件分析结果表明,冬瓜BhiOXR基因的启动子区域含有多种抗逆元件,如参与低温响应的LTR、参与防御和应激反应的TC-rich repeats、参与厌氧诱导的ARE等顺式作用元件,暗示它们可能参与非生物胁迫响应。
2.3 冬瓜BhiOXR基因在非生物胁迫下的表达分析为明确6个冬瓜BhiOXR基因在非生物胁迫下的表达特征,利用qRT-PCR研究其在低温(4℃)、高温(40℃)、盐胁迫(150 mmol/L NaCl)和干旱(10%PEG6000)等非生物胁迫下的表达特征。
2.3.1 低温胁迫 BhiOXR2a在低温处理后表达量明显降低,而BhiOXR1、BhiOXR2b、BhiOXR4和BhiOXR5均呈现先显著升高、随后下降的趋势。BhiOXR3在处理后4 h表达量轻微升高,12 h和24 h表达量显著降低(图 4)。
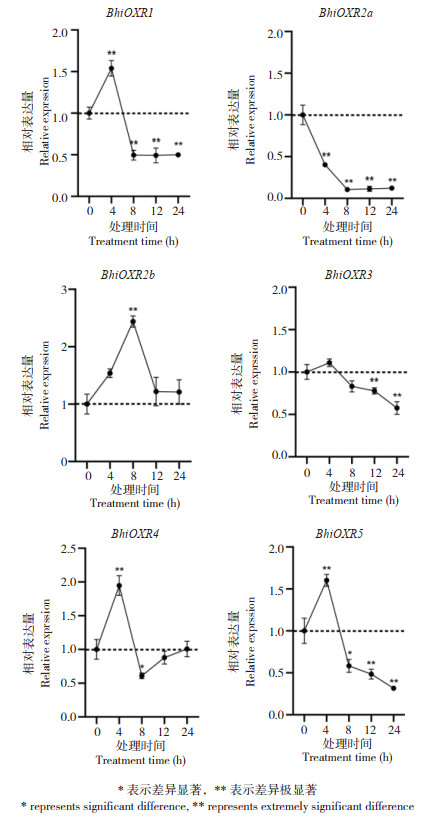
|
| 图 4 冬瓜BhiOXR家族成员在低温胁迫下的表达分析 Fig. 4 Expression analysis of wax gourd BhiOXR family members under low temperature stress |
2.3.2 高温胁迫 图 5显示,高温处理后,BhiOXR2a、BhiOXR2b和BhiOXR4的表达量有波动,但均未显著升高或降低;BhiOXR1、BhiOXR3和BhiOXR5的表达量均显著升高,并分别在24、12和12 h达到峰值。
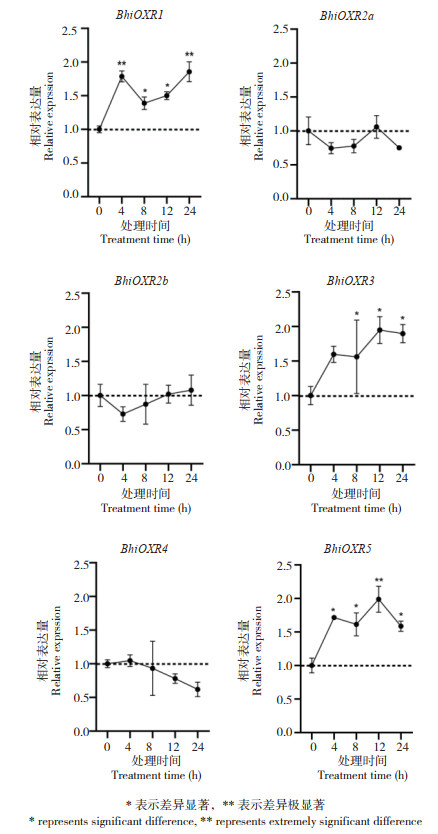
|
| 图 5 冬瓜BhiOXR家族成员在高温胁迫下的表达分析 Fig. 5 Expression analysis of wax gourd BhiOXR family members under high temperature stress |
2.3.3 盐胁迫 盐处理后,BhiOXR2a和BhiOXR4的表达量显著降低;BhiOXR2b的表达量在盐处理后4 h升高并达到峰值,随后恢复正常水平;BhiOXR3和BhiOXR5在盐处理后表达量有所下降,但降幅不大;BhiOXR1的表达量在盐处理前后没有发生显著变化(图 6)。

|
| 图 6 冬瓜BhiOXR家族成员在盐胁迫下的表达分析 Fig. 6 Expression analysis of wax gourd BhiOXR family members under salt stress |
2.3.4 干旱胁迫 BhiOXR3的表达量在干旱处理后8 h轻微降低,BhiOXR5的表达量在处理后4 h和8 h也降低,但均未发生显著降低;BhiOXR1在干旱处理前期表达无明显变化,24 h时表达量显著升高;BhiOXR2a在干旱处理下表达量先下降(12 h时达到最低),然后又急剧升高,24 h时达到峰值;BhiOXR2b的表达量在受到干旱胁迫后有所升高;BhiOXR4的表达量有所下降,处理后12h达到谷值(图 7)。
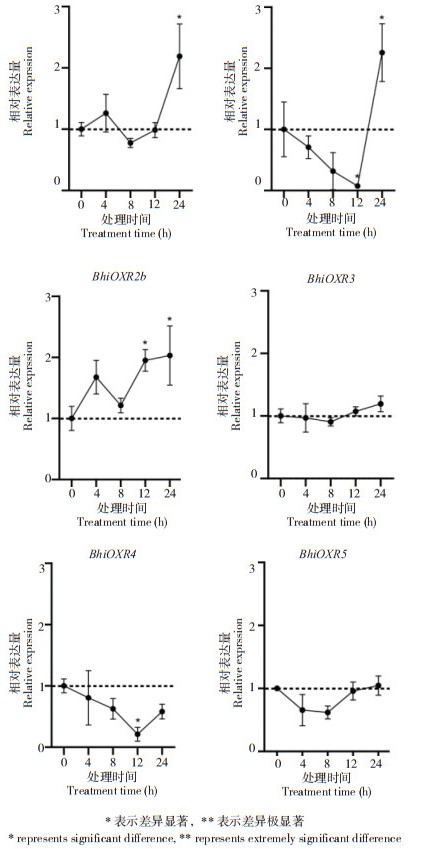
|
| 图 7 冬瓜BhiOXR家族成员在干旱胁迫下的表达分析 Fig. 7 Expression analysis of wax gourd BhiOXR family members under drought stress |
3 讨论
非生物胁迫会影响植物的生长和发育,最终会导致产量减少和品质下降[13]。处于非生物胁迫下的植物,细胞内会产生过量的活性氧(ROS)[14],这些ROS将破坏蛋白质、脂质和DNA等生物大分子物质,从而导致细胞损伤甚至死亡[15-17]。前人研究表明,OXR基因可以影响氧自由基清除基因的表达,降低细胞中的ROS水平,从而减少ROS对细胞的损伤[5]。通过对冬瓜OXR基因的全基因组鉴定,我们获得6个OXR成员,根据与拟南芥OXR基因的系统发育树,可将其分为OXR1~OXR5等5个亚族,其中OXR2亚族有2个成员、其余亚族均有1个成员;拟南芥OXR有6个成员,OXR3亚族有2个成员,其余亚族都仅有1个成员[6];菊科植物向日葵也有6个OXR成员,其中OXR1亚族有2个成员,其余亚族都有1个成员[7]。张梦云等[8]通过对7种十字花科植物的OXR基因进行全基因组鉴定,发现十字花科7种物种中的OXR基因家族成员的数量为5~11个,且位于OXR1~OXR5亚族的成员数量不一致;单子叶植物小麦有15个OXR成员,可分为Ⅰ~Ⅳ组,Ⅰ组有6个成员,Ⅱ组、Ⅲ组和Ⅳ组分别有3个成员[9]。说明OXR家族成员的个数以及不同OXR亚族成员的个数也会因物种特异性而产生差异。
基因表达趋势通常可以反映其相应功能[18-19]。Zhang等[20]通过对西瓜和甜瓜超氧化物歧化酶(SOD)基因家族的全基因组鉴定及其在胁迫下的表达模式分析,发现大多数CmSOD和ClSOD基因在低温、NaCl和PEG6000下表达明显上调,表明它们可能在应答胁迫时发挥重要作用。C2H2锌指蛋白家族(C2H2-ZFP)在植物的生长发育和非生物胁迫响应中起着重要作用,Chen等[21]从黄瓜基因组中共鉴定出101个C2H2锌指蛋白家族成员,通过qRT-PCR分析了16个C2H2-ZFP基因在低温、干旱、盐和脱落酸(ABA)胁迫下的表达特征,表明C2H2-ZFP基因可能参与不同的信号通路。为揭示小麦TaOXR基因在非生物胁迫〔NaCl、甘露醇(Mannitol)、SA、H2O2〕中的潜在功能,李寒等[9]使用qRT-PCR分析TaOXR1.6A和TaOXR2.5D的表达水平,结果显示两者均能响应氧化胁迫。
因此,为了解冬瓜BhiOXR基因在非生物胁迫应答过程中的重要作用,本研究利用qRT-PCR技术分析BhiOXR基因在4种非生物胁迫(低温、高温、盐和干旱)下的表达特征,结果表明6个BhiOXR基因均能响应特定的非生物胁迫,推测BhiOXR基因可能在抵抗非生物胁迫过程中发挥重要作用。然而,不同的BhiOXR基因响应的非生物胁迫有异同,如BhiOXR1响应低温、高温和干旱胁迫,不响应盐胁迫;BhiOXR2a、BhiOXR2b和BhiOXR4响应低温、盐和干旱胁迫,不响应高温胁迫;BhiOXR3和BhiOXR5响应低温、高温和盐胁迫,不响应干旱胁迫,这可能与基因的功能特性有关。
4 结论通过全基因组鉴定、克隆获得6个冬瓜OXR基因家族成员,并对这些成员的物理化学性质、系统发育、染色体定位、启动子、亚细胞定位以及蛋白质二级结构和三级结构等进行生物信息学分析。通过qRT-PCR分析其响应低温、高温、盐胁迫和干旱等非生物胁迫的表达特征,发现冬瓜BhiOXR基因受低温、高温、盐和干旱等非生物胁迫诱导表达,并且不同的BhiOXR基因响应的非生物胁迫有异同,由此推测6个BhiOXR成员可能在抵御不同非生物胁迫过程中所表现的抗氧化作用不尽相同。本研究结果为进一步探究冬瓜BhiOXR基因的生物学功能奠定了基础,对开展冬瓜分子育种有一定的参考作用。
| [1] |
GROVER J K, ADIGA G, VATS V, RATHI S S. Extracts of Benincasa hispida prevent development of experimental ulcers[J]. Journal of Ethnopharmacology, 2001, 78(2): 159-164. DOI:10.1016/S0378-8741(01)00334-8 |
| [2] |
VERMA V K, BEHERA T K, MUNSHI A D, PARIDA S K, MOHAPATRA T. Genetic diversity of ash gourd 〔Benincasa hispida(Thunb.)Cogn. 〕inbred lines based on RAPD and ISSR markers and their hybrid performance[J]. Scientia Horticulturae, 2007, 113(3): 231-237. DOI:10.1016/j.scienta.2007.03.019 |
| [3] |
JIANG B, LIU W R, HE X M, PENG Q W, XIE D S. Characterization and chromosomal distribution of Ty1-copia retrotransposons in wax gourd[J]. Gene, 2014, 551(1): 26-32. DOI:10.1016/j.gene.2014.08.014 |
| [4] |
谢大森, 江彪, 刘文睿, 薛舒丹, 张白鸽. 优质、抗病冬瓜多样化育种研究进展[J]. 广东农业科学, 2020, 47(11): 50-59. DOI:10.16768/j.issn.1004-874X.2020.11.006 XIE D S, JIANG B, LIU W R, XUE S D, ZHANG B G. Research progress on diversified breeding of high-quality and disease-resistant wax gourd[J]. Guangdong Agricultural Sciences, 2020, 47(11): 50-59. DOI:10.16768/j.issn.1004-874X.2020.11.006 |
| [5] |
FINELLI M J, OLIVER P L. TLDc proteins: new players in the oxidative stress response and neurological disease[J]. Mammalian Genome, 2017, 28(9): 395-406. DOI:10.1007/s00335-017-9706-7 |
| [6] |
FRANCISCO C, REGINA M, LUCILA G, NATANAEL M, SERGIO A, ANDREA M A, DANIEL H G AND ELINA W. The mitochondrial oxidation resistance protein AtOXR2 increases plant biomass and tolerance to oxidative stress[J]. Journal of Experimental Botany, 2019, 70(12): 3177-3195. DOI:10.1093/jxb/erz147 |
| [7] |
TORTI P, RAINERI J, MENCIA R, CAMPI M, GONZALEZ D H, WELCHEN E. The sunflower TLDc-containing protein HaOXR2confers tolerance to oxidative stress and waterlogging when expressed in maize plants[J]. Plant Science, 2020, 300: 110626. DOI:10.1016/j.plantsci.2020.110626 |
| [8] |
张梦云, 袁凌云, 朱世东, 侯金锋, 侯喜林, 刘同坤, 单国雷, 陈国户, 唐小燕, 汪承刚. 十字花科植物抗氧化基因(OXR)的鉴定与生物信息学分析[J/OL]. 分子植物育种: 1-11[2021-08-26]. http://kns.cnki.net/kcms/detail/46.1068.S.20210825.1923.021.html. ZHANG M Y, YUAN L Y, ZHU S D, HOU J F, HOU X L, LIU T S, DAN G L, CHEN G H, TANG X Y, WANG C G. Genome-wide identifification and analysis of oxidation resistance genes (OXR) in cruciferae[J/OL]. Molecular Plant Breeding: 1-11[2022-08-26]. http://kns.cnki.net/kcms/detail/46.1068.S.20210825.1923.021.html. |
| [9] |
李寒, 夏鹏亮, 唐艳红, 郑涛, 马东方, 李艳. 小麦OXR基因家族全基因组生物信息学及表达分析[J/OL]. 分子植物育种: 1-10[2022-04-12]. http://kns.cnki.net/kcms/detail/46.1068.S.20220411.0854.004.html. LI H, XIA P L, TANG Y H, ZHENG T, MA D F, LI Y. Genome-wide bioinformatics and expression analysis of OXR gene family in Triticum aestivum[J/OL]. Molecular Plant Breeding: 1-10[2022-04-12]. http://kns.cnki.net/kcms/detail/46.1068.S.20220411.0854.004.html. |
| [10] |
REGINA M, GABRIEL C, GEORGINA F, PABLO T, FRANCISCO C, JUTTA L M, ALVAREZ M E, WELCHEN E. OXR2 increases plant defense against a hemibiotrophic pathogen via the salicylic acid pathway[J]. Plant Physiology, 2020, 184(2): 1112-1127. DOI:10.1104/pp.19.01351 |
| [11] |
XIE D S, XU Y C, WANG J P, LIU W R, ZHOU Q, LUO S B, HUANG S W, HE X M, LI Q, PENG Q W, YANG X Y, YUAN J Q, YU J G, WANG X Y, LUCAS W J, HUANG S W, JIANG B, ZHANG Z H. The wax gourd genomes offer insights into the genetic diversity and ancestral cucurbit karyotype[J]. Nature Communications, 2019, 10(1): 51-58. DOI:10.1038/s41467-019-13185-3 |
| [12] |
CHOU K C, SHEN H B. Plant-mPLoc: a top-down strategy to augment the power for predicting plant protein subcellular localization[J]. PLoS ONE, 2017, 5(6): e11335. DOI:10.1371/journal.pone.0011335 |
| [13] |
COMBET C, BLANCHET C, GEOURJON C, DELÉAGE G. NPS@: Network protein sequence analysis[J]. Trends in Biochemical Sciences, 2000, 25(3): 147-150. DOI:10.1016/S0968-0004(99)01540-6 |
| [14] |
WATERHOUSE A, BERTONI M, BIENERT S, STUDER G, TAURIELLO G, GUMIENNY R, HEER F T, DE BEER T A P, REMPFER C, BORDOLI L, LEPORE R, SCHWEDE T. SWISSMODEL: homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(1): 296-303. DOI:10.1093/nar/gky427 |
| [15] |
MEENA K K, SORTY A M, BITLA U M, CHOUDHARY K, GUPTA P, PAREEK A, SINGH D P, PRABHA R, SAHU P K, GUPTA V K, SINGH H B, KRISHANANI K K, MINHAS P S. Abiotic stress responses and microbe-mediated mitigation in plants: the omics strategies[J]. Frontiers in Plant Science, 2017, 8: 172. DOI:10.3389/fpls.2017.00172 |
| [16] |
CZARNOCKA W, KARPIŃSKI S. Friend or foe? Reactive oxygen species production, scavenging and signaling in plant response to environmental stresses[J]. Free Radical Biology and Medicine, 2018, 122: 4-20. DOI:10.1016/j.freeradbiomed.2018.01.011 |
| [17] |
NADARAJAH K K. ROS homeostasis in abiotic stress tolerance in plants[J]. International Journal of Molecular Sciences, 2020, 21(15): 5208. DOI:10.3390/ijms21155208 |
| [18] |
SH EI K H-MOH A M A DI M, ET EM A DI N, N I K BA K H T A, FARAJPOUR M, ARAB M, MAJIDI M M. Wheatgrass germination and seedling growth under osmotic stress[J]. Agronomy Journal, 2018, 110(2): 572-585. DOI:10.2134/agronj2017.06.0364 |
| [19] |
徐磊, 刘洋. 高粱组ⅢWRKY转录因子对干旱胁迫的表达分析[J]. 广东农业科学, 2021, 48(2): 11-16. DOI:10.16768/j.issn.1004-874X.2021.02.002 XU L, LIU Y. Expression analysis of group Ⅲ WRKY transcription factors in sorghum under drought st ress[J]. Guangdong Agricultural Sciences, 2021, 48(2): 11-16. DOI:10.16768/j.issn.1004-874X.2021.02.002 |
| [20] |
ZHANG G Y, DING Q, WEI B Q. Genome-wide identification of superoxide dismutase gene families and their expression patterns under low-temperature, salt and osmotic stresses in watermelon and melon[J]. 3 Biotech, 2021, 11: 194. DOI:10.1007/s13205-021-02726-7 |
| [21] |
CHEN Y, WANG G, PAN J, WEN H F, DU H, SUN J X, ZHANG K Y, LYU D, HE H L, CAI R, PAN J S. Comprehensive genomic analysis and expression profiling of the C2H2 zinc finger protein family under abiotic stresses in cucumber(Cucumis sativus L.)[J]. Genes, 2020, 11(2): 171. DOI:10.3390/genes11020171 |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49