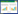文章信息
基金项目
- 国家自然科学基金(32030102);广东省重点领域研发计划项目(2018B020203003);广东省自然科学基金(2019A1515011134)
作者简介
- 许美娜(1990—),女,在读博士生,研究方向为动物遗传育种,E-mail:Meina709@163.com.
通讯作者
- 何祖勇(1981—),男,博士,副教授,研究方向为家猪生物育种,E-mail:zuyonghe@foxmail.com.
文章历史
- 收稿日期:2022-05-28
《中国畜禽遗传资源志·猪志》记载现代家猪的祖先是古代野猪。科学家们研究众多地区不同猪种的线粒体D-loop区序列多态性发现,东南亚拥有最古老的野猪群体,它们逐渐分散至不同地区,随着自然条件的改变以及人工选择,野猪的习性、体态结构和生理机能等逐渐发生变化,最终形成家猪[1]。至今家猪已成为最重要的家畜之一[2]。我国人工驯化野猪历史悠久,各地区劳动人民结合当地的自然条件、风俗习惯和社会经济条件培育出众多具有地方特色的猪种,使我国成为世界上猪种资源最为丰富的国家。目前已有76个地方猪种收录于《中国畜禽遗传资源志·猪志》。我国的地方猪品种具有肉质细嫩、肌纤维细、肌内脂肪含量高等优良肉质性状,但存在生长缓慢、早熟易肥、瘦肉率低、饲料转化率低等缺点[3]。而西方瘦肉型猪具有生长速度快、饲料利用率高、瘦肉率高等优点,正好可以弥补我国地方猪种的缺点。因此,利用西方瘦肉型猪种进行杂交可以有效改良我国地方猪种的生长及瘦肉率等性状,但同时也对其肉质性状造成不利影响。而基因编辑技术可对目标性状进行精确修饰,并可避免对其他重要经济性状带来不利影响,同时还可以减少传统杂交因世代间隔和级进杂交选育的耗时,能在最短的时间内得到符合生产需求的突破性改良品种,实现猪个体和群体水平的“基因编辑育种” [4]。
基因编辑技术是一种可以对生物体基因组DNA进行特异性识别并定点修饰的技术[5]。通过人工核酸酶定点切割DNA双链,依靠细胞自身的修复机制,可以对生物个体的基因组进行基因敲除、敲入和碱基替换等一系列人工修饰,实现基因序列的改变。目前,基因编辑技术可分为三代:第一代利用锌指核酸酶(Zinc Finger Nucleases, ZFN)。ZFN由锌指蛋白(Zinc Finger Protein, ZFP)组成的DNA识别结合域和具有限制性内切酶活性的Fok Ⅰ组成的DNA切割域两部分组成。其中DNA识别结合域赋予ZFN结合特异性,使其基因组在特定靶位点上结合,而非特异性核酸内切酶Fok Ⅰ则发挥切割功能,使靶序列的双链断裂(Double Strand Break, DSB) [6]。第二代利用类转录激活因子效应物核酸酶(Transcription Activator-Like Effector Nucleases, TALEN)。TALEN的构造与ZFN类似,由TALE基序串联成决定靶向性的DNA识别模块,与Fok Ⅰ结构域连接而成[7]。与锌指基序不同,1个TALE基序识别1个碱基对,因此串联的TALE基序与所识别的碱基对是一一对应的关系,使设计TALEN比设计ZFN更为简易[8]。第三代利用常间回文重复序列丛集(Clustered Regularly Interspaced Short Palindromic Repeats/Cas,CRISPR/Cas)系统[9]。三者都是通过人工核酸酶对基因组中特定位置产生断裂缺口,诱导生物体对受损DNA进行修复,从而产生突变。但前两项技术因成本高、操作复杂等特点而发展受限;而CRISPR/Cas系统因构造简洁,操作简便,编辑效率高且可以进行多个位点修饰而被广泛使用[10]。因此,本文主要介绍CRISPR/Cas9基因编辑技术的发展及其在家猪遗传改良中的研究进展,以期加深人们对基因编辑技术在家猪遗传育种中具有特殊作用的认识。
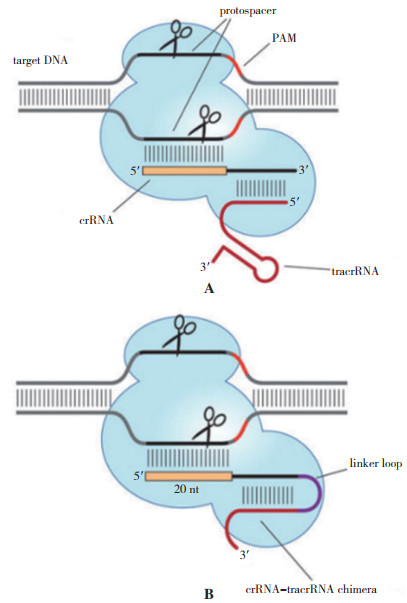
1 CRISPR/Cas9基因编辑技术的发展 1.1 CRISPR/Cas9CRISPR/Cas是一种古细菌的天然免疫系统[11]该系统由CRISPR序列与CAS(CRISPR associate system) 两部分组成。CRISPR/Cas系统可分为Type Ⅰ、Type Ⅱ和Type Ⅲ 3种类型[9] Type Ⅱ因其简易性和可操作性被开发成为使用最广泛的基因编辑工具——CRISPR/Cas9。CRISPR/Cas9来源于酿脓链菌的SF370菌株。Cas9蛋白是一种核酸内切酶,包含RuvC和HNH两个具有DNA切割活性的结构域,在CRISPR RNA(crRNA)与反式激活crRNA(Trans-activating crRNA, tracrRNA) 复合体的引导下,Cas9蛋白结合到靶序列上,HNH结构域切割与crRNA配对的单链,而RuvC结构域则切割crRNA的非互补单链,最终造成DSB[12] (图 1A)。DNA双链断裂后可引起非同源末端连接(Non-Homologous End Joining, NHEJ)或者同源重组修复(Homology-Directed Repair, HDR)。NHEJ修复的过程中往往会产生小片段DNA的插入或删除(Insertion and deletion, Indel),造成移码突变,致使基因功能丧失,从而实现基因敲除;在携带目标突变供体模板的情况下,细胞可通过HDR机制实现定点突变或插入。为了方便操作,目前普遍使用crRNA和tracrRNA嵌合一起的单链向导RNA(single-guide RNA, sgRNA)来引导核酸内切酶Cas9结合到基因组的特定位点进行切割,以实现基因编辑目的(图 1B)。
目前CRISPR/Cas9基因编辑技术在人类疾病模型构建和畜禽遗传改良等领域已产生重要的推动作用,但研究应用过程中也发现该技术的一些缺点:Cas9核酸酶对特异性靶位点进行切割后,在DSB修复过程中,HDR在与NHEJ进行竞争时往往占优势,因此基因编辑结果基本上是Indel造成的基因敲除,而难以获得HDR介导的点突变。对于大多数已知的遗传疾病和畜禽遗传变异,基因编辑的主要目的是对目标变异位点进行精确的点突变修复,而非对基因进行随机破坏。因此,研究人员试图提高HDR的效率并抑制NHEJ以提高基因编辑的准确性[13-15],同时不断研究开发可进行精确碱基修饰的基因编辑新技术。
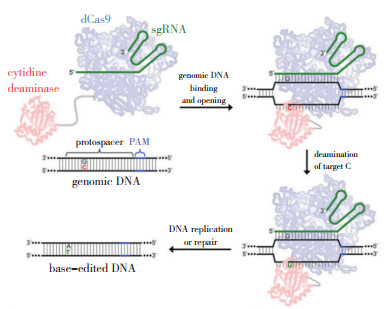
1.2 单碱基编辑技术的发展1.2.1 胞嘧啶碱基编辑系统(Cytosine Base Editor, CBE) 2016年,哈佛大学的Liu研究团队报道了一种基于CRISPR系统的单碱基编辑器(Base Editor, BE),是一种不需要引入DNA双链断裂即可进行单碱基转换的基因编辑系统CBE。CBE系统可在基因组靶位点上实现C → T碱基的替换[15]。构建CBE首先要对Cas9蛋白进行改造,通过点突变使其切割结构域失去活性而获得dead Cas9(dCas9),接着通过连接肽链将dCas9蛋白与胞嘧啶脱氨酶连接形成CBE(图 2)。CBE系统通过sgRNA的引导将dCas9锚定到基因组靶位点上,dCas9与DNA双链结合但不发生切割,此时胞嘧啶脱氨酶使靶位点上的碱基C脱去氨基变为U,当DNA发生复制时,U会被替换为T,而DNA互补链上的G相应地被替换成A,从而实现G-C碱基对到A-T碱基对的替换(图 2)。通过DNA的复制和修复,实现了不需要引入DNA双链断裂即可进行单碱基转换的编辑。
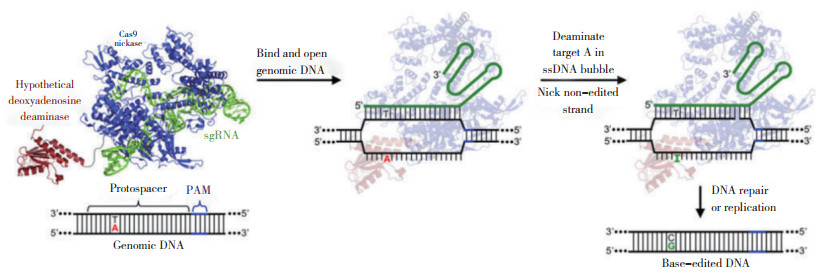
1.2.2 腺嘌呤碱基编辑系统(Adenine Base Editor, ABE) 2017年,Liu研究团队在开发CBE系统的经验基础上,进一步开发出腺嘌呤碱基编辑系统ABE。他们发现目前已知的腺嘌呤脱氨酶均不能以DNA为底物对碱基A进行脱氨,因此,他们以大肠杆菌tRNA脱氨酶(TadA)为基础,通过定向进化筛选到能作用于单链DNA的腺嘌呤脱氨酶突变体,该突变体含有A106V和D108N两个突变,被命名为TadA*[16]。通过XTEN连接肽将nCas9(D10A)切刻酶与TadA*进行连接,并在nCas9的C端加上核定位信号(Nuclear Localization Signal, NLS),形成TadA*- XTEN-nCas9-NLS,最终获得了能实现A-T碱基对到G-C碱基对转换的ABE编辑器[16]。ABE系统在工作过程中,将碱基A脱去氨基,变成I,DNA聚合酶将I识别为G,使其与C配对,经过下一轮DNA复制即可实现A-T碱基对到G-C碱基对的转换(图 3)。
单碱基编辑技术是在CRISPR/Cas9系统的基础上发展建立起来的,相比于CRISPR/Cas9介导的HDR编辑,单碱基编辑既不会诱导基因组发生DSB,也不需要额外添加DNA重组修复模板,有效降低了脱靶所造成的基因组Indel发生。在动物育种研究中,单碱基编辑技术已展现了很好的应用前景[17-19],但是该技术目前还存在一些不足,其中一个突出问题是编辑结果带有副产物,以及编辑窗口宽度受限,导致其应用范围受到一定限制[20]。
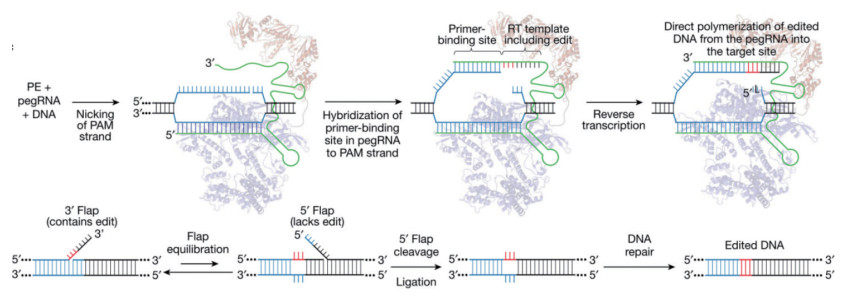
1.2.3 先导编辑系统(Prime Editor, PE) CBE和ABE组合虽然可以进行4种碱基转换(C → T, G → A, A → G, T → C),但无法对另外8种碱基转换(C → A, C → G, G → C, G → T, A → C, A → T, T → A, T → G) 以及Indel起作用。而2019年问世的先导编辑技术PE则可以在不依赖DSB和模板DNA的条件下有效实现12种碱基转换,此外还能有效实现多碱基的插入(最多可插入44 bp)和删除(最多删除80 bp) [21]。PE系统利用先导编辑向导RNA(prime editing guide RNA, pegRNA)引导Cas9切刻酶(nCas9)与逆转录酶的融合蛋白在基因组靶位点实现基因编辑[21]。其中,peg RNA由3部分组成,包括单链向导RNA(single-guide RNA, sgRNA)、引物结合位点(Prime Binding Site, PBS)和携带有靶位点编辑信息的逆转录模板(RT template including edit)。pegRNA依靠其sgRNA序列引导nCas9蛋白结合到基因组靶位点上,并切断含有PAM序列的DNA单链。断裂的DNA单链与pegRNA上的PBS序列互补结合,逆转录酶则以RT序列作为模板进行逆转录反应。反应完成后,DNA切口处出现3' flap(分支结构)和5' flap的动态平衡。其中3' flap含有目标突变,而5' flap不含目标突变但更易被结构特异性内切酶FEN1(片状核酸内切酶)切除,残余的5' flap碱基将进一步被5' 核酸外切酶EXO1切除,最后经过DNA链的连接和修复实现目标序列的替换(图 4)。
2 CRISPR/Cas9基因编辑技术在猪遗传改良中的应用
自2013年张锋研究团队在真核细胞中成功进行基因编辑以来[10],CRISPR/Cas9技术发展迅速,在动物研究领域得到广泛应用。CRISPR/Cas9技术突破了传统育种的局限性,可在较短时间内培育出传统育种方法无法育成或难以育成的动物品种,从而加快动物遗传改良进展。目前CRISPR/Cas9基因编辑技术在家猪遗传育种研究中的关键进展如下。
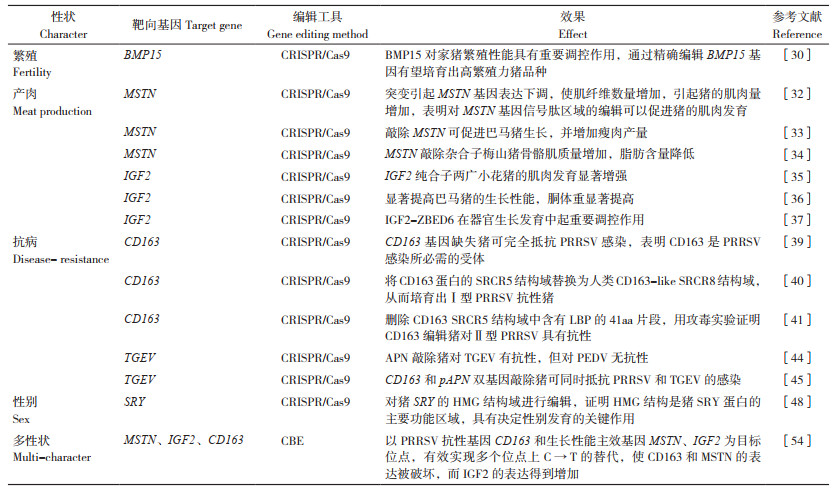
2.1 在高繁殖力育种新材料创制中的应用猪的繁殖性能水平直接决定了整个繁育体系的效率,是生猪产业发展的基础。母猪繁殖力属于低遗传力性状,通过常规育种技术获得遗传进展缓慢。因此,通过对调控繁殖性能的关键候选基因进行编辑,有望加快培育出高繁殖力猪品种。目前已鉴定到BMP15基因是调控单胎动物排卵率和产仔数的1个关键基因[22-30],但它对多胎家猪繁殖力的调控功能尚不明确[31-32]。Shi等[33]应用CRISPR/Cas9编辑BMP15外显子1区域,获得一批基因编辑大白猪,但双等位基因编辑的母猪均不育;通过对卵巢进行HE组织切片分析发现,双等位基因编辑猪卵泡中的细胞核着色较深,而且有颗粒细胞丢失和卵母细胞形态异常等现象,在次级卵泡阶段卵泡发育阻滞,导致无法产生可排卵的成熟卵泡。但编码BMP15蛋白前导肽序列产生66 bp缺失的基因编辑杂合子母猪,其初产活仔13头,明显高于供体细胞来源的英系大白母猪的繁殖性能(初产母猪窝均产活仔数为8.46±2.83;经产母猪窝均产活仔数为9.15±2.84) [34],表现出高产潜能,揭示BMP15基因对家猪繁殖性能具有重要调控作用,通过精确编辑该基因有望培育出高繁殖力猪品种。
2.2 在高产肉性能育种新材料创制中的应用肌生成抑制素(Myostatin, MSTN)是肌肉生长的负调控因子,通过抑制成肌细胞的增殖发挥作用[35] Li等[36]在两广小花猪的MSTN信号肽区域引入突变(PVD20H和GP19del),结果发现突变可引起MSTN基因表达下调,使肌纤维数量增加,促进地方猪的肌肉生成,表明MSTN基因信号肽区域的精确编辑可以促进猪的肌肉发育。Zhu等[37]通过敲除MSTN基因促进了中国巴马猪的生长,并增加猪的瘦肉产量,其中6月龄时MSTN基因编辑猪比野生型重约9.6%。Li等[38]研究发现,MSTN基因敲除杂合子梅山猪的骨骼肌重量增加9%,脂肪含量降低8.48%。胰岛素样生长因子2(IGF2)对动物胎儿发育以及出生后的生长发育均发挥重要作用。研究表明,猪IGF2基因内含子3的3072位点若发生单碱基突变(G → A),会引起IGF2基因的抑制因子ZBED6从结合基序(motif)上解离,进而提高IGF2基因的表达水平,使猪的瘦肉量增加3%~4%[39]。Liu等[40]对两广小花猪的胚胎成纤维细胞IGF2内含子3中的ZBED6结合基序进行破坏,结果可显著上调IGF2的表达,增强了猪胚胎成纤维细胞(Porcine Embryonic Fibroblast, PEFs)的成肌分化潜能和细胞增殖能力;他们以IGF2基因编辑细胞作为供体,通过体细胞克隆获得ZBED6结合基序缺失63 bp的两广小花猪纯合个体,该基因编辑猪肌肉发育显著增强,在338 d时体重比野生型猪提高32%。Xiang等[41]对IGF2基因内含子3的3072位点进行编辑,发现这些突变解除了ZBED6与其基序的结合作用,显著提高了巴马猪的生长性能,胴体重比野生型提高33.35%。Wang等[42]敲除ZBED6获得了ZBED 6-/- 瘦肉型巴马猪,其IGF2表达量显著上调,骨骼肌和内脏器官的重量显著高于野生型,进一步证明了IGF2-ZBED6在器官生长发育中的重要调控作用。
2.3 在抗病育种新材料创制中的应用疫病防控是保障生猪养殖产业健康可持续发展的关键,通过基因编辑提高家猪的抗病能力可有效提升猪场的经济效益。猪繁殖与呼吸综合征是当前猪场疾病防控最备受关注的疾病之一,该病是由猪繁殖与呼吸道综合征病毒(PRRSV)感染引起的一种高危传染病,俗称蓝耳病。PRRSV通过感染家猪的巨噬细胞或单核细胞,进一步引发怀孕母猪在妊娠后期流产或产死胎,给养猪生产造成巨大经济损失[43]。2016年Whitworth等利用CRISPR/Cas9技术培育了CD163基因编辑猪。通过攻毒实验发现,CD163基因缺失的猪可完全抵抗PRRSV感染,表明CD163是PRRSV感染所必需的受体[44]。2017年该研究团队将CD163蛋白的清道夫受体半胱氨酸5(Scavenger Receptor Cysteine Rich 5, SRCR5) 结构域替换为人类CD163-like SRCR8结构域,培育出I型PRRSV抗性猪[45]。删除CD163 SRCR5结构域中含有配体结合口袋(Ligand-binding pocket, LBP) 的41aa片段,获得基因编辑大白猪,经攻毒试验证明该基因编辑猪对Ⅱ型PRRSV具有完全抗性[46]。这些研究表明,通过编辑CD163基因可以产生PRRSV抗性猪品种,可有效消除PRRSV对生猪生产的威胁。除PRRSV外,传染性胃肠炎病毒(TGEV)也是一种具有高度接触传染性的病毒性疾病,常与猪流行性腹泻病毒(PEDV) 和猪轮状病毒(PoRV)等猪腹泻相关病毒同时感染[47]。TGEV是一种单链阳性RNA冠状病毒,以猪肠道上皮细胞为感染靶点。目前认为pAPN蛋白是介导TGEV感染的关键受体。TGEV的糖蛋白与小肠上皮细胞表面的pAPN受体结合后可介导膜融合,从而导致病毒进入到上皮细胞内[48]。Whitworth等[49]报道,APN敲除猪断奶后对TGEV有抗性,但对PEDV无抗性。Xu等[50]利用基因编辑技术同时敲除CD163和pAPN,发现双基因敲除猪(Double-gene-Knockout, DKO) 可以同时抵抗PRRSV和TGEV感染;此外,除肉色评分和铁含量外,DKO猪和野生型WT猪的生产性能、繁殖性能和猪肉营养成分含量均无显著性差异。
2.4 在性别控制上的应用仔猪阉割是生猪生产中的一个重要环节,可以减少雄性生殖系统发育的能量消耗,以最大化发挥猪的生长效能,还可消除公猪性激素产生的骚味导致的猪肉口感与品质下降。但是仔猪阉割是一项耗费精力和成本的工作,而且对仔猪会产生很大的刺激,如果操作不当容易引起感染,甚至会影响后期生长。此外,阉割还涉及动物伦理与福利问题,因此,如能通过基因编辑技术使母猪所产仔猪均为雌性个体,将有效避免仔猪阉割带来的问题。
性别控制技术是通过对动物的正常生殖过程进行人为干预,使成年雌性动物产出人们期望的单一性别后代的一门生物技术。哺乳动物Y染色体上存在一个性别决定区域,即SRY基因,SRY基因又称为睾丸决定因子(Testis Determining Factor, TDF),是哺乳动物性别决定的总开关[51-52]。研究发现,SRY基因的突变与人类及其他哺乳动物的性逆转综合征相关[51]。Kurtz[53]等使用CRISPR/Cas9技术对猪SRY的HMG结构域进行编辑,发现SRY基因编辑雄性仔猪发育出完整的雌性生殖器官,其子宫和输卵管形态与野生型母猪的相同,但卵巢相对较小,证明位于SRY基因的HMG结构是猪SRY蛋白的主要功能区域,具有决定性别发育的关键作用。
2.5 在多性状遗传改良中的应用在家猪基因组上同时实现多位点单碱基编辑,可同时对家猪的多个性状进行改良。Song等[54]以PRRSV抗性基因CD163和控制生长性能的主效基因MSTN和IGF2为靶点,在猪受精卵中注射CBE系统,有效实现了多个位点上C → T的替换。他们发现在CD163和MSTN基因中突变均形成了1个终止密码子,破坏了2个基因的表达;同时在IGF2中引入1个有益的等位基因,可增强IGF2的表达。生长性能分析结果表明,编辑猪的生长速度显著快于野生型猪,且组织切片分析表明,编辑猪肌纤维的平均横截面积较野生型猪增大约160%, 而纤维数量则降低23%。为了评估猪对病毒感染的抵抗力,使用PRRSV毒株CH-1R进行感染,结果发现编辑猪的PRRSV抗体在所有时间点均为阴性,而野生型猪则在感染7~10 d一直呈阳性。该研究结果表明多位点单碱基编辑可同时提高猪的生长性能和抗病性[54] (表 1)。
|
3 展望
目前CRISPR/Cas9以及单碱基编辑在家猪遗传育种上的应用主要侧重于编辑具有较大遗传效应的单个基因或位点,有效突破了传统育种的局限性。但目前鉴定到的影响家猪繁殖力、生长、产肉量、肉质和抗病性的关键基因和位点寥寥无几,而且这些性状还受大量微效基因控制。未来随着对包括家猪在内的动物基因组与表型组之间关系了解程度的进一步加深,多基因编辑将有可能成为改良家猪单个或多个重要经济性状的重要手段。目前CRISPR/Cas9技术虽然可以实现有限的多基因编辑,但对于同时编辑数十个甚至上百个基因或位点却无法实现,因此,未来需要改进基因编辑工具或在编辑策略上进行探索,创建一套稳定可靠的多基因编辑技术,以有效提高点突变效率,这样才有可能对控制家猪重要经济性状的多个微效基因或位点进行同时编辑,获得预期目标的遗传改良效果。
基因编辑动物能否顺利进入商业化应用,不仅受到政府主管部门监管理念与具体管理措施的影响,同时还受到公众对基因编辑技术认知和接受程度的影响。目前虽然全球多数国家对基因编辑动物的监管标准尚未明确,但学界一致认为基因编辑动物只要通过严格的生物安全评价,可有序进入商业化应用。美国食品药品监督管理局(FDA)对基因编辑动物监管严格,认为对待基因编辑动物应如同对待新药一样,须进行一系列的生物安全检测。2020年12月由美国Revivicor公司研发的基因编辑猪“GalSafe猪”,经过一系列严格的生物安全评价后获得FDA批准上市。“GalSafe猪”经CRISPR/Cas9敲除了α- 半乳糖苷寡糖的表达基因,使得对肉类过敏的人群也可以安全食用这种猪肉,为基因编辑猪的商业化应用开创了先例。2022年1月我国农业农村部发布《农业用基因编辑植物安全评价指南(试行)》,打开了基因编辑植物商品化的大门,也为基因编辑农业动物的商业化应用指引了方向。
| [1] |
FRANTZ L A, SCHRAIBER J G, MADSEN O, MEGENS H J, CAGAN A, BOSSE M, PAUDEL Y, CROOIJMANS R P, LARSON G, GROENEN M A. Evidence of long-term gene flow and selection during domestication from analyses of Eurasian wild and domestic pig genomes[J]. Nature Genetics, 2015, 47(10): 1141-1148. DOI:10.1038/ng.3394 |
| [2] |
LARSON G, DOBNEY K, ALBARELLA U, FANG M Y, MATISOOSMITH E, ROBINS J, LOWDEN S, FINLAYSON H, BRAND T, WILLERSLEV E, ROWLEY-CONWY P, ANDERSSON L, COOPER A. Worldwide phylogeography of wild boar reveals multiple centers of pig domestication[J]. Science, 2005, 307(5715): 1618-1621. DOI:10.1126/science.1106927 |
| [3] |
杨宇明. 浅谈地方猪种育种工作[J]. 中国畜禽种业, 2019, 15(6): 60. YANG Y M. Discussion on local pig breeding work[J]. The Chiense Livestock and Poulty Breeding, 2019, 15(6): 60. |
| [4] |
刘志国, 王冰源, 牟玉莲, 魏泓, 陈俊海, 李奎. 分子编写育种——动物育种的发展方向[J]. 中国农业科学, 2018, 51(12): 2398-2409. DOI:10.3864/j.issn.0578-1752.2018.12.016 LIU Z G, WANG B Y, MU Y L, WEI H, CHEN J H, LI K. Breeding by molecular writing (BMW): the future development of animaI breeding[J]. Scientia Agricultura Sinica, 2018, 51(12): 2398-2409. DOI:10.3864/j.issn.0578-1752.2018.12.016 |
| [5] |
ESVELT K M, WANG H H. Genome-scale engineering for systems and synthetic biology[J]. Molecular Systems Biology, 2013(9): 641. DOI:10.1038/msb.2012.66 |
| [6] |
钟强, 赵书红. 锌指蛋白核酸酶的作用原理及其应用[J]. 遗传, 2011, 33(2): 123-130. DOI:10.3724/sp.j.1005.2011.00123 ZHONG Q, ZHAO S H. The mechanism and application of zinc finger nucleases[J]. Hereditas (Beijing), 2011, 33(2): 123-130. DOI:10.3724/sp.j.1005.2011.00123 |
| [7] |
严爱芬, 刘婉霞, 刘芳, 唐冬生. TALEN技术研究进展[J]. 广东农业科学, 2015, 42(23): 145-150. DOI:10.16768/j.issn.1004-874X.2015.23.071 YAN A F, LIU Y X, LIU F, TANG D S. Research progress of TALEN technology[J]. Guangdong Agricultural Sciences, 2015, 42(23): 145-150. DOI:10.16768/j.issn.1004-874X.2015.23.071 |
| [8] |
CHANDRASEGARAN S, CARROLL D. Origins of programmable nucleases for genome engineering[J]. Journal of Molecular Biology, 2016, 428(5): 963-989. DOI:10.1016/j.jmb.2015.10.014 |
| [9] |
JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA J A, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [10] |
CONG L, RAN F A, COX D, LIN S L, BARRETTO R, HABIB N, HSU P D, WU X B, JIANG W Y, MARRAFFINI L A, ZHANG F. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [11] |
GRISSA I, VERGNAUD G, POURCEL C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007(8): 172. DOI:10.1186/1471-2105-8-172 |
| [12] |
HOCKEMEYER D, WANG H, KIANI S, LAI C S, GAO Q, CASSADY J P, COST G J, ZHANG L, SANTIAGO Y, MILLER J C, ZEITLER B, CHERONE J M, MENG X, HINKLEY S J, REBAR E J, GREGORY P D, URNOV F D, JAENISCH R. Genetic engineering of human pluripotent cells using TALE nucleases[J]. Nature Biotechnology, 2011, 29(8): 731-734. DOI:10.1038/nbt.1927 |
| [13] |
MALI P, YANG L, ESVELT K M, AACH J, GUELL M, DICARLO J E, NORVILLE J E, CHURCH G M. RNA-guided human genome engineering via Cas9[J]. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [14] |
DELTCHEVA E, CHYLINSKI K, SHARMA C M, GONZALES K, CHAO Y, PIRZADA Z A, ECKERT M R, VOGEL J, CHARPENTIER E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [15] |
KOMOR A C, KIM Y B, PACKER M S, ZURIS J A, LIU D R. Programmable editing of a target base in genomic DNA without doublestranded DNA cleavage[J]. Nature, 2016, 533(7603): 420-424. DOI:10.1038/nature17946 |
| [16] |
GAUDELLI N M, KOMOR A C, REES H A, PACKER M S, BADRAN A H, BRYSON D I, LIU D R. Programmable base editing of A. T to G. C in genomic DNA without DNA cleavage[J]. Nature, 2017, 551(7681): 464-471. DOI:10.1038/nature24644 |
| [17] |
ZHOU S, CAI B, HE C, WANG Y, DING Q, LIU J, LIU Y, DING Y, ZHAO X, LI G, LI C, YU H, KOU Q, NIU W, PETERSEN B, SONSTEGARD T, MA B, CHEN Y, WANG X. Programmable base editing of the sheep genome revealed no genome-wide off-target mutations[J]. Frontiers in Genetics, 2019(10): 215. DOI:10.3389/fgene.2019.00215 |
| [18] |
LIU Z, CHEN M, CHEN S, DENG J, SONG Y, LAI L, LI Z. Highly efficient RNA-guided base editing in rabbit[J]. Nature Communication, 2018, 9(1): 2717. DOI:10.1038/s41467-018-05232-2 |
| [19] |
LI G, ZHOU S, LI C, CAI B, YU H, MA B, HUANG Y, DING Y, LIU Y, DING Q, HE C, ZHOU J, WANG Y, ZHOU G, LI Y, YAN Y, HUA J, PETERSEN B, JIANG Y, SONSTEGARD T, HUANG X, CHEN Y, WANG X. Base pair editing in goat: nonsense codon introgression into FGF5 results in longer hair[J]. FEBS Journal, 2019, 286(23): 4675-4692. DOI:10.1111/febs.14983 |
| [20] |
ZHOU S, DING Y, LIU J, LIU Y, ZHAO X, LI G, ZHANG C, LI C, WANG Y, KALDS P, GAO Y, ZONG B, HUANG X, HUANG S, YU H, KOU Q, PETERSEN B, HUANG X, WANG X, MA B, CHEN Y. Highly efficient generation of sheep with a defined FecB(B) mutation via adenine base editing[J]. Genetics Selection Evolution, 2020, 52(1): 35. DOI:10.1186/s12711-020-00554-6 |
| [21] |
ANZALONE A V, RANDOLPH P B, DAVIS J R, SOUSA A A, KOBLAN L W, LEVY J M, CHEN P J, WILSON C, NEWBY G A, RAGURAM A, LIU D R. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4 |
| [22] |
GALLOWAY S M, MCNATTY K P, CAMBRIDGE L M, LAITINEN M P, JUENGEL J L, JOKIRANTA T S, MCLAREN R J, LUIRO K, DODDS K G, MONTGOMERY G W, BEATTIE A E, DAVIS G H, RITVOS O. Mutations in an oocyte-derived growth factor gene (BMP15) cause increased ovulation rate and infertility in a dosage-sensitive manner[J]. Nature Genetics, 2000, 25(3): 279-283. DOI:10.1038/77033 |
| [23] |
QIN Y, TANG T, LI W, LIU Z, YANG X, SHI X, SUN G, LIU X, WANG M, LIANG X, CONG P, MO D, LIU X, CHEN Y, HE Z. Bone morphogenetic protein 15 knockdown inhibits porcine ovarian follicular development and ovulation[J]. Frontiers in Cell and Developmental Biology, 2019(7): 286. DOI:10.3389/fcell.2019.00286 |
| [24] |
HANRAHAN J P, GREGAN S M, MULSANT P, MULLEN M, DAVIS G H, POWELL R, GALLOWAY S M. Mutations in the genes for oocytederived growth factors GDF9 and BMP15 are associated with both increased ovulation rate and sterility in Cambridge and Belclare sheep (Ovis aries)[J]. Biology of Reproduction, 2004, 70(4): 900-909. DOI:10.1095/biolreprod.103.023093 |
| [25] |
SMITH P, O W S, CORRIGAN K A, SMITH T, LUNDY T, DAVIS G H, MCNATTY K P. Ovarian morphology and endocrine characteristics of female sheep fetuses that are heterozygous or homozygous for the inverdale prolificacy gene (FecX1)[J]. Biology of Reproduction, 1997, 57(5): 1183-1192. DOI:10.1095/biolreprod57.5.1183 |
| [26] |
BRAW-TAL R, MCNATTY K P, SMITH P, HEATH D A, HUDSON N L, PHILLIPS D J, MCLEOD B J, DAVIS G H. Ovaries of ewes homozygous for the X-linked Inverdale gene (FecXI) are devoid of secondary and tertiary follicles but contain many abnormal structures[J]. Biology of Reproduction, 1993, 49(5): 895-907. DOI:10.1095/biolreprod49.5.895 |
| [27] |
BODIN L, DI PASQUALE E, FABRE S, BONTOUX M, MONGET P, PERSANI L, MULSANT P. A novel mutation in the bone morphogenetic protein 15 gene causing defective protein secretion is associated with both increased ovulation rate and sterility in Lacaune sheep[J]. Endocrinology, 2007, 148(1): 393-400. DOI:10.1210/en.2006-0764 |
| [28] |
LASSOUED N, BENKHLIL Z, WOLOSZYN F, REJEB A, AOUINA M, REKIK M, FABRE S, BEDHIAF-ROMDHANI S. FecX (Bar): A novel BMP15 mutation responsible for prolificacy and female sterility in Tunisian Barbarine Sheep[J]. BMC Genetics, 2017, 18(1): 43. DOI:10.1186/s12863-017-0510-x |
| [29] |
JUENGEL J L, DAVIS G H, MCNATTY K P. Using sheep lines with mutations in single genes to better understand ovarian function[J]. Reproduction, 2013, 146(4): R111-123. DOI:10.1530/REP-12-0509 |
| [30] |
DEMARS J, FABRE S, SARRY J, ROSSETTI R, GILBERT H, PERSANI L, TOSSER-KLOPP G, MULSANT P, NOWAK Z, DROBIK W, MARTYNIUK E, BODIN L. Genome-wide association studies identify two novel BMP15 mutations responsible for an atypical hyperprolificacy phenotype in sheep[J]. PLoS Genetics, 2013, 9(4): e1003482. DOI: 10.1371/journal.pgen.1003482PGENETICS-D-12-02801[pii].
|
| [31] |
YAN C, WANG P, DEMAYO J, DEMAYO F J, ELVIN J A, CARINO C, PRASAD S V, SKINNER S S, DUNBAR B S, DUBE J L, CELESTE A J, MATZUK M M. Synergistic roles of bone morphogenetic protein 15 and growth differentiation factor 9 in ovarian function[J]. Molecular Endocrinology, 2001, 15(6): 854-866. DOI:10.1210/mend.15.6.0662 |
| [32] |
MESTER B, RITTER L J, PITMAN J L, BIBBY A H, GILCHRIST R B, MCNATTY K P, JUENGEL J L, MCINTOSH C J. Oocyte expression, secretion and somatic cell interaction of mouse bone morphogenetic protein 15 during the peri-ovulatory period[J]. Reproduction Fertility and Development, 2015, 27(5): 801-811. DOI:10.1071/RD13336 |
| [33] |
SHI X, TANG T, LIN Q, LIU H, QIN Y, LIANG X, CONG P, MO D, LIU X, CHEN Y, HE Z. Efficient generation of bone morphogenetic protein 15-edited Yorkshire pigs using CRISPR/Cas9[J]. Biology of Reproduction, 2020, 103(5): 1054-1068. DOI:10.1093/biolre/ioaa138 |
| [34] |
曾乐平, 詹应祥. 美系、英系、新丹系长白猪繁殖性能的比较[J]. 养猪, 2002(4): 11-12. DOI:10.13257/j.cnki.21-1104/s.2002.04.003 ZENG L P, ZHAN YX. Comparison of reproductive performance of American, British and New Dan landrace pigs[J]. Swine Production, 2002(4): 11-12. DOI:10.13257/j.cnki.21-1104/s.2002.04.003 |
| [35] |
THOMAS M, LANGLEY B, BERRY C, SHARMA M, KIRK S, BASS J, KAMBADUR R. Myostatin, a negative regulator of muscle growth, functions by inhibiting myoblast proliferation[J]. Journal of Biological Chemistry, 2000, 275(51): 40235-40243. DOI:10.1074/jbc.M004356200 |
| [36] |
LI R, ZENG W, MA M, WEI Z, LIU H, LIU X, WANG M, SHI X, ZENG J, YANG L, MO D, LIU X, CHEN Y, HE Z. Precise editing of myostatin signal peptide by CRISPR/Cas9 increases the muscle mass of Liang Guang Small Spotted pigs[J]. Transgenic Research, 2020, 29(1): 149-163. DOI:10.1007/s11248-020-00188-w |
| [37] |
ZHU X X, ZHAN Q M, WEI Y Y, YAN A F, FENG J, LIU L, LU S S, TANG D S. CRISPR/Cas9-mediated MSTN disruption accelerates the growth of Chinese Bama pigs[J]. Reproduction in Domestic Animals, 2020, 55(10): 1314-1327. DOI:10.1111/rda.13775 |
| [38] |
LI W, LI R, WEI Y, MENG X, WANG B, ZHANG Z, WU W, LIU H. Effect of MSTN mutation on growth and carcass performance in Duroc×Meishan hybrid population[J]. Animals (Basel), 2020, 10(6): 932. DOI:10.3390/ani10060932 |
| [39] |
MARKLJUNG E, JIANG L, JAFFE J D, MIKKELSEN T S, WALLERMAN O, LARHAMMAR M, ZHANG X L, WANG L, SAENZ-VASH V, GNIRKE A, LINDROTH A M, BARRES R, YAN J, STROMBERG S, DE S, PONTEN F, LANDER E S, CARR S A, ZIERATH J R, KULLANDER K, WADELIUS C, LINDBLAD-TOH K, ANDERSSON G, HJALM G, ANDERSSON L. ZBED6, a novel transcription factor derived from a domesticated DNA Transposon regulates IGF2 expression and muscle growth[J]. PLoS Biology, 2009, 7(12): e1000256. DOI:10.1371/journal.pbio.1000256 |
| [40] |
LIU X, LIU H, WANG M, LI R, ZENG J, MO D, CONG P, LIU X, CHEN Y, HE Z. Disruption of the ZBED6 binding site in intron 3 of IGF2 by CRISPR/Cas9 leads to enhanced muscle development in Liang Guang Small Spotted pigs[J]. Transgenic Res, 2019, 28(1): 141-50. DOI:10.1007/s11248-018-0107-9 |
| [41] |
XIANG G H, REN J L, HAI T, FU R, YU D W, WANG J, LI W, WANG H Y, ZHOU Q. Editing porcine IGF2 regulatory element improved meat production in Chinese Bama pigs[J]. Cellular and Molecular Life Sciences, 2018, 75(24): 4619-4628. DOI:10.1007/s00018-018-2917-6 |
| [42] |
WANG D D, PAN D K, XIE B C, WANG S N, XING X Y, LIU X X, MA Y H, ANDERSSON L, WU J W, JIANG L. Porcine ZBED6 regulates growth of skeletal muscle and internal organs via multiple targets[J]. PLoS Genetics, 2021, 17(10): e1009862. DOI:10.1371/journal.pgen.1009862 |
| [43] |
PEJSAK Z, STADEJEK T, MARKOWSKA-DANIEL I. Clinical signs and economic losses caused by porcine reproductive and respiratory syndrome virus in a large breeding farm[J]. Veterinary Microbiology, 1997, 55(1/4): 317-322. DOI:10.1016/s0378-1135(96)01326-0 |
| [44] |
WHITWORTH K M, ROWLAND R R, EWEN C L, TRIBLE B R, KERRIGAN M A, CINO-OZUNA A G, SAMUEL M S, LIGHTNER J E, MCLAREN D G, MILEHAM A J, WELLS K D, PRATHER R S. Geneedited pigs are protected from porcine reproductive and respiratory syndrome virus[J]. Nature Biotechnology, 2016, 34(1): 20-22. DOI:10.1038/nbt.3434 |
| [45] |
WELLS K D, BARDOT R, WHITWORTH K M, TRIBLE B R, FANG Y, MILEHAM A, KERRIGAN M A, SAMUEL M S, PRATHER R S, ROWLAND R R R. Replacement of porcine CD163 scavenger receptor cysteine-rich domain 5 with a CD163-Like homolog confers resistance of pigs to genotype 1 but not genotype 2 porcine reproductive and respiratory syndrome virus[J]. Journal of Virology, 2017, 91(2): e01521. DOI:10.1128/JVI.01521-16 |
| [46] |
GUO C, WANG M, ZHU Z, HE S, LIU H, LIU X, SHI X, TANG T, YU P, ZENG J, YANG L, CAO Y, CHEN Y, LIU X, HE Z. Highly efficient generation of pigs harboring a partial deletion of the CD163 SRCR5 domain, which are fully resistant to porcine reproductive and respiratory syndrome virus 2 infection[J]. Frontiers in Immunology, 2019, 10: 1846. DOI:10.3389/fimmu.2019.01846 |
| [47] |
ZHANG Q, HU R, TANG X, WU C, HE Q, ZHAO Z, CHEN H, WU B. Occurrence and investigation of enteric viral infections in pigs with diarrhea in China[J]. Archives of Virology, 2013, 158(8): 1631-1636. DOI:10.1007/s00705-013-1659-x |
| [48] |
HANSEN B D L. The coronavirus transmissible gastroenteritis virus causes infection after receptor-mediated endocytosis and aciddependent fusion with an intracellular compartment[J]. Journal of Virology, 1998, 72(1): 527-534. DOI:10.1128/JVI.72.1.527-534.1998 |
| [49] |
WHITWORTH K M, ROWLAND R R R, PETROVAN V, SHEAHAN M, CINO-OZUNA A G, FANG Y, HESSE R, MILEHAM A, SAMUEL M S, WELLS K D, PRATHER R S. Resistance to coronavirus infection in amino peptidase N-deficient pigs[J]. Transgenic Research, 2019, 28(1): 21-32. DOI:10.1007/s11248-018-0100-3 |
| [50] |
XU K, ZHOU Y R, MU Y L, LIU Z G, HOU S H, XIONG Y J, FANG L R, GE C L, WEI Y H, ZHANG X L, XU C J, CHE J J, FAN Z Y, XIANG G M, GUO J K, SHANG H T, LI H, XIAO S B, LI J L, LI K. CD163 and pAPN double-knockout pigs are resistant to PRRSV and TGEV and exhibit decreased susceptibility to PDCoV while maintaining normal production performance[J]. Elife, 2020(9): e57132. DOI:10.7554/eLife.57132 |
| [51] |
WATERS P D, WALLIS M C, MARSHALL GRAVES J A. Mammalian sex——Origin and evolution of the Y chromosome and SRY[J]. Seminars in Cell & Developmental Biology, 2007, 18(3): 389-400. DOI:10.1016/j.semcdb.2007.02.007 |
| [52] |
PARMA P, RADI O. Molecular mechanisms of sexual development[J]. Sexual Development, 2012, 6(1-3): 7-17. DOI:10.1159/000332209 |
| [53] |
KURTZ S, LUCAS-HAHN A, SCHLEGELBERGER B, GOHRING G, NIEMANN H, METTENLEITER T C, PETERSEN B. Knockout of the HMG domain of the porcine SRY gene causes sex reversal in geneedited pigs[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(2): e2008743118. DOI:10.1073/pnas.2008743118 |
| [54] |
SONG R G, WANG Y, ZHENG Q T, YAO J, CAO C W, WANG Y F, ZHAO J G. One-step base editing in multiple genes by direct embryo injection for pig trait improvement[J]. Science China-Life Sciences, 2022, 65(4): 739-752. DOI:10.1007/s11427-021-2013-8 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49