文章信息
基金项目
- 国家自然科学基金(32001526);广东省基础与应用基础研究基金(2020A1515110067)
作者简介
- 李文龙(1996—),男,在读硕士生,研究方向为超级稻重要农艺性状的定向改良,E-mail:3310799522@qq.com; 刘志霞, 硕士, 副研究员, 现任广东省农业科学院水稻研究所超级稻育种研究室主任, 主要从事超级稻、优质稻新品种培育及水稻育种应用基础研究。作为主要参加人先后参加国家自然科学基金、国家科技支撑计划和重点研发课题、广东省重点领域研发项目6项, 主持省、市级科技项目8项; 主持育成审定水稻新品种7个, 作为主要参加人参与育成新品种12个; 在国内外学术期刊发表论文15篇; 2020年获广东省科技进步一等奖1项、2021年获广东省农业技术推广奖一等奖1项; 作为广东省粮食绿色高产创建专家组成员、水稻生产技术服务专家组成员, 长期深入水稻生产一线开展生产技术指导、新品种新技术示范推广等工作.
通讯作者
- 刘志霞(1981—),女,硕士,副研究员,研究方向为超级稻育种,E-mail:446594509@qq.com.
文章历史
- 收稿日期:2022-07-25
2. 华南农业大学农学院, 广东 广州 510642
2. College of Agriculture, South China Agricultural University, Guangzhou 510642, China
水稻是功能基因组学研究的模式作物,同时也作为世界上重要的粮食作物养育了全球超过半数的人口[1]。我国是目前世界上最大的稻米生产国和消费国,约60% 的人口以稻米为主食[2]。水稻生产是关系粮食安全、人民生活福祉和国家长治久安的头等大事。中国稻作文化源远流长,是水稻种植历史最为悠久的国家,也是水稻种质资源富国和稻作科技强国[2],先后引领了以“矮化育种”和“杂种优势利用”为基础的两次水稻“绿色革命”[3]。经过一代又一代稻作科学家的接续努力,水稻育种技术发展迅速,先后经历了纯系育种、杂交育种、诱变育种和分子育种等各个阶段,水稻产量记录不断刷新[2]。目前,我国水稻育种已经进入以籼粳亚种间杂种优势利用为基础的“5G时代”[4-5],将继续引领世界稻作发展。
随着经济的发展,人民生活水平不断提高,对稻米的需求逐步从“吃得饱”到“吃得好”转变,对水稻的产量、品质和抗性提出了更高的要求,高产优质多抗新品种的培育已经成为水稻育种追求的主要目标。稻米需求的升级为水稻育种技术带来了新的要求和挑战,以往的遗传改良方法如杂交育种、回交育种、诱变育种、分子标记辅助选择育种和转基因育种等,有的具有明显缺点或局限,有的面临一些实际技术问题无法大范围推广使用,已不利于水稻品种的快速迭代升级改良:杂交育种、回交育种具有后代分离大、难以快速稳定、育种周期较长、盲目性大等缺点;诱变育种具有变异方向不稳定不可控、有益突变概率低、精准性不高等缺点;分子标记辅助选择育种虽然在一定程度上解决了育种精准性的问题,但也同样面临连锁累赘无法打破、耗时费力等诸多问题;转基因育种可以通过将外源功能基因整合到受体作物的染色体上表达,达到精准定向改良作物性状的目的,但是由于其会引入外源DNA片段,引发对转基因食品安全方面的担忧而无法全面释放应用。相较于传统育种方法,新兴的CRISPR/Cas9基因编辑技术能通过预先设定的引导序列,精准地对内源目标靶基因位点进行定点突变(插入或缺失)或精准编辑(碱基替换),达到敲除或者改变目标基因功能的目的。虽然CRISPR/Cas9基因编辑技术依靠转基因的方式完成,但是由于转基因TDNA插入位点往往与编辑位点位置不同,可以在编辑后代中通过遗传分离得到完全没有转基因元件的编辑突变体。随着CRISPR/Cas9基因编辑技术不断改进和发展,编辑技术日渐稳定成熟,目前已经在动植物的功能基因研究、种质资源创制创新、优良新品种培育和定向改良中发挥重要作用。在水稻品种改良中,CRISPR-Cas9系统能快速定向稳定目标性状、缩短育种周期,展现出强大的应用潜力,有望成为未来水稻育种的重要辅助工具。因此,在当前形势下,加强对水稻基因编辑技术的研究具有十分重要的现实意义。本文主要综述了近年来CRISPR-Cas9基因编辑技术在水稻性状定向改良上的研究进展,并对该技术的未来发展前景进行探讨和展望,以期为水稻育种方法的革新与发展提供新的思路和参考。
1 CRISPR/Cas9基因编辑技术规律间隔成簇短回文重复序列相关系统(Clustered Regularly Interspaced Short Palindromic Repeats associated system,CRISPR/Cas)是细菌和古细菌体内长期演化过程中形成的一种适应性免疫防御机制,可以利用双RNA引导DNA内切酶在目标DNA中引入位点特异性双链断裂来抵御外源DNA的侵染[6]。CRISPR/Cas是继锌指核酸酶(ZFN)、转录激活样效应因子核酸酶(TALEN)等基因编辑技术之后的第三代基因编辑系统,不同于ZFN和TALEN系统的DNA识别是依赖蛋白质来完成,CRISPR/Cas编辑系统的目标靶位点识别是利用一段短RNA片段实现的。理论上Cas蛋白可以在RNA的引导下对任何一段特异的基因组DNA序列进行精准操纵,用来阐释基因功能、修复有害突变、抑制被激活的致病基因或者激活病症的抑制基因;研究者可以通过一次实验同时靶向编辑多个位点验证多个基因的功能[6]。该技术目前已经广泛应用于水稻、小麦、玉米、人类、斑马鱼、猪等动植物的基因组研究,具有设计简单、操作简便、成本低廉、精准高效等优点[6-10]。
CRISPR/Cas系统主要分为3种类型(图 1A),Ⅱ型主要在细菌基因组中被发现,Ⅲ型则主要存在于古细菌,而Ⅰ型则在细菌和古细菌中均有分布[11]。3类系统均含有靶向DNA区域、crRNA区域,I型和II型依赖于前间区序列临近基序(Protospacer Adjacent Motif,PAM),而Ⅲ型则不依赖PAM序列[11]。CRISPR/Cas9基因编辑系统属于Ⅱ型系统,是广泛应用于基因编辑、转录调控、表观遗传修饰和基因组成像等领域的一个强大基因编辑平台[6],也是目前为止研究最多最透彻、操作最简单方便的基因编辑系统。在Ⅰ型、Ⅲ型CRISPR/Cas系统中,成熟的crRNA引导几个核酸酶Cas组成的大型蛋白复合体共同发挥作用才能顺利剪切外源DNA,而Ⅱ型CRISPR/Cas系统仅需要单一的DNA内切酶Cas9蛋白配合crRNA、tracrRNA即可识别和剪切目标靶DNA,不依赖于多蛋白复合体[6, 12]。为方便使用,目前常用的CRISPR/Cas9系统经过简化将crRNA和tracrRNA合并成一个嵌合的sgRNA转录本,并完全保留了Cas9介导的序列特异性剪切功能(图 1B),可以根据实验需求设计改造sgRNA,引导Cas9蛋白到基因组上特定区段,并在PAM序列的上游剪切DNA双链形成平末端裂口[6, 11, 13]。当目的基因双链断裂后,裂口会被细胞中存在的DNA修复机制所修复。修复机制可分为同源修复(HR)和非同源末端连接(NHEJ),NHEJ修复过程中会在剪切位点产生小片段的插入或缺失引发基因编码改变或者提前终止,导致基因沉默;HR修复会恢复野生型序列;而在有供体DNA存在的情况下,断裂位置会通过HR修复方式进行同源修复精准地引起DNA片段插入或替换,达到基因敲入的目的(图 1B)[6, 11]。
2 CRISPR/Cas9基因编辑技术在水稻定向改良中的应用
性状改良是新品种不断升级迭代的源动力。传统的水稻品种改良通常采用杂交方式将各种优异等位基因聚合于受体亲本,周期漫长、费时耗力且无法打破连锁累赘,在引入优良基因的同时可能会带入诸多不利基因。如何实现目标基因的精准快速改良,克服传统育种方式的种种弊端是育种家面临的一个亟需解决的问题。作为一种新兴的基因编辑技术,由细菌免疫系统发展而来的CRISPR/Cas9仅需改造引导RNA序列即可实现基因组靶位点的定点编辑[11]。在基因编辑过程中,由于CRISPR/Cas9基因编辑植株的转基因标签插入位点往往与编辑位点不在同一位置,易于在后代中获取没有外源基因整合的编辑材料,避免了传统转基因方法导致的食品安全担忧问题。因此,CRISPR/Cas9基因编辑技术在水稻定向改良中具有巨大的应用潜力。目前,众多研究者已经利用CRISPR/Cas9基因编辑技术针对水稻中的多个重要靶标展开定向改良研究工作,取得一系列研究成果(表 1)。

|
2.1 水稻抗病性的定向改良
稻瘟病是水稻生产中的主要病害之一,全球每年因稻瘟病导致的产量损失约占水稻总产量10%~30%,稻瘟病爆发时,轻则引起水稻大幅减产,重则导致绝收[14-15]。因此,改良水稻稻瘟病抗性对水稻稳产增产具有重要意义。目前,水稻中已鉴定出超过100个稻瘟病抗性位点,其中超过30个稻瘟病抗性基因已被克隆[14, 16]。在抗性基因克隆和功能解析的基础上,通过CRISPR/Cas9基因编辑技术定点修饰抗性相关基因,可以有针对性地定向改良水稻稻瘟病抗性。Zhou等[16]在籼型光温敏不育系LK638S背景下采用CRISPR/Cas9技术对Bsr-d1、Pi21和ERF922等3个已知的稻瘟病抗性相关基因进行基因编辑研究,发现单基因突变体和3基因突变体的稻瘟病抗性均显著高于野生型受体品种,同时突变体的产量性状基本不受影响,为水稻抗病性的快速定向改良提供了范例。在粳稻Kuiku131中敲除水稻稻瘟病抗性的负调控因子OsERF922也得出类似实验结果,突变体在不影响穗数、株高、千粒质量等重要农艺性状的前提下,显著提高了稻瘟病抗性[17]。在高感稻瘟病的优质水稻品种大粒香中,利用CRISPR/Cas9基因编辑技术定点编辑稻瘟病相关基因pid3,后代无转基因标签的纯合突变体材料的稻瘟病抗性有效提高,因病损失率显著降低,达到了稻瘟病抗性定向改良的目标[15]。编码区内21 bp和48 bp的两处缺失导致pi21功能丧失,进而赋予水稻稻瘟病广谱抗性,房耀宇等[18]利用CRISPR/Cas9技术在Pi21基因的外显子区域设计2个靶标位点对该基因进行敲除研究,结果发现在敲除Pi21的突变体中该基因发生移码突变,表达量较野生型品种明显降低,稻瘟病抗性则显著提高。OsVQ25通过与E3泛素连接酶OsPUB73和转录因子OsWRKY53互作,在平衡水稻的广谱抗病性和生长发育中发挥重要功能[19]。VQ25通过抑制OsWRKY53的功能负向调控水稻抗病性,在水稻中敲除VQ25,敲除突变体的稻瘟病抗性得到显著提高,而其他农艺性状基本不受影响,表明CRISPR/Cas9技术改良作物性状可以做到精准定向、有效避免连锁累赘。
白叶枯病是一种由水稻黄单胞杆菌(Xanthomonas oryzae pv.Oryzae,Xoo)引起的毁灭性水稻病害,白叶枯病频繁爆发严重威胁水稻生产。水稻OsSWEET14基因是已知大部分Xoo菌株的易感靶标,能够被Xoo中的转录激活样效应子(TAL)通过结合其启动子区域的效应子结合元件(EBE)激活表达,从而负向调控水稻的白叶枯抗性,不同研究者分别利用CRISPR/Cas9技术定点编辑OsSWEET14的基因编码区和启动子序列的EBE元件,均获得白叶枯病抗性显著提高的水稻材料[20-21],并且可保持主要农艺性状基本不改变[20]。为改良IR24的白叶枯病抗性,郝巍等[22] 利用CRISPR/Cas9定点编辑了与白叶枯病抗性相关的Pong2-1、Pong11-1两个基因位点,发现与野生型IR24相比,突变体pong2-1和pong11-1的主要农艺性状无显著变化,但对白叶枯病的抗性显著提高,由此创制出新的白叶枯病抗性材料。Xig1(Xoo-induced gene 1)是一个受水稻白叶枯病菌诱导表达的基因,与感白叶枯病相关。在籼稻IR24背景下敲除Xig1的突变体对白叶枯病菌敏感性降低,接种14 d后突变体的病斑长度较野生型降低53%~65%,白叶枯病抗性提高且突变株系在主要农艺性状上与野生型IR24无显著差异[23]。武广珩等[24]通过CRISPR/Cas9技术敲除水稻的免疫防卫相关基因OsEDR1,白叶枯病原菌Xoo PXO99的接种侵染试验结果显示,突变体水稻植株病斑长度相较于野生型日本晴减少约50%,白叶枯病抗性明显增强。除修饰单个基因位点获得某一特定抗性外,利用CRISPR/Cas9技术编辑1个抗性基因也可以实现多种抗病性状协同改良的目标。研究发现,Pi21或ERF922的单基因敲除突变体除可显著提高水稻稻瘟病抗性外,白叶枯病的抗性也得到显著增强[16];在水稻中敲除OsVQ25能同时协同提升稻瘟病和白叶枯病的抗性[19]。
稻瘟病和白叶枯病是水稻生产中面临的主要病害,爆发严重时会造成水稻大幅减产甚至绝收[14, 20-21]。长期通过喷洒农药防控水稻病害,会造成环境污染、药害、推高水稻生产成本等一系列问题,因此,通过培育抗病新品种来改良水稻的抗病性,是解决水稻病害问题最直接、最有效、最经济的方法。通过杂交育种方法改良水稻抗病性周期过长,加上病菌进化变异速度快,往往经过多年杂交、回交导入的抗性基因刚刚稳定就已失去对新的病菌小种的抗性作用,严重制约抗病新品种的培育。前人研究表明,利用CRISPR/Cas9基因编辑技术定向改良抗病相关基因,如pid3[15]、Bsr-d1[16]、ERF922[16-17]、Pi21[16, 18]、VQ25[19]、OsSWEET14[20-21]、Pong2-1[22]、Pong11-1[22]、Xig1[23]、OsEDR1[24]等,能在基本不改变其他主要农艺性状的前提下,在短期内(两个水稻生长世代)获得纯合稳定、不含转基因标签的定向改良水稻新材料。CRISPR/Cas9基因编辑技术大大提高了育种效率、缩短了育种年限,为未来水稻抗病性的定向改良提供了参考和范例。
2.2 水稻品质的定向改良稻米品质是复杂的农艺性状,包括外观品质、加工品质、蒸煮食味品质和营养品质等,是水稻商品价值的重要衡量因素。香气被认为是优质稻米的重要指标,水稻OsBadh2基因功能的缺失导致2-乙酰-1-吡咯啉(2-AP)的积累是水稻香气形成的主要原因。研究者在不同的遗传背景下运用CRISPR/Cas9基因编辑技术对OsBadh2进行敲除研究,发现敲除突变体材料的2-AP含量均能显著提高,可以产生强烈香气,稻米食味性状得到快速精准改良[18, 25-26]。直链淀粉含量是影响水稻口感的重要因素,黄李春等[27]在粳稻日本晴背景下,利用CRISPR/Cas9技术在Wx基因编码区的C端第12、13外显子处设计2个靶标位点进行定点敲除研究,获得8种无转基因标签的纯合突变材料。与野生型相比,8种突变材料的农艺性状无显著变化,但是直链淀粉含量从16.79% 下降至3.69%~4.44%,得到直链淀粉含量极低的糯稻新种质。除编辑Wx基因的编码区,研究者还利用CRISPR/Cas9技术编辑Wx基因启动子上的关键顺式作用元件,通过调节Wx基因的表达量,进而达到改良稻米直链淀粉含量的目的[28-30]。毛兴学等[28]在籼稻保持系吉丰B背景下,定向编辑Wx基因的编码区上游序列,得到两份低直链淀粉含量的水稻新种质。Huang等[29]通过分析Wx基因启动子序列上的顺式作用元件,并选择7个靶标作为编辑位点,通过下调Wx基因的表达量调节稻米直链淀粉含量,获得6个新的Wx等位基因类型。利用CRISPR/Cas9技术编辑Wx基因的启动子序列,Zeng等[30]也在高直链淀粉含量的保持系天丰B背景下获得了直链淀粉含量不同程度降低的一系列新材料。Sun等[31]运用CRISPR/Cas9基因编辑技术对水稻淀粉分支酶基因SBEI、SBEIIb进行敲除研究,结果表明sbeI突变体与野生型相比无明显差异,而sbeIIb突变体的直链淀粉、抗性淀粉含量分别提高至25%、9.8%,创制了高直链淀粉、高抗性淀粉含量新种质。在外观品质方面,为获得优质长粒水稻,徐善斌等[32]利用CRISPR/Cas9技术将水稻的GS3和GS9基因位点进行编辑,高效地将圆粒形粳稻龙粳11改造成长粒。Cheng等[33]在粳稻日本晴背景下敲除粒形基因GS3和GL3.1,发现GS3敲除突变体呈现细长粒,同时垩白率降低;而GS3/GL3.1双敲除突变体表现为大粒和高垩白,表明敲除GS3和GL3.1可以快速改良粒形并影响稻米品质。
水稻品质是稻米商品价值的重要影响因素,是水稻不断升级改良的重要目标,同时也是调控最复杂的重要性状之一。水稻品质性状调控涉及方面广、参与基因多、调控网络复杂。传统方式对品质性状的遗传改良中,往往不同性状之间相互制约、相互影响,难以多方面协调改良。CRISPR/Cas9基因编辑技术不仅可以精准定点修饰目标基因、有效避免不利的连锁效应,还能同时编辑多个基因位点,协同改良多个性状,是复杂性状定向改良的理想工具。此外,控制水稻重要品质性状的基因往往只有一个到几个有限的等位基因类型,多样性有限,采用传统遗传改良方法创制新的等位基因类型十分困难,而CRISPR/Cas9基因编辑技术能通过编辑基因的启动子序列不同程度地上调或下调基因的表达量,从而产生一系列自然变异中不存在的新等位基因类型[28-30],以满足不同的需求。在未来水稻品质定向改良、新优异种质资源创制中具有广阔的应用前景。
2.3 水稻育性的定向改良在两系法杂交水稻中,光温敏核不育系(TGMS)是杂种优势利用的基础,TMS5是调控光温敏核不育的主效基因。梁敏敏等[34]利用CRISPR/Cas9基因编辑技术在早籼品种中早70中敲除TMS5,获得了败育稳定的温敏不育系材料。在粳稻武运粳7号背景下敲除TMS5,同样可以快速获得粳型温敏雄性不育系[35]。通过CRISPR/Cas9技术编辑TMS5基因,Zhou等[36]仅用1年时间即获得具有商业应用潜力的籼型和粳型温敏核不育系11个,大大提高了不育系的育种效率。水稻籼粳间杂种不育是亚种间杂种优势利用的主要障碍。在水稻籼粳杂种F1代,多个拷贝的Sc-i通过高表达等位抑制Sc-j导致花粉败育,利用CRISPR-Cas9敲除1~2个Sc-i基因拷贝可以有效克服籼粳杂交F1代的雄性不育[37],该研究为克服籼粳杂种不育、充分利用水稻亚种间杂种优势奠定了基础。杂种优势的固定是一系法水稻的前提和基础。Khanday等[38]研究表明,敲除REC8、PAIR1、OSD1等3个介导减数分裂的主要基因,同时异位表达BBM1能够调控水稻的无性繁殖而实现杂种优势固定。Wang等[39]通过使用CRISPR/Cas9系统在杂交水稻中同时编辑REC8、PAIR1、OSD1、MTL等4个内源基因,在杂种一代水稻中获得了克隆杂一代种子,有望实现杂种优势的固定,使一系法水稻的创制成为可能。
水稻育性是关乎产量的重要农艺性状。在杂交水稻中,不育系是杂种优势利用的基础,前人研究表明,利用CRISPR/Cas9基因编辑技术在不同的遗传背景下均可以快速创制光温敏核不育系[34-36],这将大大拓宽水稻杂种优势的利用范围。理论上通过利用CRISPR/Cas9基因编辑技术,未来我们可以将任何一个水稻材料快速定向改造成不育系应用于杂交水稻生产。籼粳杂种不育一直是水稻亚种间杂种优势利用的主要障碍,以往以籼粳架桥或寻找中性亲和基因的克服杂种不育策略,受到严重不育和资源的制约而困难重重,今后可以利用CRISPR/Cas9基因编辑技术敲除籼粳亚种间杂种不育相关位点,精准快速创制亲和性材料,将为充分利用水稻籼粳亚种间杂种优势扫清障碍。
2.4 水稻耐受非生物胁迫的定向改良除了改良上述农艺性状,CRISPR/Cas9基因编辑技术在水稻非生物胁迫相关性状改良中也展现了广阔的应用前景[40]。利用CRISPR/Cas9定向编辑水稻ALS基因,Sun等[41]成功获得不含转基因元件的抗除草剂水稻品系。Li等[42]使用CRISPR/Cas9编辑水稻EPSPS基因,在后代中获得具有除草剂草甘膦抗性的水稻。这些研究为水稻耐除草剂筛选和抗除草剂育种打下基础。研究者在水稻中编辑OsNAC041基因,结果发现突变体水稻对盐分敏感性发生变化,影响其耐盐性[43]。在籼稻品种MTU1010中敲除OsDST基因后,叶片增宽、气孔密度降低、叶片保水能力提高,水稻的抗旱性和耐盐性得到提高[44]。Chen等[45]利用CRISPR/Cas9敲除OsFTIP1基因,获得更加耐旱的水稻材料。高温是影响水稻生产的重要胁迫因素,Kan等[46]研究发现敲除水稻TT2基因能有效提高水稻的耐热性,同时发现敲除钙敏转录因子SCT1和SCT2同样可增强水稻耐热性,为耐高温水稻品种的定向改良提供了新思路。
随着全球气候变化加剧,极端天气(如超高温、极端低温)频发,加上盐渍化土壤增多,这为未来水稻品种耐受非生物胁迫提出了更高要求。CRISPR/Cas9基因编辑技术已经在水稻的耐盐性[40, 44]、除草剂抗性[41-42]、耐干旱[44-45]和耐高温[46]等性状改良中崭露头角,今后或将在更多非生物胁迫性状的定向改良中发挥更重要的作用。
2.5 水稻其他性状的定向改良抽穗期是水稻的重要农艺性状,抽穗期性状的定向改良对扩展水稻品种的区域适应性具有重要意义。Li等[47]通过设计特异靶点在7个主栽品种中定向编辑Hd2、Hd4、Hd5等3个抽穗期基因,结果显示敲除后代均能在不同程度上缩短抽穗期,达到生育期定向改良的目标。抗倒伏是保证水稻稳产的重要性状,在水稻中敲除OsRhoGDI2基因可以缩短水稻第Ⅱ、第Ⅲ茎节,显著降低株高,提高水稻的抗倒伏能力[48]。王新等[49]运用CRISPR/Cas9基因编辑技术将水稻的SD1基因敲除,突变体中GA水平降低,水稻株高降低,有效改良了优质香糯品种的抗倒性。水稻株型、穗粒数和穗型是产量相关的重要农艺性状,研究者通过编辑IPA、Gnla、DEP1等基因,定向改良了水稻株型、穗粒数和穗型等性状[50]。水稻开花时间是影响杂交水稻制种的主要因素,Wang等[51]通过敲除DFOT1基因将粳稻中花11在一天中的花时提前约2.5 h,成功改良了粳稻晚花性状,有望应用于籼粳杂交育种中以提高籼粳杂交制种的产量。随后,Xu等[52]报道了早花基因EMF1,缺失该基因能提高浆片的吸水能力,引起早花,无论在籼稻还是粳稻中敲除该基因均能提早花时2.0~2.5 h,这为不育系花时改良提供了新途径。为探索高油酸水稻种质资源的创制,任代胜等[53]利用CRISPR/Cas9基因编辑技术在粳稻日本晴中靶向敲除脂肪酸去饱和酶基因OsFAD2,敲除突变体籽粒中的油酸含量较野生型水稻提高25%~30%,为高油酸稻米育种和稻米油产业发展提供了材料基础。
CRISPR/Cas9作为新一代基因编辑技术,以其强大的编辑能力、极简便的可操作性和广阔的应用前景,逐步成为现代水稻育种过程中不可或缺的重要补充,其应用已经深入到水稻性状定向改良的各个方面(图 2),必将在水稻育种方式不断革新中发挥更加重要的作用。
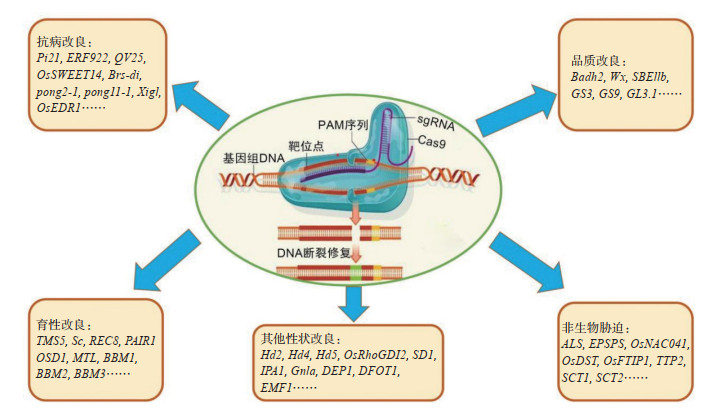
|
| 图 2 CRISPR/Cas9基因编辑技术在水稻定向改良中的应用 Fig. 2 Application of CRISPR/Cas9 gene editing technology in targeted improvement of rice |
3 展望
目前水稻育种已经进入新时代,对水稻品种的产量、品质、抗性等方面都提出了更高要求,以育种家经验为基础的传统育种方式已逐渐不能满足新时代的育种要求。CRISPR/Cas9技术以其在水稻性状定向改良中展现出来的无可比拟的精准性、可预见性和高效性,已然成为新时期水稻育种的重要辅助手段。此外,创制多样化的种质资源类型以满足不同人群的消费需求也是未来水稻育种的重要方向。传统遗传方法难以在水稻中创制新的等位基因类型,而通过修饰基因的调控区域,CRISPR/Cas9基因编辑技术能达到调节基因表达的目的,从而创制一些自然变异中不存在的新等位基因,这将在未来突破性、多样化水稻新品种的培育中具有广阔的应用前景。
目前,CRISPR/Cas9基因编辑技术对作物改良研究仍处于基础研究或技术储备阶段,真正应用到水稻生产实践中仍面临着不少挑战和问题。首先,国际上对基因编辑作物的监管普遍采用基于过程或基于产品的两种管理方法。其中,欧盟采用基于过程的监管方式,将基因编辑作物视作转基因作物监管;美国、加拿大和阿根廷等国家则是基于产品监管,将基因编辑作物视作非转基因作物监管[54]。截至目前,我国对这种经过转基因过程产生的且不带有转基因元件的基因编辑产物,尚未按照非转基因作物对待全面放开推广释放,这意味着基因编辑作物的大范围推广仍面临不确定性。其次,水稻重要性状的调控往往是涉及众多基因的调控网络,其中发挥重要功能的基因有些是正向调控,有些是负向调控。目前基于CRISPR/Cas9基因编辑技术的研究绝大部分是敲除或失活基因,对增强正向调控或功能获得性基因敲入的研究还非常少,这在很大程度上限制了CRISPR/Cas9基因编辑技术在实际育种中的应用。未来,根据需要对CRISPR/Cas9基因编辑平台与其他基因组改造技术进行深入交叉融合升级[9],进一步扩展CRISPR/Cas9基因编辑技术的应用范围,实现无转基因元件残存的基因增强和基因敲入功能,将大大扩宽该技术的育种应用。再次,目前主流应用的CRISPR/Cas9基因编辑技术,编辑靶位点设计和选择往往需要依赖PAM序列,这将严重限制敲除位点的灵活选择,难以实现任意位点的基因编辑,也将是CRISPR/Cas9基因编辑技术的主要限制因素。研究发现Ⅲ型CRISPR/Cas系统的基因编辑不需要依赖PAM序列[11],未来或将具有更加广阔的应用前景。最后,CRISPR/Cas9基因编辑技术在应用过程中会存在一定程度的脱靶现象,这将引起非目标区域的基因突变,导致编辑结果的不确定性或者偏差。以上这些因素都会影响CRISPR/Cas9基因编辑技术在实践中的大规模商业化应用。
CRISPR/Cas9基因编辑技术作为一种新的育种辅助手段,尽管还存在各种制约问题,但仍以其独特的精准改造基因能力吸引着越来越多的研究者对其开展深入研究。随着研究的不断推进和深入,上述制约问题或将迎刃而解。靶标任意选择、精准定点替换、基因敲入的实现将大大扩展CRISPR/Cas9基因编辑技术在育种中的适用范围,助推育种技术不断革新。
| [1] |
LI Y, XIAO J H, CHEN L L, HUANG X H, CHENG Z K, HAN B, ZHANG Q F, WU C Y. Rice functional genomics research: past decade and future[J]. Molecular Plant, 2018, 11(3): 359-380. DOI:10.1016/j.molp.2018.01.007 |
| [2] |
程式华. 中国水稻育种百年发展与展望[J]. 中国稻米, 2021, 27(4): 1-6. DOI:10.3969/j.issn.1006-8082.2021.04.001 CHEN S H. One-hundred years'development and prospect of rice breeding in China[J]. China Rice, 2021, 27(4): 1-6. DOI:10.3969/j.issn.1006-8082.2021.04.001 |
| [3] |
朱英国. 杂交水稻研究50年[J]. 科学通报, 2016, 61(35): 3740-3747. DOI:10.1360/N972016-01043 ZHU Y G. Fifty years of hybrid rice research in China[J]. Chinese Science Bulletin, 2016, 61(35): 3740-3747. DOI:10.1360/N972016-01043 |
| [4] |
ZHANG G Q. The next generation of rice: inter-subspecific indica-japonica hybrid rice[J]. Frontiers in Plant Science, 2022, 13: 857896. |
| [5] |
张桂权. 5G水稻的演变与发展[J]. 华南农业大学学报, 2019, 40(5): 211-216. DOI:10.7671/j.issn.1001-411X.201905075 ZHANG G Q. Evolution and development of five geneations of rice[J]. Journal of South China Agricultural University, 2019, 40(5): 211-216. DOI:10.7671/j.issn.1001-411X.201905075 |
| [6] |
JIANG F G, DOUDNA J A. CRISPR-Cas9 structures and mechanisms[J]. Annual Review of Biophysics, 2017, 46(1): 505-529. DOI:10.1146/annurev-biophys-062215-010822 |
| [7] |
HSU P D, LANDER E S, ZHANG F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278. DOI: 10.1016l.2014.05.010.
|
| [8] |
YOUNG S A, AITKEN R J, IKAWA M. Advantages of using the CRISPR/Cas9 system of genome editing to investigate male reproductive mechanisms using mouse models[J]. Asian Journal of Andrology, 2015, 17(4): 623-627. DOI:10.4103/1008-682X.153851 |
| [9] |
颜雯, 李海涛, 向华, 王晓虎. CRISPR-Cas基因组改造技术研究进展[J]. 广东农业科学, 2014, 41(2): 149-152. DOI:10.16768/j.issn.1004-874X.2014.02.002.j.issn.1004-874X.2014.02.002 YAN W, LI H T, XIANG H, WANG X H. Research progress on the genome modification technologies of CRISPR-Cas[J]. Guangdong Agricultural Sciences, 2014, 41(2): 149-152. DOI:10.16768/j.issn.1004-874X.2014.02.002.j.issn.1004-874X.2014.02.002 |
| [10] |
王敏, 黄翔, 石翾, 刘小凤, 曾检华, 刘小红, 陈瑶生, 何祖勇. 应用CRISPR/Cas9编辑广东小耳花猪MSTN基因[J]. 广东农业科学, 2017, 44(2): 141-148. DOI:10.16768/j.issn.1004-874X.2017.02.022 WANG M, HUANG X, SHI X, LIU X F, ZENG J H, LIU X H, CHEN Y S, HE Z Y. Editing MSTN gene of Guangdong Xiaoerhuapig by using CRISPR/Cas9[J]. Guangdong Agricultural Sciences, 2017, 44(2): 141-148. DOI:10.16768/j.issn.1004-874X.2017.02.022 |
| [11] |
RICHTER H, RANDAU L, PLAGENS A. Exploiting CRISPR/Cas: interference mechanisms and applications[J]. International Journal of Molecular Sciences, 2013, 14(7): 14518-14531. DOI:10.3390/ijms140714518 |
| [12] |
CHYLINSKI K, RHUN A L, CHARPENTIER E. The tracrRNA and Cas9 families of type Ⅱ CRISPR-Cas immunity systems[J]. RNA Biology, 2013, 10(5): 726-737. DOI:10.4161/rna.24321 |
| [13] |
MAKAROVA K S, KOONIN E V. Annotation and classification of CRISPR-Cas systems[M]. New York: Springer New York, 2015: 47-75. DOI:10.1007/978-1-4939-2687-94
|
| [14] |
SKAMNIOTI P, GUURR S J. Against the grain: safeguarding rice from rice blast disease[J]. Trends in Biotechnology, 2009, 27(3): 141-150. DOI:10.1016/j.tibtech.2008.12.002 |
| [15] |
吴娴, 李佳丽, 曾庆鸿, 张大双, 徐海峰, 姜雪, 宋莉, 彭强, 朱速松. 利用CRISPR/Cas9基因编辑技术改良大粒香稻瘟病抗性[J]. 种子, 2021, 40(7): 50-55. DOI:10.16590/j.cnki.1001-4705.2021.07.050 WU X, LI J, ZENG Q H, ZHANG D S, XU H F, JIANG X, SONG L, PENG Q, ZHU S S. Improvement of blast resistance of rice variety Dalixiang by CRISPR/CAS9 gene editing techology[J]. Seed, 2021, 40(7): 50-55. DOI:10.16590/j.cnki.1001-4705.2021.07.050 |
| [16] |
ZHOU Y B, XU S C, JIANG N, ZHAO X H, BAI Z A, LIU J L, YAO W, TANG Q Y, XIAO G, LYU C, WANG K, HU X C, TAN J J, YANG Y Z. Engineering of rice varieties with enhanced resistances to both blast and bacterial blight diseases via CRISPR/Cas9[J]. Plant Biotechnology Journal, 2022, 20(5): 876-885. DOI:10.1111/pbi.13766 |
| [17] |
WANG F J, WANG C L, LIU P Q, LEI C L, HAO W, GAO Y, LIU Y G, ZHAO K J. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J]. PLoS ONE, 2016, 11(4): e154027. DOI:10.1371/journal.pone.0154027 |
| [18] |
房耀宇, 杨金莲, 郭新颖, 彭好文, 覃宝祥, 刘芳, 刘丕庆, 李容柏. 利用CRISPR/Cas9技术编辑Pi21和Badh2基因改良水稻稻瘟病抗性和香味品质[J]. 分子植物育种, 2022, 1-23. FANG Y Y, YANG J L, GUO X X, PENG H W, QIN B X, LIU F, LIU P Q, LI R B. Editing Pi21 and Badh2 genes to improve rice blast resistance and fragrance quality by CRISPR/Cas9 system[J]. Molecular Plant Breeding, 2022, 1-23. |
| [19] |
HAO Z Y, TIAN J F, FANG H, FANG L, XU X, HE F, LI S Y, XIE W Y, DU Q, YOU X M, WANG D B, CHEN Q H, WANG R Y, ZUO S M, YUAN M, WANG G L, XIA L Q, NING Y S. A VQ-motif-containing protein fine-tunes rice immunity and growth by a hierarchical regulatory mechanism[J]. Cell Reports, 2022, 40(7): 111235. DOI:10.1016/j.celrep.2022.111235 |
| [20] |
ZENG X, LUO Y F, VU N T Q, SHEN S J, XIA K F, ZHANG M Y. CRISPR/Cas9-mediated mutation of OsSWEET14 in rice cv. Zhonghua11 confers resistance to Xanthomonas oryzae pv. oryzae without yield penalty[J]. BMC Plant Biology, 2020, 20(1): 311-313. DOI:10.1186/s12870-020-02524-y |
| [21] |
ZAFAR K, KHAN M Z, AMIN I, MUKHTAR Z, YASMIN S, ARIF M, EJAZ K, MANSOOR S. Precise CRISPR-Cas9 mediated genome editing in super basmati rice for resistance against bacterial blight by targeting the major susceptibility gene[J]. Frontiers in Plant Science, 2020, 11: 575. DOI:10.3389/fpls.2020.00575 |
| [22] |
郝巍, 纪志远, 郑凯丽, 孙宏达, 王福军, 唐永超, 张明伟, 赵开军, 王春连. 利用基因组编辑技术创制水稻白叶枯病抗性材料[J]. 植物遗传资源学报, 2018, 19(3): 523-530. DOI:10.13430/j.cnki.jpgr.2018.03.018 HAO W, JI Z Y, ZHENG K L, SUN H D, WANG F J, TANG Y C, ZHANG M W, ZHAO K J, WANG C L. Enhancing rice resistance to bacterial blight by genome editing[J]. Journal of Plant Genetic Resources, 2018, 19(3): 523-530. DOI:10.13430/j.cnki.jpgr.2018.03.018 |
| [23] |
郑凯丽, 纪志远, 郝巍, 唐永超, 韦叶娜, 胡运高, 赵开军, 王春连. 水稻白叶枯病感病相关基因Xig1的分子鉴定及抗病资源创制[J]. 作物学报, 2020, 46(9): 1332-1339. DOI:10.3724/SP.J.1006.2020.02013 ZHENG K L, JI Z Y, HAO W, TANG Y C, WEI Y N, HU Y G, ZHAO K J, WANG C L. Molecular identification of rice bacterial blight susceptible gene Xig1 and creation of sieseasee resistant resources[J]. Acta Agronomica Sinica, 2020, 46(9): 1332-1339. DOI:10.3724/SP.J.1006.2020.02013 |
| [24] |
武广珩, 傅仙玉. 利用CRISPR/Cas9技术编辑水稻负调控抗病基因OsEDR1及基因功能分析[J]. 应用与环境生物学报, 2019, 25(6): 1375-1380. DOI:10.19675/j.cnki.1006-687x.2019.05007 WU G H, FU X Y. Editing rice negative regulation resistance gene OsEDR1 by CRISPR/Cas9 and analysis of its function[J]. Chinese Journal of Applied and Environmental Biology, 2019, 25(6): 1375-1380. DOI:10.19675/j.cnki.1006-687x.2019.05007 |
| [25] |
杨平, 陈春莲, 姚晓云, 熊运华, 黄永萍, 胡标林, 尹建华. 利用基因编辑技术改良水稻直链淀粉含量与香味[J]. 分子植物育种, 2020, 18(3): 915-923. DOI:10.1371/journal.pone.0237018 YANG P, CHEN C L, YAO X Y, XIONG Y H, HUANG Y P, HU B L, YIN J H. Improvement of amylose and fragrance levels of rice by CRISPR/Cas9 system[J]. Molecular Plant Breeding, 2020, 18(3): 915-923. DOI:10.1371/journal.pone.0237018 |
| [26] |
ASHOKKUMAR S, JAGANATHAN D, RAMANATHAN V, RAHMAN H, PANISWAMY R, KAMBALE R, MUTHURAJAN R. Creation of novel alleles of fragrance gene OsBADH2 in rice through CRISPR/Cas9 mPalediated gene editing[J]. PLoS ONE, 2020, 15(8): e237018. DOI:10.1371/journal.pone.0237018 |
| [27] |
黄李春, 顾正文, 谈红艳, 赵微, 肖颖, 储睿, 范晓磊, 张昌泉, 李钱峰, 刘巧泉. CRISPR/Cas9技术编辑Wx基因创制新型糯稻种质[J]. 植物遗传资源学报, 2021, 22(3): 789-799. DOI:10.13430/j.cnki.jpgr.20210104003 HUANG L C, GU Z W, TAN H Y, ZHAO W, XIAO Y, CHU R, FAN X L, ZHANG C Q, LI Q F, LIU Q Q. Creating novel glutinous rice germplasms by editing Wx gene via CRISPR/Cas9 technology[J]. Journal of Plant Genetic Resources, 2021, 22(3): 789-799. DOI:10.13430/j.cnki.jpgr.20210104003 |
| [28] |
毛兴学, 郑晓钰, 孙炳蕊, 李晨, 陈文丰, 潘大建, 柳武革, 范芝兰, 王丰. 应用CRISPR/Cas9技术创制低直链淀粉含量水稻种质[J]. 植物遗传资源学报, 2022, 23(2): 583-591. DOI:10.13430/j.cnki.jpgr.20210926001 MAO X X, ZHENG X Y, SUN B R, LI C, CHEN W F, PAN D J, LIU W G, FAN Z L, WANG F. Creating novel rice germplasms with low amylose content by editing upstream sequence of Wx gene cooding region via CRISPR/Cas9 technology[J]. Journal of Plant Genetic Resources, 2022, 23(2): 583-591. DOI:10.13430/j.cnki.jpgr.20210926001 |
| [29] |
HUANG L C, LI Q F, ZHANG C Q, CHU R, GU Z W, TAN H Y, ZHAO D S, FAN X L, LIU Q Q. Creating novel Wx alleles with fine-tuned amylose levels and improved grain quality in rice by promoter editing using CRISPR/Cas9 system[J]. Plant Biotechnology Journal, 2020, 18(11): 2164-2166. DOI:10.1111/pbi.13391 |
| [30] |
ZENG D C, LIU T L, MA X L, WANG B, ZHENG Z Y, ZHANG Y L, XIE X R, YANG B W, ZHAO Z, ZHU Q L, LIU Y G. Quantitative regulation of Waxy expression by CRISPR/Cas9-based promoter and 5'UTR-intron editing improves grain quality in rice[J]. Plant Biotechnology Journal, 2020, 18(12): 2385-2387. DOI:10.1111/pbi.13427 |
| [31] |
SUN Y W, JIAO G A, LIU Z P, ZHANG X, LI J Y, GUO X P, DU W M, DU J L, FRANCIS F, ZHAO Y D, XIA L Q. Generation of high-amylose rice through CRISPR/Cas9-mediated targeted mutagenesis of starch branching enzymes[J]. Frontiers in Plant Science, 2017, 8: 298. DOI:10.3389/fpls.2017.00298 |
| [32] |
徐善斌, 郑洪亮, 刘利锋, 卜庆云, 李秀峰, 邹德堂. 利用CRISPR/Cas9技术高效创制长粒香型水稻[J]. 中国水稻科学, 2020, 34(5): 406-412. XU S B, ZHENG H L, LIU L F, BU Q Y, LI X F, ZOU D T. Improvement of grain shape and fragrance by using CRISPR/Cas9 system[J]. Chinese Journal of Rice Science, 2020, 34(5): 406-412. |
| [33] |
CHENG Y Y, ZHU A, XUE P, WEN X X, CHAO Y R, WANG B F, ZHANG Y, SHAH L, CHENG S H, CAO L Y, ZHANG Y X. Effects of GS3 and GL3.1 for gran size editing by CRISPR/Cas9 in rice[J]. Rice Science, 2020, 27(5): 405-413. DOI:10.1016/j.rsci.2019.12.010 |
| [34] |
梁敏敏, 张华丽, 陈俊宇, 戴冬青, 杜成兴, 王惠梅, 马良勇. 利用CRISPR/Cas9技术创制抗稻瘟病香型早籼温敏核不育系[J]. 中国水稻科学, 2022, 36(3): 248-258. DOI:10.16819/j.1001-7216.2022.211007 LIANG M M, ZHANG H L, CHEN J Y, DAI D Q, DU C X, WANG H M, MA L Y. Developing fragrant early indica TGMS Line with blast resistance by using CRISPR/Cas9 technoloy[J]. Chinese Journal of Rice Science, 2022, 36(3): 248-258. DOI:10.16819/j.1001-7216.2022.211007 |
| [35] |
杜茜, 费云燕, 王芳权, 许扬, 王军, 李文奇, 赵凌, 陈智慧, 梁国华, 周勇, 杨杰. 敲除TMS5基因获得温敏不育粳稻新材料[J]. 中国水稻科学, 2019, 33(5): 429-435. DOI:10.16819/j.1001-7216.2019.9035 DU Q, FEI Y Y, WANG F Q, XU Y, WANG J, LI W Q, ZHAO L, CHEN Z H, LIANG G H, ZHOU Y, YANG J. Thermo-sensitive male sterile line created by editing TMS5 gene in japonica rice[J]. Chinese Journal of Rice Science, 2019, 33(5): 429-435. DOI:10.16819/j.1001-7216.2019.9035 |
| [36] |
ZHOU H, HE M, LI J, CHEN L, HUANG Z F, ZHENG S N, ZHU L, NI E, JIANG D G, ZHAO B R, ZHUANG C X. Development of commercial thermo-sensitive genic male sterile rice accelerates hybrid rice breeding using the CRISPR/Cas9-mediated TMS5 editing system[J]. Scientific Reports, 2016, 6: 37395. DOI:10.1038/srep37395 |
| [37] |
SHEN R X, WANG L, LIU X P, WU J, JIN W W, ZHAO X C, XIE X R, ZHU Q L, TANG H W, LI Q, CHEN L T, LIU Y G. Genomic structural variation-mediated allelic suppression causes hybrid male sterility in rice[J]. Nature Communications, 2017, 8(1): 1310. DOI:10.1038/s41467-017-01400-y |
| [38] |
KHANDAY I, SKINNER D, YANG B, MERCIER R, SUNDARESAN V. A male-expressed rice embryogenic trigger redirected for asexual propagation through seeds[J]. Nature, 2019, 565(7737): 91-95. DOI:10.1038/s41586-018-0785-8 |
| [39] |
WANG C, LIU Q, SHEN Y, HUA Y F, WANG J J, LIN J R, WU M G, SUN T T, CHENG Z K, MERCIER R, WANG K J. Clonal seeds from hybrid rice by simultaneous genome engineering of meiosis and fertilization genes[J]. Nature Biotechnology, 2019, 37(3): 283-286. DOI:10.1038/s41587-018-0003-0 |
| [40] |
FARHAT S, JAIN N, SINGH N, SREEVATHSA R, DASH P K, RAI R, YADAV S, KUMAR P, SARKAR A K, JAIN A, SINGH N K, RAI V. CRISPR-Cas9 directed genome engineering for enhancing salt stress tolerance in rice[J]. Seminars in Cell and Developmental Biology, 2019, 96: 91-99. DOI:10.1016/j.semcdb.2019.05.003 |
| [41] |
SUN Y W, ZHANG X, WU C, HE Y B, MA Y Z, HOU H, GUO X P, DU W Mu W, ZHAO Y D, XIA L Q. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of aetolactate synthase[J]. Molecular Plant, 2016, 9(4): 628-631. DOI:10.1016/j.molp.2016.01.001 |
| [42] |
LI J, MENG X B, ZONG Y, CHEN K L, ZHANG H W, LIU J Y, LI J Y, GAO C X. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9[J]. Nature Plants, 2016, 2: 16139. DOI:10.1038/nplants.2016.139 |
| [43] |
WANG B, ZHONG H H, ZHANG H H, WANG X, LIU B L, YANG L J, HAN X Y, YU D H, ZHENG X L, WANG C G, SONG W Q, CHEN C B, ZHANG Y. Targeted mutagenesis of NAC transcription factor gene, OsNAC041, leading to salt sensitivity in rice[J]. Rice Science, 2019, 26(2): 98-108. DOI:10.1016/j.rsci.2018.12.005 |
| [44] |
SANTOSH K V V, VERMA R K, YADAV S K, YADAV P, WATTS A, RAO M V, CHINNUSAMY V. CRISPR-Cas9 mediated genome editing of drought and salt tolerance (OsDST) gene in indica mega rice cultivar MTU1010[J]. Physiology and Molecular Biology of Plants, 2020, 26(6): 1099-1110. DOI:10.1007/s12298-020-00819-w |
| [45] |
CHEN Y, SHEN J, ZHANG L, QI H Y, YANG L J, WANG H Y, WANG J X, WANG Y X, DU H, TAO Z, ZHAO T, DENG P C, SHU Q Y, QIAN Q, YU H, SONG S Y. Nuclear translocation of OsMFT1 that is impeded by OsFTIP1 promotes drought tolerance in rice[J]. Molecular Plant, 2021, 14(8): 1297-1311. DOI:10.1016/j.molp.2021.05.001 |
| [46] |
KAN Y, MU X R, ZHANG H, GAO J, SHAN J X, YE W W, LIN H X. TT2 controls rice thermotolerance through SCT1-dependent alteration of wax biosynthesis[J]. Nature Plants, 2022, 8(1): 53-67. DOI:10.1038/s41477-021-01039-0 |
| [47] |
LI X F, ZHOU W J, REN Y K, TIAN X J, LV T X, WANG Z Y, FANG J, CHU C C, YANG J, BU Q Y. High-efficiency breeding of early-maturing rice cultivars via CRISPR/Cas9-mediated genome editing[J]. Journal of Genetics Genomics, 2017, 44(3): 175-178. DOI:10.1016/j.jgg.2017.02.001 |
| [48] |
王凯婕, 安文静, 刘亚菲, 刘迪, 冯连杰, 王俊杰, 黄俊骏, 刘肖飞, 梁卫红. CRISPR/Cas9技术编辑OsRhoGDI2基因导致水稻半矮化[J]. 生物工程学报, 2020, 36(4): 707-715. DOI:10.13345/j.cjb.190333 WANG K J, AN W J, LIU Y F, LIU D, FENG L J, WANG J J, HUANG J J, LIU X F, LIANG W H. Disuption of OsRhoGDI2 by CRISPR/CAS9 technology leads to semi-dwarf in rice[J]. Chinese Journal of Biotechnology, 2020, 36(4): 707-715. DOI:10.13345/j.cjb.190333 |
| [49] |
王新, 韩悦, 冯璇, GUL N, 罗亮, 刘芳, 覃宝祥, 刘耀光, 李容柏. 应用CRISPR-Cas9基因编辑技术改良传统优质糯稻品种[J]. 分子植物育种, 2019, 17(19): 6332-6342. DOI:10.13271/j.mpb.017.006332 WANG X, HAN Y, FENG X, GUL N, LUO L, LIU F, QIN B X, LIU Y G, LI R B. Improvement of a traditional high-quality glutinous rice variety by CRISPR-Cas9 gene editing system[J]. Molecular Plant Breeding, 2019, 17(19): 6332-6342. DOI:10.13271/j.mpb.017.006332 |
| [50] |
LI M, LI X X, ZHOU Z J, WU P Z, FANG M C, PAN X P, LIN Q P, LUO W B, WU G J, LI H Q. Reassessment of the four yield-related genes Gn1a, DEP1, GS3, and IPA1 in rice using a CRISPR/Cas9 System[J]. Frontiers in Plant Science, 2016, 7: 377. DOI:10.3389/fpls.2016.00377 |
| [51] |
WANG M M, ZHU X P, PENG G Q, LIU M L, ZHANG S Q, CHEN M H, LIAO S T, WEI X Y, XU P, TAN X Y, LI F P, LI Z C, DENG L, LUO Z L, ZHU L Y, ZHAO S, JIANG D G, LI J, LIU Z L, XIE X R, WANG S K, WU A M, ZHUANG C X, ZHOU H. Methylesterification of cell-wall pectin controls the diurnal flower-opening times in rice[J]. Molecular Plant, 2022, 15(6): 956-972. DOI:10.1016/j.molp.2022.04.004 |
| [52] |
XU P Z, WU T K, ALI A, ZHANG H Y, LIAO Y X, CHEN X Q, TIAN Y H, WANG W M, FU X D, LI Y, FAN J, WANG H, TIAN Y F, LIU Y T, JIANG Q S, SUN C H, ZHOU H, WU X J. EARLY MORNING FLOWERING1 (EMF1) regulates the floret opening time by mediating lodicule cell wall formation in rice[J]. Plant Biotechnology Journal, 2022, 20(8): 1441-144. DOI:10.1111/pbi.13860 |
| [53] |
任代胜, 刘浩, 乔保建. 基于CRISPR/Cas9基因编辑技术靶向敲除OsFAD2创制高油酸水稻突变体[J]. 广东农业科学, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 REN D S, LIU H, QIAO B J. Creation of high oleic acid rice mutant by targeted knockout of OsFAD2 via CRISPR/Cas9 genome editing technology[J]. Guangdong Agricultural Sciences, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 |
| [54] |
GAO C X. Genome engineering for crop improvement and future agriculture[J]. Cell, 2021, 184(6): 1621-1635. DOI:10.1016/j.cell.2021.01.005 |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49





