文章信息
基金项目
- 广州市科技计划项目“珠江科技新星”专题(201806010094);广东省科技计划项目(2021B1212050020)
作者简介
-
潘俊峰,博士,副研究员,现任广东省农业科学院水稻研究所副所长,2018年入选广州市“珠江科技新星”。主要从事水稻高产、氮高效与抗倒伏机理研究,主持广东省科技计划、广州市科技计划等课题项目9项,作为主要参加人承担国家863计划、国家重点研发计划、农业行业专项、国际科技合作等项目15项。以第一作者在Physiologia Plantarum、Agricultural Water Management、《中国水稻科学》等国内外学术期刊发表科技论文11篇,其中SCI论文3篇,参编专著1部。参与研发水稻“三控”施肥技术,作为主要完成人获广东省科技进步一等奖和神农中华农业科技二等奖各1项;参与研发水稻低碳高产栽培技术,该技术具有低投入、低排放、高产出等优势,2017年被国家发改委列入重点推广技术目录。
潘俊峰(1983—),男,博士,副研究员,研究方向为水稻高产高效生理,E-mail:junfeng401@163.com.
通讯作者
- 崔克辉(1967—),男,博士,教授,研究方向为作物生理与栽培,E-mail:cuikehui@mail.hzau.edu.cn.
文章历史
- 收稿日期:2022-07-18
2. 华中农业大学植物科技学院/作物遗传改良国家重点实验室/农业农村部长江中游作物生理生态与耕作重点实验室,湖北 武汉 430070
2. College of Plant Science and Technology, Huazhong Agricultural University/National Key Laboratory of Crop Genetic Improvement/Key Laboratory of Crop Ecophysiology and Farming System in the Middle Reaches of the Yangtze River, Ministry of Agriculture and Rural Affairs, Wuhan 430070, China
【研究意义】水稻是我国主要粮食作物,其高产与稳产对我国粮食安全意义重大。氮是植物需求量最大的养分之一。在水稻生长发育过程中,合理施用氮肥可增加产量,氮营养供应不充足则会导致水稻减产[1],而过量施用氮肥会导致环境污染等问题[2-3]。水稻叶绿素含量是对氮肥施用最为敏感的表型性状之一,与植株光合作用密切相关;研究水稻叶绿素含量的遗传机制,对氮高效的高产水稻品种选育具有重要意义[4-6]。【前人研究进展】水稻叶片叶绿素属于多基因调控的数量性状,其合成与降解易受环境影响[7]。利用不同遗传群体分析苗期、抽穗期和灌浆期等时期不同部位叶片的水稻叶绿素含量,已成功定位了超过600个QTL,分布于12条染色体上[7-10]。QTL定位最终目的是用于基因鉴定和育种应用,以往研究大多采用RFLP、SSR和STS标记构建遗传连锁图谱,鉴定的主效QTL检出率偏低且鉴定位点数量也较少[9]。随着基因组测序技术的快速发展与应用,构建Bin图谱用于QTL检测、候选基因筛选和分子标记开发逐步受到关注[9, 11-12]。已有研究在同一试验点经2年重复试验,利用Bin图谱检测到2个控制叶绿素含量的QTL,并筛选到9个候选基因[9]。前人也在不同氮处理下定位到14个控制叶宽的SNP位点,并鉴定出20个与高氮和低氮响应有关的SNP位点,同时研究者建议,对易受环境影响的性状开展高密度Bin图谱定位分析时,结合多处理、多年、异地环境的分析更能提高QTL定位的精准度[13]。利用高密度Bin图谱开展QTL定位,精确度高,是深入研究重要复杂生理性状遗传规律的有效方法[9]。【本研究切入点】叶绿素含量是容易受环境因素影响的数量性状[14],尤其易受氮肥施用量的影响。然而经多年份、多时期开展不同氮肥施用量条件下的有关叶绿素含量精细QTL定位的研究仍鲜有报道。【拟解决的关键问题】本研究采用不同年份、地点的珍汕97和明恢63水稻重组自交系群体为材料,测定不施氮和正常施氮条件下水稻叶片叶绿素含量,开展不同发育时期叶片叶绿素含量高密度Bin图谱QTL定位分析,旨在寻找一些新的、稳定的叶绿素含量相关位点,以期为水稻叶绿素含量相关基因的鉴定和高光效水稻分子标记育种改良提供有利靶标。
1 材料与方法 1.1 试验材料供试水稻材料为珍汕97(ZS97)和明恢63(MH63)重组自交系(RIL),由华中农业大学作物遗传改良国家重点实验室水稻研究团队提供。该群体共210个家系,从F11中随机选取113个家系用于本研究。
RIL高密度图谱包括1 619个重组Bin标记,各染色体Bin标记数为79~217个,总长1 625.5 cM,平均约1.0 cM/bin,标记间平均物理距离约为230 kb[11]。本研究采用的Bin标记详细数据和图谱均由华中农业大学作物遗传改良国家重点实验室水稻研究团队提供。
1.2 大田试验本试验分别于2006年5—10月在湖北省孝感市新铺镇(30 °56'' N、113 °54'' E)、2007年5—10月在湖北省武穴市大金镇(29 °51'' N、115°33'' E)进行。两地稻田表层25 cm土壤主要养分分别为:新铺镇pH值(1∶1土水比)5.01,有机质含量30.06 g/kg,全氮含量1.39 g/kg(2006年);大金镇pH值(1∶1土水比)5.37,有机质含量22.76 g/kg,全氮含量1.09 g/kg(2007年)。
采用秧盘育秧,种植密度16.7 cm×20.0 cm,划行移栽,每个家系种植1个小区,小区面积4.8 m2(2.0 m×2.4 m)。小区采用随机区组排列,设置正常氮水平(2006:130 kg/hm2;2007:135 kg/hm2)和低氮水平(0 kg/hm2)处理,每个处理3次重复,肥料运筹参照文献[15]。精细管理并全程防治病虫害,成熟期挂防鸟网避免产量损失。两年的试验设计与管理措施保持一致。
1.3 叶绿素含量测定分别于移栽后30 d(30 Days after transplanting,30 DAT)和抽穗期(Heading,HD),采用502型SPAD(Soil and Plant Analyzer Development)叶绿素仪进行水稻叶片叶绿素含量测定。移栽后30 d,测定各小区除边行外植株的主茎1.5叶,分别测定叶片的中部和中部上、下3.0 cm处,取3处平均值表示该叶片SPAD值,每小区重复测定10片叶。由于RIL群体家系的抽穗期相差较大,各小区抽穗10% 定义为抽穗期。抽穗期直接测定植株主茎剑叶的SPAD值,其他操作与移栽后30 d的检测方法相同。
采用Statistix 9.0软件进行数据分析,差异显著性检验用独立样本t检验和单因子方差分析(one-way ANOVA,LSD)。
1.4 QTL定位利用QTL IciMapping V3.4软件,采用完备区间作图法(Inclusive Composite Interval Mapping,ICIM)在全基因组范围内进行扫描[16],扫描步长为1.0 cM,分别检测不同处理和不同发育时期控制叶绿素含量的QTL。以LOD ≥ 2.5作为阈值检测QTL,计算每个QTL的贡献率和加性效应。QTL的命名方法参照McCouch等[17]公布的规则。
2 结果与分析 2.1 不同氮处理下RIL群体和亲本不同发育时期的叶绿素表型变异由表 1可知,2006、2007年RIL群体和亲本均表现为低氮条件下的叶片叶绿素含量显著低于正常氮处理。亲本叶片叶绿素含量两年均表现为母本珍汕97高于父本明恢63;在移栽后30 d,同一亲本相同处理的不同年份差异不大;在抽穗期,2007年明恢63的叶片叶绿素含量显著高于2006年。RIL群体中的叶绿素含量分布均表现为连续的正态分布,并且有超亲分离现象,呈现数量性状的特点(图 1)。
|

|
| 图 1 不同氮处理RIL群体及其亲本在不同发育时期的叶绿素含量分布 Fig. 1 Distribution of chlorophyll contents in RIL population and its parents at different development stages under different nitrogen treatments |
2.2 不同年份和不同发育时期水稻叶片叶绿素含量相关性分析
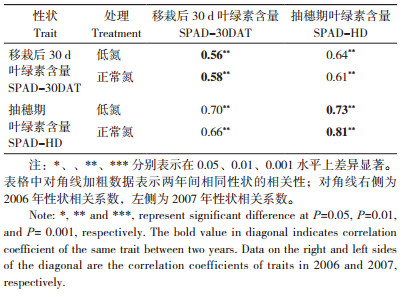
移栽后30 d、抽穗期的水稻叶片叶绿素含量相关系数分别达到极显著水平(表 2)。此外,叶绿素含量在不同氮处理、不同发育时期均表现出显著相关性,两年表现一致,表明两年叶绿素含量的环境稳定性较好。
|
2.3 水稻叶片叶绿素含量的QTL检测
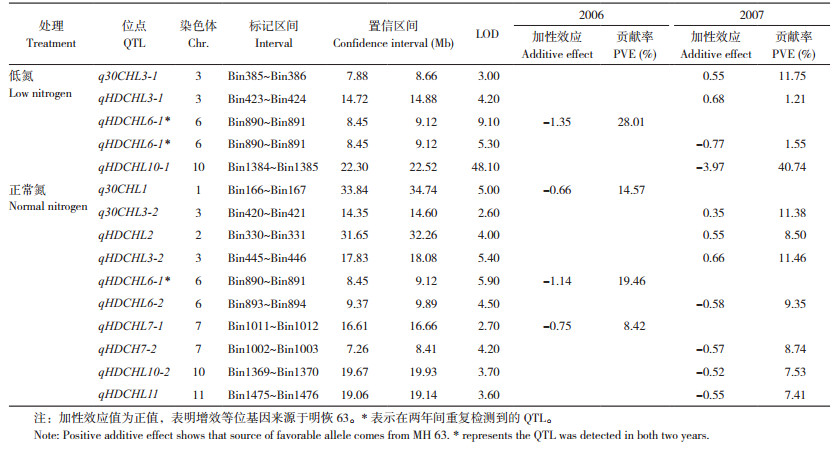
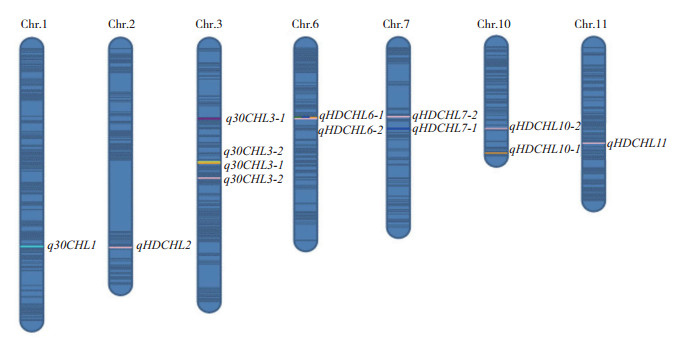
低氮处理下叶片叶绿素含量,两年共检测到5个QTL(2006年1个、2007年4个),分别位于第3、6、10号染色体;正常氮处理下叶片叶绿素含量,两年共检测到10个QTL(2006年4个、2007年11个),分别位于第1、2、3、6、7、10、11号染色体(表 3),正常氮处理下检测到的QTL明显多于低氮处理。在第4、5、8、9号染色体上没有检测到控制叶绿素含量的QTL。15个QTL在染色体上的分布如图 2所示。
|
|
| qHDCHL6-1被重复检测到3次,图中仅标注1次 qHDCHL6-1 was repeatedly detected three times, and was marked only one time 图 2 水稻叶绿素含量QTL在染色体上的分布 Fig. 2 Locations of QTL for chlorophyll content in rice on chromosomes |
第1、2、11号染色体上分别检测到1个QTL。其中,q30CHL1位于第1染色体,在2006年低氮处理水稻叶片中被检测到,影响移栽后30 d的叶绿素含量,解释14.57% 的表型变异,增效基因来自珍汕97;qHDCHL2位于第2号染色体,在2007年正常氮处理水稻叶片中被检测到,等位基因来自明恢63,解释8.50% 的表型变异;qHDCHL11位于第11号染色体,在2007年正常氮处理水稻叶片中被检测到,对表型的贡献率为7.41%。
第3染色体上共检测到4个QTL,均于2007年的水稻叶片中被检测到,等位基因均来自明恢63;在低氮处理叶片中检测到2个QTL,q30CHL3-1影响移栽后30 d的叶绿素含量,解释11.75% 的表型变异,qHDCHL3-1影响抽穗期的叶绿素含量,解释1.21% 的表型变异。另外两个QTL(q30CHL3-2和qHDCHL3-2)在正常氮处理水稻叶片中被检测到,分别解释11.38% 和11.46% 的表型变异。
第6染色体上共检测到4个QTL,均影响抽穗期剑叶的叶绿素含量,且等位基因均来自珍汕97;在2006年的水稻叶片中检测到2个QTL,低氮和正常氮处理下各1个,均位于染色体8.45~9.12 Mb处,命名为qHDCHL6-1,低氮和正常氮处理检测到的QTL分别解释28.01% 和19.46% 的表型变异;在2007年的水稻叶片中也检测到2个QTL,qHDCHL6-1在低氮水平下同样被检测到,解释1.55% 的表型变异,qHDCHL6-2在正常氮水平下检测到,解释9.35% 的表型变异。qHDCHL6-1在2年低氮处理下和2006年正常氮水平下均被检测到,表现出较强的稳定性。
第7染色体上共检测到2个QTL,均为正常氮水平下影响抽穗期剑叶叶绿素含量的QTL,且等位基因均来自珍汕97;qHDCHL7-1于2006年的水稻叶片中被检测到,可解释8.42% 的表型变异;qHDCH7-2于2007年的水稻叶片中被检测到,可解释8.74% 的表型变异。
第10染色体上共检测到2个QTL,均于2007年的水稻叶片中被检测到,增效等位基因均来自珍汕97;qHDCHL10-1在低氮处理下被检测到,是所有QTL中效应最大的位点,解释40.74% 的表型变异;qHDCHL10-2在正常氮处理下被检测到,可解释7.53% 的表型变异。
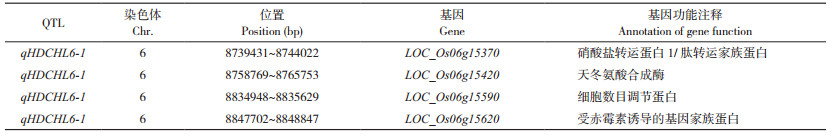
2.4 主效QTL区段中候选基因的筛选利用水稻基因组注释数据库(http://rice.uga.edu/)对重复检测到的qHDCHL6-1区间内的基因进行功能注释。qHDCHL6-1位于第6号染色体的8.45~9.12 Mb处,经查询,该区间内共有113个注释基因,除去不转录蛋白、逆转录转座子蛋白、修复蛋白、假定蛋白等相关基因89个,共筛选到4个有关叶绿素代谢和影响水稻叶色的候选基因(表 4)。
LOC_Os06g15370已被克隆,为硝酸盐转运子/肽转运蛋白基因OsNPF3.1,该基因通过调控水稻株高、抽穗期和千粒重,影响水稻氮肥利用效率。LOC_Os06g15420已被克隆,为天冬酰胺合成酶基因OsAS2。LOC_Os06g15590注释为类似细胞数目调节蛋白,影响细胞增殖和生长,负向调控器官的形态发育。LOC_Os06g15620注释为受赤霉素诱导的基因家族蛋白(GAST),GAST家族在植物激素信号转导、叶片发育及生物与非生物胁迫响应等方面发挥重要调控作用[18]。
3 讨论 3.1 本研究定位的QTL中已鉴定的功能基因前人利用不同的定位群体和标记类型检测到大量控制不同时期、不同处理以及不同部位叶片叶绿素含量的QTL,但这些位点对表型的贡献率均较低、主效基因较少,表明水稻叶片叶绿素含量的遗传背景较为复杂[7-8]。本研究采用高密度Bin连锁图谱,从两年的水稻叶片中共检测到15个控制移栽后30 d和抽穗期叶片叶绿素含量的QTL,其中7个对表型的贡献率超过10%,表明设置不同氮肥处理更有利于揭示叶绿素含量的遗传规律。
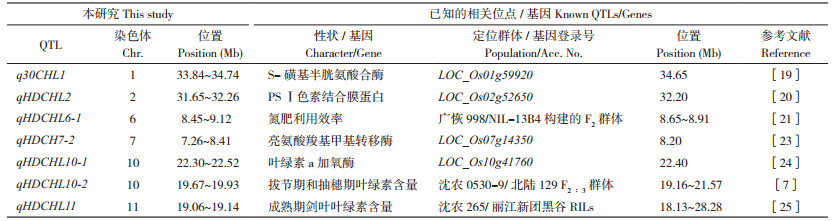
本研究检测到的QTL与前人定位或克隆的QTL有交叉重叠(表 5)。例如,q30CHL1位于第1染色体33.84~34.74 Mb处,在该区域已经克隆S- 磺基半胱氨酸合酶基因(GRA78),该基因主要功能是直接调控叶绿体内基粒和类囊体的生长发育,在水稻苗期和分蘖期分别调控Chla、Chlb和总叶绿素含量性状,突变体的叶绿素含量在苗期和分蘖期分别比野生型降低63.6% 和32.5%[19]。前人在qHDCHL2区域发现了1个光系统I(PSI)色素结合膜蛋白(Lhca),该蛋白与PSI结合形成的复合体对太阳能具有极高的转化效率[20]。qHDCHL6-1位于第6染色体8.45~9.12 Mb处,前人利用F2群体在该区段定位到一个与氮肥利用效率密且相关的qNUE6 位点,利用近等基因系进行生理分析发现,该位点存在2个与拟南芥AtNPF3.1和ASN2高度同源的基因[21]。qHDCH7-2位于第7染色体7.26~8.41 Mb处,该区域已经克隆了亮氨酸羧基甲基转移酶基因(OsMTS1),该基因突变会导致水稻叶倾角增大、叶片叶绿素含量降低40%、叶片早衰和产量显著降低[22],过表达则能够提高水稻产量15.69%[23]。qHDCHL10-1对表型的贡献率为40.74%,前人在该区段鉴定到2个Chla加氧酶基因OsCAO1和OsCAO2,它们在染色体上是串联的,Chla加氧酶承担催化Chla合成Chlb的功能,OsCAO1在有光条件下起主要作用,而OsCAO2主要在黑暗或无光照条件下发挥作用[24]。本研究中,qHDCHL10-1仅在低氮条件下检测到,该基因是否受到低氮处理的强烈诱导目前仍不清楚。qHDCHL10-2在正常氮处理下检测到,前人用籼粳交群体在该区域定位到拔节期和抽穗期叶绿素含量稳定表达的qCJ10和qCH10。前人利用粳粳交RIL在第11号染色体定位到成熟期剑叶叶绿素含量相关的QTL,与qHDCHL11有重叠片段[25]。
|
3.2 qHDCHL6-1是控制叶绿素含量的主效QTL
本研究结果显示,在正常氮处理水稻叶片中检测到的QTL数量明显多于低氮处理,表明低氮处理限制了部分QTL的表达。前人研究建议,对氮肥敏感的数量性状应加强在多环境下的分析[13]。qHDCHL6-1在2006年抽穗期的两个氮处理中均被检测到,在2006年的正常氮处理中也被检测到,表明该位点属于控制抽穗期叶绿素含量的稳定表达的QTL。通过水稻基因组注释数据库比对发现,该区间中存在4个候选基因,其中LOC_Os06g15370和LOC_Os06g15420两个基因已克隆,分别为硝酸盐转运子/肽转运蛋白基因OsNPF3.1和天冬酰胺合成酶基因OsAS2,两个基因均对氮素吸收利用具有重要作用[26-27];低氮处理会刺激OsAS2和OsNPF3.1表达,增加水稻对氮素的吸收利用[26, 28],本研究结果也验证了这一点。LOC_Os06g15620是GAST家族基因,该基因已在2009年克隆,被命名为OsGSR1,是GA信号的正调节因子,在油菜素内酯(BR)和GA信号通路中起重要作用,并介导两者互作,在器官形态建成中正向调控细胞分裂,此外还密切参与植物组织抵抗生物胁迫和氧化还原反应[18, 29],但LOC_Os06g15620对叶片叶绿素含量的调控机理目前仍不清楚。LOC_Os06g15590为类似细胞数目调节蛋白基因,该基因仍未被克隆,但在第2染色体上的同源基因OsCNR1(LOC_Os02g52550)已被克隆,其负调控器官细胞的数量和大小[30],该基因是否调控叶绿体大小或叶片厚度,目前仍不清楚。因此,推测qHDCHL6-1作为控制叶绿素含量的主效QTL,OsGSR1和LOC_Os06g15590有可能参与调控叶片细胞的分化、排布和器官形态建成。综上所述,qHDCHL6-1是多方面调控水稻叶片叶绿素含量的热点区间,具有深入挖掘的潜力,同时对高光效、氮高效水稻品种改良也具有重要应用价值。
4 结论本研究以珍汕97×明恢63重组自交系(F11)113个家系为QTL分析的试验材料,在大田栽培条件下,设置低氮和正常氮两种氮肥处理,分别在两年和两个发育时期测定叶片叶绿素含量,利用高密度Bin遗传图谱和完备区间作图法共检测到15个控制叶片叶绿素含量的QTL。其中,低氮处理共检测到5个QTL,正常氮处理共检测到10个QTL。发现1个低氮和正常氮水平下稳定表达的QTL qHDCHL6-1,等位基因来自珍汕97,控制抽穗期剑叶叶绿素含量,在该区段筛选到4个候选基因。
| [1] |
潘俊峰, 钟旭华, 黄农荣, 刘彦卓, 田卡, 梁开明, 彭碧琳, 傅友强, 胡香玉. 不同栽培模式对华南双季晚稻产量和氮肥利用率的影响[J]. 浙江农业学报, 2019, 31(6): 857-868. DOI:10.3969/j.issn.1004-1524.2019.06.01 PAN J F, ZHONG X H, HUANG N R, LIU Y Z, TIAN K, LIANG K M, PENG B L, FU Y Q, HU X Y. Effects of different cultivation patterns on grain yield and nitrogen use efficiency of rice in South China[J]. Acta Agriculturae Zhejiangensis, 2019, 31(6): 857-868. DOI:10.3969/j.issn.1004-1524.2019.06.01 |
| [2] |
GUO J H, LIU X J, ZHANG Y, SHEN J L, HAN W X, ZHANG W F, CHRISTIE P, GOULDING K W T, VITOUSEK P M, ZHANG F S. Significant acidification in major Chinese croplands[J]. Science, 2010, 327(5968): 1008-1010. DOI:10.1126/science.1182570 |
| [3] |
LIANG K M, ZHONG X H, JUNFENG PAN J F, HUANG N R, LIU Y Z, PENG B L, FU Y Q, HU X Y. Reducing nitrogen surplus and environmental losses by optimized nitrogen and water management in double rice cropping system of South China[J]. Agriculture, Ecosystems & Environment, 2019, 286: 106680. DOI:10.1016/j.agee.2019.106680 |
| [4] |
ZHANG Q F. Strategies for developing green superrice[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(42): 16402-16409. DOI:10.1073/pnas.0708013104 |
| [5] |
MOOSE S P, MUMM R H. Molecular plant breeding as the foundation for 21st century crop improvement[J]. Plant Physiology, 2008, 147(3): 969-977. DOI:10.1104/pp.108.118232 |
| [6] |
MCALLISTER C H, BEATTY P H, GOOD A G. Engineering nitrogen use efficient crop plants: the current status[J]. Plant Biotechnology Journal, 2012, 10(9): 1011-1025. DOI:10.1111/j.1467-7652.2012.00700.x |
| [7] |
刘进, 王嘉宇, 姜树坤, 徐正进. 水稻叶绿素含量动态QTL分析[J]. 植物生理学报, 2012, 48(6): 577-583. DOI:10.13592/j.cnki.ppj.2012.06.001 LIU J, WANG J Y, JIANG S K, XU Z J. Detection and analysis of dynamic QTL of leaf chlorophyll content in rice (Oryza sativa L.)[J]. Plant Physiology Journal, 2012, 48(6): 577-583. DOI:10.13592/j.cnki.ppj.2012.06.001 |
| [8] |
阿加拉铁, 曾龙军, 薛大伟, 胡江, 曾大力, 高振宇, 郭龙彪, 李仕贵, 钱前. 水稻灌浆期不同阶段叶绿素含量的QTL分析[J]. 作物学报, 2008, 34(1): 61-66. DOI:10.3724/SP.J.1006.2008.00061 JIA L T, ZENG L J, XUE D W, HU J, ZENG D L, GAO Z Y, GUO L B, LI S G, QIAN Q. QTL analysis for chlorophyll content in four grain-filling stage in rice[J]. Acta Agronomica Sinica, 2008, 34(1): 61-66. DOI:10.3724/SP.J.1006.2008.00061 |
| [9] |
赵凌, 张勇, 魏晓东, 梁文化, 赵春芳, 周丽慧, 姚姝, 王才林, 张亚东. 利用高密度Bin图谱定位水稻抽穗期剑叶叶绿素含量QTL[J]. 中国农业科学, 2022, 55(5): 825-836. DOI:10.3864/j.issn.0578-1752.2022.05.001 ZHAO L, ZHANG Y, WEI X D, LIANG W H, ZHAO C F, ZHOU L H, YAO S, WANG C L, ZHANG Y D. Mapping of QTLs for chlorophyll content in flag leaves of rice on high-density Bin map[J]. Scientia Agricultura Sinica, 2022, 55(5): 825-836. DOI:10.3864/j.issn.0578-1752.2022.05.001 |
| [10] |
姚晓云, 杨平, 黄永萍, 彭志勤, 刘进, 吴延寿, 邹国兴, 尹建华. 水稻叶绿素含量主效QTL定位[J]. 江西农业学报, 2022, 34(2): 8-14. DOI:10.19386/j.cnki.jxnyxb.2022.02.002 YAO X Y, YANG P, HUANG Y P, PENG Z Q, LIU J, WU Y S, ZOU G X, YIN J H. Mapping of major QTLs for chlorophyll content in rice[J]. Acta Agriculturae Jiangxi, 2022, 34(2): 8-14. DOI:10.19386/j.cnki.jxnyxb.2022.02.002 |
| [11] |
YU H H, XIE W B, WANG J, XING Y Z, XU C G, LI X H, XIAO J H, ZHANG Q F. Gains in QTL detection using an ultra-high density SNP map based on population sequencing relative to traditional RFLP/SSR markers[J]. PLOS ONE, 2011, 6(3): e17595. DOI:10.1371/journal.pone.0017595 |
| [12] |
王朝欢, 宋博文, 余思佳, 肖武名, 黄明. 基于全基因组测序构建水稻RIL群体遗传图谱[J]. 华南农业大学学报, 2021, 42(2): 44-50. DOI:10.7671/j.issn.1001-411X.202006039 WANG C H, SONG B W, YU S J, XIAO W M, HUANG M. Construction of a genetic map of rice RILs based on whole genome sequencing[J]. Journal of South China Agricultural University, 2021, 42(2): 44-50. DOI:10.7671/j.issn.1001-411X.202006039 |
| [13] |
高易宏, 燕金香, 涂政军, 冷语佳, 陈龙, 黄李超, 代丽萍, 张光恒, 朱丽, 胡江, 任德勇, 郭龙彪, 钱前, 王丹英, 曾大力. 不同氮处理下水稻剑叶叶宽的全基因组关联分析[J]. 中国农业科学, 2017, 50(14): 2635-2646. DOI:10.3864/j.issn.0578-1752.2017.14.001 GAO Y H, YAN J X, TU Z J, LENG Y J, CHEN L, HUANG L C, DAI L P, ZHANG G H, ZHU L, HU J, REN D Y, GUO L B, QIAN Q, WANG D Y, ZENG D L. Genome-Wide association analysis on flag leaf width under different nitrogen levels in rice[J]. Scientia Agricultura Sinica, 2017, 50(14): 2635-2646. DOI:10.3864/j.issn.0578-1752.2017.14.001 |
| [14] |
杨树明, 刘关所, 张素华. 不同生长环境下水稻孕穗期叶绿素QTL定位[J]. 云南大学学报(自然科学版), 2017, 39(4): 684-690. DOI:10.7540/j.ynu.20170035 YANG S M, LIU G S, ZHANG S H. Identification of QTL for chlorophyll contents at booting stage of rice under different growing environments[J]. Journal of Yunnan University (Natural Sciences Edition), 2017, 39(4): 684-690. DOI:10.7540/j.ynu.20170035 |
| [15] |
PAN J F, CUI K H, WEI D, HUANG J L, XIANG J, NIE L X. Relationships of non-structural carbohydrates accumulation and translocation with yield formation in rice recombinant inbred lines under two nitrogen levels[J]. Physiologia Plantarum, 2011, 141(4): 321-331. DOI:10.1111/j.1399-3054.2010.01441.x |
| [16] |
MENG L, LI H H, ZHANG L Y, WANG J K. IciMapping: Integrated software for genetic linkage map construction and quantitative trait locus mapping in bi-parental populations[J]. Crop Journal, 2015, 3(3): 269-283. DOI:10.1016/j.cj.2015.01.001 |
| [17] |
MCCOUCH S R, CHO Y G, YANO M, PAUL E, BLINSTRUB M, MORISHIMA H, KINOSHITA T, MCCOUCH S R, CHO Y G, PAUL E, MORISHIMA H, CHO Y G, YANO M, MCCOUCH S, YANO P E, KINOSHITA T, MCCOUCH S, CHO Y, PAULE E, BLINSTRUE M, MORISHIMA H M, MCCOUCH S R, KINOSHITA T. Report on QTL nomenclature[J]. Rice Genet Newsletter, 1997, 14: 11-13. |
| [18] |
李相伯, 史双月, 朱韵, 陶亚军, 杨泽峰, 梁国华, 周勇. 植物GAST家族基因功能研究进展[J]. 扬州大学学报(农业与生命科学版), 2019, 40(2): 25-32. DOI:10.16872/j.cnki.1671-4652.2019.02.004 LI X B, SHI S Y, ZHU Y, TAO Y J, YANG Z F, LIANG G H, ZHOU Y. Progress in the functions of GAST family genes in plants[J]. Journal of Yangzhou University (Agricultural and Life Science Edition), 2019, 40(2): 25-32. DOI:10.16872/j.cnki.1671-4652.2019.02.004 |
| [19] |
WANG Y, ZHONG P, ZHANG X Y, LIU J Q, ZHANG C Y, YANG X R, WAN C M, LIU C Q, ZHOU H, YANG B, SUN C H, DENG X J, WANG P R. GRA78 encoding a putative S-sulfocysteine synthase is involved in chloroplast development at the early seedling stage of rice[J]. Plant Science, 2019, 280: 321-329. DOI:10.1016/j.plantsci.2018.12.019 |
| [20] |
UMATE P. Genome-wide analysis of the family of light-harvesting chlorophyll a /b-binding proteins in Arabidopsis and rice[J]. Plant Signaling & Behavior, 2010, 5(12): 1537-1542. DOI:10.4161/psb.5.12.13410 |
| [21] |
YANG X H, XIA X Z, ZHANG Z Q, NONG B X, ZENG Y, XIONG F Q, WU Y Y, GAO J, DENG G F, LI D T. QTL mapping by whole genome re-sequencing and analysis of candidate genes for nitrogen use efficiency in rice[J]. Frontiers in Plant Science, 2017(8): 1634. DOI:10.3389/fpls.2017.01634 |
| [22] |
TAMIRU M, TAKAGI H, ABE A, YOKOTA T, KANZAKI H, OKAMOTO H, SAITOH H, TAKAHASHI H, FUJISAKI K, OIKAWA K, UEMUR A A, NATSUME S, JIKUMARU Y, MATSUUR A H, UMEMURA K, TERRY M A, TERAUCHI R. A chloroplast-localized protein LESION AND LAMINA BENDING affects defence and growth responses in rice[J]. New Phytologist, 2016, 210(4): 1282-1297. DOI:10.1111/nph.13864 |
| [23] |
HONG Y B, ZHANG Y X, SINUMPORN S, YU N, ZHAN X D, SHEN X H, CHEN D B, YU P, WU W X, LIU Q N, CAO Z Y, ZHAO C D, CHENG S H, CAO L Y. Premature leaf senescence 3, encoding a methyltransferase, is required for melatonin biosynthesis in rice[J]. The Plant Journal, 2018, 95(5): 877-891. DOI:10.1111/tpj.13995 |
| [24] |
LEE S, KIM J H, YOO E S, LEE C H, HIROCHIK A H, AN G. Differential regulation of chlorophyll a oxygenase genes in rice[J]. Plant Molecular Biology, 2005, 57(6): 805-818. DOI:10.1007/s11103-005-2066-9 |
| [25] |
姜树坤, 张喜娟, 徐正进, 陈温福. 粳稻叶绿素含量QTL与其合成降解相关基因的比较分析[J]. 作物学报, 2010, 36(3): 376-384. DOI:10.3724/SP.J.1006.2010.00376 JIANG S K, ZHANG X J, XU Z J, CHEN W F. Comparison between QTLs for chlorophyll content and genes controlling chlorophyll biosynthesis and degradation in Japonica rice (Oryza sativa L.)[J]. Acta Agronomica Sinica, 2010, 36(3): 376-384. DOI:10.3724/SP.J.1006.2010.00376 |
| [26] |
OHASHI M, ISHIYAMA K, KOJIMA S, KONISHI N, NAKANO K, KANNO K, HAYAKAWA T, YAMAYA T. Asparagine Synthetase1, but not Asparagine Synthetase2, is responsible for the biosynthesis of asparagine following the supply of ammonium to rice roots[J]. Plant and Cell Physiology, 2015, 56(4): 769-778. DOI:10.1093/pcp/pcv005 |
| [27] |
YANG X H, NONG B X, CHEN C, WANG J R, XIA X Z, ZHANG Z Q, WEI Y, ZENG Y, FENG R, WU Y Y, GUO H, YAN H F, LIANG Y T, LIANG S H, YAN Y, LI D T, DENG G F. OsNPF3.1, a member of the NRT1/P TR family, increases nitrogen use efficiency and biomass production in rice[J]. Crop Journal, 2022. DOI:10.1016/j.cj.2022.07.001 |
| [28] |
DAVID L C, BERQUIN P, KANNO Y, SEO M, DANIEL V F, FERRARIO M S. Navailability modulates the role of NPF3.1, a gibberellin transporter, in GA-mediated phenotypes in Arabidopsis[J]. Planta, 2016, 244(6): 1315-1328. DOI:10.1007/s00425-016-2588-1 |
| [29] |
WANG L, WANG Z, XU Y Y, JOO S H, KIM S K, XUE Z, XU Z H, WANG Z Y, CHONG K. OsGSR1 is involved in crosstalk between gibberellins and brassinosteroids in rice[J]. The Plant Journal, 2009, 57(3): 498-510. DOI:10.1111/j.1365-313X.2008.03707.x |
| [30] |
RUAN B P, SHANG L G, ZHANG B, HU J, WANG Y X, LIN H, ZHANG A P, LIU C L, PENG Y L, ZHU L, REN D Y, SHEN L, DONG G J, ZHANG G H, ZENG D L, GUO L B, QIAN Q, GAO Z Y. Natural variation in the promoter of TGW2 determines grain width and weight in rice[J]. New Phytologist, 2020, 227(2): 629-640. DOI:10.1111/nph.16540 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49