文章信息
基金项目
- 云浮市2021年省乡村振兴战略专项(2021020608);广东省重点领域研发计划项目(2022B0202110002);国家自然科学基金青年科学基金(32102539);广东省农业科学院协同创新中心项目(XT202217);国家肉鸡产业技术体系岗位科学家项目(CARS-41)
作者简介
- 李晓娇(1997—),女,在读硕士生,研究方向为家禽遗传育种与繁殖,E-mail:lxjleslie@163.com.
通讯作者
- 罗成龙(1981—),男,博士,研究员,研究方向为动物遗传育种与繁殖,E-mail:luochenglong@gdaas.cn.
文章历史
- 收稿日期:2022-10-26
2. 广东省农业科学院动物科学研究所/畜禽育种国家重点实验室/广东省畜禽育种与营养研究重点实验室,广东 广州 510640
2. Institute of Animal Science, Guangdong Academy of Agricultural Sciences/State Key Laboratory of Livestock Breeding/Guangdong Key Laboratory of Livestock Breeding and Nutrition Research, Guangzhou 510640, China
【研究意义】基因编辑技术对高效获得特定表型的畜禽新品种具有重要意义,而畜禽功能基因的挖掘和开发是选育新品种的重要手段。成簇的规律间隔短回文重复序列(CRISPR/Cas9)是当前应用最广泛的基因编辑技术[1-2]。近年来,通过应用CRISPR/Cas9技术对哺乳动物细胞、活体进行精确基因编辑和基因功能研究,显著加快了生物学、医学相关研究的效率。基于CRISPR/ Cas9的基因工程技术与细胞工程技术相结合,可以不断挖掘动物的功能基因,丰富动物遗传资源,推进动物育种进程。本研究建立稳定表达Cas9蛋白的鸡成纤维细胞系(DF-1)将有利于推进鸡功能基因的挖掘工作,对加快明确鸡育种进程的主效基因具有重要意义。【前人研究进展】CRISPR/Cas9系统中sgRNA引导Cas9蛋白造成DNA双链断裂(Double-Strand Breakage,DSB),并诱导DNA自我修复,包括同源重组修复(Homology Directed Repair,HDR)、非同源末端链接(Non-homologous End Joining,NHEJ)和单链退火(Single-stranded Annealing, SSA)3种方式[3-4]。利用DNA自我修复的特性,可在DSB时精确敲除、插入或替换特定的碱基。CRISPR/ Cas9技术具有高效、应用简单且成本低的优点,因此在动物和植物的遗传育种研究中发挥重要作用[5-7]。为了快速确认CRISPR/Cas9系统的编辑能力,一般采用报告系统便于结果的观察[8-9]。SSA修复机制是一种重要的DNA重组方式,双链DNA断裂产生3'ss DNA悬臂末端,当两端存在同向的同源序列时,同源序列与悬臂末端通过碱基互补配对拟合在一起,再经过核酸内切酶切除多余的3' 末端、聚合酶补齐双链缺口、连接酶连接最终完成修复。近年来基于SSA修复机制的报告系统相继被报道[9-13],其修复效率比NHEJ高10倍[14]。Oishi等[15]在禽细胞上利用SSA报告载体判断sgRNA活性,针对卵清蛋白(OVA)和卵胞浆样蛋白(OVM)分别设计4条sgRNA,将Cas9蛋白和sgRNA共表达载体与SSA报告载体共同转染293T细胞,快速筛选出活性最好的sgRNA序列,在后续研究中用于产生基因编辑鸡,且无出现脱靶效应。黄思嘉等[16]通过双荧光报告载体试验证明了成对敲除载体的工作效率远高于单敲除载体,采用成对的AMHR2敲除载体共转染DF-1细胞,结果显示AMHR2基因的突变率达到60%。【本研究切入点】应用SSA报告系统是快速检测sgRNA活性的有效手段,而在鸡细胞上利用SSA报告载体检测稳转Cas9细胞系活性还未见报道。获得有稳定切割活性的稳转Cas9蛋白细胞系非常重要,在稳转细胞系中同时引入SSA报告载体系统和sgRNA表达载体,sgRNA能快速引导Cas9蛋白切割SSA报告载体中的靶片段,从而快速判断Cas9核酸酶活性,得到具有活性的稳转Cas9蛋白的细胞系。【拟解决的关键问题】与哺乳动物细胞系相比,鸡DF-1细胞转染效率较低,通过慢病毒感染DF-1细胞筛选稳转Cas9的阳性细胞,并结合SSA报告载体验证其活性,可为后续在DF-1细胞上开展高通量筛选以及功能基因验证奠定基础。
1 材料与方法 1.1 试验材料DF-1、293T、DF-1-EGFP-Cas9细胞和DH5α感受态细胞由广东省农业科学院动物科学研究所保存;Lenti-Blast-Cas9、psPAX2、PMD2.G由华中农业大学动物科技学院赠送,pYP152、pCMV-SSA-mCherry-Hind Ⅲ由中国科学院上海生命科学研究院神经科学研究所赠送。转染试剂LipofectamineTM3000、杀稻瘟菌素(Blasticidin,下文简写为Blast)、限制性内切酶Bbs Ⅰ、Hind Ⅲ以及细胞培养用的基础培养基DMEM、PBS缓冲液和细胞培养胎牛血清(FBS)购于赛默飞世尔科技(中国)有限公司,慢病毒感染增强试剂聚凝胺Polybrene购于西格玛奥德里奇(上海)贸易有限公司;AG高保真DNA聚合酶购于湖南艾科瑞生物工程有限公司,In fusion同源重组酶和DNA Ligation Mix购于宝日医生物技术(北京)有限公司,质粒中量提取试剂盒购于Omega Bio-tek广州飞扬生物工程有限公司,血液/细胞/组织基因组DNA提取试剂盒和琼脂糖凝胶核酸纯化回收试剂盒购于天根生化(北京)科技有限公司,LB肉汤购于环凯微生物有限公司,氨苄青霉素购于北京索莱宝科技有限公司,Cas9蛋白抗体购于艾博抗(上海)贸易有限公司,HRP标记的山羊抗兔IgG购于赛信通(上海)生物试剂有限公司。
1.2 试验方法1.2.1 细胞培养 293T细胞和DF-1细胞均以含10% FBS的DMEM培养基为完全培养基,分别置于37 ℃、含5% CO2的培养箱和39 ℃、含5% CO2的培养箱中培养。
1.2.2 Blast工作浓度筛选 在6孔板上接种DF-1细胞,当细胞长至密度为80%~90% 时加入含有Blast的完全培养基(DMEM+10%FBS),将1 mg/mL的Blast溶液用不加抗生素的完全培养基分别稀释为0、2、4、6、8、10 μg/mL,每天观察细胞生长状态,每隔48 h更换1次新鲜的筛选培养基。
1.2.3 稳转Cas9的DF-1细胞构建 将293T细胞铺板在10 cm培养皿上,生长至密度为70%~80% 时转染。按照LipofectamineTM3000转染试剂说明书转染慢病毒三质粒系统,转染质粒的总量为24 μg,3种质粒的比例为Lenti -Blast-Cas9 ︰ psPAX2 ︰ PMD2.G=3 ︰ 2 ︰ 1,转染前更换新鲜的完全培养基,转染6 h后换液,并分别在转染48、72 h后收集慢病毒上清液,-80℃保存。之后将DF-1细胞接种于6孔板,生长至密度为40% 左右用慢病毒感染细胞,慢病毒︰完全培养基=1 ︰ 1,加入终浓度为8 μg/mL的Polybrene进行培养,以不加入慢病毒为对照。感染24 h后换为完全培养基,48 h后加入最适工作浓度Blast的完全培养基,隔天换液,直至对照处理细胞全部死亡。之后更换为含最适抗生素浓度1/2的完全培养基继续培养细胞。
1.2.4 单克隆细胞株的筛选 采用有限稀释法筛选单克隆细胞株。用0.1% 胰蛋白酶消化单层DF-1细胞,制成细胞悬液,用完全培养基将细胞稀释成1 000 cell/mL后,取200 μL细胞悬液加入96孔板的A1孔,其他孔加入100 μL含2 μg/mL Blast的筛选培养基,吸取100 μL A1孔的细胞加至A2孔,以此类推稀释到A8孔。再向A1~A8孔各加入100 μL培养基,混匀后分别取100 μL加入B1~B8孔,逐步稀释至L1~L8孔。观察选择只有单个细胞的孔,继续培养2~3周,当细胞长满后在48孔板扩大培养,依次传代到12孔板、6 cm皿、10 cm皿并冻存细胞。
1.2.5 PCR扩增Cas9核酸酶DNA片段 用SnapGene软件分析Lenti-Blast-Cas9慢病毒载体图谱和序列(图 1),针对载体中的Cas9蛋白设计引物(Lenti Cas9-F:5'ATGGACAAGAAGTACAGCATCGGC3';Lenti Cas9-R:5'ACAGGTCGATCCGTGTCTCGTA3'),由生工生物工程(上海)股份有限公司合成。提取单克隆细胞株的基因组DNA,进行PCR扩增Cas9片段,PCR反应体系参考AG高保真DNA聚合酶说明书,反应程序为:95 ℃预变性3 min;95 ℃变性30 s、60 ℃退火30 s、72 ℃延伸4 min,共35个循环。扩增产物用1% 琼脂糖凝胶电泳检测。
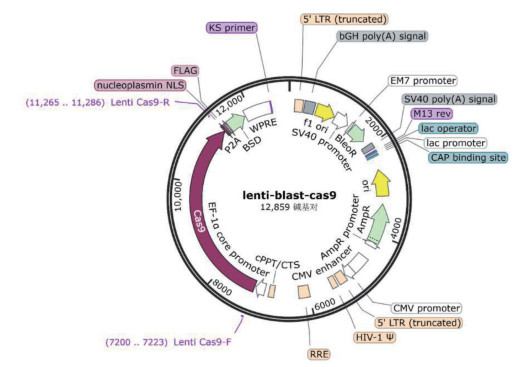
|
| 图 1 Lenti-Blast-Cas9载体图谱 Fig. 1 Lenti-Blast-Cas9 vector map |
1.2.6 Western blot验证Cas9蛋白表达 将阳性单克隆细胞在6孔板中传代,同时设置阴性和阳性对照,其中阴性对照为没有做任何处理的DF-1细胞,阳性对照为DF-1-EGFP-Cas9细胞,培养48 h后弃去培养基,用PBS清洗2次,然后分别在细胞中加入配制好的RIPA蛋白裂解液,在冰上裂解20 min后加入5×上样缓冲液,在100 ℃金属浴中变性10 min。之后用8% SDS-PAGE分离胶于90 V下恒压电泳2 h分离蛋白样品,300 mA恒流转膜2 h,室温下用含5%BSA的TBST封闭1 h,一抗在4 ℃下过夜孵育(一抗稀释比例为1 ︰ 20 000),用1× TBST洗膜3次后在常温下孵育二抗(二抗稀释比例为1 ︰ 5 000),洗膜3次后显影观察结果,用Image J软件分析目的蛋白和内参蛋白灰度值,计算Cas9蛋白相对表达量,用GraphPad Prism 9软件绘制散点图。
1.2.7 sgRNA表达载体构建 在线(http://crispor.tefor.net/)设计靶向OVA基因的sgRNA序列,引物(OVA sgRNA top:5'caccGCCATGCCAAT GAGAACATCT3',OVA sgRNA bottom:5'aaacAGA TGTTCTCATTGGCATGGC3',小写字母为Bbs Ⅰ粘性末端)由生工生物工程(上海)股份有限公司合成,纯化方式为PAGE。用ddH2O将引物稀释到100 nmol/mL,在PCR管中加入引物对各5 μL,在PCR仪中退火形成寡聚核苷酸链,退火程序为95℃ 5 min、65℃ 1 h。sgRNA表达载体以pYP152为骨架,在U6启动子后带有Bbs Ⅰ酶切位点,用于插入sgRNA序列。将pYP152载体用Bbs Ⅰ在37℃下酶切1 h,线性化后用5 μL DNA Ligation Mix连接sgRNA退火产物,反应条件为16℃ 1 h。将连接产物转化DH5α、涂板,第2天挑菌送出测序。
1.2.8 SSA报告载体构建 SSA修复机制可以快速灵敏地检测稳转Cas9细胞株的切割活性,依赖于同源重组的SSA修复机制如图 2所示。
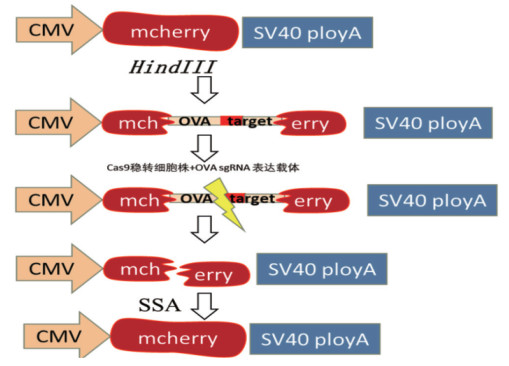
|
| 图 2 SSA修复过程示意图 Fig. 2 SSA repair process schematic diagram |
SSA报告载体以pCMV-SSA-mCherry- Hind Ⅲ为骨架,用软件snapGene设计OVA靶片段引物,该片段需要包含OVA sgRNA序列,并添加报告载体pCMV-SSA-mCherry-Hind Ⅲ的酶切位点两端15 bp同源臂(OVA SSA F:5'caggactcctccctgATCCTTACATTTTCACTGTTCTGCTG3',OVA SSA R:5'ccttggtcaccttcaCCTCTGAGCTATGCAGTTTCCAA3',小写字母为Hind Ⅲ酶切位点两端15 bp同源臂)。PCR扩增OVA靶片段,反应程序为:95℃预变性3 min;95 ℃变性30 s、60 ℃退火30 s、72 ℃延伸30 s,共35个循环。对报告载体用 Hind Ⅲ在37℃下酶切15 min,用天根DNA回收试剂盒纯化靶片段PCR产物和报告载体酶切产物,之后用In fusion酶连接,连接体系为:PCR纯化产物200 ng、酶切产物50 ng、5× In-fusion HD Enzyme Premix 2 μL,补水至10 μL,在50℃下反应15 min,连接产物转化DH5α感受态细胞,涂板,第2天挑选单克隆菌送出测序。
1.2.9 DF-1稳转Cas9蛋白细胞培养与转染 对稳转Cas9蛋白的DF-1细胞用含2 μg /mL Blast的筛选培养基在39℃下培养,细胞长满后将细胞接种到24孔板,当细胞密度达到70%~80% 左右时进行转染试验,转染试剂为LipfectamineTM 3000,按照说明书操作,sgRNA表达载体和SSA报告载体比例为1 ︰ 1,总量为1 μg。设置DF-1仅转染pCMV-OVA-SSA-mCherry-Hind Ⅲ的阴性对照,DF-1共转Lenti -Blast-Cas9、pYP152-OVA-sgRNA以及pCMV-OVA-SSA-mCherry-Hind Ⅲ的阳性,其他4株单克隆细胞株共转pYP152-OVA-sgRNA和pCMV-OVA-SSA-mCherry-Hind Ⅲ,转染48 h后在荧光显微镜下观察荧光。
2 结果与分析 2.1 Blast最佳工作浓度用含有不同浓度Blast的培养基培养DF-1细胞,以培养7 d后细胞全部死亡的Blast浓度为DF-1稳转Cas9细胞株筛选的最适工作浓度,结果(图 3)表明,Blast浓度为4 μg/mL时,培养7 d后细胞全部死亡,因此,选择4 μg/mL为Blast的最适工作浓度。

|
| 图 3 不同浓度Blast培养DF-1细胞7 d后的细胞状态(4×) Fig. 3 Cell status of DF-1 cells after 7 days of culture with different concentrations of Blast (4×) |
2.2 单克隆细胞株的筛选
利用含4 μg/mL Blast的培养基培养7 d后阴性对照细胞全部死亡,慢病毒感染组还有少量存活细胞,继续培养3 d后,换为含2 μg/mL Blast的培养基维持筛选,再培养1周后明显观察到慢病毒感染组形成多个单克隆细胞(图 4)。用有限稀释法以10个96孔板将这些单克隆细胞进行稀释,最终获得27株单克隆细胞株。
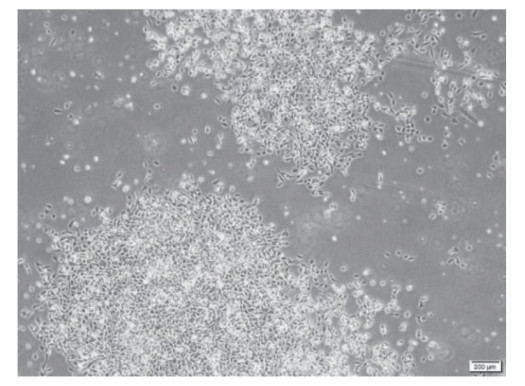
|
| 图 4 稳定表达Cas9蛋白的DF-1阳性细胞 Fig. 4 DF-1 positive cells of stably expressing Cas9 protein |
2.3 单克隆细胞株Cas9蛋白表达验证
提取单克隆细胞株的DNA,PCR扩增Cas9核酸酶DNA片段,琼脂糖凝胶电泳结果显示,单克隆细胞株在4 140 bp处均有明显的目的条带,而阴性的DF-1细胞未检测到任何条带(图 5A)。提取细胞总蛋白,Western blot结果表明,挑选的DF-1稳转株均表达Cas9蛋白,且DF-1细胞不表达(图 5B)。用Image J分析灰度值后,计算目的蛋白与内参蛋白的比值并做归一化处理,使用GraphPad Prism 9得到稳转Cas9蛋白细胞系的Cas9蛋白表达量散点图(图 5C),这些稳转株的Cas9表达水平均不一致,选择其中几株表达较高的细胞株扩大培养,用于后续的SSA修复活性检测。
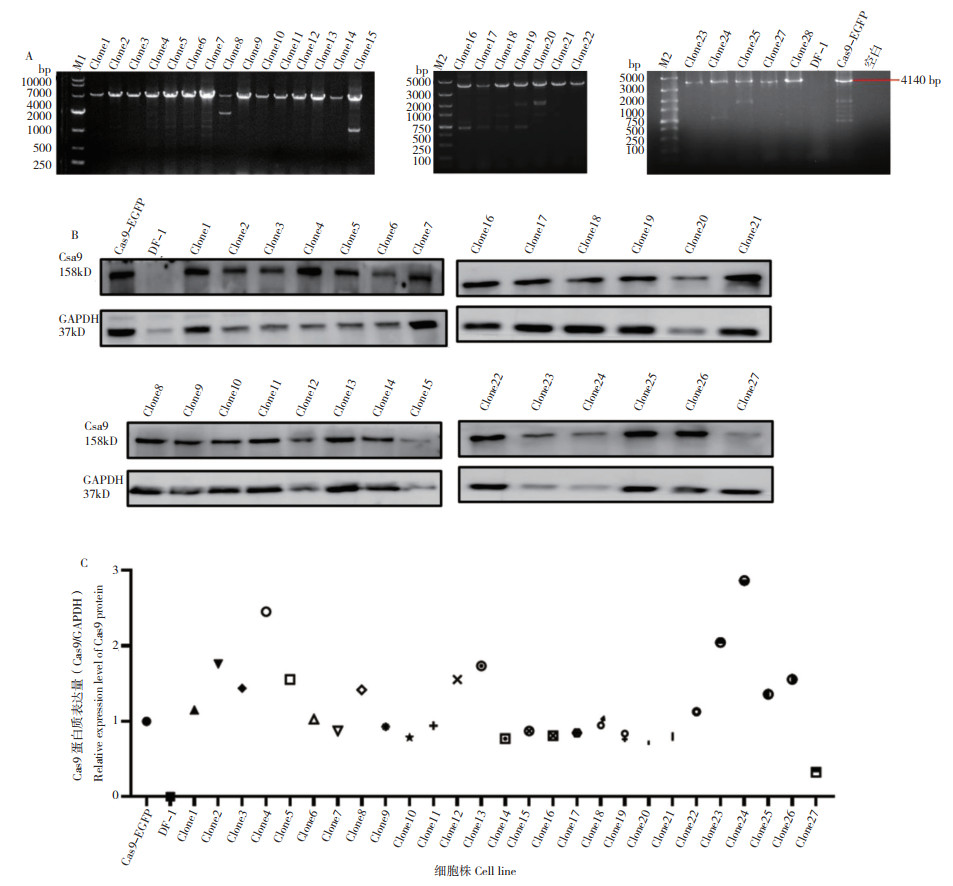
|
| A:PCR扩增单克隆细胞株中的Cas9 DNA片段(M1:DL10000 Maker,M2:DL5000 Maker);B:Western blot鉴定单克隆细胞株中Cas9蛋白表达;C:各单克隆细胞株Cas9蛋白表达量分析 A: PCR amplification of Cas9 DNA fragments in monoclonal cell lines (M1: DL10000Maker, M2: DL5000Maker); B: Western blot identification of Cas9 protein expression in monoclonal cell lines; C: Cas9 protein expression in each monoclonal cell line quantitative analysis 图 5 单克隆细胞株Cas9蛋白表达验证 Fig. 5 Validation of Cas9 protein expression in a monoclonal cell line |
2.4 sgRNA表达载体与SSA报告载体的构建
为了构建sgRNA表达载体,用Bbs Ⅰ酶切pYP152线性化载体(图 6A),与退火的OVA sgRNA序列相连,转化感受态DH5α后在氨苄抗性固体培养基上培养,挑取单克隆菌落,由天一辉远生物科技有限公司测序,结果显示OVA sgRNA序列插入pYP152载体的位置、方向和序列均正确,成功构建OVA sgRNA表达载体。PCR扩增OVA靶片段,琼脂糖凝胶电泳验证条带正确,切胶,用试剂盒回收DNA片段(图 6C),与线性化的pCMV-mCherry-SSA-Hind Ⅲ载体连接,转化后挑取单克隆菌落,菌液测序结果与克隆载体进行比对,结果(图 6D)显示有408 bp插入,该OVA靶片段包含正确的sgRNA序列和对应的PAM位点,成功构建SSA报告载体。所构建的pYP152-OVA sgRNA表达载体和pCMV-OVA-SSA-mCherry-Hind Ⅲ报告载体用于后续共转检测稳转Cas9蛋白细胞的切割活性。
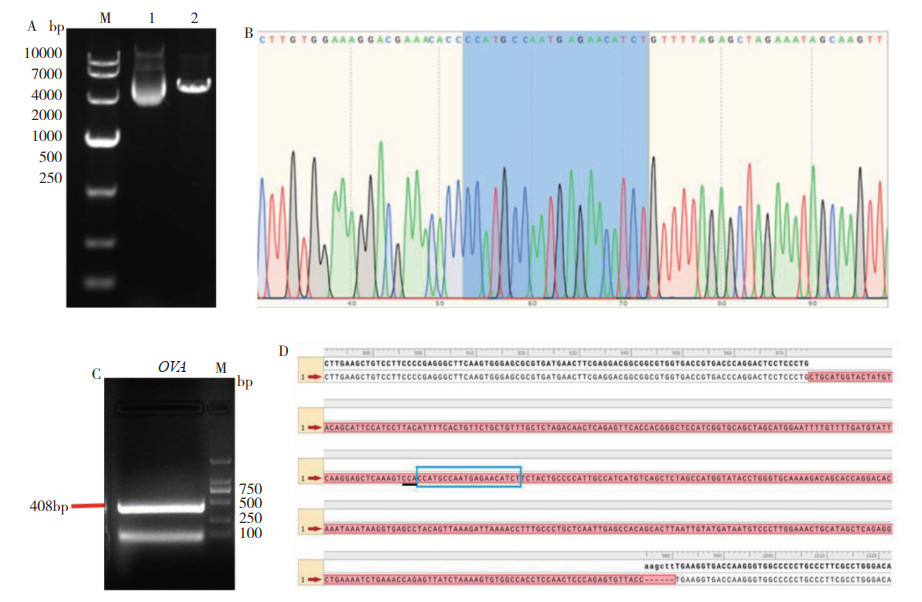
|
| A:Bbs Ⅰ酶切pYP152载体,M为DNA Maker,1为pYP152载体质粒,2为Bbs Ⅰ酶切pYP152载体质粒产物;B:pYP152-OVA-sgRNA载体sgRNA靶位点测序结果;C:含同源臂的OVA靶片段PCR扩增结果;D:pCMV-OVA-SSA-mCherry-Hind Ⅲ载体构建中OVA靶序列测序结果,红色部分为408 bp含有sgRNA靶位点的OVA序列,黑色划线为PAM位点,蓝色方框为sgRNA序列 A: pYP152 vector digested by Bbs I, M is DNA Maker, 1 is pYP152 vector plasmid, 2 is the product of pYP152 vector plasmid digested by Bbs I; B: Sequencing result of sgRNA target site of pYP152-OVA-sgRNA vector; C: PCR amplification results of OVA target fragments containing homologous arms; D: OVA target sequence sequencing results of pCMV-OVA-SSA-mCherry-Hind Ⅲ vector construction, the red part is the 408 bp OVA sequence containing sgRNA target site, the black line is the PAM site, and the blue box is the sgRNA sequence 图 6 sgRNA表达载体和SSA报告载体构建结果 Fig. 6 Construction results of sgRNA expression vector and SSA reporter vector |
2.5 基于SSA报告载体检测稳转细胞株活性
将构建好的sgRNA表达载体和SSA报告载体共转稳定表达Cas9蛋白的DF-1细胞系,转染48 h后通过荧光显微镜观察每株细胞的SSA修复情况。由图 7可见,单转报告载体的细胞不表达红色荧光,说明SSA报告载体在插入一段外源序列后不表达红色荧光,而瞬时转染慢病毒载体Lenti-Blast-Cas9、sgRNA表达载体、SSA报告载体以及其他DF-1稳转株均有不同程度的红色荧光,表明筛选到的稳转Cas9蛋白的DF-1细胞系均可表达Cas9蛋白,且在sgRNA的引导下发挥切割DNA的作用,并诱发细胞的SSA修复机制,恢复红色荧光。

|
| BF、FITC和Red分别为荧光显微镜白光、绿色荧光和红色荧光拍摄下的细胞状态;阴性对照是转染pYP152-OVA-sgRNA和pCMV-OVA-SSA-mCherry-Hind Ⅲ的DF-1细胞;阳性对照是转染Lenti-Blast-Cas9、pYP152-OVA-sgRNA和pCMV-OVA-SSA-mCherry-Hind Ⅲ的DF-1细胞;Clone21、Clone25、Clone26和Clone4均为转染了pYP152-OVA-sgRNA和pCMV-OVA-SSA-mCherry-Hind Ⅲ的稳转Cas9蛋白的单克隆细胞 BF, FITC, and Red represent cell status captured by fluorescence microscopy under bright light, green fluorescence, and red fluorescence, respectively; Negative control is DF-1 cells transfected with pYP152-OVA-sgRNA and pCMV-OVA-SSA-mCherry-Hind Ⅲ; Positive control is DF-1 cells transfected with Lenti-Blast-Cas9, pYP152-OVA-sgRNA and pCMV-OVA-SSA-mCherry-Hind Ⅲ; Clone21, Clone25, Clone26, and Clone4 are monoclonal cells stably transfected with Cas9 protein, pYP152-OVA-sgRNA, and pCMV-OVA-SSA-mCherry-Hind Ⅲ 图 7 稳定表达Cas9蛋白细胞株转染结果(10×) Fig. 7 Transfection results of cell lines of stably expressing Cas9 protein (10×) |
3 讨论
慢病毒属于逆转录病毒,可以感染处于分裂期和非分裂期的多种类型细胞,常被用于携带大片段的外源基因,在逆转录酶的作用下高效整合到目的细胞基因组中[17],构成稳定表达外源基因的细胞系。DF-1细胞作为大多数禽类病毒易感细胞,常被用于研究禽病产生经过和发病机制[18]。本研究使用慢病毒载体Lenti-Blast-Cas9与其他两个慢病毒辅助质粒,在293T细胞中转染包装产生Cas9慢病毒,该慢病毒载体携带Blast抗性标签,感染DF-1细胞后筛选出具有Blast抗性的稳定表达Cas9蛋白的细胞,结合SSA报告载体证明该细胞具有产生DSB的能力,表明成功构建了稳定表达Cas9蛋白的DF-1细胞,这为今后研究鸡的功能基因以及构建鸡全基因组CRISPR/Cas9敲除细胞库奠定基础。
近年来,基于报告系统检测基因编辑系统中核酸酶、sgRNA活性的方式逐渐兴起。报告系统通常包括报告细胞和报告载体,其工作原理是利用基因编辑系统在报告基因编码区引起DSB,之后通过NHEJ、HDR或SSA修复抑制或者恢复报告基因表达,报告基因可选抗生素抗性基因、荧光蛋白等,可以直观、快速并灵敏地观察结果[14, 19]。SSA修复由同源重组介导且不需要额外引入模板链,在DSB位点存在重复序列时,双链断裂的末端会暴露单链DNA,这些单链DNA上的反向重复序列会通过互补配对形成环状DNA结构,再由内切酶和DNA聚合酶发挥作用使DNA双链断裂得到修复[20-21],从而恢复SSA载体中报告基因的表达[22]。本研究筛选到Blast阳性单克隆细胞后,为了快速检测稳转Cas9蛋白细胞的活性,采用SSA报告系统,实验流程简单,只需将已确定有活性的OVA sgRNA序列和构建含有OVA靶片段的SSA报告载体共转染稳定表达Cas9蛋白的细胞,48 h后即可直接观察到mCherry恢复表达的情况,SSA报告载体系统应用于检测核酸酶活性,避免了复杂的实验过程,从而节约时间和成本。
通过基因敲除或插入研究基因功能是必不可少的生物技术手段。CRISPR/Cas9系统适用于编辑任何细胞类型的基因,目前在人和小鼠方面已经建立了较为成熟的应用体系[23-25],采用CRISPR/Cas9系统研究猪的功能基因(如CD163、MSTN等)也已经有突破性进展[26-29],相关功能基因的敲除均产生了具有特定表型的猪。近年也有利用CRISPR/Cas9技术进行鸡已知功能基因的研究,研究表明,W38纯合敲除鸡可以抵抗禽白血病侵袭[30];DMRT1的敲除证明该基因与鸡睾丸发育至关重要[31-32]。目前已知的鸡生长性能、器官发育以及抗病性等相关功能基因还较少。CRISPR/Cas9系统在禽类研究方面的应用近几年才开始,且面临sgRNA脱靶、编辑效率低以及CRISPR递送方式困难等问题[33]。本研究结果表明,SSA报告载体系统在检测Cas9核酸酶活性方面发挥重要作用。在应用CRISPR/ Cas9进行基因编辑时,往往需要设计多条sgRNA进行验证,最终选择1条活性最高的sgRNA进行后续研究[34],因此后续可以利用SSA报告载体系统并结合稳转Cas9蛋白的DF-1细胞来快速筛选切割效率高的sgRNA。本研究中稳转Cas9蛋白的DF-1细胞不仅可以用于功能基因验证,还可用于高通量筛选功能基因。
4 结论本研究利用慢病毒载体系统获得长期稳定表达Cas9蛋白的鸡DF-1细胞系。鉴于SSA报告载体系统的高效性,本研究构建了SSA报告载体,结合CRISPR/Cas9系统的作用快速检测稳转细胞的修复活性。SSA报告系统结果显示,上述建立的稳定表达Cas9蛋白的DF-1细胞具有不同水平的SSA修复能力,表明本研究得到了稳定表达Cas9蛋白且具有Cas9核酸酶活性的DF-1细胞系,可为后续在鸡细胞水平上进行功能基因筛选及鉴定等相关研究奠定基础。
| [1] |
LIU Q, YANG F, ZHANG J, LIU H, RAHMAN S, ISLAM S, MA W, SHE M. Application of CRISPR/Cas9 in crop quality improvement[J]. International Journal of Molecular Sciences, 2021, 22(8): 4206. DOI:10.3390/ijms22084206 |
| [2] |
XU X, CHEMPARATHY A, ZENG L, KEMPTON H R, SHANG S, NAKAMURA M, QI L S. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing[J]. Molecular Cell, 2021, 81(20): 4333-4345. DOI:10.1016/j.molcel.2021.08.008 |
| [3] |
JASIN M, HABER J E. The democratization of gene editing: Insights from site-specific cleavage and double-strand break repair[J]. DNA Repair (Amst), 2016, 44: 6-16. DOI:10.1016/j.dnarep.2016.05.001 |
| [4] |
HER J, BUNTING S F. How cells ensure correct repair of DNA double-strand breaks[J]. Journal of Biological Chemistry, 2018, 293(27): 10502-10511. DOI:10.1074/jbc.TM118.000371 |
| [5] |
任代胜, 刘浩, 乔保建. 基于CRISPR/Cas9基因编辑技术靶向敲除OsFAD2创制高油酸水稻突变体[J]. 广东农业科学, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 REN D S, LIU H, QIAO B J. Targeted knockout of OsFAD2 based on CRISPR/Cas9 gene editing technology to create high oleic rice mutants[J]. Guangdong Agricultural Sciences, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 |
| [6] |
林春姿, 谢华斌, 黄其伟, 高上, 王加峰. 利用CRISPR/Cas9技术定向编辑水稻OsIQD1基因[J]. 广东农业科学, 2022, 49(8): 1-10. DOI:10.16768/j.issn.1004-874X.2022.08.001 LIN C Z, XIE H B, HUANG Q W, GAO S, WANG J F. Targeted editing of rice OsIQD1 gene using CRISPR/Cas9 technology[J]. Guangdong Agricultural Sciences, 2022, 49(8): 1-10. DOI:10.16768/j.issn.1004-874X.2022.08.001 |
| [7] |
许美娜, 朱奕舟, 林思远, 陈瑶生, 何祖勇. CRISPR/Cas9基因编辑技术在猪育种中的研究进展[J]. 广东农业科学, 2022, 49(8): 87-96. DOI:10.16768/j.issn.1004-874X.2022.08.011 XU M N, ZHU Y Z, LIN S Y, CHEN Y S, HE Z Y. Research progress of CRISPR/Cas9 gene editing technology in pig breeding[J]. Guangdong Agricultural Sciences, 2022, 49(8): 87-96. DOI:10.16768/j.issn.1004-874X.2022.08.011 |
| [8] |
姜爱文, 郭鸿运, 吴望军, 刘红林. 利用Cas9蛋白/sgRNA体系提高猪胎儿成纤维细胞基因组编辑效率[J]. 南京农业大学学报, 2022, 24(5): 1-13. DOI:10.7685/jnau.202112034 JIANG A W, GUO H Y, WU W J, LIU H L. Improving the genome editing efficiency of pig fetal fibroblasts using Cas9 protein/ sgRNA system[J]. Journal of Nanjing Agricultural University, 2022, 24(5): 1-13. DOI:10.7685/jnau.202112034 |
| [9] |
刘灵康, 郑喜邦. 基于CRISPR/Cas9介导的SSA修复途径检测核酸酶活性的研究//2019中国畜牧兽医学会兽医外科学分会第十届会员代表大会暨第24次学术研讨会[C]. 南宁: 中国畜牧兽医学会, 2019: 452-465. DOI: 10.26914/c.cnkihy.2019.049652. LIU L K, ZHENG X B. Detection of nuclease activity based on CRISPR/ Cas9-mediated SSA repair pathway//2019 The 10th Member Congress and 24th Academic Symposium of the Veterinary Surgery Branch of the Chinese Society of Animal Husbandry and Veterinary Medicine[C]. Nanning: Chinese Society of Animal Husbandry and Veterinary Medicine, 2019: 452-465. DOI: 10.26914/c.cnkihy.2019.049652. |
| [10] |
WANG C, CHENG J K W, ZHANG Q, HUGHES N W, XIA Q, WINSLOW M M, CONG L. Microbial single-strand annealing proteins enable CRISPR gene-editing tools with improved knock-in efficiencies and reduced off-target effects[J]. Nucleic Acids Research, 2021, 49(6): e36. DOI:10.1093/nar/gkaa1264 |
| [11] |
BAE W, HONG S, PARK M S, JEONG H K, LEE M H, KOO H S. Single-strand annealing mediates the conservative repair of double-strand DNA breaks in homologous recombination-defective germ cells of Caenorhabditis elegans[J]. DNA Repair (Amst), 2019, 75: 18-28. DOI:10.1016/j.dnarep.2019.01.007 |
| [12] |
YANG Y, LIU S, CHENG Y, NIE L, LYU C, WANG G, ZHANG Y, HAO L. Highly efficient and rapid detection of the cleavage activity of Cas9/gRNA via a fluorescent reporter[J]. Applied Biochemistry and Biotechnology, 2016, 180(4): 655-667. DOI:10.1007/s12010-016-2122-8 |
| [13] |
RAMAKRISHNA S, CHO S W, KIM S, SONG M, GOPALAPPA R, KIM J S, KIM H. Surrogate reporter-based enrichment of cells containing RNA-guided Cas9 nuclease-induced mutations[J]. Nature Communication, 2014, 5: 3378. DOI:10.1038/ncomms4378 |
| [14] |
KUHAR R, GWIAZDA K S, HUMBERT O, MANDT T, PANGALLO J, BRAULT M, KHAN I, MAIZELS N, RAWLINGS D J, SCHARENBERG A M, CERTO M T. Novel fluorescent genome editing reporters for monitoring DNA repair pathway utilization at endonuclease-induced breaks[J]. Nucleic Acids Research, 2014, 42(1): e4. DOI:10.1093/nar/gkt872 |
| [15] |
OISHI I, YOSHⅡ K, MIYAHARA D, KAGAMI H, TAGAMI T. Targeted mutagenesis in chicken using CRISPR/Cas9 system[J]. Scientific Reports, 2016, 6: 23980. DOI:10.1038/srep23980 |
| [16] |
黄思嘉, 祝梦琦, 李鹏程, 张智英, 魏泽辉. 利用CRISPR/Cas9技术对鸡AMHR2基因进行精确编辑[J]. 家畜生态学报, 2020, 41(6): 12-18. DOI:10.3969/j.issn.1673-1182.2020.003 HUANG S J, ZHU M Q, LI P C, ZHANG Z Y, WEI Z H. Precise editing of chicken AMHR2 gene using CRISPR/Cas9 technology[J]. Journal of Domestic Animal Ecology, 2020, 41(6): 12-18. DOI:10.3969/j.issn.1673-1182.2020.003 |
| [17] |
BORSOTTI C, BORRONI E, FOLLENZI A. Lentiviral vector interactions with the host cell[J]. Current Opinion in Virology, 2016, 21: 102-108. DOI:10.1016/j.coviro.2016.08.016 |
| [18] |
ANAM S, RAHMAN S U, ALI S, SAEED M, GOYAL S M. Comparative growth kinetic study of Newcastle disease virus, infectious bursal disease virus and avian influenza virus in chicken embryo fibroblast and DF-1 cell lines[J]. Polish Journal of Veterinary Sciences, 2021, 24(2): 287-292. DOI:10.24425/pjvs.2021.137664 |
| [19] |
AUBERT M, RYU B Y, BANKS L, RAWLINGS D J, SCHARENBERG A M, JEROME K R. Successful targeting and disruption of an integrated reporter lentivirus using the engineered homing endonuclease Y2 I-AniI[J]. PLoS One, 2011, 6(2): e16825. DOI:10.1371/journal.pone.0016825 |
| [20] |
MASHIKO D, FUJIHARA Y, SATOUH Y, MIYATA H, ISOTANI A, IKAWA M. Generation of mutant mice by pronuclear injection of circular plasmid expressing Cas9 and single guided RNA[J]. Scientific Reports, 2013, 3: 3355. DOI:10.1038/srep03355 |
| [21] |
LI X, BAI Y, CHENG X, KALDS P G T, SUN B, WU Y, LYU H, XU K, ZHANG Z. Efficient SSA-mediated precise genome editing using CRISPR/Cas9[J]. FEBS Journal, 2018, 285(18): 3362-3375. DOI:10.1111/febs.14626 |
| [22] |
ZHOU Y, LIU Y, HUSSMANN D, BRøGGER P, AL-SAAIDI R A, TAN S, LIN L, PETERSEN T S, ZHOU G Q, BROSS P, AAGAARD L, KLEIN T, RøNN S G, PEDERSEN H D, BOLUND L, NIELSEN A L, SøRENSEN C B, LUO Y. Enhanced genome editing in mammalian cells with a modified dual-fluorescent surrogate system[J]. Cellular and Molecular Life Sciences, 2016, 73(13): 2543-2563. DOI:10.1007/s00018-015-2128-3 |
| [23] |
SHALEM O, SANJANA N E, HARTENIAN E, SHI X, SCOTT D A, MIKKELSON T, HECKL D, EBERT B L, ROOT D E, DOENCH J G, ZHANG F. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343(6166): 84-87. DOI:10.1126/science.1247005 |
| [24] |
CONG L, ZHANG F. Genome engineering using CRISPR-Cas9 system[J]. Methods in Molecular Biology, 2015, 1239: 197-217. DOI:10.1007/978-1-4939-1862-1_10 |
| [25] |
ZHANG Z, ELSAYED A K, SHI Q, ZHANG Y, ZUO Q, LI D, LIAN C, TANG B, XIAO T, XU Q, CHANG G, CHEN G, ZHANG L, WANG K, WANG Y, JIN K, WANG Y, SONG J, CUI H, LI B. Crucial genes and pathways in chicken germ stem cell differentiation[J]. Journal of Biological Chemistry, 2015, 290(21): 13605-13621. DOI:10.1074/jbc.M114.601401 |
| [26] |
ZHAO C, LIU H, XIAO T, WANG Z, NIE X, LI X, QIAN P, QIN L, HAN X, ZHANG J, RUAN J, ZHU M, MIAO Y L, ZUO B, YANG K, XIE S, ZHAO S. CRISPR screening of porcine sgRNA library identifies host factors associated with Japanese encephalitis virus replication[J]. Nature Communication, 2020, 11(1): 5178. DOI:10.1038/s41467-020-18936-1 |
| [27] |
ZHANG J Q, GUO J X, WU X J, WANG Y, YUE W, L I ZH, WANG W, WEI X F, WU X L, SHEN Q Y, LEI A M, WEI H J, HUA J L. Optimization of sgRNA expression strategy to generate multiplex gene-edited pigs[J]. Zoological Research, 2022, 43(6): 1005. DOI:10.24272/j.issn.2095-8137.2022.244 |
| [28] |
WHITWORTH K M, LEE K, BENNE J A, BEATON B P, SPATE L D, MURPHY S L, SAMUEL M S, MAO J, O'GORMAN C, WALTERS E M, MURPHY C N, DRIVER J, MILEHAM A, MCLAREN D, WELLS K D, PRATHER R S. Use of the CRISPR/Cas9 system to produce genetically engineered pigs from in vitro-derived oocytes and embryos[J]. Biology of Reproduction, 2014, 91(3): 78. DOI:10.1095/biolreprod.114.121723 |
| [29] |
ZHU X X, ZHAN Q M, WEI Y Y, YAN A F, FENG J, LIU L, LU S S, TANG D S. CRISPR/Cas9-mediated MSTN disruption accelerates the growth of Chinese Bama pigs[J]. Reproduction in Domestic Animals, 2020, 55(10): 1314-1327. DOI:10.1111/rda.13775 |
| [30] |
KOSLOVA A, TREFIL P, MUCKSOVA J, REINISOVA M, PLACHY J, KALINA J, KUCEROVA D, GERYK J, KRCHLIKOVA V, LEJCKOVA B, HEJNAR J. Precise CRISPR/Cas9 editing of the NHE1 gene renders chickens resistant to the J subgroup of avian leukosis virus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(4): 2108-2112. DOI:10.1073/pnas.1913827117 |
| [31] |
WOODCOCK M E, GHEYAS A A, MASON A S, NANDI S, TAYLOR L, SHERMAN A, SMITH J, BURT D W, HAWKEN R, MCGREW M J. Reviving rare chicken breeds using genetically engineered sterility in surrogate host birds[J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(42): 20930-20937. DOI:10.1073/pnas.1906316116 |
| [32] |
IOANNIDIS J, TAYLOR G, ZHAO D, LIU L, IDOKO-AKOH A, GONG D, LOVELL-BADGE R, GUIOLI S, MCGREW M J, CLINTON M. Primary sex determination in birds depends on DMRT1 dosage, but gonadal sex does not determine adult secondary sex characteristics[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(10): 24696-24699. DOI:10.1073/pnas.2020909118 |
| [33] |
KHWATENGE C N, NAHASHON S N. Recent advances in the application of CRISPR/Cas9 gene editing system in poultry species[J]. Frontiers in Genetics, 2021, 12: 627714. DOI:10.3389/fgene.2021.627714 |
| [34] |
BAI Y, HE L, LI P, XU K, SHAO S, REN C, LIU Z, WEI Z, ZHANG Z. Efficient genome editing in chicken DF-1 cells using the CRISPR/ Cas9 system[J]. G3-Genes Genom Genet, 2016, 6(4): 917-923. DOI:10.1534/g3.116.027706 |
(责任编辑 崔建勋)
 2023, Vol. 50
2023, Vol. 50




