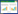文章信息
基金项目
- 广东省自然科学基金项目(2019A1515011825);广东省水稻育种新技术重点实验室2020年开放课题
作者简介
- 林春姿(1996-),女,在读硕士生,研究方向为水稻抗稻瘟病研究,E-mail:1162825005@qq.com.
通讯作者
- 王加峰(1979-),男,博士,副研究员,研究方向为水稻抗病分子育种,E-mail:jfwang@scau.edu.cn.
文章历史
- 收稿日期:2022-12-11
【研究意义】水稻(Oryza sativa L.) 是世界上重要的粮食作物,全球有近50% 的人以水稻为主食[1]。随着人口不断增加,粮食需求也相应增加,因此关注粮食生产安全显得尤为重要[2]。目前,传统的育种方法仍为提高水稻产量作出巨大贡献。但当前,农业正面临人口快速增长、全球气候变化、病虫害和其他环境危害等多重挑战[3],因此,迫切需要更先进、更完善的技术,开发出产量潜力更高的水稻新品种,增强生物胁迫抗性。多胺氧化酶(Polyamine oxidases,PAOs)是氧化分解多胺(Polyamines,PAs)的一种重要酶,在维持植物体内多胺的动态平衡中起着重要作用[4]。研究显示,PAs主要作用于亚精胺(Spermidine, Spd)和精胺(Spermine, Spm)等高级多胺,多胺氧化酶具有多个家族,根据功能不同PAOs大致可以分为3类:第一类可催化亚精胺与精胺,生成4- 氨基正丁醛或3- 氨丙基-4- 氨基正丁醛,同时生成1, 3- 丙二胺与H2O2,这些PAOs仅存在于植物中[5-7];第二类通过氧化动物体内N- 乙酰基衍生物二级氨基外侧面的碳以及植物体内非乙酰化的多胺,使PAs反向转化[8];第三类包含多胺氧化酶结构域,但没有氧化分解多胺的作用[9-13]。【前人研究进展】已有研究报道,植物受到干旱、盐渍化、高温低温、病原物侵染等逆境胁迫时,植物体内的多胺含量会发生显著变化,说明多胺的合成与代谢参与了植物对逆境胁迫的响应[5, 14]。而PAOs作为一种氧化分解多胺的重要酶,可能通过调节体内多胺水平来应对逆境胁迫。此外,PAOs氧化PAs过程中会产生H2O2,而H2O2是生物体细胞在代谢中产生的一种活性氧(Reactive oxygen species,ROS),适宜浓度的ROS具有免疫防御功能,也能作为第二信使调节细胞信号通路[15]。PAOs在烟草中已被证明是氧爆发的关键因素,其衍生产物H2O2可直接导致细胞程序性死亡(Programmed cell death, PCD) [16]。灰霉病菌丝内PAOs活性的增加会生成大量H2O2,进而对菌丝体造成氧化损伤,并阻碍其生长[17]。SlPAO4通过触发H2O2介导的Spd增加番茄对盐碱胁迫的耐受性[18]。PAs代谢可以提高玉米黄曲霉病抗性[19]。OsPAO4可促进γ- 氨基丁酸(GABA)的生成,而γ- 氨基丁酸转氨酶(GABA-T)催化GABA转化为琥珀酸半醛,且OsGABA-T表达受稻瘟菌所诱导[20]。此外,OsPAO5被证实是水稻中胚轴伸长的负调控因子,定点突变能促进直播水稻的胚轴伸长、提高出苗速率,同时还可以提高粒重等产量性状,敲除该基因后会导致其乙烯含量升高[21]。RNAi抑制草莓FaPAO5的表达,可促进Spd、Spm和脱落酸(Abscisic acid, ABA)的积累,同时抑制H2O2的产生[22]。水稻OsPAO4属于PAOs家族的一员,可促进GABA的生成[23],但在水稻抗病免疫反应中的作用尚未见报道。【本研究切入点】基因组编辑技术的准确性和效率不断提高[24]。现有的基因组编辑系统包括锌指核酸酶系统(Zinc-finger nucleases,ZFNs)、转录激活子样效应核酸酶系统(Transcription activator-like effector nucleases,TALENs)和聚类规则间隔短回文重复相关蛋白系统(CRISPR/Cas)。ZFNs和TALENs由于系统组装技术困难、目标范围窄、效率低、成本高,逐渐被CRISPR/Cas系统所取代[25]。CRISPR/Cas9系统是目前应用最广泛的Ⅱ型基因组编辑技术[26]。在过去几年里,应用CRISPR/Cas9编辑系统进行水稻遗传改良,显著提高了水稻的产量、品质及抗病能力[27–29]。此外,CRISPR/Cas9编辑系统显著推动水稻功能基因、转录调控及水稻分子育种研究[30–33]。【拟解决的关键问题】本研究利用CRISPR/Cas9技术对OsPAO4基因实行定点编辑,构建水稻OsPAO4的突变体材料。研究结果可为后续开展OsPAO4基因功能及作物抗逆育种奠定重要基础。
1 材料与方法 1.1 试验材料供试水稻材料为粳稻Pik-H4 NIL(本实验室利用诱变株系H4与丽江新团黑谷LTH构建的含有抗稻瘟病基因Pik-H4的近等基因系)。Cas9- gRNA表达盒载体pGTR和pRGEB32购自中国北京Addgene公司。试验所需的编辑靶点引物、核酸测序服务分别由生工生物工程(上海)股份有限公司、北京擎科生物科技有限公司完成。Bsa Ⅰ内切酶、Fok Ⅰ内切酶、T4 DNA连接酶购自NEB(北京)有限公司,Phanta Super-Fidelity DNA Polymerase高保真酶购自南京诺唯赞生物科技股份有限公司。大肠杆菌DH5α、根癌农杆菌EHA105均购自中国北京擎科生物科技有限公司。
1.2 OsPAO4蛋白功能预测分析首先采用NCBI-BLAST分析OsPAO4蛋白的保守结构域,然后利用ExPASy-Protparam在线平台分析OsPAO4蛋白的理化性质,最后分别应用SOMPA和SWISS-MODE对OsPAO4蛋白的二级结构与三级结构进行预测分析。
1.3 OsPAO4基因靶点选择及gRNA设计利用CRISPR-GE网站(http://skl.scau. edu.cn/targetdesign/) 对OsPAO4基因gRNA靶位点进行设计:首先按照NGG序列,与OsPAO4基因序列进行对比后筛选靶点,然后在PLN02268结构域(第2外显子)处设计1个靶点(5'-ACCATTGAGAGGCAATGTGC-3')。通过Cas-OFFinder(http://www.rgenome.net/casoffinder/)分析和水稻基因组对比分析验证确定OsPAO4的目的靶点,以检验其特异性。
1.4 CRISPR/Cas9表达载体构建CRISPR/Cas9敲除载体构建参考Xie等[34]方法。根据靶点序列设计相应引物(表 1)。以pGTR载体为模板,分别用L5AD5-F引物和OsPAO4-TARGET-R引物、OsPAO4-TARGET-F引物和L3AD5-R引物扩增相应片段。对扩增片段分别进行纯化后,用Bsa Ⅰ、T7连接酶组装,然后再用引物(S5AD5-NEW-F和S3AD5-NEW-R) 对组装产物进行PCR扩增。扩增产物纯化后用Fok Ⅰ单酶切,最终得到gRNA- 靶点-tRNA片段,进一步通过重组方法将该gRNA片段克隆至Cas9蛋白表达载体pRGEB32,把重组载体转化大肠杆菌DH5α后,挑取几个单菌落摇菌,提取质粒,进行双酶切鉴定和菌落PCR,挑取符合预期片段大小的PCR产物送至北京擎科生物有限公司测序,根据测序结果分析得到OsPAO4基因编辑载体。
1.5 表达载体遗传转化及阳性植株鉴定
选用饱满一致的Pik-H4 NIL种子诱导出愈伤组织,采用农杆菌介导的方法侵染水稻愈伤组织,经潮霉素筛选、分化获得转基因抗性植株。利用CTAB法提取T0代植株DNA,以特异性潮霉素引物(HPT-F/HPT-R)进行PCR扩增(表 1),可扩增出501 bp条带的植株,此为转基因阳性株系。
1.6 突变位点PCR扩增检测及突变类型分析为检测OsPAO4编辑靶点的突变情况,分别在靶点的上下游设计测序引物OsPAO4-T1-F、OsPAO4-T1-R(表 1),对提取的水稻基因组DNA(见1.5中所述)进行PCR扩增(目的片段长度为483 bp),扩增片段送至北京擎科生物有限公司进行测序分析,将测序结果与OsPAO4基因组序列进行比对,分析鉴定转基因植株的基因突变类型。
1.7 过氧化氢酶(Catalase,CAT)活性测定利用CAT测定试剂盒(南京建成生物工程研究所,中国)测定样品中CAT活性。具体操作步骤依据说明书进行:准确称取水稻叶片,用剪刀剪碎后置于研钵中,按照质量体积比1 : 9加入9倍体积的生理盐水,然后在冰浴条件下将叶片研磨至匀浆。将匀浆液用离心管收集后,用冷冻离心机在4℃、10 000 r/min下离心10 min,取10% 上清匀浆进行测定,然后按照说明书分别制作对照管、测定管,3次重复,计算公式如下:
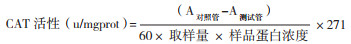
|
OsPAO4蛋白的理化性质分析结果表明,该蛋白包含487个氨基酸,分子量为52.91 kD,理论等电点(pI)和总平均亲水性分别为5.29和-0.041,不稳定系数为32.73,表明OsPAO4蛋白偏酸性且亲水、稳定。OsPAO4蛋白中丰度最高的氨基酸为亮氨酸(Leu),含有49个且占比达10.1%,
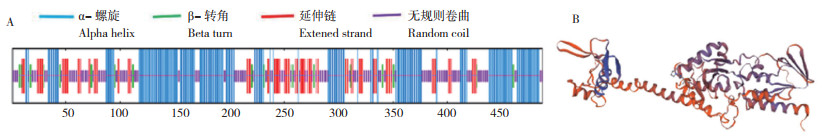
其次是丙氨酸(Ala)和缬氨酸(Val),分别含有47和42个,各占氨基酸总数的9.7% 和8.6%。OsPAO4蛋白保守结构域分析结果(图 1) 显示其在第24位到第458位氨基酸之间具有一个典型的PLN02268保守结构域,表明该基因属于PAOs家族。进一步预测其蛋白二级结构与三级结构,发现OsPAO4蛋白中包含42.30% 的α-螺旋、5.75% 的β- 转角、18.07% 延伸链以及33.88% 无规则卷曲(图 2)。

|
| 图 1 OsPAO4蛋白保守结构域预测 Fig. 1 Conserved domains prediction of OsPAO4 protein |
|
| 图 2 OsPAO4蛋白二级结构(A)与三级结构(B)预测 Fig. 2 Prediction of the secondary (A) and tertiary (B) structures of OsPAO4 protein |
2.2 gRNA靶点选择和序列设计
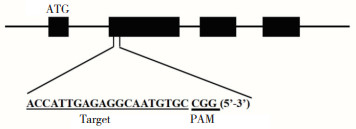
OsPAO4位于水稻第4号染色体上,基因组序列全长为3 392 bp,CDS为1 464 bp,含有9个外显子和8个内含子,其中2号外显子最长、为430 bp,1号外显子最短、仅28 bp。为破坏OsPAO4蛋白PLN02268保守结构域的功能,利用CRISPR-GE网站(http://skl.scau.edu.cn/targetdesign/)在OsPAO4第2个外显子区域设计一个靶点(5'-AC CATTGAGAGGCAATGTGC CGG - 3')进行定点编辑(图 3)。
|
| 图 3 OsPAO4基因结构和靶位点位置 Fig. 3 Gene structure of OsPAO4 and the target site location |
2.3 CRISPR/Cas9表达载体构建
将组装好的gRNA表达盒替换Cas9载体骨架中的gRNA scaffold片段,得到pRGEB32- OsPAO4-gRNA表达载体(图 4A)。挑取几个单菌落,摇菌,提取质粒进行双酶切鉴定及菌落PCR鉴定,符合要求的单菌落进行测序鉴定。利用引物pRGEB32-CX-F对载体上的靶点gRNA表达盒进行测序,结果显示,靶点gRNA表达盒序列正确克隆至pRGEB32上,可用于下一步农杆菌介导的水稻遗传转化(图 4B)。
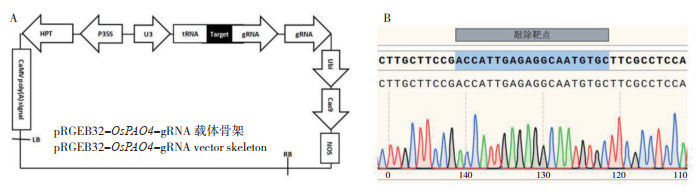
|
| 图 4 pRGEB32-OsPAO4-gRNA表达载体组装(A)及靶点测序鉴定(B) Fig. 4 Assembly diagram (A) and target identification sequences (B) inserted in pRGEB32-OsPAO4-gRNA recombinant vector |
2.4 转基因阳性植株鉴定
利用农杆菌介导法将pRGEB32-OsPAO4- gRNA重组表达载体转化到Pik-H4 NIL的愈伤组织,经再分化、抗性筛选得到29个转化株系,提取每个株系的基因组DNA,利用PCR扩增检测潮霉素(Hygromycin,HPT Ⅱ)抗性基因。结果(图 5)显示,共有25株转基因植株可以检测到潮霉素抗性基因,表明成功将外源质粒pRGEB32- OsPAO4-gRNA转化到水稻中,转化率约86.2%。
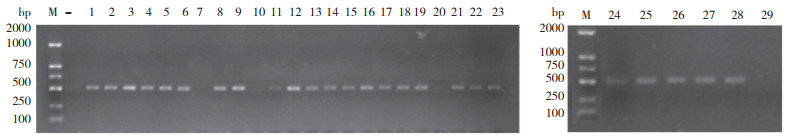
|
| “M”代表marker,“-”代表以野生型DNA为模板的阴性对照,泳道1~29代表OsPAO4对应的第1~29株转基因株系 "M" represents marker. "-" represents the negative control using wild-type DNA as template. Lane "1-29" represent the 1-29 transgenic plants corresponding to OsPAO4 图 5 OsPAO4转基因株系潮霉素抗性基因(HPT Ⅱ) PCR鉴定 Fig. 5 HPT Ⅱ identification of the positive transgenic OsPAO4 plants by PCR amplification |
2.5 突变类型分析
为明确OsPAO4的编辑类型,对获得的25株T0代转基因阳性植株的目标基因靶点区域序列设计特异性引物(OsPAO4-T1-F与OsPAO4- T1-R),并进行PCR扩增和测序分析(图 6~ 图 7)。对比野生型Pik-H4 NIL中该基因序列,参考OsPAO4转基因株系中靶点附近序列的测序峰图,并利用解码网站(http://skl.scau.edu.cn/dsdecode/) 对全部转基因植株测序结果进行解码分析。结果(表 2)表明,25株中有3株为纯合突变,18株为双峰,4株检测结果与野生型一致、即靶点处未发生基因编辑。在纯合突变体ospao4-3中该基因存在2个碱基缺失,导致氨基酸移码突变,并未引起提前终止,但是后面氨基酸排列发生较大改变;ospao4-17中发生单碱基的缺失同时导致移码突变,使突变位点后16位氨基酸处发生终止,变成仅有27个氨基酸的小肽;ospao4-24中靶标处发生单碱基的的插入突变,导致翻译提前终止、蛋白变短,仅包含17个氨基酸。综上所述,纯合突变株系在T0代时目的基因功能可能完全丧失,还需结合后续试验进行验证,而杂合突变则需要等T1、T2代分离后继续测序分析。
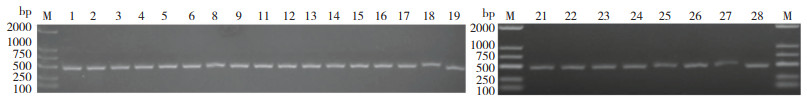
|
| “M”代表marker;泳道上的数字代表OsPAO4对应的转基因株系 "M" represents marker. The numbers in the lane represent the transgenic plants corresponding to OsPAO4 图 6 OsPAO4突变位点PCR扩增检测 Fig. 6 PCR amplification detection of OsPAO4 mutation site |
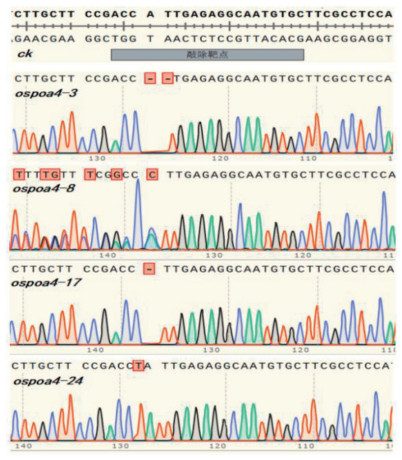
|
| 图 7 部分转基因株系OsPAO4靶点区域序列的测序比对分析 Fig. 7 Sequencing comparison analysis chart of the OsPAO4 target region sequences of some transgenic plants |
2.6 籽粒颖壳颜色表型分析
对T0代植株的生长表型及籽粒颖壳颜色调查结果发现,杂合类型植株(ospao4-8)的籽粒结实率未发生变化,但植株分蘖明显增多,且颖壳颜色由紫色变为深灰色,而其他突变类型的杂合株系表型均没有明显改变(图 8),可能杂合突变影响了花青素的积累,具体发生机制需要结合T1、T2代表型进行分析。

|
| 图 8 野生型Pik-H4NIL(A)和杂合株系ospao4-8/OsPAO4(B)的籽粒颜色 Fig. 8 Glume color of the wild-type Pik-H4 NIL (A) and a ospao4-8/OsPAO4 mutant (B) |
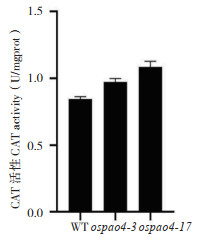
2.7 CAT活性分析
PAOs氧化PAs过程中会产生H2O2,而H2O2是生物体细胞在代谢中产生的一种活性氧(ROS),适宜浓度的ROS能作为第二信使调节细胞信号通路,激活植物防御功能[14]。而CAT可分解H2O2,检测转基因株系ospao4-3、ospao4-17和野生型植株Pik-H4 NIL中CAT活性有助于了解植株体内H2O2水平。已知CAT可以将H2O2分解为H2O和O2,使细胞免遭H2O2毒害。本研究结果(图 9)发现,ospao4突变会导致CAT活性升高,因此推测ospao4突变会促进H2O2分解,降低ROS水平,进而增强水稻敏感性。
|
| 图 9 野生型和ospao4突变体中CAT活性 Fig. 9 CAT activity in wild type and ospao4 mutants |
3 讨论
生物和非生物胁迫可造成粮食作物产量大幅降低,提高植物的抗逆性是当前育种的主要目标。其中CRISPR/Cas9系统因其简单、对靶标切割的高特异性、不涉及复杂的蛋白质化学和普遍适用性而被育种研究人员广泛接受[35],已在水稻、玉米、小麦等作物重要性状的改良中得到应用[36]。
PAs是一种重要的植物生长调节物质。在植物受到病原入侵时,体内迅速积累大量PAs并相互转化,一部分PAs由二胺氧化酶(Diamine oxidase, DAO)氧化,DAOs主要催化氧化腐胺(Putrescine, Put) 和尸胺(Cadaverine, Cad) 产生4- 氨基丁醛、氨气(Ammonia,NH3) 和H2O2[37-38];另一部分被PAOs氧化分解,引起下游相关抗病基因和细胞死亡相关基因的表达等来响应这些生物胁迫[39],如下游物质γ- 氨基丁酸(GABA) 在环境胁迫下迅速积累。内源GABA可影响植物生长;外源GABA可参与植物信号转导、转录调控、激素生物合成,刺激ROS产生(适宜浓度),进而增强植物抗病性[40]。PAs在PAOs催化下的最终产物为H2O2,但在PAs代谢过程中,参与调控植物免疫途径的基因仍未可知。目前已在水稻中发现7种PAOs基因[8],但关于OsPAO4功能的报道较少,因此研究OsPAO4的功能也有利于进一步解析PAs在抗逆方面的作用。此外,前人研究表明ROS是防御稻瘟病菌侵染的关键因素[8]。PAOs活性会影响细胞内PAs水平,导致H2O2水平的改变,进而影响ROS爆发[4],所以PAOs可能与水稻抗病性相关。
本研究通过对OsPAO4蛋白进行保守结构域预测,发现OsPAO4蛋白拥有PAOs家族高度保守的PLN02268结构域。为探究OsPAO4基因的生物学功能,利用CRISPR/Cas9技术在PLN02268结构域设计单编辑靶点(第2外显子),对抗稻瘟病近等基因系Pik-H4 NIL中的OsPAO4基因进行定点编辑,成功获得25株阳性转基因T0代植株。通过对靶点区域序列测序,发现T0代存在多种突变类型,如纯合突变、杂合突变、双等位突变等,单靶点编辑对OsPAO4基因也具有较高的编辑效率。进一步分析发现,这些突变体中大部分因缺失或插入的碱基数不是3的倍数,从而产生移码突变或导致蛋白翻译提前终止。同时,本研究获得一种杂合突变类型ospao4-8,其颖壳呈深灰色(原始材料Pik-H4 NIL颖壳为紫色),推测ospao4基因杂合突变影响了颖壳中花青素的积累。植物中花青素的产生受到光、温度、碳水化合物、植物激素和胁迫等多种因素的影响[41],PAs与乙烯合成前体均为S- 腺苷甲硫氨酸(S-Adenosylmethionine, SAM) [42],ospao4的杂合突变可能部分降低了PAs的水平、促进了乙烯的产生,进而促进水稻的成熟及花青素的积累。此外,有研究指出,氮含量会影响花青素的合成,而PAs可以替代无机氮促进其生长[43],因此,ospao4杂合突变的颖壳颜色变化可能是由于PAs水平的改变间接影响了花青素的积累,进而改变颖壳颜色。
已有研究表明,PAs氧化产生的H2O2参与了超敏反应(Hypersensitive response,HR)途径,从而使得细菌对病原体具有耐受性[44]。外源施加Spm可增强拟南芥对黄瓜花叶病毒的抗性,而利用PAOs抑制剂可抑制防御基因的激活,以上实验均证实PAO参与抗性反应[45]。由于PAOs活性的改变会影响细胞内PAs水平,进而导致H2O2水平发生改变[4]。而CAT可促使H2O2降解,因此测定CAT活性有助于间接了解突变体植物体内H2O2的水平。本研究CAT活性测定结果显示,突变体中CAT活性相较于野生型有所升高,可能是由于ospao4突变后会促使植物体内H2O2被降解和清除,降低ROS水平,进而增强了水稻对病原菌的敏感性,初步证实OsPAO4可能通过影响PAs水平调控H2O2的积累,进而参与水稻抗病反应。
综上所述,利用CRISPR/Cas9技术获得了类型较为丰富的ospao4突变体,为研究水稻OsPAO4基因介导的抗病分子机制提供了丰富的突变材料。CRISPR/Cas9技术可以定点编辑植物的内源基因,产生多样的突变体,获得丰富的水稻育种材料,将突变体经过不断回交等一系列手段消除转基因的痕迹,结合传统的作物育种,将极大加快育种进程。值得关注的是,发展非组织培养依赖的基因编辑技术,并降低编辑系统的脱靶效应仍是该技术推广应用的关键,需大力投入和积极开展相关研究。
4 结论本研究利用CRISPR/Cas9基因编辑技术,设计靶向OsPAO4基因PLN02268结构域(第2外显子)处的单靶点,对OsPAO4基因进行定点编辑,并获得ospao4纯合突变体材料。ospao4杂合突变体ospao4-8在幼苗时期的长势与野生型Pik-H4 NIL大致相同,但植株分蘖明显增多,且籽粒颜色由紫色变为深灰色,但其他突变类型的杂合株系表型未发生明显改变。同时,与野生型Pik-H4 NIL相比,ospao4突变体中CAT活性有所升高,H2O2被分解和清除,推测OsPAO4可能参与水稻基础免疫过程的调控,为进一步深入揭示OsPAO4基因的功能和分子调控机制奠定了重要基础。
| [1] |
BIRLA D S, MALIK K, SAINGER M, CHAUDHARY D, JAIWAL R, JAIWAL P K. Progress and challenges in improving the nutritional quality of rice (Oryza sativa L.)[J]. Critical Reviews in Food Science and Nutrition, 2017, 57(11): 2455-2481. DOI:10.1080/10408398.2015.1084992 |
| [2] |
MILOVANOVIC V, SMUTKA L. Asian countries in the global rice market[J]. Acta Universitatis Agriculturae et Silviculturae Mendelianae Brunensis, 2017, 65: 679-688. DOI:10.11118/actaun201765020679 |
| [3] |
MISHRA R, JOSHI R K, ZHAO K. Genome editing in rice: Recent advances, challenges, and future implications[J]. Frontiers in Plant Science, 2018, 9: 1361. DOI:10.3389/fpls.2018.01361 |
| [4] |
YU Z, JIA D, LIU T. Polyamine oxidases play various roles in plant development and abiotic stress tolerance[J]. Plants, 2019, 8(6): 184. DOI:10.3390/plants8060184 |
| [5] |
朱雪峰. 棉花GhPAO3基因抗旱功能初步分析[D]. 石河子: 石河子大学, 2019. ZHU X F. Preliminary analysis of drought resistance function of cotton GhPAO3 gene[D]. Shihezi: Shihezi University, 2019. |
| [6] |
CERVELLI M, BIANCHI M, CONA A, CROSATTI C, STANCA M, ANGELINI R, FEDERICO R, MARIOTTINI P. Barley polyamine oxidase isoforms 1 and 2, a peculiar case of gene duplication: Barley polyamine oxidase isoform 1 gene expression[J]. FEBS Journal, 2006, 273(17): 3990-4002. DOI:10.1111/j.1742-4658.2006.05402.x |
| [7] |
MOSCHOU P N, PASCHALIDIS K A, ROUBELAKIS-ANGELAKIS K A. Plant polyamine catabolism: The state of the art[J]. Plant Signaling & Behavior, 2008, 3(12): 1061-1066. DOI:10.4161/psb.3.12.7172 |
| [8] |
ONO Y, KIM D W, WATANABE K, SASAKI A, NIITSU M, BERBERICH T, KUSANO T, TAKAHASHI Y. Constitutively and highly expressed Oryza sativa polyamine oxidases localize in peroxisomes and catalyze polyamine back conversion[J]. Amino Acids, 2012, 42(2-3): 867-876. DOI:10.1007/s00726-011-1002-3 |
| [9] |
SHI Y, LAN F, MATSON C, MULLIGAN P, WHETSTINE J R, COLE P A, CASERO R A, SHI Y. Histone demethylation mediated by the nuclear amine oxidase homolog lsd1[J]. Cell, 2004, 119(7): 941-953. DOI:10.1016/j.cell.2004.12.012 |
| [10] |
SPEDALETTI V, POLTICELLI F, CAPODAGLIO V, SCHININA M E, STANO P, FEDERICO R, TAVLADORAKI P. Characterization of a lysine-specific histone demethylase from Arabidopsis thaliana[J]. Biochemistry, 2008, 47(17): 4936-4947. DOI:10.1021/bi701969k |
| [11] |
LUO M, HUNG F Y, YANG S G, LIU X C, WU K Q. Histone lysine demethylases and their functions in plants[J]. Plant Molecular Biology Reporter, 2014, 32(2): 558-565. DOI:10.1007/s11105-013-0673-1 |
| [12] |
MOSAMMAPARAST N, SHI Y. Reversal of histone methylation: Biochemical and molecular mechanisms of histone demethylases[J]. Annual Review of Biochemistry, 2010, 79(1): 155-179. DOI:10.1146/annurev.biochem.78.070907.103946 |
| [13] |
FORTES A M, AGUDELO-ROMERO P, PIMENTEL D, ALKAN N. Transcriptional modulation of polyamine metabolism in fruit species under abiotic and biotic stress[J]. Frontiers in Plant Science, 2019, 10: 816. DOI:10.3389/fpls.2019.00816 |
| [14] |
KEREN I, LAPIDOT M, CITOVSKY V. Coordinate activation of a target gene by KDM1C histone demethylase and OTLD1 histone deubiquitinase in Arabidopsis[J]. Epigenetics, 2019, 14(6): 602-610. DOI:10.1080/15592294.2019.1603982 |
| [15] |
HUANG H, ULLAH F, ZHOU D X, YI M, ZHAO Y. Mechanisms of ROS regulation of plant development and stress responses[J]. Frontiers in Plant Science, 2019, 10: 800. DOI:10.3389/fpls.2019.00800 |
| [16] |
YODA H, YAMAGUCHI Y, SANO H. Induction of hypersensitive cell death by hydrogen peroxide produced through polyamine degradation in tobacco plants[J]. Plant Physiology, 2003, 132(4): 1973-1981. DOI:10.1104/pp.103.024737 |
| [17] |
杨康, 陈健, 辛爱景, 蔡金霞, 石志琦, 杨立飞. 麝香草酚抑制灰霉菌的作用机理: PAO-H2O2系统[J]. 应用生态学报, 2020, 31(7): 2441-2448. DOI:10.13287/j.1001-9332.202007.031 YANG K, CHEN J, XIN A J, CAI J X, SHI Z Q, YANG L F. Mechanism of thymol inhibiting Botrytis cinerea: PAO-H2O2 system[J]. Chinese Journal of Applied Ecology, 2020, 31(7): 2441-2448. DOI:10.13287/j.1001-9332.202007.031 |
| [18] |
YANG J Y, WANG P J, LI S Z, LIU T, HU X H. Polyamine oxidase triggers H2O2-mediated spermidine improved oxidative stress tolerance of tomato seedlings subjected to saline-alkaline stress[J]. International Journal of Molecular Sciences, 2022, 23(3): 1625. DOI:10.3390/ijms23031625 |
| [19] |
MAJUMDAR R, MINOCHA R, LEBAR M D, RAJASEKARAN K, LONG S, CARTER-WIENTJES C, MINOCHA S, CARY J W. Contribution of maize polyamine and amino acid metabolism toward resistance against Aspergillus flavus infection and aflatoxin production[J]. Frontiers in Plant Science, 2019, 10: 692. DOI:10.3389/fpls.2019.00692 |
| [20] |
WU C X, ZHOU S Y, ZHANG Q, ZHAO W S, PENG Y L. Molecular cloning and differential expression of an γ-aminobutyrate transaminase gene, OsGABA-T, in rice (Oryza sativa) leaves infected with blast fungus[J]. Journal of Plant Research, 2006, 119(6): 663-669. DOI:10.1007/s10265-006-0018-3 |
| [21] |
LYU Y S, SHAO G N, JIAO G A, SHENG Z H, XIE L H, HU S K, TANG S Q, WEI X J, HU P S. Targeted mutagenesis of POLYAMINE OXIDASE 5 that negatively regulates mesocotyl elongation enables the generation of direct-seeding rice with improved grain yield[J]. Molecular Plant, 2021, 14(2): 344-351. DOI:10.1016/j.molp.2020.11.007 |
| [22] |
MO A, XU T, BAI Q, SHEN Y Y, GAO F, GUO J X. FaPAO5 regulates Spm/Spd levels as a signaling during strawberry fruit ripening[J]. Plant Direct, 2020, 4(5)[2023-02-03]. https://onlinelibrary.wiley.com/doi/10.1002/pld3.217 DOI: 10.1002/pld3.217.
|
| [23] |
BRIKIS C J, ZAREI A, CHIU G Z, DEYMAN K L, LIU J Y, TROBACHER C P, HOOVER G J, SUBEDI S, DEEII J R, BOZZO G G, SHELP B J. Targeted quantitative profiling of metabolites and gene transcripts associated with 4-aminobutyrate (GABA) in apple fruit stored under multiple abiotic stresses[J]. Horticulture Research, 2018, 5(1): 61. DOI:10.1038/s41438-018-0069-3 |
| [24] |
JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA J A, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821. DOI:10.1126/science.1225829 |
| [25] |
李文龙, 栾鑫, 张强, 余宁, 冯晓敏, 刘志霞. 基于CRISPR/Cas9基因编辑技术的水稻定向改良研究进展[J]. 广东农业科学, 2022, 49(9): 114-124. DOI:10.16768/j.issn.1004-874X.2022.09.012 LI W L, LUAN X, ZHANG Q, YU N, FENG X M, LIU Z X. Target improvement of rice based on CRISPR/Cas9 gene editing technology[J]. Guangdong Agricultural Sciences, 2022, 49(9): 114-124. DOI:10.16768/j.issn.1004-874X.2022.09.012 |
| [26] |
CONG L, RAN F A, COX D, LIN S, BARRETTO R, HABIB N, HSU P D, WU X, JIANG W, MARRAFFINI L A, ZHANG F. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819-823. DOI:10.1126/science.1231143 |
| [27] |
ZHOU J P, XIN X H, HE Y, CHEN H Q, LI Q, TANG X, ZHONG Z H, DENG K J, ZHENG X L, AKHER S A, CAI G Z, QI Y P, ZHANG Y. Multiplex QTL editing of grain-related genes improves yield in elite rice varieties[J]. Plant Cell Reports, 2019, 38(4): 475-485. DOI:10.1007/s00299-018-2340-3 |
| [28] |
WANG F J, WANG C L, LIU P Q, LEI C L, HAO W, GAO Y, LIU Y G, ZHAO K J. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J]. PloS one, 2016, 11(4): e0154027. DOI:10.1371/journal.pone.0154027 |
| [29] |
SHEN L, HUA Y F, FU Y P, LI J, LIU Q, JIAO X Z, XIN G W, WANG J J, WANG X C, YAN C J, WANG K J. Rapid generation of genetic diversity by multiplex CRISPR/Cas9 genome editing in rice[J]. Science China Life Sciences, 2017, 60(5): 506-515. DOI:10.1007/s11427-017-9008-8 |
| [30] |
林春姿, 谢华斌, 黄其伟, 高上, 王加峰. 利用CRISPR/Cas9技术定向编辑水稻OsIQD1基因[J]. 广东农业科学, 2022, 49(8): 1-10. DOI:10.16768/j.issn.1004-874X.2022.08.001 LIN C Z, XIE H B, HUANG Q W, GAO S, WANG J F. Targeted editing of OsIDQ1 gene in rice based on CRISPR/Cas9 technology[J]. Guangdong Agricultural Sciences, 2022, 49(8): 1-10. DOI:10.16768/j.issn.1004-874X.2022.08.001 |
| [31] |
段敏, 谢留杰, 高秀莹, 唐海娟, 黄善军, 潘晓飚. 利用CRISPR/Cas9技术创制广亲和水稻温敏雄性不育系TB52S[J/OL]. 中国水稻科学, 2023https//kns.cnki.net/kcms/detail//33.1146.S.20230106.1517.011.html. [2013-01-09]. DUAN M, XIE L J, GAO X Y, TANG H J, HUANG S J, PAN X B. Construction of wide compatibility thermo-sensitive genic male sterility TB52S in rice based on CRISPR/Cas9 technology[J/OL]. Chinese Journal of Rice Science, 2023https//kns.cnki.net/kcms/detail//33.1146.S.20230106.1517.011.html. [2013-01-09]. |
| [32] |
牛淑琳, 鞠培娜, 周冠华, 戴南平, 周晋军, 谢先芝, 郑崇珂. 利用CRISPR/Cas9技术编辑OsRR22基因创制耐盐水稻种质资源[J]. 山东农业科学, 2023, 55(2): 30-35. DOI:10.14083/j.issn.1001-4942.2023.02.004 NIU S L, JU P N, ZHOU G H, DAI N P, ZHOU J J, XIE X Z, ZHENG C K. Creation of salt-tolerant rice germplasm by editing OsRR22 gene via CRISPR/Cas9[J]. Shandong Agricultural Sciences, 2023, 55(2): 30-35. DOI:10.14083/j.issn.1001-4942.2023.02.004 |
| [33] |
李景芳, 温舒越, 赵利君, 陈庭木, 周振玲. 基于CRISPR/Cas9技术创制耐盐香味水稻[J/OL]. 中国水稻科学, 2023http://kns.cnki.net/kcms/detail//33.1146.S.20230106.1454.010.html. [2023-01-09]. LI J F, WEN S Y, ZHAO L J, CHEN T M, ZHOU Z L. Production of salt-tolerant and fragrant rice based on CRISPR/Cas9 technology[J]. Chinese Journal of Rice Science, 2023http://kns.cnki.net/kcms/detail//33.1146.S.20230106.1454.010.html. [2023-01-09]. . |
| [34] |
XIE K, MINKENBERG B, YANG Y. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system[J]. Proceedings of the National Academy of Sciences, 2015, 112(11): 3570-3575. DOI:10.1073/pnas.1420294112 |
| [35] |
ZAIDI S S E A, MUKHTAR M S, MANSOOR S. Genome editing: Targeting susceptibility genes for plant disease resistance[J]. Trends in Biotechnology, 2018, 36(9): 898-906. DOI:10.1016/j.tibtech.2018.04.005 |
| [36] |
王福军, 赵开军. 基因组编辑技术应用于作物遗传改良的进展与挑战[J]. 中国农业科学, 2018, 51(1): 1-16. DOI:10.3864/j.issn.0578-1752.2018.01.001 WANG F J, ZHAO K J. Progress and challenge of crop genetic improvement via genome editing[J]. Scientia Agricultura Sinica, 2018, 51(1): 1-16. DOI:10.3864/j.issn.0578-1752.2018.01.001 |
| [37] |
ALCÁZAR R, ALTABELLA T, MARCO F, BORTOLOTTI C, REYMOND M, KONCZ C, CARRASCO P, TIBURCIO A F. Polyamines: molecules with regulatory functions in plant abiotic stress tolerance[J]. Planta, 2010, 231(6): 1237-1249. DOI:10.1007/s00425-010-1130-0 |
| [38] |
MOSCHOU P N, WU J, CONA A, TAVLADORAKI P, ANGELINI R, ROUBELAKIS-ANGELAKIS K A. The polyamines and their catabolic products are significant players in the turnover of nitrogenous molecules in plants[J]. Journal of Experimental Botany, 2012, 63(14): 5003-5015. DOI:10.1093/jxb/ers202 |
| [39] |
GHOLIZADEH F, MIRZAGHADERI G. Genome-wide analysis of the polyamine oxidase gene family in wheat (Triticum aestivum L.) reveals involvement in temperature stress response[J]. PloS One, 2020, 15(8): e0236226. DOI:10.1371/journal.pone.0236226 |
| [40] |
PODLEŠÁKOVÁ K, UGENA L, SPíCHAL L, DOLEZAL K, DEDIEGO N. Phytohormones and polyamines regulate plant stress responses by altering GABA pathway[J]. New Biotechnology, 2019, 48: 53-65. DOI:10.1016/j.nbt.2018.07.003 |
| [41] |
KIM J S, LEE B H, KIM S H, OK K H, CHO K Y. Response to environmental and chemical signals for anthocyanin biosynthesis in nonchlorophyllous corn (Zea mays L.) leaf[J]. Journal of Plant Biology, 2006, 49(1): 16-25. DOI:10.1007/BF03030784 |
| [42] |
MARTIN-TANGUY J. Metabolism and function of polyamines in plants: recent development (new approaches)[J]. Plant Growth Regulation, 2001, 34: 135-148. DOI:10.1023/A:1013343106574 |
| [43] |
TAKAHASHI T. Plant polyamines[J]. Plants (Basel), 2020, 9(4): 511. DOI:10.3390/plants9040511 |
| [44] |
FU X Z, CHEN C W, WANG Y, LIU J H, MORIGUCHI T. Ectopic expression of MdSPDS1 in sweet orange (Citrus sinensis Osbeck) reduces canker susceptibility: involvement of H2O2 production and transcriptional alteration[J]. BMC Plant Biology, 2011, 11: 55. DOI:10.1186/1471-2229-11-55 |
| [45] |
MITSUYA Y, TAKAHASHI Y, BERBERICH T, MIYAZAKI A, MATSUMURA H, TAKAHASHI H, TERAUCHI R, KUSAANO T. Spermine signaling plays a significant role in the defense response of Arabidopsis thaliana to cucumber mosaic virus[J]. Journal of Plant Physiology, 2009, 166(6): 626-643. DOI:10.1016/j.jplph.2008.08.006 |
(责任编辑 张辉玲)
 2023, Vol. 50
2023, Vol. 50