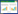文章信息
基金项目
- 广东省基础与应用基础研究基金(2019A1515110745);广东省科技专项资金(2020A06013);广东海洋大学科研启动经费资助项目(R19025,R19029)
作者简介
- 谭建彬(1994-),男,在读硕士生,研究方向为害虫生物防治,E-mail: Tanjianbin2022@163.com.
通讯作者
- 徐进(1984-),男,博士,讲师,研究方向为害虫生物防治,E-mail: xujin@gdou.edu.cn.
文章历史
- 收稿日期:2023-03-20
【研究意义】小菜蛾(Plutella xylostella)属鳞翅目菜蛾科,是一种严重危害十字花科蔬菜的世界性害虫,具有极强的繁殖潜力[1-2]。全球每年由小菜蛾造成的经济损失达40亿~50亿美元,在我国造成的损失高达7.7亿美元[3-4]。长期使用广谱型化学杀虫剂对小菜蛾进行防治,使其已对包括苏云金杆菌(Bacillus thuringiensis)在内的大部分的杀虫剂产生抗性[5-7]。在农药的“双减”政策下,寻找新型绿色环保的防治方法变得十分紧迫。RNAi被称为“农业上又一次新的科技革命”,已应用于基因功能研究和害虫防治,其通过注入特定的双链RNA分子来抑制目标基因的表达,在鞘翅目、鳞翅目、半翅目等昆虫中取得了成功[8],鉴定和分析小菜蛾清道夫受体(Scavenger receptor, SR)基因的功能对于开发有效的小菜蛾RNAi农药具有重要意义。通过这种方法,可以提高RNAi技术的效率和特异性,从而更好地防控小菜蛾,减少经济损失。
【前人研究进展】 SR基因家族在凋亡细胞清理、机体免疫防御及感受信息素等生理过程中有着重要作用。细胞程序性死亡的关键步骤是凋亡细胞的吞噬清除,这一过程对于维持体内环境平衡、免疫选择及组织形态和功能的重塑都至关重要。有研究将CD36包含在B类SR基因家族中进行分类,包括CD36、LIMP Ⅱ和SR家族B(Scavenger receptor class B member, SCRBs),也有研究将其分为五大类。本研究将其分为3类,即SCRBs、感受神经元膜蛋白(Sensory neuron membrane proteins,SNMPs)以及Croquemort(Catcher of death)[9-10]。在昆虫中,关于CD36的研究主要集中在果蝇和家蚕。果蝇的CD36基因主要有两种:一种是Croquemort基因,主要介导吞噬并在凋亡细胞的清除中发挥作用;另一种是SNMP基因,主要控制昆虫触角的许多嗅觉感受神经表达。SNMPs在昆虫中有广泛研究,尤其是在鳞翅目和双翅目昆虫中研究最为充分,其主要分为SNMP1和SNMP2两大亚家族, 它们位于嗅觉神经元细胞树突末上,是引起嗅觉的重要元件,昆虫通过嗅觉来定位自身位置、寻找食物和居住地,并具有躲避天敌、趋避危险、交配和哺育后代等作用。在家蚕研究中,通过对家蚕全基因组的免疫研究鉴定分析了13个SR-B基因,证实了家蚕的黄茧基因C(Yellow cocoon)是SCRBs家族的一个成员,编码Cameo2蛋白,并且鉴定得到另一个与β- 胡萝卜素转运相关的蛋白[11]。然而,清道夫受体B类I基因(SRB Ⅰ)还能结合磷脂类物质,这一生理过程是巨噬细胞消除凋亡细胞的重要步骤,并且SRB Ⅰ还是模式识别受体,可识别多种不同的微生物结构物质,如LPS(革兰氏阴性菌的细胞壁组成成分),在机体先天性免疫中发挥作用[12-14]。CD36在机体抗真菌防御中也发挥作用,有研究证实小鼠中CD36结合β葡聚糖后利于抵抗新型隐球菌的感染。CD36还可以作为信号传递分子结合钳合蛋白、胶原等,完整的CD36特异抗原等能够激活血小板,诱发单核细胞和血小板的突发性氧化作用。SR也参与先天性宿主防御细菌的感染,Stuart等[13]研究发现,利用连续高通量RNA干涉方法筛选果蝇SR基因家族中Croquemort分支有特异性抑制不同菌的吞噬。此外,SR基因家族还被指出可能参与脂质转移、脂蛋白代谢和类胡萝卜素转运,这与其跨膜蛋白性质有关。因此,SR基因家族是昆虫生存中重要的几个基因家族之一。
【本研究切入点】RNAi农药技术发展亟待解决的关键问题在于寻找绿色农药新靶标。目前国内大部分昆虫先天免疫研究都集中在体液免疫中,涉及抗菌肽的PRRs、PGRPs、BGRPs受体以及Toll等信号途径,这些途径都调控抗菌肽的表达和酚氧化酶活化的级联反应。SR则是一类位于细胞表面的跨膜糖蛋白,通过识别病原相关分子模式(PAMPs)调控溶酶体对病原物进行吞噬和清除。然而,以SR为特异性靶标的RNAi农药研究在国内外仍然是一个未开发的领域。【拟解决的关键问题】本研究采用生物信息学方法,从全基因组到蛋白质水平鉴定小菜蛾SR家族成员,并将小菜蛾B类清道夫受体基因家族同源蛋白分为SCRBs、SNMPs、Croquemort共3大类,填补小菜蛾SR家族成员信息空白,探究小菜蛾SR在天然免疫防御中的调控机制,为其功能研究及构建外源dsRNA抑制小菜蛾天然免疫的虫生真菌工程提供基础,并为筛选害虫防治的绿色RNAi农药靶标提供数据支撑。
1 材料与方法 1.1 小菜蛾SR基因家族成员全基因组鉴定从NCBI数据库下载由单倍体(Flye)组装染色体水平的小菜蛾基因组、所有蛋白序列、CDS及注释文件(RefSeq:GCF_932276165.1),及其他5个物种的基因组文件和蛋白pep序列:家蚕(RefSeq:GCF_014905235.1)、冈比亚按蚊(RefSeq:GCF_000005575.2)、黑腹果蝇(RefSeq:GCF_000001215.4)、烟草天蛾(RefSeq:GCF_014839805.1)、赤拟谷盗(RefSeq:GCF_000002335.3)。
在pfam网站(http://pfam.xfam.org/)获取CD36 HMM模型(隐马尔可夫模型),利用hmmer v3.3.2软件(http://hmmer.org/)以CD36结构域(PF01130)对小菜蛾基因组进行同源蛋白筛选,e-value阈值小于1E-20,将筛选得到的蛋白序列用mafft v7.310(https://mafft.cbrc.jp/alignment/software/)软件进行多序列比对[15],使用默认参数,将利用trimal v1.4.1(https://github.com/scapella/trimal.git)过滤掉比对蛋白文件gap区域的杂乱信号,以trimal后的文件构建新的hmm模型重新搜索小菜蛾基因组文件,新的筛选e-value阈值为小于1E-3,得到20个SR家族成员蛋白。在NCBI搜索昆虫SR基因家族的氨基酸序列构建blast数据库,利用blastp v2.12.0(ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATES)以构建的blast数据库搜索小菜蛾基因组的同源蛋白,筛选的e-value阈值为1E-5,选取分数大于70分的蛋白序列,共得到25个蛋白序列。利用diamond v2.0.11(http://github.com/bbuchfink/diamond/)[16], 以小菜蛾的所有蛋白序列构建数据库,在NCBI网站上搜索昆虫的氨基酸序列构建数据库,筛选的e-value阈值为1E-5,得到27个SR蛋白。取3种软件的蛋白序列去重得到12个小菜蛾SR基因家族的蛋白序列,依次按CD36px1~ CD36px12命名。
将上述鉴定的所有蛋白在CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)、pfam网站以及InterProScan(http://www.ebi.ac.uk/interpro/)、SMART(http://smart.embl-heidelberg.de/)上验证结构域。鉴定发现小菜蛾的12个SR基因家族蛋白均含有CD36结构域。
1.2 SR基因家族疏水性、信号肽及跨膜螺旋区域分析运用ProtScale(https://web.expasy.org/protscale/)进行蛋白疏水性分析,运用TMHMM(http://www.cbs.dtu.dk/services/TMHMM)、Phobius(https://phobius.sbc.su.se/)预测蛋白质跨膜螺旋区(Transmem-Branehelicalsegments,TMHs),通过ProtParam(http://web.expasy.org/protparam/)分析分子量和等电点(pI),信号肽通过SignalP-5.0程序(http://www.cbs.dtu.dk/services/SignalP)进行预测。
1.3 SR基因家族系统进化树构建将小菜蛾、家蚕、冈比亚按蚊、黑腹果蝇、烟草天蛾、赤拟谷盗中鉴定的SR蛋白保留最长转录本并去重共获得117个SR蛋白,通过mafft进行多序列比对后,用trimal过滤比对序列文件的gap区域的杂乱信号,然后通过iqtree v1.6.12- Linux(http://www.iqtree.org)构建系统进化树[17],选择最大似然法(ML)建树,采用最佳蛋白模型LG+R5,bootstrap重复次数1 000。利用ITOL(https://itol.embl.de/)完成进化树注释。
1.4 SR基因家族蛋白质二级结构预测及三级结构建模使用SOPMA(https://npsaprabi.ibcp.fr/cgibin/secpred_sopma.pl)预测SR蛋白的二级结构,width设置为70,window width设置为17,相似性阈值设置为6,在swiss-model(https://swissmodel.expasy.org/)上提交SR蛋白的序列建模,以GMQE选择最佳模型进行三级结构预测[18]。
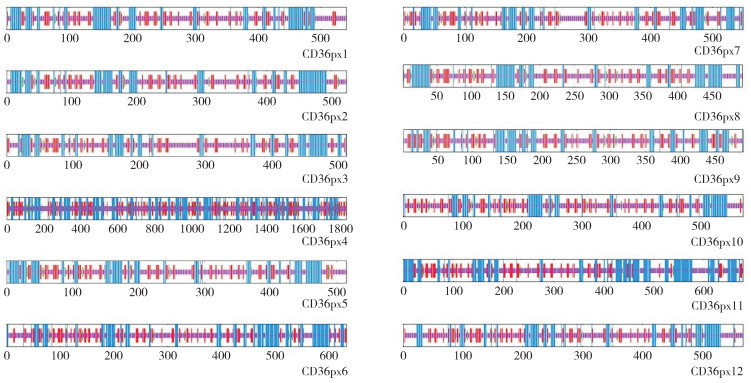
1.5 SR基因家族磷酸化位点分析使用NetPhos3.1 Serve(http://www.cbs.dtu.dk/services/NetPhos/)预测蛋白质磷酸化位点,选择经典方式预测3种氨基酸, 分析丝氨酸、苏氨酸、酪氨酸磷酸化位点数量。
1.6 SR基因家族蛋白质序列基序分析使用MEME(https://meme-suite.org/meme/)对蛋白质序列的基序进行分析,最小motif宽度为6个氨基酸,最大motif宽度为200个氨基酸,最大基序检索数值设为20,其余参数使用默认值。
2 结果与分析 2.1 小菜蛾SR基因家族蛋白质疏水性、信号肽及跨膜螺旋区域分析蛋白质的亲水性是维持其稳定性和功能的基础。由表 1可知,小菜蛾SR家族蛋白等电点介于5.08~8.94,表明它们具有不同的空间结构和功能;以CD36px1为例对蛋白质疏水性结果进行说明,得分正值为疏水性,负值为亲水性,得分的绝对值越大代表疏水性/ 亲水性越强。CD36px1中的第21位点疏水性得分3.400,表明具有较高疏水性,第311位疏水性得分较低(-2.267),表明具有较高的亲水性。12个蛋白的疏水性最低值为-3.589,最高值为3.833,表明该家族的蛋白质包含着多个亲水性和疏水性区域。跨膜螺旋区域分析结果显示,该家族的蛋白质均有两个跨膜螺旋区,表明该12个蛋白存在跨膜结构,为跨膜蛋白。信号肽分析仅有CD36px1、CD36px1有信号肽。
|
2.2 小菜蛾SR基因家族系统进化树构建
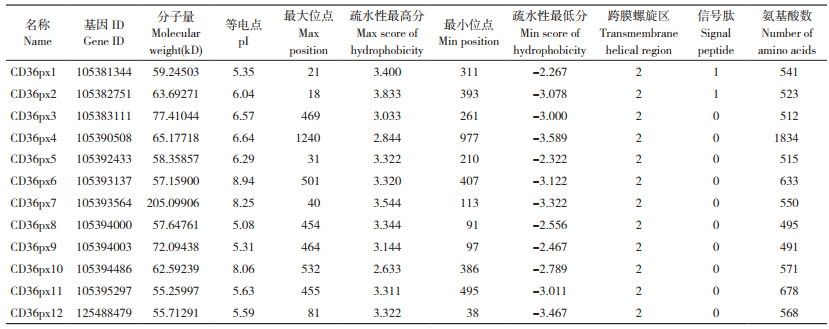
基于其他5类已鉴定的蛋白分类结果,将小菜蛾SR基因家族划分为3大类,系统进化树结果显示,CD36px3、CD36px5、CD36px6、CD36px7、CD36px10、CD36px11、CD36px12属于SCRBs分支,CD36px8、CD36px9属于Croquemort分支,CD36px1、CD36px2、CD36px4属于SNMPs分支(图 1)。小菜蛾SR蛋白以SCRBs分支为主,大部分位于同一进化枝上,表明其在进化中具有较强的保守性。
|
| 图 1 小菜蛾SR基因家族系统进化树 Fig. 1 Phylogenetic tree of SR gene family in Plutella xylostella |
2.3 小菜蛾SR基因家族蛋白质序列基序分析
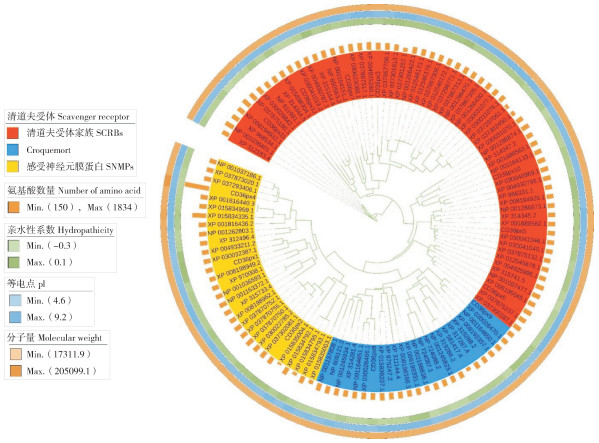
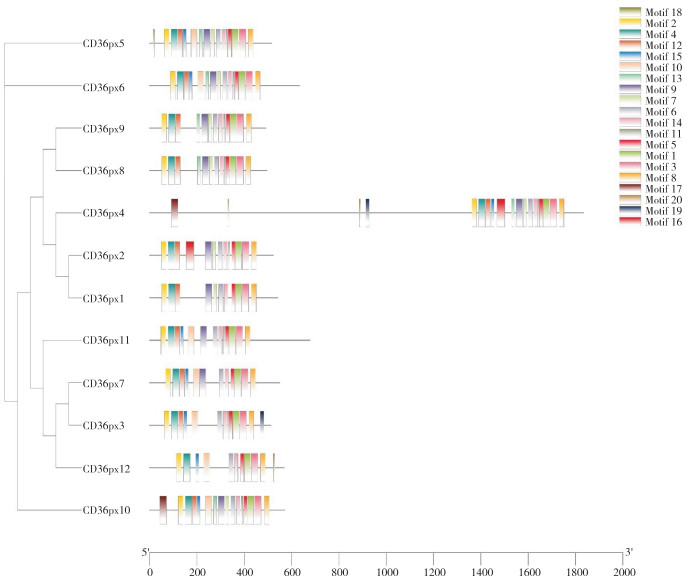
通过对12个CD36蛋白序列进行保守基序分析,获得前20个motif,依次命名为motif1~ motif20。结果(图 2)显示,大部分motif分布基本一致,表明其功能相似;不同分支上的motif类型及数量有所差异,表明其功能发生了一定分化,具有多样性。其中motif1、motif2、motif3、motif4、motif6、motif8、motif14等7个基序存在于所有蛋白序列中,分布范围最广、数量最多,具有非常强的保守特性;同时,motif19、motif20两个基序仅存在于CD36px4中,推测其在进化中可能有利于嗅觉特异性功能的保守进化。
|
| 图 2 小菜蛾SR基因家族蛋白质保守基序预测 Fig. 2 Prediction of conserved motifs of SR proteins in Plutella xylostella |
2.4 小菜蛾SR基因家族蛋白质二级结构预测及三级结构建模
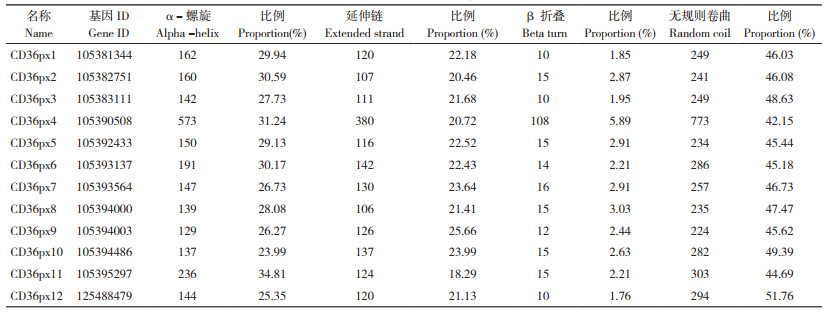
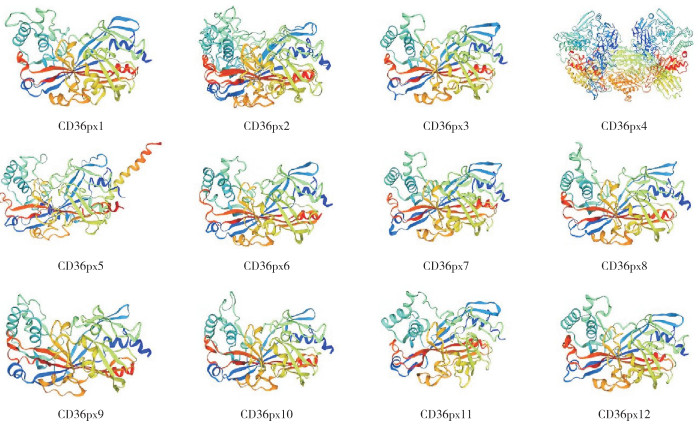
小菜蛾SR基因家族蛋白质二级结构分析结果(图 3)表明,12条CD36蛋白序列最主要的二级结构为无规则卷曲,α- 螺旋和延伸链比例相近,为次主要结构。不同SR蛋白的4种结构比例皆相近(表 2),推测该蛋白家族可能结构相似、蛋白质组成相近。三级结构建模结果(图 4)显示该蛋白家族结构相似:一端由多个α- 螺旋接1~3个β- 折叠区域,另一端由1~3个α- 螺旋组成及包括大量无规则卷曲,与二级结构预测一致。但CD36px4和CD36px5的三级结构较为特殊,表现了SR基因家族的多样性,推测这两个SR蛋白可能会有更加丰富的功能。
|
| 图 3 小菜蛾SR基因家族蛋白质二级结构预测 Fig. 3 Prediction of secondary structure of SR proteins in Plutella xylostella |
|
|
| 图 4 小菜蛾SR基因家族蛋白质三级结构预测 Fig. 4 Prediction of the tertiary structure of SR proteins in Plutella xylostella |
2.5 小菜蛾SR基因家族蛋白磷酸化位点预测分析
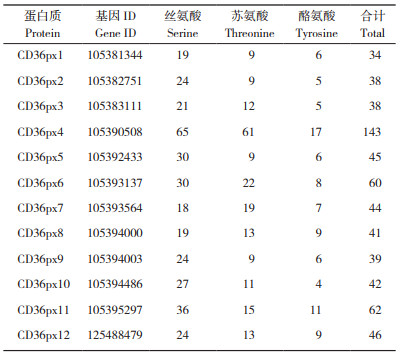
蛋白磷酸化是调节蛋白质自身活性和功能的最重要、最关键、最普遍的机制之一,对于生长发育、细胞周期和信号传导至关重要。在真核生物中,磷酸基团可以附着在丝氨酸、苏氨酸和酪氨酸残基上[19],表 3结果表明,小菜蛾SR蛋白中存在着34~143个丝氨酸、苏氨酸及酪氨酸潜在磷酸化位点。预测的磷酸化位点主要以丝氨酸形式存在,其次是苏氨酸,酪氨酸的磷酸化位点最少。CD36px1含有19个丝氨酸、9个苏氨酸和6个酪氨酸,磷酸化位点最少;CD36px4含有65个丝氨酸、61个苏氨酸和17个酪氨酸,磷酸化位点最多,与其序列长度有一定关系。
|
3 讨论
随着高通量测序技术的发展,三代测序技术和Canu、Flye等组装软件相继问世,许多高质量基因组在EMBL、GenBank等数据库中发布。由Flye组装的染色体水平小菜蛾基因组,为全基因组范围内鉴定小菜蛾SR家族提供了便利[20]。SR主要在巨噬细胞、血管内皮细胞和脂肪细胞等组织中表达,参与动脉粥样硬化、炎症反应、宿主防御、血管生成以及凋亡细胞的清除等重要生理过程[21]。目前,对SR的功能研究主要集中在哺乳动物,而昆虫领域中的相关研究非常有限,主要集中于探究家蚕和果蝇的生理过程及功能[22]。小菜蛾是一种为害严重的十字花科害虫[23],对于小菜蛾SR的研究鲜有报道。然而,SR基因在小菜蛾的生长发育和先天免疫应答中发挥了重要作用,不同的SR基因在小菜蛾的免疫系统中具有不同的功能或作用方式,增加了小菜蛾的免疫谱[24]。因此,通过预测蛋白质二、三级结构,帮助解释该基因的突变位点和酶催化位置及机理,可进一步推断其蛋白质结构与功能之间的关系[25]。研究蛋白质的空间结构,确定其结合位点和作用机制,有助于深入了解该基因在小菜蛾免疫防御和生长发育等生理过程中的作用机理,对于深入研究昆虫的生理过程以及探索新的农药具有重要意义。RNAi是一种常见的基因调控方式,通过介导基因沉默可导致昆虫发育异常甚至死亡[26-27],也可对特定基因的mRNA转录进行干扰或降解来抑制或增强其表达水平,从而实现对生物过程的调控[28-29]。在害虫防治领域,RNAi已成为一种绿色、高效的农药防治方法[30],但其应用受限于可靶向的RNAi靶标基因。基于SR基因功能的深入研究,发现其在昆虫生长发育过程中发挥重要作用,可以将其作为RNAi的靶标基因用于害虫防治。
本研究在筛选SR基因作为RNAi靶标的过程中,为寻找新型RNAi农药提供了重要的数据支撑和理论基础。下一步可通过基因克隆、转录组学、蛋白质组学、RNAi和基因编辑等技术,深入研究SR基因的功能和调控机制,并验证其作为RNAi靶标的可行性和有效性,系统地分析SR基因的生物学特性和RNAi靶向效应,为绿色RNAi农药的设计与开发提供更全面、精准的参考依据。
4 结论本研究通过全基因组水平鉴定和分析小菜蛾SR基因家族成员,共发现12个SR基因家族成员。疏水性、信号肽和跨膜螺旋区域分析结果显示其均为跨膜型蛋白质。系统发育树结果显示,12个小菜蛾SR基因家族成员分SNMPs分支、Croquemort分支、SCRBs分支3大类。其中大部分基因家族成员属于SCRBs,具有识别多种微生物结构物质的能力,在小菜蛾的天然免疫防御和与真菌拮抗过程中发挥作用。其次是SNMPs分支,它们与嗅觉受体结合,使嗅觉受体对特定的化学物质更敏感,参与昆虫的信息传递和行为反应过程[31]。蛋白质二、三级结构分析结果表明,它们蛋白质组成和结构相似,但二级结构数量的差异可能会影响蛋白质功能的细化。保守基序分析发现它们有相似类型的motif,表明在基因结构和蛋白质组成方面具有保守性。此外,磷酸化位点以丝氨酸为主要类型,可能通过磷酸化实现凋亡细胞吞噬清除的生物学功能。综上所述,SR基因家族成员在小菜蛾的生长发育和先天免疫过程中有着重要作用,可成为RNAi农药的备选靶标,通过RNAi技术来抑制其表达,从而控制小菜蛾的为害。
| [1] |
尹飞, 李振宇, SAMINA S, 林庆胜. P450基因在氯虫苯甲酰胺不同抗性品系小菜蛾中的表达及功能分析[J]. 中国农业科学, 2022, 55(13): 2562-2571. DOI:10.3864/j.issn.0578-1752.2022.13.007 YIN F, LI Z Y, SAMINA S, LIN Q S. Expression and functional analysis of P450 gene in diamondback moth (Plutella xylostella) with different resistance to chloramphenicol[J]. Scientia Agricultura Sinica, 2022, 55(13): 2562-2571. DOI:10.3864/j.issn.0578-1752.2022.13.007 |
| [2] |
SHI Y, LIN G L, FU X L, MIKE K, GUY S, LIU T X. Cocoon-Spinning behavior and 20-Hydroxyecdysone regulation of Fibroin genes in Plutella xylostella[J]. Frontiers in Physiology, 2020, 11: 574800ss. DOI:10.3389/fphys.2020.574800 |
| [3] |
LI Z Y, XIA F, LIU S S, YOU M S, MICHAEL J F. Biology, ecology, and management of the Diamondback Moth in China[J]. Annual Review of Entomology, 2016, 61: 277-296. DOI:10.1146/annurevento-010715-023622 |
| [4] |
MYRON P Z, ASAD S, REHAN S, DAVID A, LIU S S, MICHAEL J F. Estimating the economic cost of one of the world's major insect pests, Plutella xylostella (Lepidoptera: Plutellidae): just how long is a piece of string?[J]. Journal of Economic Entomology, 2012, 105: 1115-1129. DOI:10.1603/EC12107 |
| [5] |
XU J, XU X X, MUHAMMAD S, LI S Z. The entomopathogenic fungi Isaria fumosorosea plays a vital role in suppressing the immune system of Plutella xylostella: RNA-Seq and DGE analysis of immunity-related genes[J]. Frontiers in Microbiology, 2017, 8: 1421. DOI:10.3389/fmicb.2017.01421 |
| [6] |
YIN Q, LU Q, SONG P P, TUN Y J. Molecular mechanisms conferring asymmetrical cross-resistance between tebufenozide and abamectin in Plutella xylostella[J]. Journal of Asia-Pacific Entomology, 2019, 22: 189-193. DOI:10.1016/j.aspen.2018.12.015 |
| [7] |
ANSA B, MUHAMMAD B S A, SHOAIB H, MAMUNA L. Status of insecticide resistance in Plutella xylostella (Linnaeus) (Lepidoptera: Plutellidae) from 1997 to 2019: cross-resistance, genetics, biological costs, underlying mechanisms, and implications for management[J]. Phytoparasitica, 2022, 50: 465-485. DOI:10.1007/s12600-021-00959-z |
| [8] |
BURAND J, HUNTER W B. RNAi: future in insect management[J]. Journal of Invertebrate Pathology, 2013, 112: 68-74. DOI:10.1016/j.jip.2012.07.012 |
| [9] |
ANEYEN P, GORDON S, GARDON S. Macrophage scavenger receptors and host-derivedligands[J]. Methods, 2007, 43(3): 207-217. DOI:10.1016/j.ymeth.2007.06.004 |
| [10] |
RICHARD G V, NATALIE S, RACHEL L, RICHARD A F. The insect SNMP gene family[J]. Insect Biochemistry and Molecular Biology, 2009, 39(7): 448-456. DOI:10.1016/j.ibmb.2009.03.007 |
| [11] |
OKUMURA T, JAMIESON G A. Platelet glycocalicin. I. Orientation of glycoproteins of the human platelet surface[J]. Journal of Biological Chemistry, 1976, 251(19): 5944-5949. DOI:10.1016/S0021-9258(17)33043-0 |
| [12] |
TANAKA H, ISHIBASHI J, FUJITA K, NAKAJIMA Y. A genomewide analysis of genes and gene families involved in innate immunity of Bombyx mori[J]. Insect Biochemistry and Molecular Biology, 2008, 38(12): 1087-1110. DOI:10.1016/j.ibmb.2008.09.001 |
| [13] |
STUART L M, DENG J, SILVER J M, TAKAHASHI K. Response to Staphylococcus aureus requires CD36-mediated phagocytosis triggered by the COOH-terminal cytoplasmic domain[J]. Journal of Cell Biology, 2005, 170: 477-485. DOI:10.1083/jcb.200501113 |
| [14] |
SAKUDOH T, IIZUKA T, NARUKAWA J, SEZUTSU H. A CD36-related transmembrane protein is coordinated with an intracellular lipid-binding protein in selective carotenoid transport for cocoon coloration[J]. Journal of Biological Chemistry, 2010, 285(10): 7739-7751. DOI:10.1074/jbc.M109.074435 |
| [15] |
JOHN R, LI S L, AMADA K M, STANDLEY D M, KATOH K. MAFFT-DASH: integrated protein sequence and structural alignment[J]. Nucleic Acids Research, 2019, 47(1): 5-10. DOI:10.1093/nar/gkz342 |
| [16] |
BUCHFINK B, REUTER K, DROST H G. Sensitive protein alignments at tree-of-life scale using DIAMOND[J]. Nature Methods, 2021, 18: 366-368. DOI:10.1038/s41592-021-01101-x |
| [17] |
MINH B Q, SCHMIDT H A, CHERNOMOR O, SCHREMPF D, MICHAELW H, ARNDT V H, ROBERT L. IQ-TREE 2: new models and efficient methods for phylogenetic inference in the genomic era[J]. Molecular Biology and Evolution, 2020, 37: 1530-1534. DOI:10.1101/849372 |
| [18] |
WATERHOUSE A, BERTONI M, BIENERT S, STUDER G. SWISSMODEL: homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(1): 296-303. DOI:10.1093/nar/gky427 |
| [19] |
ESTELLA N, ELISE D, ELAFORGE E, RASMUS H P, KAREN S. How phosphorylation impacts intrinsically disordered proteins and their function[J]. Essays in Biochemistry, 2022, 66(7): 901-913. DOI:10.1042/EBC20220060 |
| [20] |
ZUO Y, CAROL V V, CORENTIN M, ANGEL M. De novo genome assembly and functional annotation for Fusarium langsethiae[J]. BMC Genomics, 2022, 23(1): 1-10. DOI:10.1101/2021.04.13.439621 |
| [21] |
赵羽卒, 张奎, 唐梅, 徐曼, 李重阳, 潘光照, 申利, 崔红娟, 杨丽群. 家蚕B类清道夫受体BmSCRB8基因的克隆及表达[J]. 生物工程学报, 2016, 32(10): 1408-1421. DOI:10.13345/j.cjb.160023 ZHAO Y Z, ZHANG K, TANG M, XU M, LI C Y, PAN G Z, SHEN L, CUI H J, YANG L Q. Cloning and expression of the BmSCRB8 gene for the B-class scavenger receptor in silkworm[J]. Journal of Biotechnology, 2016, 32(10): 1408-1421. DOI:10.13345/j.cjb.160023 |
| [22] |
申利, 许川, 张蕾, 张奎, 毛景欣, 潘光照, 李重阳, 胡仁建, 杨丽群, 崔红娟. 家蚕C类清道夫受体BmSR-C的基因克隆、原核表达及亚细胞定位[J]. 昆虫学报, 2018, 61(10): 1132-1144. DOI:10.16380/j.kcxb.201810.002 SHEN L, XU C, ZHANG L, ZHANG K, MAO J X, PAN G Z, LI C Y, HU R J, YANG L Q, CUI H J. Gene cloning, prokaryotic expression and subcellular localization of Class C scavenger receptor BmSR-C in silkworm[J]. Acta Entomologica Sinica, 2018, 61(10): 1132-1144. DOI:10.16380/j.kcxb.201810.002 |
| [23] |
李振宇, 肖勇, 冯夏. 广东蔬菜害虫综合治理研究进展[J]. 广东农业科学, 2020, 47(12): 90-97. DOI:10.16768/j.issn.1004-874X.2020.12.010 LI Z Y, XIAO Y, FENG X. Research progress on comprehensive management of vegetable pests in Guangdong[J]. Guangdong Agricultural Sciences, 2020, 47(12): 90-97. DOI:10.16768/j.issn.1004-874X.2020.12.010 |
| [24] |
ETEBARI K, HUSSAIN M, ASGARI S. Suppression of scavenger receptors transcription by parasitoid factors[J]. Developmental & Comparative Immunology, 2012, 38(4): 517-524. DOI:10.1016/j.dci.2012.08.006 |
| [25] |
JOHN J, RICHARD E, AIEXANDER P, TIM G. Highly accurate protein structure prediction with AlphaFold[J]. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2 |
| [26] |
陈咏琪, 尹延萍, 冯嘉伟, 白新宇, 李庆荣, 钟仰进, 杨婉莹. 家蚕RNAi效率相关核酸酶基因对RNAi效率的影响[J]. 昆虫学报, 2023(3): 1-9. DOI:10.16380/j.kcxb.2023.03.004 CHEN Y Q, YIN Y P, FENG J W, BAI X Y, LI Q R, ZHONG Y J, YANG W Y. The effect of silkworm RNAi efficiency related nuclease gene on RNAi efficiency[J]. Acta Insectae Sinica, 2023(3): 1-9. DOI:10.16380/j.kcxb.2023.03.004 |
| [27] |
WANG Y B, ZHANG H, LI H C, MIAO X X. Second-generation sequencing supply an effective way to screen RNAi targets in large scale for potential application in pest insect control[J]. Public Library of Science, 2011, 6(4): e18644. DOI:10.1371/journal.pone.0018644 |
| [28] |
袁瑞玲, 郑传伟, 冯丹, 王艺璇, 杜春花, 陈鹏. 细菌表达dsRNA介导桔小实蝇Flightin基因的RNA干扰[J]. 广东农业科学, 2020, 47(2): 118-124. DOI:10.16768/j.issn.1004-874X.2020.02.016 YUAN R L, ZHENG C W, FENG D, WANG Y X, DU C H, CHEN P. RNA interference of bacteria expressing dsRNA mediated Flightin gene of Bactrocera dorsalis[J]. Guangdong Agricultural Sciences, 2020, 47(2): 118-124. DOI:10.16768/j.issn.1004-874X.2020.02.016 |
| [29] |
杨雨航, 黄新意, 翁群芳. 昆虫不育分子研究进展[J]. 广东农业科学, 2020, 47(9): 94-104. DOI:10.16768/j.issn.1004-874X.2020.09.013 YANG Y H, HUANG X Y, WENG Q F. Research progress on insect infertility molecules[J]. Guangdong Agricultural Sciences, 2020, 47(9): 94-104. DOI:10.16768/j.issn.1004-874X.2020.09.013 |
| [30] |
LI X, LIU X G, LU W H, YIN X M. Application progress of plantmediated RNAi in pest control[J]. Frontiers in Bioengineering and Biotechnology, 2022, 10: 963026. DOI:10.3389/fbioe.2022.963026 |
| [31] |
CASSAU S, KRIEGER J. The role of SNMPs in insect olfaction[J]. Cell and Tissue Research, 2021, 383: 21-33. DOI:10.1007/s00441-020-03336-0 |
(责任编辑 陈丽娥)
 2023, Vol. 50
2023, Vol. 50