文章信息
基金项目
- 江西省烟草公司项目(2020.006);国家自然科学基金青年基金(122018AZ0330);上海烟草集团项目(D2019052)
作者简介
- 管成伟(1989—),男,硕士,农艺师,研究方向为烟草微生物资源应用,E-mail:guanxwei@163.com.
通讯作者
- 张超群(1982—),男,硕士,高级农艺师,研究方向为烟草病虫害综合防治技术,E-mail:chaoqunyc@163.com.
文章历史
- 收稿日期:2023-05-06
2. 中国烟草总公司郑州烟草研究院,河南 郑州 450001;
3. 江西省烟草公司抚州市公司,江西 抚州 344000;
4. 抚州市烟草公司黎川分公司,江西 抚州 344600
2. Zhengzhou Tobacco Science Institute of China National Tobacco Corporation, Zhengzhou 450001, China;
3. Fuzhou Company of Jiangxi Tobacco Corporation, Fuzhou 344000, China;
4. Lichuan Branch Company of Fuzhou Tobacco Company, Fuzhou 344600, China
【研究意义】青枯病菌也叫青枯雷尔氏菌(Ralstonia solanacearum),是引发青枯病的病原菌,具有侵染力强、寄主范围广的特点,目前已知能够侵染50多科200多种作物,烟草就是其中之一。青枯病菌还有明显的生理分化现象[1-2],来源于不同地区和不同寄主植物的病原菌在寄主范围、致病力和菌系特性等方面差异很大[3-4]。我国烟草青枯菌属于生理小种1、生化型Ⅲ和Ⅳ,且均属于演化型Ⅰ[4]。尽管我国烟草青枯菌的生理小种、生化型及演化型等群体结构较为单一,但同一演化型的青枯菌致病力也存在较大差异[5-10],给青枯病的防治带来很大困难。截至目前,针对烟草青枯病防治,国内尚未有较为广泛有效的技术方法。因此,了解烟草青枯菌致病力差异的内在基础,对于探索更有针对性的防控措施、提高烟草青枯病的防治效果具有重要意义。【前人研究进展】烟草青枯病菌的致病力不但与温度、湿度、pH值以及营养条件等环境因素密切相关,还与病原菌的内在编码基因有关,其中主要的内在编码基因有Ⅲ型和Ⅵ型分泌系统、胞外多糖(Extracellular polysaccharide,EPS)合成、鞭毛合成等基因(簇)[3-4, 11]。目前已经有包括GMI1000在内的多个青枯菌株系的基因组测序已经完成,通过基因组比较发现,不同青枯病菌细胞内部的巨大质粒上携带着大量特有的基因,大小变异幅度达到26%[4, 12-13], 这些特有基因赋予不同青枯病菌株不同的生理特性和侵染能力。【本研究切入点】虽然众多研究学者已证实青枯病菌株的致病力差异主要与编码Ⅲ型和Ⅵ型分泌系统、胞外多糖合成、鞭毛合成等基因(簇)有关,但由于青枯病菌之间遗传物质变化和差异大,同一区域内关系相近的青枯病菌株在致病力和基因序列方面仍存在不同,这些内在差异直接影响青枯病防治药剂的防治效果。因此,比较不同致病力青枯病菌菌株之间基因序列上的差异,探索针对青枯病菌的遗传差异的防治药剂和技术,对提高青枯病防效具有重要意义。【拟解决的关键问题】本研究从江西抚州广昌县头陂镇和南丰县太源乡种植的云烟87上分离得到2株序列变种为13的青枯病菌,为首次在江西抚州地区发现能够侵染烟草的青枯病菌序列变种13[14]。通过对2株致病力有差异的菌株进行基因组测序, 比对其编码基因和编码蛋白的差异,探索2株菌株之间致病力差异的内在基础,以期为根据致病力差异制定防治措施、开发针对性青枯病防治药剂、提高防治烟草青枯病防治效果提供理论参考。
1 材料与方法 1.1 青枯病菌的分离与鉴定青枯病菌菌株RsTP2和RsTY2于2016年7月分别分离自抚州市广昌县和南丰县的发病烟草植株。将采集的青枯病烟草植株茎杆于室内切成5 cm长的茎段,置于盛有湿纱布的塑料袋内3~5 h,将溢出的白色菌液挑入盛有2~3 mL无菌水试管中研磨制成菌悬液,静置15 min后用移菌环移取菌悬液并在牛肉膏蛋白胨选择培养基平板上划线[15](选择培养基含放线菌酮50 mg/L、氯霉素10 mg/L和硫酸多粘菌素B 10 mg/L),28 ℃恒温培养48 h,挑取2个单菌落进行纯化,分别命名为RsTP2和RsTY2,于4 ℃冰箱内斜面保存备用。采用青枯病菌种特异引物对759/760对菌株RsTP2和RsTY2的egl基因片段进行PCR鉴定[14],同时将菌株RsTP2和RsTY2置于中国工业微生物保藏中心(http://www.china-cicc.org/)保藏,保藏号分别为CICC24306和CICC24305。
1.2 侵染试验将青枯病菌株接入LB液体培养基(胰蛋白胨10 g/L、酵母提取物5 g/L、氯化钠10 g/L)中,30 ℃、200 r/min,过夜培养至浑浊状态,离心后菌体采用无菌水重悬,采用紫外分光光度计检测菌液浓度,菌液稀释至1.0×108 CFU/mL用于侵染试验。
侵染试验在郑州烟草研究院温室进行。测试品种为当地主栽品种云烟87,该品种对青枯病抗性为中感。当云烟87苗龄4~5周时,采用伤根灌菌液法分别接种1.0×108 CFU/mL的青枯病菌液,以清水处理为对照,每个处理侵染36株苗。28~30 ℃条件下培养2周后调查发病情况,病情指数和发病率调查按照国家标准GB/T23222-2008进行。
1.3 青枯病菌基因组DNA提取青枯病菌基因组的提取采用Ezup柱式细菌基因组DNA抽提试剂盒(生工生物工程(上海) 股份有限公司)进行,提取方法参照试剂盒说明书进行操作。提取的DNA可立即进行下一步实验或-20 ℃保存。
1.4 青枯病菌基因组测序采用Covaris ME220(美国Covaris公司)将提取的DNA片段化打断,以Agencourt AMPure XP核酸纯化试剂盒(德国Beckman Coulter公司)将破碎后的DNA片段浓缩回收。使用Next® UltraTM DNA文库制备试剂盒(英国NEB公司)进行建库和连接接头,磁珠分选纯化连接产物,接着采用Next Q5 Hot Start HiFi PCR预混液(英国NEB公司)进行文库的扩增和扩增产物磁珠纯化,使用Qubit®2.0(美国Invitrogen公司)进行文库浓度测定,使用Agilent 2100生物分析仪(美国Agilent公司)进行文库长度分布检测,采用Hiseq XTen平台(美国Illumina公司)进行测序。
1.5 基因序列的生物信息学分析与基因注释获得测序数据后,采用FastQC软件对原始数据进行质量评估,过滤处理后使用IDBA_UD对各样本有效序列进行拼接组装[16-17],获得contigs,综合评定多个Kmer的组装结果,采用Prodigal对最佳拼接结果进行ORF预测[18],并将长度≥ 100 bp的基因翻译成氨基酸序列。采用CD-HIT软件对基因预测结果进行去冗,获得非冗余基因集[19]。采用Bowtie2将各样本序列比对到非冗余基因集序列上[20-21]。将获得的基因和蛋白序列与相关数据库(包括NR)进行比对,获得基因的功能及物种注释信息[22-24]。
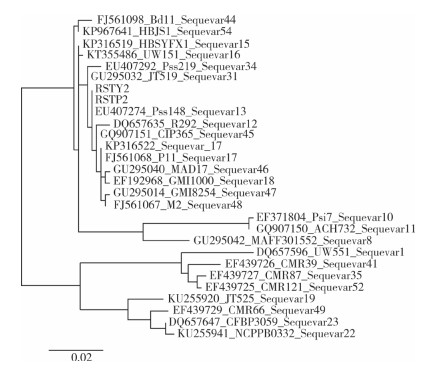
2 结果与分析 2.1 菌株RsTP2和RsTY2的分离与鉴定菌株RsTP2和RsTY2的PCR鉴定结果如图 1,其扩增产物出现在青枯病菌预期位置(281 bp),同时将其回收纯化送生工生物工程(上海) 股份有限公司进行测序,序列比对结果表明其均为青枯病菌。将菌株RsTP2和RsTY2的egl基因扩增测序结果提交NCBI数据库,获得编号分别为MG669274和MG669275,将其与亚洲分支序列变种12、13、15、17、34和44等序列变种egl基因进行比对分析,MG669274和MG669275序列与来自序列变种13菌株JT523的AF295252序列相似度为100%,确定菌株RsTP2和RsTY2均为亚洲分支演化型Ⅰ中的序列变种13(图 2)。

|
| M: Marker;NTC: 阴性对照 M: Marker; NTC: No template control 图 1 菌株RsTP2和RsTY2的PCR鉴定结果 Fig. 1 PCR identification results of strains RsTP2和RsTY2 |
|
| 图 2 菌株RsTP2和RsTY2与相关序列变种egl序列的系统发育分析 Fig. 2 Phylogenetic analysis based on egl gene sequences of strains RsTP2 and RsTY2 with related sequence variants |
2.2 菌株RsTP2和RsTY2对云烟87的致病力测试
将浓度为1.0×108 CFU/mL的菌株RsTP2和RsTY2菌液以伤根灌菌液法接种云烟87,2周后调查发病率和病情指数。结果(图 3)显示,未接种青枯病菌的对照烟苗生长正常,未发生青枯病;而接种菌株RsTP2的云烟87烟苗叶片基本上全部凋萎,发病率为100%、病情指数达76.9;接种菌株RsTY2的云烟87烟苗发病率为100%、病情指数达35.6。由此表明,菌株RsTP2和RsTY2对云烟87的致病力有显著差异,菌株RsTP2致病力较强,菌株RsTY2致病力较弱。

|
| 大写英文字母不同者表示差异显著 Different uppercase letters represent significant differences 图 3 云烟87接种菌株RsTP2和RsTY2的发病情况 Fig. 3 Disease incidence of Yunyan 87 inoculated with strains RsTP2 and RsTY2 |
2.3 菌株RsTP2和RsTY2的基因组测序
将菌株RsTP2和RsTY2进行基因组测序,总共获得785 MB和787 MB数据,其Clean Data Q30(%)分别为92.52% 和92.23%,基因组框架图总共获得5.43 MB和5.23 MB,分别预测编码基因4 967和4 801个,平均基因长度分别为965和970 bp(表 1)。测序数据已上传至NCBI,菌株RsTP2和RsTY2基因组序列登记号分别为SAMN35628657和SAMN35628658。
2.4 菌株RsTP2和RsTY2的差异基因筛选
为探究菌株RsTP2和RsTY2致病力差异的分子机制,对两者的差异基因进行筛选,将菌株RsTP2和RsTY2编码基因的蛋白序列进行BLASTP分析,结果表明菌株RsTY2有52个蛋白序列与菌株RsTP2的44个蛋白存在差异,其相似度在50.00%~99.88%。基于生物学过程的GO分析结果表明,其差异基因更多地参与含氮化合物代谢、杂环化合物代谢、芳香族化合物代谢、蛋白定位与细胞运输等过程(图 4)。其中,凝集素(Hemagglutinin)类蛋白数量最多,RsTY2有10个蛋白,菌株RsTP2有9个蛋白,其序列相似度在55.00%~99.61%;其次是细胞溶素分泌激活蛋白(Family hemolysin secretion activation protein),在菌株RsTP2和RsTY2各有4个蛋白序列差异较大;穿孔素家族蛋白(Phage holin family protein)在两者之间也存在较大差异,各有4个蛋白序列差异较大(表 2)。

|
| 图 4 基于生物学过程的菌株RsTP2和RsTY2差异基因GO分析 Fig. 4 GO analysis of strains RsTP2 and RsTY2 differential genes based on biological processes |
2.5 菌株RsTP2和RsTY2凝集素家族的系统发育分析
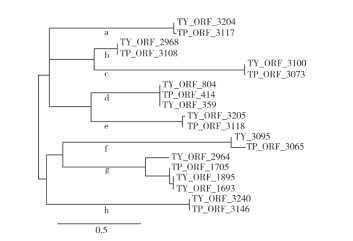
将菌株RsTP2和RsTY2的差异凝集素蛋白进行比对后构建系统发育树,结果(图 5)表明,以0.5遗传距离进行划分,可以将其分成a~h共8个亚类,其中d亚类和g亚类包含凝集素蛋白最多、分别为3个和4个,其他亚类均为2个。可见,凝集素蛋白在菌株RsTP2和RsTY2之间差异蛋白较多、序列差异较大,可能是其致病力差异的因素之一。
|
| 图 5 菌株RsTP2和RsTY2的凝集素基因遗传多样性分析 Fig. 5 Genetic diversity analysis of lectin genes in strains RsTP2 and RsTY2 |
3 讨论
烟草青枯病菌具有很强的侵染性,目前已知能够侵染450余种寄主植物,其致病性主要与编码Ⅲ型和Ⅵ型分泌系统、胞外多糖合成、鞭毛合成等基因(簇)有关[4]。然而,即使同属于一个亚型或序列变种的青枯病菌,不同菌株之间的致病性仍然存在较大差异。Remenant等[25]从泛基因组学的角度对6个基因组的比对结果显示,青枯菌核心基因组的权重仅占28%,非必需基因组占35%,各菌株拥有数量不等的菌株特异基因,因此,各菌株独有的特异基因可能是导致菌株间致病力差异较大的重要原因[26]。
本研究从江西抚州广昌县头陂镇和南丰县太源乡等地种植的云烟87上均分离到青枯雷尔氏菌菌株RsTP2和RsTY2,egl测序表明其为亚洲分支序列变种13,侵染试验表明菌株RsTP2对云烟87致病力较强、菌株RsTY2致病力较弱。分析两者基因组序列发现,菌株RsTY2的52个蛋白序列与RsTP2菌株的44个蛋白存在差异,其相似度在50.00%~99.88% 之间,其中凝集素类蛋白数量最多,其次是细胞溶素分泌激活蛋白和穿孔素家族蛋白。编码凝集素的基因广泛存在于动植物病原菌中,如青枯雷尔氏菌的基因组中就有27个与凝集素相关的编码基因,这些基因编码的蛋白主要作用是促使病原菌附着到寄主细胞表面[27-28],已有研究证明其在青枯雷尔氏菌(R. solanacearum)[12]、百日咳杆菌(Bordetella pertussis)[29]、流感嗜血杆菌(Haemophilus infl uenzae)[30]、苛养木杆菌(Xylella fastidiosa)[31]等致病菌侵入寄主细胞中起着重要作用。细胞溶素对致病菌在寄主细胞表面的黏连和侵染、寄主组织中细胞间的生存及毒力发挥方面起着重要作用,而细胞溶素分泌激活蛋白可促进细胞溶素的分泌、进而促进侵染发生[32-33]。穿孔素(Holin)可协助自溶素(Autolysin) 作用于宿主细胞的细胞壁,并通过切割肽聚糖聚合物中的各种键来发挥作用,具体作用取决于自溶素的类型[34-35]。由此可见,凝集素、细胞溶素分泌激活蛋白和穿孔素家族蛋白等与青枯病菌的侵染能力和致病能力高度相关。
4 结论本研究从广昌县头陂镇和南丰县太源乡的青枯病发病烟株上分离到青枯雷尔氏菌菌株RsTP2和RsTY2,egl测序证明其为亚洲分支序列变种13,侵染试验表明菌株RsTP2对云烟87致病力较强、菌株RsTY2致病力较弱。通过对比属于同一序列变种、但致病力存在差异的2株青枯病菌RsTP2和RsTY2基因序列,发现菌株RsTY2的52个蛋白序列与菌株RsTP2的44个蛋白存在差异,其相似度在50.00%~99.88%。差异基因更多地参与含氮化合物代谢、杂环化合物代谢、芳香族化合物代谢、蛋白定位与细胞运输等过程。其中,编码凝集素类蛋白数量差异最大,其次是细胞溶素分泌激活蛋白和穿孔素家族蛋白,这3类蛋白与青枯病菌的吸附侵染能力相关性较高。凝集素类蛋白、细胞溶素分泌激活蛋白和穿孔素家族蛋白3类基因编码的蛋白序列差异可能是导致RsTP2和RsTY2致病力差异的重要原因。
| [1] |
方树民, 顾钢, 纪成灿, 陈顺辉, 张炳生, 陈玉森. 烟草青枯菌致病型及分布的研究[J]. 中国烟草学报, 2002, 8(3): 40-43. DOI:10.3321/j.issn:1004-5708.2002.03.008 FANG S M, GU G, JI C C, CHEN S H, ZHANG B S, CHEN Y S. A study on the pathogenic types and distribution of tobacco bacterial wilt[J]. Acta Tabacaria Sinica, 2002, 8(3): 40-43. DOI:10.3321/j.issn:1004-5708.2002.03.008 |
| [2] |
佘小漫, 何自福. 作物青枯病研究进展[J]. 广东农业科学, 2020, 47(12): 82-89. DOI:10.16768/j.issn.1004-874X.2020.12.009 SHE X M, HE Z F. Research progress on crop bacterial wilt[J]. Guangdong Agricultural Sciences, 2020, 47(12): 82-89. DOI:10.16768/j.issn.1004-874X.2020.12.009 |
| [3] |
汪汉成, 余婧, 蔡刘体, 陆宁. 温度、湿度、接菌量及pH对烟草青枯病菌致病力的影响[J]. 中国烟草科学, 2017, 38(5): 8-12. DOI:10.13496/j.issn.1007-5119.2017.05.002 WANG H C, YU J, CAI L T, LU N. The effects of temperature, humidity, inoculation amount, and pH on the pathogenicity of tobacco bacterial wilt[J]. Chinese Tobacco Science, 2017, 38(5): 8-12. DOI:10.13496/j.issn.1007-5119.2017.05.002 |
| [4] |
徐进, 冯洁. 植物青枯菌遗传多样性及致病基因组学研究进展[J]. 中国农业科学, 2013, 46(14): 2902-2909. DOI:10.3864/j.issn.0578-1752.2013.14.006 XU J, FENG J. Research progress on genetic diversity and pathogenic genomics of plant bacterial wilt[J]. Scientia Agricultura Sinica, 2013, 46(14): 2902-2909. DOI:10.3864/j.issn.0578-1752.2013.14.006 |
| [5] |
向忠明, 叶建如, 顾钢. 南方五省烟草青枯病菌系组成与分布[J]. 延边大学农学学报, 2001, 23(3): 170-173. DOI:10.3969/j.issn.1004-7999.2001.03.004 XIANG Z M, YE J R, GU G. Composition and distribution of tobacco bacterial wilt strains in the five southern provinces[J]. Agricultural Science Journal of Yanbian University, 2001, 23(3): 170-173. DOI:10.3969/j.issn.1004-7999.2001.03.004 |
| [6] |
谢锐鸿, 巫升鑫. 粤、闽、贵烟草青枯病菌分离株及其致病力[J]. 微生物学通报, 2014(9): 1800-1806. DOI:10.13344/j.microbiol.china.130652 XIE R H, WU S X. Isolation and pathogenicity of bacterial wilt strains from Guangdong, Fujian, and Guizhou tobacco[J]. Microbiology China, 2014(9): 1800-1806. DOI:10.13344/j.microbiol.china.130652 |
| [7] |
徐进, 顾刚. 福建烟草青枯菌演化型及生化变种鉴定研究[J]. 中国烟草学报, 2010(6): 66-71. DOI:10.3969/j.issn.1004-5708.2010.06.013 XU J, GU G. Study on the evolutionary type and biochemical variant identification of tobacco bacterial blight in Fujian province[J]. Acta Tabacaria Sinica, 2010(6): 66-71. DOI:10.3969/j.issn.1004-5708.2010.06.013 |
| [8] |
刘海龙, 黎妍妍. 湖北地区烟草青枯菌系统发育分析[J]. 中国烟草科学, 2015, 36(2): 81-86. DOI:10.13496/j.issn.1007-5119.2015.02.015 LIU H L, LI Y Y. Phylogenetic analysis of tobacco bacterial wilt in Hubei region[J]. Chinese Tobacco Science, 2015, 36(2): 81-86. DOI:10.13496/j.issn.1007-5119.2015.02.015 |
| [9] |
刘颖, 唐元满, 张淑婷, 郭兵, 谷纪涛, 丁伟. 重庆烟区青枯雷尔氏菌生化变种及序列变种的特性研究[J]. 中国烟草学报, 2016, 22(4): 75-82. DOI:10.16472/j.chinatobacco.2015.527 LIU Y, TANG Y M, ZHANG S T, GUO B, GU J T, DING W. Study on the characteristics of biochemical and sequential variants of Ralstonia solanacearum in Chongqing tobacco region[J]. Acta Tabacaria Sinica, 2016, 22(4): 75-82. DOI:10.16472/j.chinatobacco.2015.527 |
| [10] |
苏慧慧, 李涛, 黎振兴, 李植良, 王永飞, 徐小万. 广州地区番茄青枯病菌的系统发育分析及致病力评价[J]. 热带作物学报, 2014, 35(11): 2284-2288. SU H H, LI T, LI Z X, LI Z L, WANG Y F, XU X W. Phylogenetic analysis and pathogenicity evaluation of tomato bacterial wilt in Guangzhou[J]. Chinese Journal of Tropical Crops, 2014, 35(11): 2284-2288. |
| [11] |
张泳, 贺红, 李巧, 王邦伟. 广藿香青枯病菌致病相关基因的筛选及分析[J]. 中药新药与临床药理, 2022, 33(2): 249-254. ZHANG Y, HE H, LI Q, WANG B W. Screening and analysis of virulence-related genes of Ralstonia solanacearum isolated from pogostemon cablin[J]. Traditional Chinese Drug Research and Clinical Pharmacology, 2022, 33(2): 249-254. |
| [12] |
SALANOUBAT M, GENIN S, ARTIGUENAVE F, GOUZY J, MANGENO ST, ARLAT M, BILLAULT A, BROTTIER P, CAMUS J C, CATTOLICO L, CHANDLER M, CHOISNE N, CLAUDEL-RENARD C, CUNNAC S, DEMANGE N, GASPIN C, LAVIE M, MOISAN A, ROBERT C, SAURIN W, SCHIEX T, SIGUIER P, THéBAULT P, WHALEN M, BOUCHER C A. Genome sequence of the plant pathogen Ralstonia solanacearum[J]. Nature, 2002, 415: 497-502. DOI:10.1038/415497a |
| [13] |
GENIN S, BOUCHER C. Lessons learned from the genome analysis of Ralstonia solanacearum[J]. Annual Review of Phytopathology, 2004, 42(42): 107. DOI:10.1146/annurev.phyto.42.011204.104301 |
| [14] |
胡利伟, 牟文君, 王皓, 郭建华, 冯小虎, 奚家勤, 薛超群, 张友武, 张志高, 王爱国, 李琰琰, 王利兵, 周汉平, 尹启生, 宋纪真. 江西省抚州烟区青枯雷尔氏菌的分离鉴定与遗传多样性分析[J]. 烟草科技, 2018, 51(12): 1-9, 22. DOI:10.16135/j.issn1002-0861.2018.0074 HU L W, MOU W J, WANG H, GUO J H, FENG X H, XI J Q, XUE C Q, ZHANG Y W, ZHANG Z G, WANG A G, LI Y Y, WANG L B, ZHOU H P, YIN Q S, SONG J Z. Isolation, identification, and genetic diversity analysis of Ralstonia solanacearum in Fuzhou tobacco region, Jiangxi province[J]. Tobacco Science & Technology, 2018, 51(12): 1-9, 22. DOI:10.16135/j.issn1002-0861.2018.0074 |
| [15] |
WICKER E, GRASSART L, CORANSON-BEAUDU R, MIAN D, GUILBAUD C, FEGAN M, PRIOR P. Ralstonia solanacearum strains from martinique (French west indies) exhibiting a new pathogenic potential[J]. Applied and Environmental Microbiology, 2007, 73(21): 6790. DOI:10.1128/AEM.00841-07 |
| [16] |
BOLGER A M, LOHSE M, USADEL B. Trimmomatic: A flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [17] |
PENG Y, LEUNG H C M, YIU S M, CHIN F Y. IDBA-UD: A de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth[J]. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [18] |
LIU Y, GUO J, HU G, ZHU H. Gene prediction in metagenomic fragments based on the SVM algorithm[J]. BMC Bioinformatics, 2013, 14(5): 12. DOI:10.1126/science.109385 |
| [19] |
LI W, GODZIK A. Cd-hit: A fast program for clustering and comparing large sets of protein or nucleotide sequences[J]. Bioinformatics, 2006, 22(13): 1658-1659. DOI:10.1093/bioinformatics/btl158 |
| [20] |
LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [21] |
LI H, HANDSAKER B, WYSOKER A, FENNELL T, RUAN J, HOMER N, MARTH G, ABECASIS G, DURBIN R. 1000 genome project data processing subgroup: The sequence alignment/map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078-2079. DOI:10.1093/bioinformatics/btp352 |
| [22] |
ALTSCHUL S F, MADDEN T L, SCHÄFFER A A, ZHANG J H, ZHANG Z, MILLER W, LIPMAN J D. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389-3402. DOI:10.1093/nar/25.17.3389 |
| [23] |
KANEHISA M, SATO Y, MORISHIMA K. BlastKOALA and GhostKOALA: KEGG tools for functional characterization of genome and metagenome sequences[J]. Journal of Molecular Biology, 2016, 428(4): 726-731. DOI:10.1016/j.jmb.2015.11.006 |
| [24] |
EDDY S R. A new generation of homology search tools based on probabilistic inference[J]. Genome Informatics, 2009, 23(1): 205. DOI:10.1142/9781848165632_0019 |
| [25] |
REMENANT B, COUPAT-GOUTALAND B, GUIDOT A, CELLIER G, WICKER E, ALLEN C, FEGAN M, PRUVOST O, ELBAZ M, CALTEAU A, SALVIGNOL G, MORNICO D, MANGENOT S, BARBE V, MéDIGUE C, PRIOR P. Genomes of three tomato pathogens within the Ralstonia solanacearum species complex reveal significant evolutionary divergence[J]. BMC Genomics, 2010, 11: 1-16. DOI:10.1186/1471-2164-11-379 |
| [26] |
贵彩英, TAHIRA S, 谢文松, 卓涛, 户勋, 邹华松, 范晓静. 青枯菌GMI1000效应蛋白RipQ对致病力的作用研究[J]. 植物病理学报, 2022, 52(5): 801-812. DOI:10.13926/j.cnki.apps.000777 GUI C Y, TAHIRA S, XIE W S, ZHUO T, HU X, ZOU H S, FAN X J. The role of effector RipQ for virulence in Ralstonia solanacearum GMI1000[J]. Acta Phytopathologica Sinica, 2022, 52(5): 801-812. DOI:10.13926/j.cnki.apps.000777 |
| [27] |
DAS D, VERMA S K, RAY S K. An altered G + C% region within potential filamentous hemagglutinin open reading frames of Ralstonia solanacearum[J]. Current Science, 2005, 88(5): 786. DOI:10.1126/science.1105370 |
| [28] |
GAMBLIN S J, VACHIERI S G, XIONG X, ZHANG J, MARTIN S R, SKEHEL J J. Hemagglutinin structure and activities[J]. Cold Spring Harbor Perspectives in Medicine, 2021, 11(10): a038638. DOI:10.1101/cshperspect.a038638 |
| [29] |
SMITH A M, GUZMAN C A, WALKER M J. The virulence factors of Bordetella pertussis: A matter of control[J]. FEMS Microbiology Reviews, 2001, 25: 309-333. DOI:10.1111/j.1574-6976.2001.tb00580.x |
| [30] |
SIMPSON A J, REINACH F C, ARRUDA P. The genome sequence of the plant pathogen Xylella fastidiosa[J]. Nature, 2000, 406: 151-157. DOI:10.1038/35018003 |
| [31] |
ROJAS C M, HAM J H, DENG W L, DOYLE J J, COLLMER A. HecA, a member of a class of adhesin produced by diverse pathogenic bacteria, contributes to the attachment, aggregation, epidermal cell killing, and virulence phenotypes of Erwinia chrysanthemi EC16 on Nicotiana cievelandii seedlings[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99: 13142-13147. DOI:10.1073/pnas.202358699 |
| [32] |
HACHANI A, WOOD T E, FILLOUX A. Type Ⅵ secretion and anti-host effectors[J]. Current Opinion in Microbiology, 2016, 29: 81-93. DOI:10.1016/j.mib.2015.11.006 |
| [33] |
KALINDAMAR S, ABDELHAMED H, KORDON A O, PINCHUK L M, KARSI A. Hemolysin co-regulated family proteins Hcp1 and Hcp2 contribute to Edwardsiella ictaluri pathogenesis[J]. Frontiers in Veterinary Science, 2021, 8: 681609. DOI:10.3389/fvets.2021.681609 |
| [34] |
CAHILL J, YOUNG R. Chapter two-phage lysis: Multiple genes for multiple barriers[J]. Advances in Virus Research, 2019, 103: 33-70. DOI:10.1016/bs.aivir.2018.09.003 |
| [35] |
VIDOR C J, HAMIOT A, WISNIEWSKI J, MATHIAS R A, DUPUY B, AWAD M, LYRAS D. A highly specific holin-mediated mechanism facilitates the secretion of lethal toxin TcsL in Paeniclostridium sordellii[J]. Toxins, 2022, 14(2): 124. DOI:10.3390/toxins14020124 |
(责任编辑 陈丽娥)
 2023, Vol. 50
2023, Vol. 50






