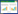文章信息
基金项目
- 国家自然科学基金(32202465);广东省自然科学基金(2023A1515012687);韶关学院博士科研启动项目(99000613)
作者简介
- 朱云娜,博士,讲师,农艺师,韶关学院生物与农业学院专任教师,主要从事氮素营养对蔬菜生长发育与品质形成的调控机理与应用研究。近5年主持和参与国家自然科学基金、广东省自然科学基金、广东省教育厅项目、韶关市科技项目等12项。以第一或通信作者在国内外期刊发表学术论文20篇;授权实用新型专利3件、外观专利2件。朱云娜(1982—),女,博士,讲师,研究方向为蔬菜生理与分子生物学,E-mail:zhuyn326@126.com; 刘建国,博士,助理实验师,韶关学院生物与农业学院实验中心副主任、广东绿乡实业博士工作站站长,主要从事土壤质量提升与生物肥料利用研究。近5年主持和参与广东省自然科学基金、广东省教育厅项目、韶关市科技项目等8项。以第一或通信作者在国内外期刊发表学术论文11篇,参编教材1部;获授权国家实用新型专利3件、外观专利2件.
通讯作者
- 刘建国(1981—),男,博士,助理实验师,研究方向为园艺作物生理生态,E-mail:jgliu@sgu.edu.cn.
文章历史
- 收稿日期:2023-08-18
【研究意义】菜心(Brassica campestris L. ssp. chinensis var. utilis Tsen et Lee)又名菜薹,属于十字花科芸薹属芸薹种白菜亚种[1],是我国华南地区种植面积最大的一种薹茎类蔬菜。菜心因其生长周期短、适应能力强、复种指数高、口感脆嫩、风味独具一格、营养丰富等众多优点,受到消费者喜爱[2]。然而,像其他绿叶蔬菜一样,菜心对氮肥需求量大,提高其氮利用率则对菜心生产具有重要意义[1, 3]。已有研究表明,跨膜转运蛋白受蛋白磷酸化调控,蛋白磷酸化在氮信号、氮代谢等方面发挥重要作用[4-5]。因此,揭示蛋白磷酸化机理对生产中调控菜心氮素吸收、转运、代谢等过程具有重要的指导意义。CIPK23激酶已成为根系响应各种环境胁迫的主要枢纽,在提高作物产量和品质方面具有巨大潜力[6-7]。【前人研究进展】钙调磷酸酶B类似蛋白的互作蛋白激酶CIPK(CBL-interacting protein kinase)是植物特有的一类丝氨酸、苏氨酸蛋白激酶,含有N-端激酶催化域和NAF结构域两个典型的保守结构域[8]。早在1998年,Zhu等[9]采用遗传筛选和图位克隆方法大规模筛选拟南芥盐过敏感(Salt overly sensitive)表型突变体时就发现了CBL-CIPK信号系统的相关基因,并命名为SOS1(Na+/H+ antiporter)、SOS2(CIPK24)和SOS3(CBL4),三者形成SOS3-SOS2-SOS复合体,从而介导植物非生物胁迫应答[9-11]。后续研究证实了拟南芥中存在一种同源基因,与动物与酵母中的CNB(B subunit of calcineurin)和NCS(Neuronal calcium sensor)具有较高相似性,且能够通过介导Ca2+来应答盐等胁迫,因此将该类基因命名为CBL(Calcineurin B-like)基因[12-13]。相关基因家族分析表明,拟南芥中分别有25个CIPK和10个CBL成员[14];水稻中分别有30个CIPK和10个CBL成员[14-15]。此后,在玉米、葡萄、大豆、高粱等物种中也报道过不同数量的CIPK和CBL家族成员[16-19]。除参与高盐、低温、干旱等逆境胁迫[20-21],植物CIPK成员在调控矿质营养转运方面也具有重要作用[6-7]。前人研究表明,拟南芥CIPK23、CBL1/9受低浓度NO3–诱导表达,形成CBLCIPK复合体调控硝转运蛋白NPF6.3/NRT1.1(PTR family 6.3/Nitrate transporter 1.1)磷酸化水平,从而促进NO3–的吸收与运输[5];而在高NH4+条件下,拟南芥CBL1与CIPK23形成复合体,通过调控铵转运蛋白AMT1.1和AMT1.2(Ammonium transporter)磷酸化水平,从而影响植株根系对NH4+的吸收与运输[22]。外界NH4+浓度变化会引起胞浆游离Ca2+含量变化,影响CBL蛋白活性,激活的CBL与激酶CIPK23结合形成复合体,将CIPK23转移到质膜上,从而调控AMT1三聚体的磷酸化水平,进一步调控细胞对NH4+的吸收[23]。本课题组前期研究发现,拟南芥AtCIPK23与菜心BcAMT1.1、BcAMT1.2存在互作[24];且菜心BcCBL1和BcCBL9表达受不同氮素形态响应[25]。【本研究切入点】目前,在菜心中关于CIPK23基因和其功能的研究尚未有相关报道,且菜心CIPK23对不同氮素的响应也尚不清楚。【拟解决的关键问题】本文从菜心中克隆CIPK23,并对其编码蛋白的生物学信息进行分析,研究其组织表达特性以及对氮源的响应,以期为研究菜心CIPK23的生物学功能以及对氮素的吸收与运输等提供一定的理论依据。
1 材料与方法 1.1 试验材料供试菜心品种为‘油绿501’(来自广州市农业科学研究院),于2021年3—5月种植于韶关学院生物与农业学院生态园玻璃温室内。
1.2 试验方法1.2.1 组织特异性分析 菜心种子用NaClO消毒后,用无菌水冲洗干净,在育苗海绵块中育苗,待幼苗长至三叶一心时移栽,待植株种子结荚后,分别取其根、茎、叶、叶柄、薹茎、花蕾、花、荚果等组织[25]。每种组织均设3次重复,所有样品均用液氮速冻,放入-80 ℃冰箱保存备用。
1.2.2 缺氮、供氮处理 菜心移苗后预培养4 d,用去离子水冲洗植株根系,分别对植株进行缺氮、供氮处理。缺氮处理:将植株移至不含任何氮源的改良霍格兰营养液[24]中培养48 h;供氮处理:缺氮处理后,再移栽到0.1、1、4、8 mmol/L NaNO3/NH4Cl不同氮源营养液中处理2 h后,分别取菜心叶片、根系。所有样品均设3次重复,每个重复随机取4~6株。样品经液氮速冻后,放在-80 ℃冰箱备用。
1.2.3 总RNA提取与cDNA合成 参考Eastep® Super总RNA提取试剂盒的说明书提取样品总RNA。参考PrimeScriptTM RT reagent Kit with gDNA Eraser试剂说明书步骤合成cDNA。
1.3 测试项目及方法1.3.1 基因克隆 从NCBI数据库搜索拟南芥AtCIPK23基因序列,将该序列在白菜(Brassica rapa)基因组进行blast比对,获得同源性最高的CIPK基因BrCIPK23(XM_009104459.3)序列设计引物(CIPK23-F:ATGGCTTCTCGA TCAACAC;CIPK23-R:TCAAGAAGCAGCCACTG CACC),以菜心cDNA为模板克隆BcCIPK23的CDS全长序列。使用TaKaRa公司的高保真酶Prime STAR Max Premix克隆目的基因片段,PCR反应程序为:98 ℃预变性3 min,98 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,35个循环,72 ℃延伸5 min。回收扩增产物,与pMD20-T载体连接,转化DH5α,挑选阳性克隆进行测序。
1.3.2 生物信息学分析 利用ExPASY(https://web.expasy.org/)在线网站分析菜心CIPK23编码氨基酸的基本理化性质,利用SWISS-MODEL分析其蛋白三维结构,利用NCBI网站上CDD在线工具分析其保守结构域,利用STRING(https://string-db.org/)分析其互作蛋白网络。
1.3.3 qRT-PCR检测 将各组织或处理的cDNA用RNase-Free ddH2O稀释5倍作为模板,以序列(F: CGCAAGGGTCAGTTAGCAGTT,R: ATCA AGAAGCAGCCACAGTA)为引物,参考TB GreenR Premix Ex TaqTM说明书,利用Bio-RAD公司的CFX connect TM实时荧光定量PCR仪进行qRTPCR检测。以GADPH和Actin2作为内参基因[25],使用2-∆∆Ct方法计算基因相对表达量[26]。
2 结果与分析 2.1 菜心BcCIPK23基因克隆采用同源克隆方法,以菜心cDNA为模板,以CIPK23-F、CIPK23-R为引物进行PCR扩增,得到CDS片段长度为1 450 bp左右,电泳图如图 1所示。将回收的PCR产物与T载体连接进行测序,根据测序结果分析,所得到的片段长度为1 449 bp。将该序列在NCBI数据库进行比对,发现该CDS片段与拟南芥(NM_001332883)、甘蓝(XM_013743437)、白菜(XM_009104459)、甘蓝型油菜(NM_001315872.1)CIPK23核苷酸序列相似,相似率分别为88.41%、96.84%、98.14% 和96.82%,表明所克隆的基因为菜心CIPK23,将其命名为BcCIPK23。
|
| M:DNA marker;1:BcCIPK23基因扩增条带 M: DNA marker; 1: BcCIPK23 gene amplification band 图 1 PCR产物琼脂糖凝胶电泳图 Fig. 1 Agarose gel electrophoretogram of PCR product |
2.2 菜心BcCIPK23编码氨基酸的生物信息学分析
2.2.1 菜心BcCIPK23序列及蛋白理化性质分析 对所克隆的菜心BcCIPK23序列进行开放阅读框预测,结果表明该基因序列的最大开放阅读框为1 449 bp,编码482个氨基酸序列。多重序列比对结果显示,BcCIPK23与白菜、甘蓝型油菜CIPK23的相似度较高,分别为98.55%、98.76%,而与拟南芥CIPK23序列的相似性较低、为92.50%(图 2)。

|
| BrCIPK23:白菜;BnCIPK23:甘蓝型油菜;AtCIPK23:拟南芥 BrCIPK23: Brassica rapa; BnCIPK23: Brassica napus; AtCIPK23: Arabidopsis thaliana 图 2 BcCIPK23编码的氨基酸序列多重比对 Fig. 2 Multiple alignment of amino acids sequences encoded by BcCIPK23 |
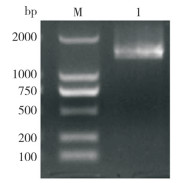
利用Expasy在线工具(http://web.expasy.org/)分析结果表明,BcCIPK23的分子量为53.41 kD,原子组成为C2379H3824N642O717S16,理论等电点(pI)为9.28,脂肪酸系数为80.91,不稳定性指数(Ⅱ)为32.52,平均亲水率为-0.38。这表明菜心BcCIPK23蛋白是稳定的亲水性蛋白。利用CELLO v.2.5在线网站(http://cello.life.nctu.edu.tw/)预测菜心BcCIPK23的亚细胞定位,结果显示,该蛋白可能定位于细胞质中;通过TMHMM 2.0(https://services.healthtech.dtu.dk/services/TMHMM-2.0)对该蛋白进行跨膜结构分析,发现该蛋白不存在跨膜结构(图 3)。
|
| 图 3 菜心BcCIPK23跨膜结构域预测 Fig. 3 Prediction transmembrane domain of BcCIPK23 from flowering Chinese cabbage |
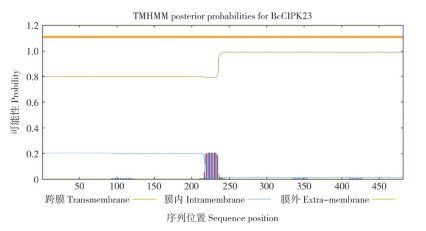
2.2.2 菜心BcCIPK23次级结构、亲疏水性与磷酸化位点分析 利用SOPMA对BcCIPK23蛋白二级结构进行预测,结果表明该蛋白包含α-螺旋(Alpha helix)、β-折叠(Extended strand)、β-转角(Beta turn)和随机卷曲(Random coil),占比分别为39.42%、15.15%、9.96% 和35.48%(图 4A)。该蛋白序列主要为α-螺旋和随机卷曲,β-折叠、β-转角散布于蛋白结构。使用SWISSMODEL在线软件预测菜心BcCIPK23的三级结构,同源建模结果显示如图 4B,结果与二级结构预测类似。利用Protscale预测菜心BcCIPK23的亲疏水性,结果表明该蛋白为亲水性蛋白(图 4C)。利用SignalP 5.0预测菜心BcCIPK23氨基酸序列,结果表明该氨基酸序列无信号肽,为非分泌型蛋白。利用NetPhos 3.1对BcCIPK23潜在磷酸化位点进行分析表明,该序列包含有多个潜在磷酸化位点,最多的磷酸化位点为丝氨酸磷酸化位点(Ser)、达27个,其次是苏氨酸磷酸化位点(Thr)、为19个,而酪氨酸磷酸化位点(Tyr)为5个(图 4D),这表明BcCIPK23能够被丝氨酸、苏氨酸、酪氨酸激酶磷酸化,从而实现其功能调控。
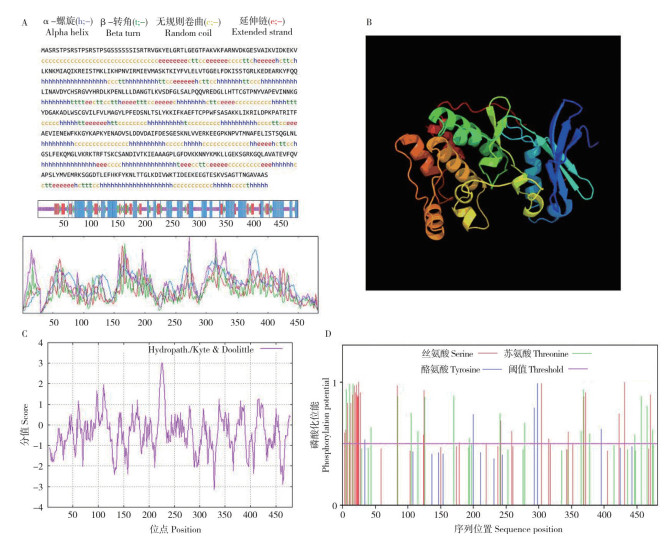
|
| A:蛋白二级结构分析;B:蛋白三级结构分析;C:亲疏水性分析;D:蛋白磷酸化位点分析 A: Secondary structure analysis; B: Tertiary structure analysis; C: Analysis of hydrophilicity and hydrophobicity; D: Phosphorylation potential analysis 图 4 菜心BcCIPK23的生物信息学分析 Fig. 4 Bioinformatic analysis of BcCIPK23 from flowering Chinese cabbage |
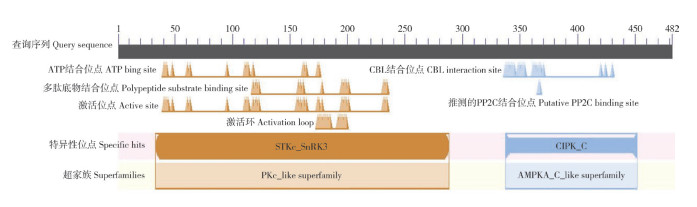
2.2.3 菜心BcCIPK23保守结构域分析 利用NCBI网站CCD在线软件(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)分析BcCIPK23蛋白序列的保守结构域,结果(图 5)显示,在该蛋白的33~288位氨基酸区域是蛋白激酶超家族的STKc-SnRK3保守结构域;337~452位氨基酸区域是CBL互作蛋白激酶CIPK-C末端调节结构域〔C-terminal regulatory domain of calcineurin B-Like (CBL)-interacting protein kinases〕,这个结构域是CIPK家族蛋白与CBL蛋白相互作用的部位,属于腺苷酸激活蛋白激酶超基因家族成员。因此,推测本研究克隆的菜心BcCIPK23所编码蛋白具有CIPK蛋白功能。
|
| 图 5 BcCIPK23蛋白序列的保守结构域分析 Fig. 5 Analysis of conserved domains of BcCIPK23 protein sequences |
2.3 菜心BcCIPK23系统进化树分析
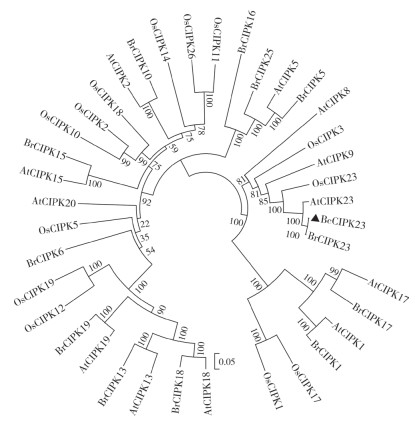
将菜心BcCIPK23与拟南芥、白菜、水稻等植物中的CIPK家族成员的蛋白序列进行Neighbor-Joining系统进化树分析。结果(图 6)显示,菜心CIPK23与拟南芥、白菜、水稻等物种的CIPK23成员聚类在同一分支中,其中菜心BcCIPK23与白菜BrCIPK23、拟南芥AtCIPK23的遗传距离较近,该分支不包含CIPK家族其他成员,表明植物CIPK23成员间的进化关系较近、保守性较强。
|
| At: Arabidopsis thaliana,拟南芥;Bc: Brassica campestris, 菜心;Br: Brassica rape,白菜;Os: Oryza sativa, 水稻 图 6 菜心BcCIPK23和其他物种CIPK成员的系统进化树分析 Fig. 6 Phylogenetic analysis of BcCIPK23 in flowering Chinese cabbage and CIPKs in other species |
2.4 BcCIPK23在菜心不同组织器官的表达分析
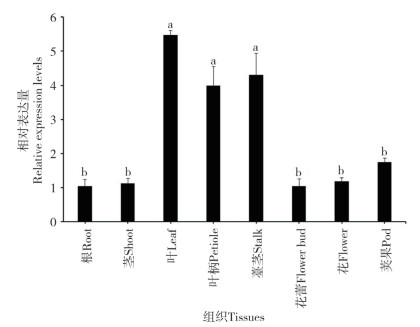
由图 7可知,菜心BcCIPK23在菜心各器官中均有表达,且在菜心叶、叶柄、薹茎等组织中高表达,显著高于在其他组织中的表达量,但3个组织间无明显差异;该基因在菜心根、茎、花蕾、花、荚果等组织器官中的表达量较低,仅为叶片表达量的18.95%~311.93%,菜心BcCIPK23在这些组织中的表达量无明显差别。
|
| 以菜心根系中的BcCIPK23表达量为1计算各组织中的相对表达量;柱上小写英文字母不同者表示差异显著(P < 0.05) Relative expression levels in different tissues of flowering Chinese cabbage were calculated by the expression of BcCIPK23 in root as 1; Different lowercase letters above the columns represent significant differences (P < 0.05) 图 7 BcCIPK23在菜心不同组织器官的表达分析 Fig. 7 Expression analysis of BcCIPK23 in different tissues of flowering Chinese cabbage |
2.5 菜心BcCIPK23对不同氮源的响应
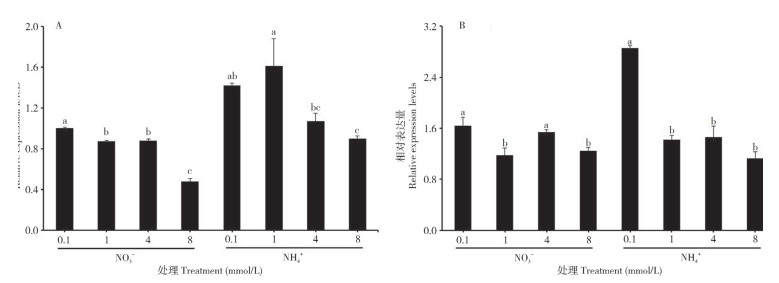
由图 8可知,无论在菜心根系还是叶片,BcCIPK23表达量均受氮源种类及浓度的影响。随着NO3–浓度的增加,BcCIPK23在菜心根系中的表达量呈明显下降趋势,与0.1 mmol/L NO3–处理相比,1~8 mmol/L NO3–处理显著降低了菜心BcCIPK23表达量,仅为0.1 mmol/L NO3–处理下表达量的47.72%~87.43%,但4、8 mmol/L NO3–处理之间无显著差异(图 8A);菜心根系中BcCIPK23对不同浓度NH4+的响应呈现出不同趋势,与0.1 mmol/L NH4+处理相比,BcCIPK23表达水平随NH4+浓度的增加呈现先增加后降低的变化趋势,其中BcCIPK23在1 mmol/L NH4+处理下表达量最高、为0.1 mmol/L NH4+处理的1.14倍,4、8 mmol/L NH4+处理的菜心BcCIPK23表达量分别为0.1 mmol/L NH4+处理的75.26%、63.07%(图 8A)。
|
| A:根;B:叶片;以0.1 mmol/L NO3-处理下菜心根系中的BcCIPK23表达量为1计算各处理中相对表达量;柱上小写英文字母不同者表示差异显著(P < 0.05) A: Root; B: Leaf; Relative expression levels in different treatments of flowering Chinese cabbage were calculated by the expression of BcCIPK23 in root under 0.1 mmol/L NO3– as 1; Different lowercase letters above the columns represent significant differences (P < 0.05) 图 8 菜心BcCIPK23对不同氮源种类和浓度的响应 Fig. 8 Response of BcCIPK23 in flowering Chinese cabbage to different nitrogen sources and concentrations |
在菜心叶片中,BcCIPK23表达量也明显受不同氮源水平的影响。与0.1 mmol/L NO3–处理相比,1~8 mmol/L NO3–处理可不同程度地降低菜心BcCIPK23的表达水平,降幅为6.14%~28.40%,其中4 mmol/L NO3–处理与0.1 mmol/L NO3–处理之间差异不显著(图 8B);随着NH4+浓度的增加,BcCIPK23在菜心叶片中的表达量呈现明显下降趋势,1~8 mmol/L NO3–处理的菜心BcCIPK23表达量仅为0.1 mmol/L NO3–处理的39.32%~51.31%(图 8B)。由此可见,菜心BcCIPK23表达受不同氮水平及氮源种类的影响,暗示菜心BcCIPK23基因可能与菜心氮素吸收利用相关。
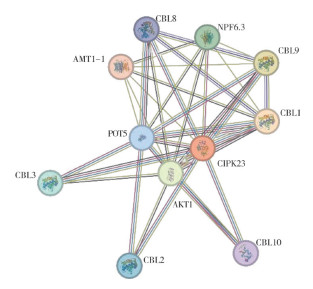
2.6 菜心BcCIPK23互作蛋白网络的构建选用模式植物拟南芥为受体物种,利用STRING在线软件绘制了菜心BcCIPK23蛋白的互作蛋白网络图。结果(图 9)表明,菜心BcCIPK23不仅可与植物类钙调磷酸酶B亚基蛋白(CBL1、CBL2、CBL3、CBL8、CBL9、CBL10)存在互作,还可与钾离子通道蛋白AKT1、钾转运蛋白POT5、硝转运蛋白NPF6.3、铵转运蛋白AMT1-1等蛋白存在互作。
|
| AMT1-1:铵转运蛋白1-1;NPF6.3:硝转运蛋白6.3;CBL1、CBL2、CBL3、CBL8、CBL9、CBL10:类钙调磷酸酶B亚基蛋白;AKT1:钾通道蛋白1;POT5:钾转运蛋白5 AMT1-1: Ammonium transporter 1-1; NPF6.3: Protein NRT1/PTR family 6.3; CBL1, CBL2, CBL3, CBL9 and CBL10: Calcineurin B-like protein; AKT1: Potassium channel 1; POT5: Potassium transporter 5 图 9 菜心BcCIPK23的互作蛋白网络 Fig. 9 Network for the interaction proteins with BcCIPK23 in flowering Chinese cabbage |
3 讨论
本研究通过同源克隆从菜心获得一个完整的BcCIPK23基因,其CDS序列全长为1 450 bp,编码482个氨基酸残基。菜心BcCIPK23具有N-端激酶催化域和NAF结构域两个典型的保守结构域,属于CIPK基因家族成员。这与前人在甘蔗、玉米、葡萄、大豆、高粱中报道的CIPKs基因结果[16-19]一致。通过进化树分析,所有植物CIPK23成员均聚类在同一分支,且该分支不包含CIPK家族的其他成员,表明植物CIPK23成员具有较高的保守性,菜心BcCIPK23与拟南芥AtCIPK23、白菜BrCIPK23亲缘关系较近,同源性均在92.50% 以上。
前人研究表明,多数CIPKs基因在植物不同组织器官中表达水平不同,甘蔗SsCIPK23基因在根系中表达量较高,在叶、茎中表达量较低[27];玉米ZmCIPK24-2基因在雄蕊、叶、茎中高表达,而在玉米其他部位低表达[28]。本研究中,BcCIPK23在菜心根、茎、叶、叶柄、薹茎、花蕾、花、荚果等组织中均有表达,但其在菜心叶片、叶柄、薹茎等绿色组织中的表达量显著高于根、茎、花蕾、花、荚果等非绿色组织器官,暗示着BcCIPK23可能在菜心组织发育中发挥作用。
CIPK是植物信号传导中的关键蛋白激酶,广泛参与植物生长发育调控和干旱、盐胁迫和营养胁迫等逆境胁迫应答[7, 20-21]。据研究,拟南芥AtCIPK23与CBL1/9形成CBL-CIPK复合体参与拟南芥氮信号通路调控[22]。本研究结果表明,菜心BcCIPK23基因表达水平明显受NO3–浓度影响,总体表现为:在低氮(0.1~1 mmol/L)条件下,无论根系还是叶片,菜心BcCIPK23基因表达量均最高,随着供氮水平的增加,其表达量呈现出明显下降趋势,尤其是8 mmol/L NO3–处理的菜心根系中BcCIPK23表达量仅为0.1 mmol/L NO3–水平下的47.76%。前人研究表明,在低氮条件下,CIPK23通过调控NRT1.1/NPF6.3蛋白Thr-101磷酸化水平,将NO3–由低亲和性转化为高亲和性,从而促进植物根系对低氮的吸收[5, 7, 29]。利用STRING在线网站预测,发现菜心BcCIPK23与NRT1.1/NPF6.3蛋白也存在互作关系,故而推测CIPK23在菜心中也可能通过与NRT1.1/NPF6.3蛋白互作来调控NO3–的吸收和转运。
在植物中,CIPK23表达水平除受外界NO3–水平的影响外,还受NH4+水平的影响[22]。本研究中,菜心BcCIPK23基因表达水平明显受不同浓度NH4+的影响,除1 mmol/L NH4+处理下菜心根系BcCIPK23基因表达量高于0.1 mmol/L NH4+处理外,无论在根系还是叶片中,BcCIPK23基因表达水平均随着NH4+浓度的增加呈现出明显下降趋势。在拟南芥中,向缺氮4 d的幼苗供2 mmol/L NH4+,AtCIPK23表达随着供NH4+时间的延长而上调表达,但在供NH4+ 2 h时,其表达水平才与缺氮4 d时的表达水平存在显著差异[22]。
已有研究指出,蛋白激酶CIPK23本身不具有活性,需要与CBL结合形成CBL-CIPK复合体才具有活性,从而参与植物生长发育、逆境胁迫、矿质元素吸收等过程[30]。本研究结果表明,菜心BcCIPK23与CBL1、CBL2、CBL3、CBL4、CBL9、CBL10等CBL基因家族成员存在互作。本课题组前期研究发现菜心BcCBL1、BcCBL9表达水平也明显受不同氮素水平影响,其中BcCBL1表达随NH4+浓度的增加而下降,BcCBL9随NH4+浓度的增加而显著增加[25],这与拟南芥AtCBL1、AtCBL9的研究结果并不一致,在拟南芥中,随着NH4+处理时间延长,前者表达水平急剧上调然后下降,后者表达水平仅呈现小幅增加[22],可能是由于本研究未考虑相同氮浓度处理不同时间对其表达水平的影响。拟南芥突变体cipk23植株在供NH4+后仍能检测到一定程度的蛋白磷酸化水平,AMT蛋白可能还受到其他激酶的调控[22]。研究表明,除CIPK23外,CIPK15也可作为NH4+受体蛋白参与调控AMT蛋白的磷酸化水平,从而影响NH4+的吸收和转运,以避免高NH4+环境对植物所造成的毒害[31]。
由此可见,对NH4+、NO3–两种氮源而言,菜心BcCIPK23表达均受氮素水平的明显影响,可能参与不同氮素条件下氮素吸收、转运等过程,但在高NH4+条件下,可能还存在其他CIPK蛋白激酶调节AMT复合体的磷酸化水平,从而影响根系对NH4+的吸收。
4 结论本研究从菜心中克隆得到1个新的全长基因BcCIPK23,属于CIPK基因家族成员,其表达量在一定程度上与生长活跃的器官同步变化,暗示菜心BcCIPK23可能在菜心不同组织发育过程中发挥作用;菜心BcCIPK23表达受到不同氮源形态及浓度的影响。菜心BcCIPK23基因在调控植物生长发育和氮代谢过程中发挥重要作用,可为揭示菜心BcCIPK23在菜心氮代谢途径中的作用提供基础数据。
| [1] |
夏岩石, 洪豪, 赖景诗, 苏伟生, 李荣华, 张润霖, 李光光, 郭培国. 夏冬两季广州菜心的硝酸盐含量及营养品质分析[J]. 广东农业科学, 2018, 45(4): 28-33. DOI:10.16768/j.issn.1004-874X.2018.04.005 XIA Y S, HONH H, LAI J S, SU W S, LI R H, ZHANG R L, LI G G, GUO P G. Analysis of nitrate and nutrition of flowering Chinese cabbage in summer and winter in Guangzhou city[J]. Guangdong Agricultural Sciences, 2018, 45(4): 28-33. DOI:10.16768/j.issn.1004-874X.2018.04.005 |
| [2] |
陈汉才, 吴增祥, 林悦欣, 沈卓, 黎庭耀, 杨易, 周轩, 张艳. 广东菜心、芥蓝研究现状与展望[J]. 广东农业科学, 2021, 48(9): 62-71. DOI:10.16768/j.issn.1004-874X.2021.09.007 CHEN H C, WU Z X, LIN Y X, SHEN Z, LI T Y, YANG Y, ZHOU X, ZHANG Y. Research status and prospect of flowering Chinese cabbage and Chinese kale in Guangdong[J]. Guangdong Agricultural Sciences, 2021, 48(9): 62-71. DOI:10.16768/j.issn.1004-874X.2021.09.007 |
| [3] |
ZHU Y N, QI B F, HAO Y W, LIU H C, SUN G W, CHEN R Y, SONG S W. Appropriate NH4+/NO3– ratio triggers plant growth and nutrient uptake of flowering Chinese cabbage by optimizing the pH value of nutrient solution[J]. Frontiers in Plant Science, 2021, 12: 656144. DOI:10.3389/fpls.2021.656144 |
| [4] |
LANQUAR V, LOQUÉ D, HORMANN F, YUAN L X, BOHNER A, ENGELSBERGER W R, LALONDE S, SCHULZE W X, VON WIRÉN N, FROMMER W B. Feedback inhibition of ammonium uptake by a phospho-dependent allosteric mechanism in Arabidopsis[J]. The Plant Cell, 2009, 21(11): 3610-3622. DOI:10.1105/tpc.109.068593 |
| [5] |
HO C, TSAY Y. Nitrate, ammonium, and potassium sensing and signaling[J]. Current Opinion in Plant Biology, 2010, 13(5): 604-610. DOI:10.1016/j.pbi.2010.08.005 |
| [6] |
SATHEE L, KRISHNA G K, ADAVI S B, JHA S K, JAIN V. Role of protein phosphatases in the regulation of nitrogen nutrition in plants[J]. Physiology and Molecular Biology of Plants, 2021, 27(12): 2911-2922. DOI:10.1007/s12298-021-01115-x |
| [7] |
RÓDENAS R, VÉRT G. Regulation of root nutrient transporters by CIPK23: 'One kinase to rule them all'[J]. Plant and Cell Physiology, 2021, 62(4): 553-563. DOI:10.1093/pcp/pcaa156 |
| [8] |
MAO J J, MO Z J, YUAN G, XIANG H Y, VISSER R G F, BAI Y L, LIU H B, WANG Q, VAN DER LINDEN C G. The CBL-CIPK network is involved in the physiological crosstalk between plant growth and stress adaptation[J]. Plant, Cell & Environment, 2023, 46(10): 3012-3022. DOI:10.1111/pce.14396 |
| [9] |
ZHU J K, LIU J, XIONG L. Genetic analysis of salt tolerance in Arabidopsis: evidence for a critical role of potassium nutrition[J]. The Plant Cell, 1998, 10(7): 1181-1191. DOI:10.1105/tpc.10.7.1181 |
| [10] |
QUINTERO F J, OHTA M, SHI H, ZHU J K, PARDO J M. Reconstitution in yeast of the Arabidopsis SOS signaling pathway for Na+ homeostasis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(13): 9061-9066. DOI:10.1073/pnas.132092099 |
| [11] |
QIU Q S, GUO Y, DIETRICH M A, SCHUMAKER K S, ZHU J K. Regulation of SOS1, a plasma membrane Na+/H+ exchanger in Arabidopsis thaliana, by SOS2 and SOS3[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(12): 8436-8441. DOI:10.1073/pnas.132092099 |
| [12] |
KUDLA J, XU Q, HARTER K, GRUISSEM W, LUAN S. Genes for calcineurin B-like proteins in Arabidopsis are differentially regulated by stress signals[J]. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(8): 4718-4723. DOI:10.1073/pnas.96.8.4718 |
| [13] |
AMTMANN A, ARMENGAUD P. The role of calcium sensorinteracting protein kinases in plant adaptation to potassium-deficiency: new answers to old questions[J]. Cell Research, 2007, 17(6): 483-485. DOI:10.1038/cr.2007.49 |
| [14] |
YU Y, XIA X, YIN W, ZHANG H. Comparative genomic analysis of CIPK gene family in Arabidopsis and Populus[J]. Plant Growth Regulation, 2007, 52(2): 101-110. DOI:10.1007/s10725-007-9165-3 |
| [15] |
KOLUKISAOGLU U, WEINL S, BLAZEVIC D, BATISTIC O, KUDLA J. Calcium sensors and their interacting protein kinases: Genomics of the Arabidopsis and rice CBL-CIPK signaling networks[J]. Plant Physioliogy, 2004, 134(1): 43-58. DOI:10.1104/pp.103.033068 |
| [16] |
CHEN X F, GU Z M, XIN D D, HAO L, LIU C J, HUANG J, MA B J, ZHANG H S. Identification and characterization of putative CIPK genes in maize[J]. Journal of Genetics and Genomics, 2011, 38(2): 77-87. DOI:10.1016/j.jcg.2011.01.005 |
| [17] |
刘涛, 王萍萍, 何红红, 梁国平, 卢世雄, 陈佰鸿, 毛娟. 草莓CIPK基因家族的鉴定与表达分析[J]. 园艺学报, 2020, 47(1): 127-142. DOI:10.16420/j.issn.0513-353x.2019-0219 LIU T, WANG P P, HE H H, LIANG G P, LU S X, CHEN B H, MAO J. Identification and expression analysis of CIPK gene family in strawberry[J]. Acta Horticulturae Sinica, 2020, 47(1): 127-142. DOI:10.16420/j.issn.0513-353x.2019-0219 |
| [18] |
ZHU K K, CHEN F, LIU J Y, CHEN X L, HEWEZI T, CHENG Z M. Evolution of an intron-poor cluster of the CIPK gene family and expression in response to drought stress in soybean[J]. Scientific Reports, 2016, 6(1): 628225. DOI:10.1038/srep28225 |
| [19] |
LI L B, ZHANG Y R, LIU K C, NI Z F, FANG Z J, SUN Q X, GAO J W. Identification and bioinformatics analysis of SnRK2 and CIPK family genes in sorghum[J]. Agricultural Sciences in China, 2010, 9(1): 19-30. DOI:10.1016/S1671-2927(09)60063-8 |
| [20] |
周国华. 水稻冷诱导蛋白激酶基因CIPK07g的克隆与表达分析[J]. 广东农业科学, 2015, 42(21): 150-155. DOI:10.16768/j.issn.1004-874X.2015.21.002 ZHOU G H. Cloning and expression analysis of cold induced proteinkinase gene CIPK07g in rice (Oryza sativa L.)[J]. Guangdong Agricultural Sciences, 2015, 42(21): 150-155. DOI:10.16768/j.issn.1004-874X.2015.21.002 |
| [21] |
MA X, LI Q H, YU Y N, QIAO Y M, HAQ S U, GONG Z H. The CBLCIPK pathway in plant response to stress signals[J]. International Journal of Molecular Sciences, 2020, 21(16): 5668. DOI:10.3390/ijms21165668 |
| [22] |
STRAUB T, LUDEWIG U, NEUHÄUSER B. The kinase CIPK23 inhibits ammonium transport in Arabidopsis thaliana[J]. The Plant Cell, 2017, 29(2): 409-422. DOI:10.1105/tpc.16.00806 |
| [23] |
STRAUB T. Plant ammonium transporter (AMT) integration in regulatory networks[D]. Stuttgar: University of Hohenheim, 2016.
|
| [24] |
朱云娜. 菜心BcAMT1s在铵硝营中调控铵吸收的作用机理研究[D]. 广州: 华南农业大学, 2018. DOI: 10.27152/d.cnki.ghanu.2018.000007. ZHU Y N. Mechanisms for ammonium transporters (BcAMT1s) to regulate ammonium absorption under the mixture of ammonium and nitrate in choysum[D]. Guangzhou: South China Agricultural University, 2018. DOI: 10.27152/d.cnki.ghanu.2018.000007. |
| [25] |
朱云娜, 符质, 冯慧敏, 王斌, 沈雪晴, 汤婉君, 刘建国. 2个菜心CBL基因的克隆、生物信息学分析及其表达特性分析[J]. 山东农业科学, 2023, 55(7): 1-9. DOI:10.14083/j.issn.1001-4942.2023.07.001 ZHU Y N, FU Z, FENG H M, WANG B, SHEN X Q, TANG W J, LIU J G. Cloning, bioinformation analysis and expression analysis of two CBL genes in flowering Chinese cabbage[J]. Shandong Ag ricultural Sciences, 2023, 55(7): 1-9. DOI:10.14083/j.issn.1001-4942.2023.07.001 |
| [26] |
LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [27] |
凌秋平, 曾巧英, 胡斐, 陈健文, 吴嘉云, 李奇伟, 齐永文. 甘蔗CIPK23基因克隆及对低钾、干旱、盐胁迫的响应[J]. 分子植物育种, 2015, 13(6): 1329-1335. DOI:10.13271/j.mpb.013.001329 LING Q P, ZENG Q Y, HU F, CHEN J W, WU J Y, LI Q W, QI Y W. Molecular cloning and expression analysis of CIPK23 gene from sugarcane under low potassium, drought and salt stress[J]. Molecular Plant Breeding, 2015, 13(6): 1329-1335. DOI:10.13271/j.mpb.013.001329 |
| [28] |
李健, 王逸茹, 张凌霄, 孙明昊, 秦阳, 郑军. 玉米ZmCIPK24-2基因在盐胁迫应答中的功能研究[J]. 作物学报, 2020, 46(9): 1351-1358. DOI:10.3724/SP.J.1006.2020.03008 LI J, WANG Y R, ZHANG L X, SUN M H, QIN Y, ZHENG J. Functional analysis of ZmCIPK24-2 gene from maize in response to salt stress[J]. Acta Agronomica Sinica, 2020, 46(9): 1351-1358. DOI:10.3724/SP.J.1006.2020.03008 |
| [29] |
SUN J, BANKSTON J R, PAYANDEH J, HINDS T R, ZAGOTTA W N, ZHENG N. Crystal structure of the plant dual-affinity nitrate transporter NRT1.1[J]. Nature, 2014, 507(7490): 73-77. DOI:10.1038/nature13074 |
| [30] |
HASHIMOTO K, KUDLA J. Calcium decoding mechanisms in plants[J]. Biochimie, 2021, 93(12): 2054-2059. DOI:10.1016/j.biochi.2011.05.019 |
| [31] |
CHEN H Y, CHEN Y N, WANG H Y, LIU Z T, FROMMER W B, HO C H. Feedback inhibition of AMT1 NH4+-transporters mediated by CIPK15 kinase[J]. BMC Biology, 2020, 18(1): 1-13. DOI:10.1186/s12915-020-00934-w |
(责任编辑 马春敏)
 2023, Vol. 50
2023, Vol. 50