文章信息
基金项目
- 国家自然科学基金(3227286,31972544)
作者简介
- 敖广宇(2003—),男,在读本科生,研究方向为动物科学,E-mail:923700668@qq.com.
通讯作者
- 罗文(1988—),男,博士,副教授,研究方向为家禽遗传育种,E-mail:luowen729@scau.edu.cn.
文章历史
- 收稿日期:2023-10-01
【研究意义】随着遗传育种研究和人工选择的不断改进,现代肉鸡具有生长发育快、体重大,适合集约化、规模化养殖等特点[1]。但腹脂沉积过高、过快引起胴体品质下降的现象已逐渐成为肉鸡养殖中普遍存在的弊端[2]。适当的腹脂沉积可以提高鸡肉口感和风味,但腹脂沉积过多会降低饲料转化率和肉鸡屠宰率,影响养殖效益。而且现代工艺对肉鸡腹脂组织的加工较为复杂,加工成本较高,脂肪组织一般作为废弃物处理,对环境造成影响。因此,在保持肉鸡良好生长性能的基础上,改善腹脂过度沉积、培育低脂系肉鸡已成为肉鸡产业亟需解决的问题[3-4]。
【前人研究进展】研究表明,家禽的腹脂沉积主要通过脂肪细胞数量的增多和脂肪细胞体积的肥大两种方式,前者主要发生在家禽生长发育阶段的前期,在生长后期脂肪细胞数量趋于稳定,主要通过脂肪细胞自身的肥大增加腹脂沉积[5]。随着测序技术的发展,转录组测序技术在肉鸡腹脂沉积选育方面发挥了重要作用。陈家辉等[6]利用转录组测序技术对鸡腹脂和肝脏组织中的基因进行了差异表达分析,发现高脂饲养可能通过激活肉鸡脂肪组织的细胞周期来促进前脂肪细胞增殖,进而增加肉鸡的腹脂沉积。可变剪接是转录后水平上调节基因表达的重要机制。在真核生物中,基于基因转录生成的mRNA前体(PremRNA)可通过不同的剪接方式形成多个成熟的mRNA,极大地丰富了真核生物的蛋白质组多样性和基因表达调控的复杂性[7]。可变剪接对于脂肪细胞分化和脂质代谢起到不可或缺的作用[8]。【本研究切入点】目前国内大多数研究都只探索基因表达水平对家禽性状的影响,而忽略了转录本结构的作用。在肉鸡腹脂沉积过程中的可变剪接事件及调控这些事件的主要遗传因子仍未知。【拟解决的关键问题】本研究在传统的基因表达分析基础上增加了可变剪接分析,并将显著差异表达和可变剪接基因进行联合分析,对剪接因子在该过程中的作用进行探索。本研究基于高脂饲喂肉鸡和普通饲喂肉鸡腹脂和肝脏组织的转录组二代测序结果,对不同饲喂处理肉鸡的2种组织进行基因表达和可变剪接比较分析,以期揭示高脂饲喂如何影响肝脏和腹脂的RNA可变剪接,探明RNA可变剪接在腹脂沉积和肝脏脂质代谢中的调控作用,找出影响肉鸡脂质代谢和腹脂沉积的关键基因和转录本。
1 材料与方法 1.1 试验材料试验肉鸡为体重相近的8周龄纯种杏花母鸡,来源于华南农业大学科研禽场,随机分成2组(每组6只),分别饲喂高脂饲料(含40% 碳水化合物、25% 脂肪和20% 蛋白质)和普通饲料(含41% 碳水化合物、5% 脂肪和22% 蛋白质),饲料由北京博泰宏达生物技术有限公司提供。连续饲喂两周后,对肉鸡进行称重和屠宰,采集肝脏组织和腹脂组织,液氮速冻后置于-80 ℃冰箱中保存备用[6]。
1.2 试验方法试验数据来源于试验肉鸡的肝脏和腹脂样品总RNA。利用深圳华大基因公司BGI-500测序平台进行转录组测序,所有序列数据均保存在NCBI'S Gene Expression Omnibus (GEO,http://www.ncbi.nim.nih.gov/geo),登录号为GSE128340。
1.2.1 可变剪接事件的鉴别 利用STAR(2.7.10b)软件[9]识别比对序列数据,参考基因组为Gallus gallus 6.0(http://ftp.ensembl.org/pub/release-106/fasta/gallus_gallus/dna/),比对结果通过Samtools(1.17)软件[10]进行排序。利用rMATS(4.1.2)软件[11]鉴别5种可变剪接事件,包括跳跃外显子(Skipping exon,SE)、可变的5' 和3' 剪接位点(Alternative 5' splice site,A5SS; Alternative 3' splice site,A3SS)、保留内含子(Retained intron,RI)、互斥外显子(Mutually exclusive exon,MXE)。本研究采用Junction Count only(JC,only)定量方式,以满足PSI差值的绝对值≥ 0.1且P ≤ 0.05这两个条件的特征基因作为显著差异剪接基因[12]。基于STAR软件比对的结果,利用DESeq2(1.41.10)软件[13]进行差异表达分析,以满足log2FoldChange的绝对值> 1且P ≤ 0.05这两个条件的特征基因作为显著差异表达基因[14]。
1.2.2 差异可变剪接基因功能富集分析 采用ClusterProfiler(4.9.3)软件[15]在Rstudio (4.3.0) 上进行GO功能注释和KEGG通路富集分析,以P ≤ 0.05作为筛选显著富集通路的标准;使用Metascape(https://www.metascape.org/)在线网站[16]对差异可变剪接基因进行富集分析。
1.2.3 差异剪接基因与差异表达基因联合分析 基于可变剪接事件的鉴别结果,对腹脂和肝脏组织中显著差异表达和剪接基因进行联合分析。利用在线网站(https://www.metascape.org/)绘制火山图和韦恩图。基于DESeq2软件KEGG通路富集和Metascape富集分析。
1.2.4 代表性差异剪接事件可视化分析 使用Rmats2sashimiplot(2.0.4)软件[11]可视化分析高脂饲喂下脂肪形成关键基因对可变剪接的影响,绘制出腹脂和肝脏组织中关键基因发生的可变剪接事件。基于STAR软件生成bam文件,通过rMAPS2(http://rmaps.cecsresearch.org/)在线网站[17],绘制出剪接因子对剪接事件调控作用的可视图谱。
2 结果与分析 2.1 不同饲喂处理肉鸡腹脂和肝脏组织的显著差异剪接基因统计分析为了探究高脂饲喂下肉鸡腹脂和肝脏组织中发生的差异可变剪接事件,对高脂饲喂肉鸡和普通饲喂肉鸡中腹脂和肝脏组织的转录组数据进行可变剪接分析。相比普通饲喂肉鸡,高脂饲喂肉鸡腹脂、肝脏组织中分别有372、235个显著可变剪接事件,SE类型是最丰富的可变剪接类型(图 1)。对转录组数据进行基因表达和可变剪接联合分析,发现高脂饲喂肉鸡的腹脂组织中有233个显著差异表达基因、349个显著差异剪接基因,通过韦恩图发现共有4个交集基因;肝脏组织中有276个显著差异表达基因,224个显著差异剪接基因,通过韦恩图发现共有2个交集基因。
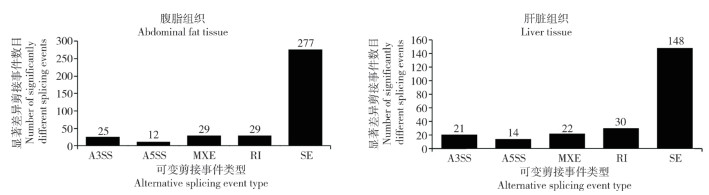
|
| 图 1 肉鸡腹脂和肝脏组织显著差异可变剪接事件类型统计 Fig. 1 Statistics of significantly differential alternative splicing event types in abdominal fat and liver tissues of broiler |
2.2 不同饲喂处理肉鸡腹脂组织的差异剪接基因功能富集分析
为了揭示参与肉鸡腹脂沉积的差异剪接基因所涉及的生物学进程,对获得的腹脂组织显著差异剪接基因进行GO功能注释、KEGG通路富集分析和Metascape富集分析。GO功能注释结果(图 2A)显示,这些基因显著富集于细胞分裂、细胞发育生长等细胞增殖相关的生物学进程。KEGG通路富集结果(图 2B)显示,这些基因显著富集于Rap1信号通路、甘油磷脂代谢和胰岛素信号通路。Metascape富集结果(图 2C)显示,这些基因显著富集于细胞形态发生、细胞生长、细胞分裂等细胞增殖相关生物学进程,暗示高脂饲养可能激活了肉鸡腹脂组织中前脂肪细胞的增殖。在这些富集基因中发现4个与细胞增殖相关的基因,包括Btk、Ctnnb1、Pard3和Fat1。
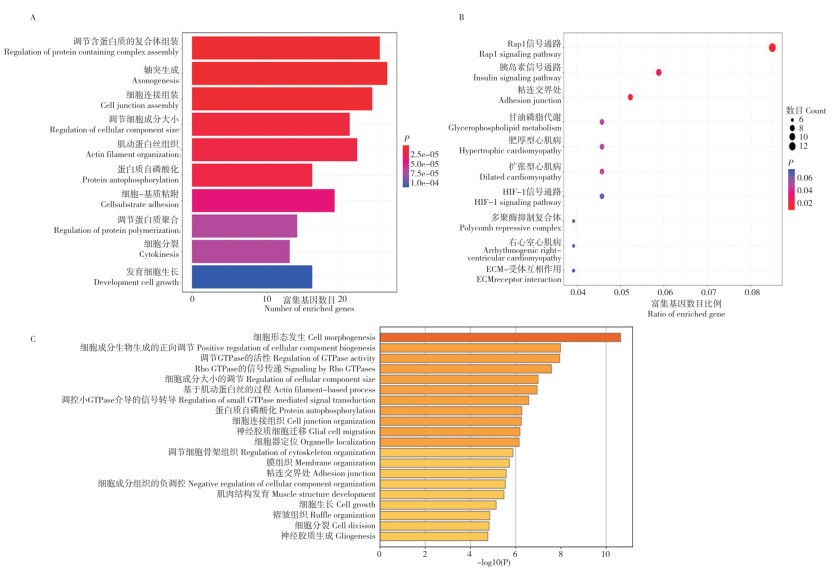
|
| A:GO功能注释,展示显著性前10的条目;B:KEGG通路富集分析,展示显著性前10的条目;C:Metascape富集分析,展示显著性前20的条目;各富集条目筛选条件均为P ≤ 0.05 A: GO functional annotation, the top ten significant entries are selected for display; B: KEGG pathway enrichment analysis, the top ten significant entries are selected for display; C: Metascape enrichment analysis, the top twenty entries are selected for display; The screening condition for each enriched entry is P ≤ 0.05 图 2 肉鸡腹脂组织差异剪接基因功能富集分析 Fig. 2 Functional enrichment analysis of differential splicing genes of abdominal fat tissue in broiler |
2.3 不同饲喂处理肉鸡肝脏组织的差异剪接基因功能富集分析
肝脏是动物体脂肪代谢的重要器官,对获得的肝脏组织显著差异剪接基因进行GO功能注释、KEGG通路富集分析和Metascape富集分析。GO功能注释结果(图 3A)显示,这些基因显著富集于组蛋白乙酰化、蛋白质内部氨基酸乙酰化等细胞代谢相关生物学进程。KEGG通路富集结果(图 3B)显示,这些基因富集于胰岛素信号通路、胰高血糖素信号通路和AMPK信号通路等脂肪生成相关通路。Metascape富集分析结果(图 3C)显示,这些基因显著富集于细胞间通讯、组蛋白乙酰化、mRNA加工等生物学进程,此外还富集于胰岛素信号通路,表明高脂饲养可能激活了肝脏组织中脂肪生成相关细胞代谢。
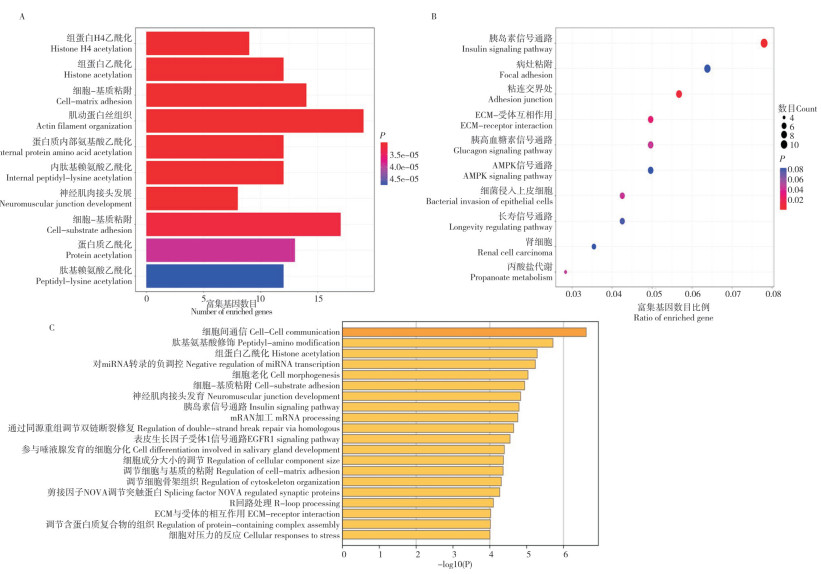
|
| A:GO功能注释,展示显著性前10的条目;B:KEGG通路富集分析,展示显著性前10的条目;C:Metascape富集分析,展示显著性前20的条目;各富集条目筛选条件均为P ≤ 0.05 A: GO functional annotation, the top ten significant entries are selected for display; B: KEGG pathway enrichment analysis, the top ten significant entries are selected for display; C: Metascape enrichment analysis, the top twenty entries are selected for display; The screening condition for each enriched entry is P ≤ 0.05 图 3 肉鸡肝脏组织差异剪接基因功能富集分析 Fig. 3 Functional enrichment analysis of differential splicing genes of liver tissue in broiler |
2.4 不同饲喂处理肉鸡腹脂组织的差异表达基因分析
对高脂饲喂肉鸡和普通饲喂肉鸡的腹脂组织进行差异表达基因分析, 结果(图 4A)发现,高脂饲喂肉鸡相比普通饲喂肉鸡有248个基因表达显著上调,60个基因表达显著下调。对所有显著差异表达基因进行GO功能注释,发现这些基因显著富集于染色体分离、核分裂、细胞周期正调控等与细胞增殖相关的生物学进程(图 4B);KEGG通路富集分析发现,这些基因显著富集于细胞周期通路和类固醇生物合成和P53信号通路等(图 4C);Metascape富集分析发现,富集结果与GO功能注释和KEGG富集结果相符,富集于有丝分裂细胞周期、有丝分裂等与细胞增殖相关生物学进程,以及富集于类固醇的代谢等生物学进程(图 4D)。综上,高脂饲喂可能激活了肉鸡腹脂组织前脂肪细胞的增殖。
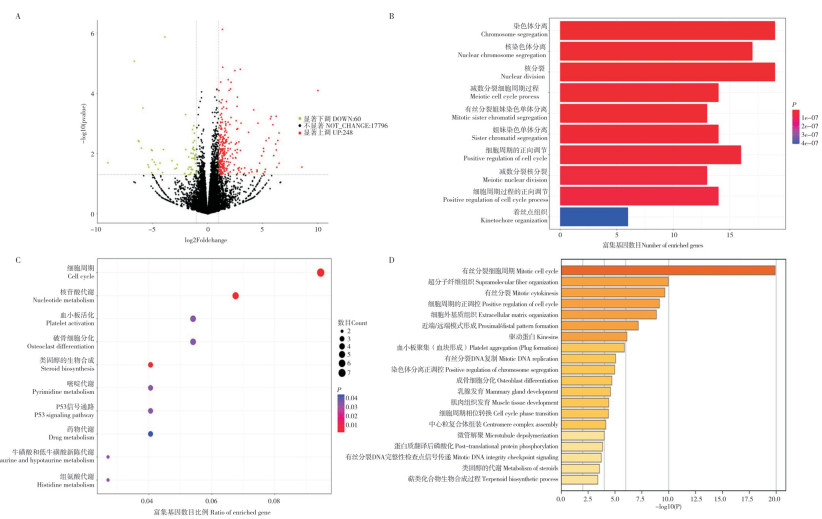
|
| A:火山图;B:GO功能注释,展示显著性前10的条目;C:KEGG通路富集分析,展示显著性前10的条目;D:Metascape富集分析,展示显著性前前20的条目;各富集条目筛选条件均为P ≤ 0.05 A: Volcano plot; B: GO functional annotation, the top ten significant entries are selected for display; C: KEGG pathway enrichment analysis, the top ten significant entries are selected for display; D: Metascape enrichment analysis, the top twenty entries are selected for display. The screening condition for each enriched entry is P ≤ 0.05 图 4 肉鸡腹脂组织差异表达基因功能富集分析 Fig. 4 Functional enrichment analysis of differentially expressed genes of abdominal adipose tissue in broiler |
2.5 不同饲喂处理肉鸡肝脏组织的差异表达基因分析
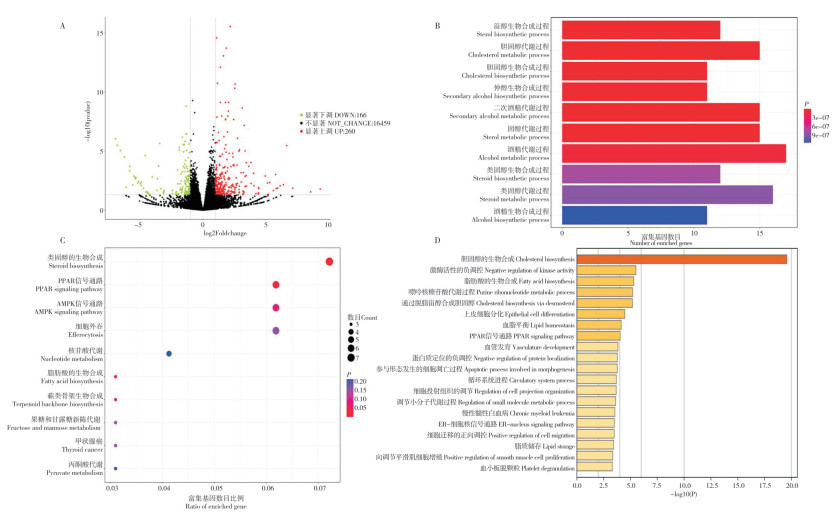
对高脂饲喂肉鸡和普通饲喂肉鸡的肝脏组织进行差异表达基因分析,结果(图 5A)发现,高脂饲喂肉鸡相比普通饲喂肉鸡有260个基因表达显著上调,有166个基因表达显著下调。对所有显著差异表达基因进行GO功能富集分析,发现这些基因显著富集于类固醇相关的生物合成和代谢进程(图 5B);KEGG通路富集分析发现,这些基因显著富集于类固醇生物合成、PPAR信号通路、AMPK信号通路(图 5C);Metascape富集分析发现,这些基因显著富集于胆固醇生物合成、脂肪酸生物合成、脂质贮存等脂肪生成相关生物学进程(图 5D),暗示高脂饲喂条件可能激活了肝脏组织脂肪生成相关细胞的增殖。
|
| A:火山图;B:GO功能注释,展示显著性前10的条目;C:KEGG通路富集分析,展示显著性前10的条目;D:Metascape富集分析,展示显著性前前20的条目;各富集条目筛选条件均为P ≤ 0.05 A: Volcano plot; B: GO functional annotation, the top ten significant entries are selected for display; C: KEGG pathway enrichment analysis, the top ten significant entries are selected for display; D: Metascape enrichment analysis, the toptwenty entries are selected for display; The screening condition for each enriched entry is P ≤ 0.05 图 5 肉鸡肝脏组织差异表达基因功能富集分析 Fig. 5 Functional enrichment analysis of differentially expressed genes of liver in broiler |
2.6 高脂饲喂对脂肪形成关键基因可变剪接的影响
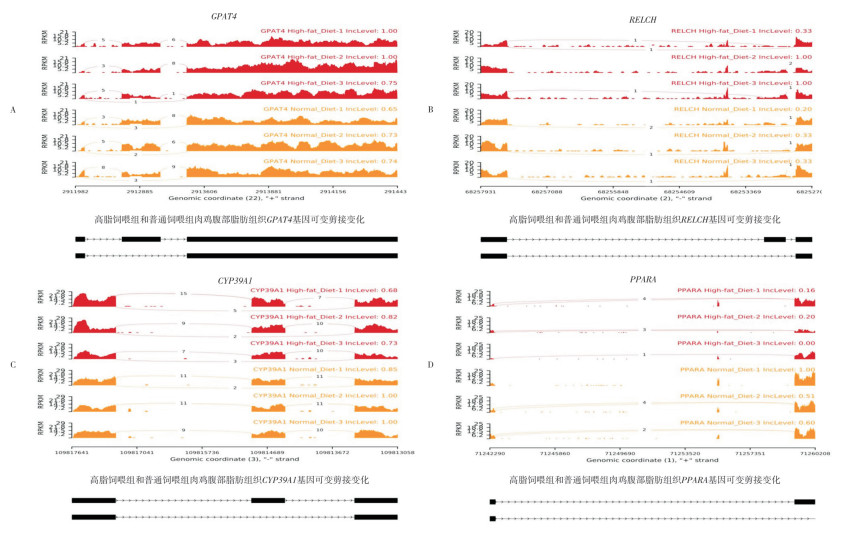
为了探究高脂饲喂对脂肪生成中关键基因可变剪接的调控,将高脂饲喂肉鸡和普通饲喂肉鸡腹脂和肝脏组织中发生的显著差异可变剪接事件进行汇总。通过基因注释和PSI值,寻找到4个参与影响脂肪生成的基因,包括GPAT4、RELCH、CYP39A1和PPARA。将4个基因的可变剪接差异进行可视化处理(图 6),发现基因GPAT4、RELCH在高脂饲喂条件下可变剪接情况上调,基因CYP39A1、PPARA在高脂饲喂条件下可变剪接情况下调;主要为SE类型可变剪接事件,与可变剪接事件的统计结果相符,印证了SE型可变剪接在高脂饲喂条件下发生显著的差异表达。推断在高脂饲喂条件下,腹脂沉积主要依靠SE型可变剪接事件来调节。
|
| 红色图形为高脂饲喂处理,橙色图形为普通饲喂处理;图底黑色横线为该转录本结构 The red graphs are for the high-fat-fed group and the orange graphs are for the normal-fed group; The black horizontal line at the bottom of the graph shows the structure of this transcription 图 6 代表性差异可变剪接基因的可视化 Fig. 6 Visualisation of representative differentially variable splicing genes |
2.7 调控脂肪形成的重要剪接因子可视化分析
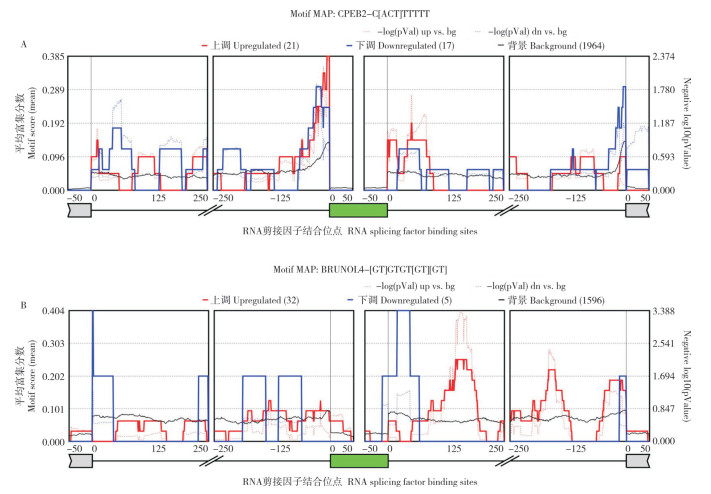
对腹脂和肝脏组织中显著调节可变剪接事件剪接因子进行可视化分析,比较高脂饲喂肉鸡和普通饲喂肉鸡腹脂组织SE型剪接因子调控可变剪接事件的过程,发现有21个剪接因子对靶基因剪接呈正向调节、17个剪接因子对靶基因剪接呈抑制调节(图 7A)。对于CPEB2剪接因子,发现其对靶基因的剪接主要富集于内含子区域,较少富集于外显子区域。当促进靶基因剪接时,CPEB2剪接因子在主要靶基因上游侧内含子区域富集;当抑制靶基因剪接时,CPEB2剪接因子同样主要富集于靶基因上游侧内含子区域(图 7A)。比较高脂饲喂肉鸡和普通饲喂肉鸡肝脏组织SE型剪接因子调控可变剪接事件的过程,发现有32个剪接因子对靶基因剪接呈正向调节、5个剪接因子对靶基因剪接呈抑制调节(图 7B)。对于BRUNOL4剪接因子,发现其对靶基因剪接的调控作用主要富集于内含子区域。当促进靶基因剪接时,BRUNOL4剪接因子主要富集于靶基因下游侧内含子区域;当抑制靶基因剪接时,BRUNOL4剪接因子同样主要富集于靶基因下游侧内含子区域(图 7B)。综上,高脂饲喂肉鸡脂肪沉积性状可能与剪接因子CPER2和BRUNOL4对靶基因的调控作用有关。
|
|
A: CPEB2 B: BRUNOL4 图中红线为剪接因子对靶基因正向调控作用,蓝线为剪接因子对靶基因负向调控作用;绿色方框为外显子区域,黑色直线为内含子区域 The red line in the graph shows the positive regulatory effect of splicing factors on target genes, and the blue line shows the negative regulatory effect of splicing factors on target genes; The green box is the exon region and the black straight line is the intron region 图 7 代表性剪接因子对靶基因调控作用的可视化 Fig. 7 Visualisation of the role of representative splicing factors in the regulation of target genes |
3 讨论
可变剪接是多细胞真核生物转录组高度复杂的重要原因,丰富了生物体蛋白质和表型性状的多样性[18]。蛋白质参与生物体的许多生命过程,它们与蛋白质、RNA、DNA等其他物质相互作用发挥生物功能[19-21]。剪接因子是一种反式作用元件,可识别或结合一些在内含子或外显子上的正调控(剪接增强子)或负调控(剪接沉默子)的顺式作用元件,在不同组织或不同发育阶段选择不同剪接位点产生不同的剪接方式[22]。本研究通过对重要剪接因子调控靶基因的过程进行可视化分析,发现有大量的剪接因子和靶基因互作,并显著调节靶基因的表达,推断剪接因子和靶基因的相互作用在肉鸡腹脂沉积调节过程中发挥重要功能。
本研究在高脂饲喂肉鸡腹脂沉积的过程中检测到许多可变剪接事件,以SE类型可变剪接事件最为丰富,在腹脂组织中大量富集,推断在高脂饲喂条件下SE类型可变剪接事件主要参与肉鸡腹脂沉积过程。Lu等[23]和Chen等[24]研究发现SE类型可变剪接是主要的剪接模式。本研究在探究高脂饲喂对脂肪形成关键基因可变剪接的影响中发现,多数转录本可变剪接类型也为SE类型,推断SE类型可变剪接可能是肉鸡腹脂沉积可变剪接调节的主要模式。
肉鸡腹脂沉积的调控受到多种因素影响,大量的基因、剪接因子、转录本、脂肪代谢相关激素都对鸡脂肪生成过程具有调控作用[25]。PPAR为Ⅱ型受体超家族成员,包括3个亚型(PPARα、PPARβ/δ、PPARγ)。研究表明,PPAR参与许多细胞内外脂类代谢过程,如肝脏组脂肪酸分解、脂蛋白组装和转运等,也能调控微粒体、过氧化物酶体β氧化过程中起重要作用的酶类基因表达,参与机体脂肪代谢过程[26]。除了PPAR信号通路外,胰岛素信号通路也在脂肪代谢过程中发挥重要作用。研究表明,胰岛素信号通路和PPAR信号通路可以相互作用参与到脂肪生成的过程中,进而调节机体脂肪代谢[27]。本研究发现高脂饲喂肉鸡和普通饲喂肉鸡腹脂和肝脏组织中显著差异表达和可变剪接基因显著富集于细胞分裂、细胞生长等细胞增殖相关生物学进程和蛋白质乙酰化、mRNA加工等细胞代谢相关生物学进程,还富集于甘油磷脂代谢、胰岛素信号通路、胰高血糖素信号通路和AMPK信号通路等脂肪生成相关通路。
显著差异剪接基因富集结果暗示高脂饲喂可能通过激活肉鸡腹脂组织前脂肪细胞的增殖和肝脏组织脂肪生成相关细胞代谢途径,进而引起肉鸡腹脂沉积;推断高脂饲喂可能通过促进前脂肪细胞增殖和相关脂肪细胞代谢,参与到可变剪接调控过程,从而导致肉鸡腹脂沉积。本研究还检测到大量剪接因子对靶基因的调控作用,部分剪接因子在高脂饲喂条件下发生显著差异表达,推断高脂饲喂可能通过作用于剪接因子对RNA的剪接作用,影响RNA可变剪接导致肉鸡腹脂沉积。
随着二代测序技术的快速发展,可变剪接分析的技术手段越来越成熟。本研究比较高脂饲喂肉鸡和普通饲喂肉鸡中肝脏和腹脂组织的可变剪接分析结果,筛选出4个脂肪形成关键基因包括:GPAT4、RELCH、CYP39A1、PPARA。研究表明这些基因都参与脂肪组织的生成代谢。刘亿兰[28]研究发现,GPAT4在小鼠脂肪组织和肝脏中高表达,通过基因过表达和基因敲除实验证实GPAT4对肥胖、肝脏脂肪变性的发生发展具有重要作用。Sobajima等[29]研究表明,RELCH通过膜束缚促进非囊泡性胆固醇从再循环内体到反面高尔基体网状结构的转运。Han等[30]在小鼠肠道微生物与脂质代谢失调的机制研究中,通过对小鼠肝脏组织进行转录组测序,发现肠道微生物群的消耗改变了参与胆固醇和脂肪酸代谢的基因表达包括CYP39A1,认为小鼠肝脏脂质的积累可能与CYP39A1基因的差异表达有关。Zhou等[31]通过皮下注射皮质酮的方式,观察肉鸡内脏脂肪的沉积,发现PPARA出现以m6A介导的平移抑制机制,认为平移抑制机制有助于皮质酮诱导的鸡内脏脂肪沉积。GPAT4、RELCH、CYP39A1、PPARA基因也与肝脏组织中胆固醇的含量密切相关。Cao等[32]研究发现,在饮食诱导的肥胖中GPAT4基因的表达和胆固醇稳态密切联系。Zhang等[33]研究发现,RELCH对胆固醇的转运有调控作用。Lathe等[34]研究表明,在肝脏中由CYP7A和CYP39A1介导胆固醇的1α-羟基化是胆汁酸合成和代谢消除的限速步骤。Aibara等[35]研究认为PPARA的表达显著影响肝细胞增殖。本研究同样发现GPAT4、RELCH、CYP39A1、PPARA基因在肉鸡腹脂和肝脏组织的可变剪接事件中显著差异表达,结合前人研究成果,推断这4个基因可能是与肉鸡腹脂沉积相关的潜在可变剪接基因。
4 结论本研究对高脂饲喂肉鸡和普通饲喂肉鸡的肝脏和腹脂组织分别进行了基因表达分析和可变剪接分析,初步揭示出高脂饲喂调控肉鸡肝脏和腹脂的RNA可变剪接调控规律。根据差异剪接基因的富集结果发现,差异剪接基因显著富集于细胞增殖、代谢相关生物学进程和甘油磷脂代谢、胰岛素信号通路、AMPK信号通路等脂肪生成相关通路,推断高脂饲喂可能通过影响基因可变剪接过程,进而激活前脂肪细胞的增殖和脂肪生成相关细胞代谢,最终导致肉鸡的腹脂沉积现象。在显著差异剪接基因中还发现了GPAT4、RELCH、CYP39A1、PPARA等4个潜在的通过可变剪接调节肉鸡腹脂沉积的剪接基因。探究可变剪接调控过程中剪接因子对靶基因的调控作用,发现大量剪接因子显著调控靶基因表达,如CPER2和BRUNOL4剪接因子,推断剪接因子通过对靶基因的调控作用参与肉鸡的腹脂沉积过程。
| [1] |
余春林, 蒋小松, 刘益平, 杜华锐, 尹华东, 杨朝武. 2020年肉鸡遗传育种研究进展[J]. 中国家禽, 2021, 43(9): 1-5. DOI:10.16372/j.issn.1004-6364.2021.09.001 YU C L, JIANG X S, LIU Y P, DU H R, YIN H D, YANG C W. Advance on genetics and breeding for broiler in 2020[J]. China Poultry, 2021, 43(9): 1-5. DOI:10.16372/j.issn.1004-6364.2021.09.001 |
| [2] |
刘福柱. 肉鸡腹脂沉积的遗传及其选择进展[J]. 甘肃畜牧兽医, 1990(5): 28-29, 35. DOI:10.15979/j.cnki.cn62-1064/s.1990.05.020 LIU F Z. Genetics of abdominal fat deposition in broiler chickens and progress in its selection[J]. Gansu Animal Husbandry and Veterinary, 1990(5): 28-29, 35. DOI:10.15979/j.cnki.cn62-1064/s.1990.05.020 |
| [3] |
郎倩倩, 肖昊, 王丽, 高开国. 优质鸡腹脂选择方法研究进展[J]. 广东农业科学, 2019, 46(9): 120-127. DOI:10.16768/j.issn.1004-874X.2019.09.017 LANG Q Q, XIAO H, WANG L, GAO K G. Research progress on selection methods of abdominal fat of high quality chicken[J]. Guangdong Agricultural Sciences, 2019, 46(9): 120-127. DOI:10.16768/j.issn.1004-874X.2019.09.017 |
| [4] |
田卫华, 聂瑞雪, 张文慧, 凌遥, 田浩宇, 张博, 张浩, 吴常信. 肉鸡腹脂遗传规律及基因调控研究进展[J]. 农业生物技术学报, 2023, 31(11): 2387-2400. DOI:10.3969/j.issn.1674-7968.2023.11.016 TIAN W H, NIE R X, ZHANG W H, LING Y, TIAN H Y, ZHANG B, ZHANG H, WU C X. Research progress on genetic characteristics and genes regulation of abdominal fat in broiler chickens[J]. Journal of Agricultural Biotechnology, 2023, 31(11): 2387-2400. DOI:10.3969/j.issn.1674-7968.2023.11.016 |
| [5] |
张平平, 毋文静, 李勇, 刘铀, 刘艳芬. 动物前脂肪细胞研究进展[J]. 广东农业科学, 2011, 38(6): 137-139, 148. DOI:10.16768/j.issn.1004-874X.2011.06.076 ZHANG P P, WU W J, LI Y, LIU Y, LIU Y F. Advances in the study of animal preadipocytes[J]. Guangdong Agricultural Sciences, 2011, 38(6): 137-139, 148. DOI:10.16768/j.issn.1004-874X.2011.06.076 |
| [6] |
陈家辉, 任学义, 李丽敏, 卢诗意, 程湉, 谭量天, 梁少东, 何丹林, 罗庆斌, 聂庆华, 张细权, 罗文. 转录组测序揭示细胞周期通路参与鸡腹脂沉积[J]. 遗传, 2019, 41(10): 962-973. DOI:10.16288/j.yczz.19-098 CHEN J H, REN X Y, LI L M, LU S Y, CHENG T, TAN L T, LIANG S D, HE D L, LUO Q B, NIE Q H, ZHANG X Q, LUO W. The cell cycle pathway regulates chicken abdominal fat deposition as revealed by transcriptome sequencing[J]. Hereditas, 2019, 41(10): 962-973. DOI:10.16288/j.yczz.19-098 |
| [7] |
韩林宏, 江海洋. 植物选择性剪接研究进展[J]. 分子植物育种, 2020, 18(10): 3259-3265. DOI:10.13271/j.mpb.018.003259 HAN L H, JIANG H Y. The Research progress of alternative splicing in plant[J]. Molecular Plant Breeding, 2020, 18(10): 3259-3265. DOI:10.13271/j.mpb.018.003259 |
| [8] |
骞鑫, 马海明, 何俊, 徐康, 张跃博. 可变剪接的表观遗传学调控机制及其在脂肪代谢中的作用研究进展[J]. 中国畜牧杂志, 2021, 57(2): 46-53. DOI:10.19556/j.0258-7033.20201103-02 QIAN X, MA H M, HE J, XU K, ZHANG Y B. Advances in the epigenetic regulation mechanism of alternative splicing and its role in lipid metabolism[J]. Chinese Journal of Animal Science, 2021, 57(2): 46-53. DOI:10.19556/j.0258-7033.20201103-02 |
| [9] |
DOBIN A, DAVIS C A, SCHLESINGER F, DRENKOW J, ZALESKI C, JHA S, BATUT P, CHAISSON M, GINGERAS T R. STAR: Ultrafast universal RNA-seq aligner[J]. Bioinformatics (Oxford, England), 2013, 29(1): 15-21. DOI:10.1093/bioinformatics/bts635 |
| [10] |
DANECEK P, BONFIELD J K, LIDDLE J, MARSHALL J, OHAN V, POLLARD M O, WHITWHAM A, KEANE T, MCCARTHY S A, DAVIES R M, LI H. Twelve years of SAMtools and BCFtools[J]. Gigascience, 2021, 10(2): giab008. DOI:10.1093/gigascience/giab008 |
| [11] |
SHIHAO S, JUW W P, LU Z X. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(51): E5593-E5601. DOI:10.1073/pnas.1419161111 |
| [12] |
BYUN S, HAN S, ZHENG Y, PLANELLES V, LEE Y. The landscape of alternative splicing in HIV-1 infected CD4 T-cells[J]. BMC Medical Genomics, 2020, 13(S5): 38. DOI:10.1186/s12920-020-0680-7 |
| [13] |
LOVE M, HUBER W, ANDERS S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550. DOI:10.1186/s13059-014-0550-8 |
| [14] |
王金玉. 基于统计学习的甲状腺癌分期特征及预后基因选取研究[D]. 济南: 山东财经大学, 2022. DOI: 10.27274/d.cnki.gsdjc.2022.000023. WANG J Y. Statistical learning based on thyroid cancer staging characteristic genes and prognostic genes selection study[D]. Jinan: Shandong University of Finance and Economics, 2022. DOI: 10.27274/d.cnki.gsdjc.2022.000023. |
| [15] |
YU G C, WANG L G, HAN Y, HE Q Y. ClusterProfiler: An R package for comparing biological themes among gene clusters[J]. OMICS A Journal of Integrative Biology, 2012, 16(5): 284-287. DOI:10.1089/omi.2011.0118 |
| [16] |
ZHOU Y, ZHOU B, PACHE L, CHANG M, KHODABAKHSHI A H, TANASEICHUK O, BENNER C, CHANDA S K. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nature Communication, 2019, 10(1): 1523. DOI:10.1038/s41467-019-09234-6 |
| [17] |
HWANG J Y, JUNG S, KOOK T L, ROUCHKA E C, BOK J, PARK J W. rMAPS2: An update of the RNA map analysis and plotting server for alternative splicing regulation[J]. Nucleic Acids Research, 2020, 48(W1): W300-W306. DOI:10.1093/nar/gkaa237 |
| [18] |
疏泽, 王立贤, 王立刚. 可变剪接及其在畜禽育种中的研究与应用[J]. 畜牧兽医学报, 2020, 51(12): 2911-2920. DOI:10.11843/j.issn.0366-6964.2020.12.001 SHU Z, WANG L X, WANG L G. Research progress of alternative splicing and its application in livestock and poultry breeding[J]. Acta Veterinaria et Zootechnica Sinica, 2020, 51(12): 2911-2920. DOI:10.11843/j.issn.0366-6964.2020.12.001 |
| [19] |
ULE J, BLENCOWE B J. Alternative splicing regulatory networks: Functions, mechanisms, and evolution[J]. Molecular Cell, 2019, 76(2): 329-345. DOI:10.1016/j.molcel.2019.09.017 |
| [20] |
YAN Y, HUANG T. The interactome of protein, DNA, and RNA[J]. Methods Molecular Biology, 2023, 2695: 89-110. DOI:10.1007/978-1-0716-3346-5_6 |
| [21] |
李莹, 瞿浩, 何静怡, 刘天飞, 王劼, 王艳, 舒鼎铭, 罗成龙. 鸡胚来源的外泌体不同提取方法比较[J]. 广东农业科学, 2020, 47(5): 93-101. DOI:10.16768/j.issn.1004-874X.2020.05.013 LI Y, QU H, HE J Y, LIU T F, WANG J, WANG Y, SHU D M, LUO C L. Comparison of different extraction methods of exosomes from chicken embryos[J]. Guangdong Agricultural Sciences, 2020, 47(5): 93-101. DOI:10.16768/j.issn.1004-874X.2020.05.013 |
| [22] |
万敬员, 张力, 叶笃筠. 选择性剪接调控机制的研究进展[J]. 医学分子生物学杂志, 2003, 25(6): 342-345. DOI:10.3870/j.issn.1672-8009.2003.06.008 WAN J Y, ZHANG L, YE D Y. Advances in selective splicing regulatory mechanisms[J]. Journal of Medical Molecular Biology, 2003, 25(6): 342-345. DOI:10.3870/j.issn.1672-8009.2003.06.008 |
| [23] |
LU Y, TAN L, XIE J, CHENG L, WANG X. Distinct microglia alternative splicing in Alzheimer's disease[J]. Aging (Albany NY), 2022, 14(16): 6554-6566. DOI:10.18632/aging.204223 |
| [24] |
CHEN Q, HUANG C, SU Y, ZHAO Q, PU Y, HE X, JIANG L, MA Y, ZHAO Q, YE S. Transcriptomic analysis reveals mRNA and alternative splicing events in ovine skeletal muscle satellite cells during proliferation and differentiation[J]. Animals (Basel), 2023, 13(6): 1076. DOI:10.3390/ani13061076 |
| [25] |
李姣, 胡群, 罗佩, 李文平. 肝脏脂质代谢关键转录因子研究进展[J]. 动物医学进展, 2016, 37(4): 90-93. DOI:10.16437/j.cnki.1007-5038.2016.04.020 LI J, HU Q, LUO P, LI W P. Progress on key transcription factors in liver lipid metabolism[J]. Progress in Veterinary Medicine, 2016, 37(4): 90-93. DOI:10.16437/j.cnki.1007-5038.2016.04.020 |
| [26] |
李昌庆, 苏瑛. PPAR家族和PGC-1α与动物遗传育种[J]. 广东农业科学, 2009(8): 174-176, 182. DOI:10.16768/j.issn.1004-874X.2009.08.067 LI C Q, SU Y. PPAR family, PGC-1α and animal genetic breeding[J]. Guangdong Agricultural Sciences, 2009(8): 174-176, 182. DOI:10.16768/j.issn.1004-874X.2009.08.067 |
| [27] |
ABDALLA B A, CHEN J, NIE Q, ZHANG X. Genomic insights into the multiple factors controlling abdominal fat deposition in a chicken model[J]. Frontiers in Genetics, 2018, 9: 262. DOI:10.3389/fgene.2018.00262 |
| [28] |
刘亿兰. 敲除肝脏GPAT4改善小鼠胰岛素抵抗及非酒精性脂肪性肝病研究[D]. 福州: 福建医科大学, 2020. DOI: 10.27020/d.cnki.gfjyu.2020.000482. LIU Y L. The Study of liver-specific GPAT4 knockout improves insulin resistance and nonalcoholic fatty liver disease in mice[D]. Fuzhou: Fujian Medical University, 2020. DOI: 10.27020/d.cnki.gfjyu.2020.000482. |
| [29] |
SOBAJIMA T, YOSHIMURA S I, MAEDA T, MIYATA H, MIYOSHI E, HARADA A. The Rab11-binding protein RELCH/KIAA1468 controls intracellular cholesterol distribution[J]. Journal of Cell Biology, 2018, 217(5): 1777-1796. DOI:10.1083/jcb.201709123 |
| [30] |
HAN H, WANG M, ZHONG R, YI B, SCHROYEN M, ZHANG H. Depletion of gut microbiota inhibits hepatic lipid accumulation in high-fat diet-fed mice[J]. International Journal of Molecular Sciences, 2022, 23(16): 9350. DOI:10.3390/ijms23169350 |
| [31] |
ZHOU Z, ZHANG A, LIU X, YANG Y, ZHAO R, JIA Y. m6A-Mediated PPAR A translational suppression contributes to corticosterone-induced visceral fat deposition in chickens[J]. International Journal of Molecular Sciences, 2022, 23(24): 15761. DOI:10.3390/ijms232415761 |
| [32] |
CAO J, PEREZ S, GOODWIN B, LIN Q, PENG H, QADRI A, ZHOU Y, CLARK R W, PERREAULT M, TOBIN J F, GIMENO R E. Mice deleted for GPAT3 have reduced GPAT activity in white adipose tissue and altered energy and cholesterol homeostasis in diet-induced obesity[J]. American Journal of Physiology, Endocrinology and Metabolism, 2014, 306(10): E1176-E1187. DOI:10.1152/ajpendo.00666.2013 |
| [33] |
ZHANG R, LI X, MA Y, LIU Y, ZHANG Y, CHENG X, NING Z. Identification of candidate genomic regions for thermogelled egg yolk traits based on a genome-wide association study[J]. Poultry Science, 2023, 102(3): 102402. DOI:10.1016/j.psj.2022.102402 |
| [34] |
LATHE R. Steroid and sterol 7-hydroxylation: Ancient pathways[J]. Steroids, 2002, 67(12): 967-977. DOI:10.1016/s0039-128x(2)00044-2 |
| [35] |
AIBARA D, TAKAHASHI S, YAGAI T, KIM D, BROCKER C N, LEVI M, MATSUSUE K, GONZALEZ F J. Gene repression through epigenetic modulation by PPARA enhances hepatocellular proliferation[J]. iScience, 2022, 25(5): 104196. DOI:10.1016/j.isci.2022.104196 |
(责任编辑 崔建勋)
 2024, Vol. 51
2024, Vol. 51




