文章信息
基金项目
- 广东省自然科学基金(2021A1515110411);广东省农业科学院新兴团队项目(202131TD);广州市重点研发计划项目(2024B03J1363);广东省农业科学院农业生物基因研究中心创新基金(202203)
作者简介
-
薛皦,博士,助理研究员,广东省农业科学院农业生物基因研究中心作物基因资源发掘与利用团队骨干成员,主要从事水稻抗病机理研究与基因编辑技术研发。近3年主持国家自然科学基金、广东省自然科学基金、中国博士后科学基金、广州市重点研发计划等项目6项;以第一或共同第一作者在《Nature Plants》《Journal of Integrative Plant Biology》《Science China Life Sciences》《Genes》《Analytical Biochemistry》《Scientific Reports》等期刊发表SCI论文6篇;以第一发明人获授权国家发明专利1件;获广东省农业科学院科学技术奖一等奖1项
薛皦(1986—),女,博士,助理研究员,研究方向为作物遗传资源发掘与利用,E-mail: xuejiao@gdaas.cn.
通讯作者
- 于洋(1987—),男,博士,副研究员,研究方向为作物遗传资源发掘与利用,E-mail: yuyang@gdaas.cn.
文章历史
- 收稿日期:2023-11-10
作物种质创新与分子育种是生命科学和农学研究的重要内容,也是国家战略性、基础性核心任务之一。近年来,作物育种面临着遗传基础狭窄、种质资源同质化等日趋严峻的问题,这不仅限制了优质、高产品种的高效培育,而且对作物病虫害抗性、环境适应性和农业发展的可持续性带来极大挑战[1]。农作物性状的遗传稳定性受到基因型和环境压力的共同影响。维持遗传多样性是农作物在不同环境压力下适应和生存的基础,因此在染色体水平解析遗传变异的形成与调控机制对作物的遗传改良实践至关重要。
染色体重排是指基因组中染色体的结构重新组合,导致基因座在染色体上的位置发生变化。得益于近年来基因组学、细胞生物学和分子遗传学的迅速发展,人们可以更深入地了解作物种质资源中自然发生的染色体变异及其生物学功能。不仅如此,植物染色体重排技术作为一种重要的遗传工程手段也备受关注,利用该技术可以人为主动、高效精准地控制植物细胞中的染色体结构,加速作物的演化进程,为优良品种的选育提供多样化的遗传资源和可供利用的物质基础。染色体重排技术的应用不仅有望提高作物的产量和品质,还可能改善作物的抗病性和环境适应性,从而为全球粮食安全和农业可持续发展作出重要贡献。
染色体重排研究在动植物中已得到广泛报道,这一领域的深入研究不仅丰富了我们对生物遗传学的认识,同时也为作物遗传改良和育种领域带来新的机遇。然而,针对植物染色体重排在不同作物遗传改良中的具体应用,进行系统分类和总结将有助于更全面地了解这一技术的现有研究进展和发展方向。本文将深入探讨植物染色体重排的基本概念、技术方法及其在作物遗传育种中的应用,同时展望该技术的应用前景,为研究人员提供系统的理论支持和实践参考。通过对植物染色体重排技术的深入了解,研究人员和育种家可以更好地认识和利用植物的基因组,为解决全球粮食安全和环境可持续发展等重大挑战提供创新解决方案。
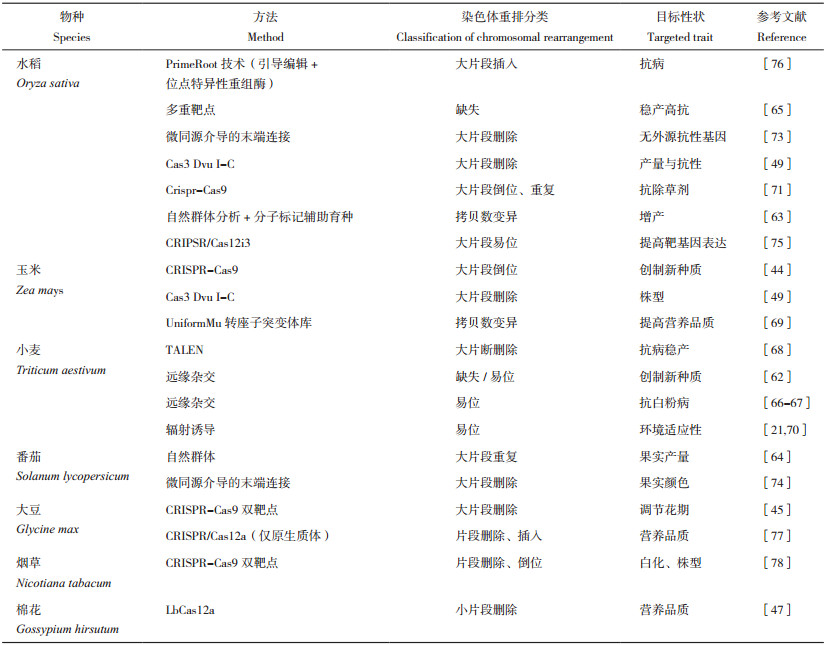
1 植物染色体重排的基本概念染色体重排是一种重要的基因组变异形式,通常发生于染色体断裂或损伤的情况下。在重新连接过程中,染色体片段可能会以异常的方式连接起来,导致染色体结构变异和重新组合。染色体重排包括插入(Insertion)、缺失(Deletion)、重复(Duplication)、拷贝数变异(Copy number variations,CNVs)、倒位(Inversion)和易位(Translocation)(图 1),每种形式都可能对植物基因组产生不同程度的影响[2-4]。其中,插入是指在染色体片段上加入新的DNA片段,可为单个或多个核苷酸,甚至是一大段核苷酸序列;缺失是指染色体上的某些DNA片段丢失或缺失,可能包括一个或多个基因,将导致染色体上的基因组结构发生变化;重复是指染色体上产生一个或多个与自身DNA片段相同区段(可以小到几个碱基,也可以大到一个主要染色体区域)的一种变异,所有生物体、尤其是在植物基因组中会出现大量重复的现象;拷贝数变异是指特定DNA片段的拷贝数发生变化,这些结构差异可能是通过重复、缺失或其他变化产生的,并可能影响DNA链的长度,导致个体间基因拷贝数差异;倒位是指当一个片段在一条染色体内断裂并以相反的方向重新连接时,染色体发生倒位,DNA可能会在这个过程中丢失,也可能不会丢失,但可导致基因的排列顺序发生改变;易位是指当一条染色体断裂,染色体碎片重新附着在其他不同的染色体上时,发生遗传学易位,易位可发生在染色体之间或染色体内部,导致染色体上的特定基因位置发生改变[5-8]。
|
| 图 1 染色体重排方式 Fig. 1 Chromosomal rearrangement modes |
2 植物染色体重排的发生机制和技术手段 2.1 染色体重排的发生机制
在植物中,自发性的染色体重排主要与物种形成、基因组演化及杂合体的遗传隔离有关,可能导致杂交不育、着丝点变化、新的开放阅读框形成、基因破坏、染色体片段移除、表达谱改变等[9-12]。植物自发性染色体重排通常被认为是由遗传漂变、环境压力或者其他未知因素引起的。通过对亚洲栽培稻、非洲栽培稻等不同水稻品种进行测序和构建水稻泛基因组的研究表明,水稻种群中存在大量结构变异和基因拷贝数变异,这些变异对调控水稻农艺性状、推动水稻进化与驯化有着至关重要的影响[13]。
染色体内或染色体间的变异可自然发生,并影响物种的形成和适应性演化等过程。染色体重排可由不同的DNA修复机制介导,通常DNA双链断裂(Double-strand breaks,DSB)可以通过两种主要途径修复:非同源末端连接(Non-homologous end joining,NHEJ)和同源重组(Homologous recombination,HR),HR依赖于利用同源模板,而NHEJ则是将断裂的DNA末端重新连接。如果DSB在修复时存在足够多的同源性区域,则可能导致非等位基因同源重组(Nonallelic homologous recombination,NAHR),这种机制能够在基因组中引起大量的重排事件,形成重复的染色体变异[14]。例如,基因组重复区域发生DSB可能导致这些重复区域频繁发生重排事件,增强了基因组的不稳定性,增加了新的遗传变异产生几率。
2.2 创制染色体重排的技术手段作物育种依赖减数分裂过程中同源染色体间的互换以产生新的等位基因组合。杂交可组合有利性状、消除不利性状[15]。然而,由于自然进程中交叉互换的频率和分布受到严格限制且难以控制,染色体上的很多部分不参与遗传交换。因此,理想的重组结果非常有限,连锁阻力往往不可避免[16]。通过现代生物技术诱导植物染色体重排不仅可以加快育种进程,还可以为基因组研究和生物技术应用提供重要支持,有助于促进作物遗传育种和生物技术领域的发展和进步。为更高效地操控染色体并创造染色体重排,近年来发展了多种诱导植物染色体重排的技术方法。
2.2.1 传统的杂交方法 传统的杂交方法是通过人为选择和交配具有不同染色体结构和基因型的植物,利用杂交后代的染色体重新组合引入新的遗传变异。这种方法依赖于杂交过程中染色体的交换和重组,使得杂交后代产生新的染色体组合和遗传变异。常见的方法如染色体片段代换系的构建及应用[17-19]。
2.2.2 物理化学诱变法 物理化学诱变是通过利用物理因素(如辐射)或化学诱变剂(如甲基磺酸乙酯、EMS)来诱导植物基因组突变。这些突变包括染色体重排(如片段缺失、倒位、易位等),以及基因突变(如碱基替换、插入、缺失等)。电离辐射可导致染色体直接断裂,也可产生DNA损伤,并在之后的复制、修复过程中引发变异。利用EMS可诱导小麦-华山新麦草染色体易位[20]。用花粉辐射法可大量生产硬粒小麦- 簇毛麦二倍体的属间染色体易位[21],为小麦育种创造了新的种质资源。
2.2.3 转座子诱导法 通过诱导转座子可以促进染色体片段的移动和重排,产生新的遗传变异和基因组结构变化。在物种演化进程中,转座子的扩增和重复在玉米[22-23]、棉花[24]、辣椒[25]、拟南芥[26]、水稻[27]等多个物种的驯化和适应性演化进程中发挥了重要作用。此外,Ac/Ds转座子系统还与序列特异重组系统Cre/Lox联合使用,进行染色体结构的靶向诱变,目前已在烟草、拟南芥等植物中诱导了染色体的缺失、倒位和易位等突变[28-31]。
2.2.4 基因编辑技术诱导法 基因编辑技术引领了染色体重排的新时代,利用这些技术可以精准地修改染色体上的基因或序列,诱导染色体重排。这一领域的演进经历了从设计蛋白质结构与目标DNA序列结合、引发DNA双链切割诱导细胞修复进行编辑的阶段,到以CRISPR系统为标志,利用RNA引导蛋白与目标DNA序列结合,通过RNA-DNA互补配对实现基因组定点编辑的发展历程。
(1)早期的核酸酶编辑技术:锌指核酸酶(Zinc finger nucleases,ZFN)和转录激活因子样效应核酸酶(Transcription activator-like effector nuclease,TALEN)是早期发展的核酸酶修饰、改造基因组的工具酶。利用ZFN技术,在拟南芥中可实现串联排列基因的缺失和倒位[32],还在烟草中成功删除了4.3 kb的大片段[33]。在水稻愈伤组织中使用2对TELENs产生了相同类型的染色体重排,但是未能遗传到下一代植株[34]。此外,归巢核酸内切酶I-SceI也被应用于植物染色体重排的诱导。在拟南芥两个不同的染色体上诱导2个DSB,可以交换2个染色体臂,导致易位[35]。
(2)CRISPR-Cas系统:CRISPR-Cas系统是最新的基因编辑技术,可以更高效、精准地改变染色体上特定基因或序列的排列顺序或结构[36]。近年来,不同类型的CRISPR系统被广泛地应用于植物染色体重排操作中。HEI10是水稻重组关键基因,利用CRISPR-Cas9技术对HEI10的启动子区或5' UTR区进行编辑,突变株遗传重组频率相应发生改变[37]。基于CRISPR-Cas系统对单个或多个基因进行基因编辑已经成为常规操作,但在植物中同时引入多个DSB位点以实现染色体重排的研究尚处于起步阶段[38-40]。真核生物基因组中广泛的、大量的存在染色体上串联排列的同源基因,称为Tandemly arrayed genes(TAG)。在成对的gRNA引导下,CRISPR介导的TAG敲除会引发高频的染色体缺失/ 倒位双等位变异[41],为多位点染色体重排提供了潜在工具。通过设计组织特异启动子驱动Cas9表达、双sgRNA设计等方法,研究人员在拟南芥、玉米、大豆等物种中成功实现了染色体大片段的倒位、删除等变异[42-45]。除了基于Ⅱ型CRISPR的Cas9系统,其他类型CRISPR也被开发利用。其中CRISPR-Cas12a/CRISPR-Cas12b系统具有分子量更小、脱靶率低等优势,而CRISPR-Cas3系统在不同作物中表现出更高的大片段敲除效率。Li等[46-47]利用CRISPR/Cpf1(Cas12a)系统在棉花中实现了靶向删除。Wang等[48]利用Cas12b(C2c1)系统在陆地棉中成功地靶向删除。Li等[49]利用I型CRISPR的Cas3 Dvu I-C系统进行双靶点设计,可在玉米和水稻中有效地删除大片段。此外,大规模、全局性、基因组水平的染色体重排已在哺乳动物细胞中得以实现,Liu等[50]使用CRISPR-Cas9靶向人类基因组多拷贝的逆转座子LINE-1/Alu,诱发了大规模的基因组DSB和染色体重排事件,然而此类技术在植物中尚未见报道,未来在植物中进行大规模染色体重排操作仍有广阔的挖掘空间。
上述方法在植物育种和基因组研究中扮演着重要角色。利用这些技术,研究人员可以根据特定需求改变染色体的序列排列,为改变植物基因组的结构和功能提供多样化的工具和途径。
3 植物染色体重排的鉴定方法 3.1 细胞生物学检测方法染色体核型分析是最传统的细胞遗传学检测方法之一。该方法主要是选取细胞分裂中期的染色体,经过染色和显带处理后,通过显微镜观察染色体核型的形态变化、数量变化及任何可见的重排现象,如倒位、插入、缺失等。但由于是细胞水平的检测,无法以更高的分辨率检测基因组碱基层面的序列变异[51]。荧光原位杂交技术(Fluorescence in situ hybridization,FISH)也常被用于植物染色体重排的可视化鉴定,其原理是将荧光素(如生物素、地高辛等)直接或间接标记的探针与待测样本中的核酸序列按照碱基互补配对的原则进行杂交,经洗涤后直接在荧光显微镜下观察,可用于检测染色体上的缺失、重排或插入等事件[52-54]。
3.2 分子遗传学检测方法分子标记是指可反映生物个体或物种种群间基因组差异的特征DNA片段,包括SNP(Single nucleotide polymorphism)标记分析、SSR(Simple sequence repeat)标记分析、AFLP(Amplified fragment length polymorphism)标记分析等。具有数量多、多态性高、信息量大、取材不受限、基因组分布均匀、检测成本低等特点。然而,分子标记技术通常需要对目标序列进行特定的扩增和检测,因此需要预先设计特定位点,对于未知的染色体重排事件或不稳定的遗传变异可能无法提供有效的检测[55]。
3.3 高通量测序及生物信息学分析法基于二代、三代测序技术对个体或群体进行基因组重测序或基因组从头组装,可以获得不同作物物种的遗传资源多样性,并构建该物种的“泛基因组” [56-57]。通过比较基因组学分析方法对不同物种、不同个体间的基因组序列进行比较,可以帮助研究人员揭示染色体重排事件在物种进化和遗传变异中的作用和意义。Qin等[13]挑选了33个具有高度代表性的水稻样本,利用第三代基因测序技术,对其中的31个样本进行了长片段测序、高质量基因组组装和基因注释,通过与已报道的‘日本晴’和‘蜀恢498’的参考基因组系统比较分析,共发现171 072个染色体结构性变异和22 549个基因拷贝数变异。Zhang等[58]通过三代测序引入长读长数据,创新构建了水稻泛基因组,与二代测序短读长数据相比,包含了604 Mb的新序列,明显增加了基因组的完整性和信息量。Shang等[59]基于三代长读长数据从头组装了251份高质量水稻基因组序列,并将鉴定到的159 491个染色体结构变异信息整合为图形化的变异图谱。上述研究为深入理解作物基因组的结构和变异提供了丰富的信息资源。
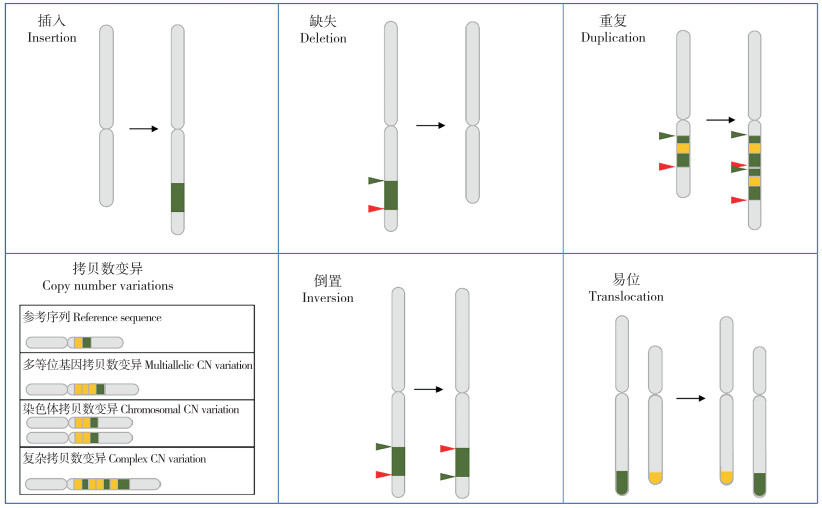
4 植物染色体重排在作物遗传育种中的应用 4.1 提高农作物的遗传多样性近年来,全球农业面临着人口增长、气候变化、土地资源减少及有害生物增加等挑战,作物育种是应对这些挑战的重要途径之一。在人类长期的驯化和选育中,栽培水稻的遗传基因出现明显的同质化,导致丰富性和多样性减少(图 2A)。尽管已育成的品种在产量和品质方面表现较佳,但却引发遗传基础狭窄化的问题,为全球农业可持续发展带来极大的风险和不确定性。作物的“从头驯化”是近年来发展出的一种作物育种新策略,在应对未来农业挑战方面具有重大潜力[60-61](图 2B)。与“从头驯化”相比,利用现代生命科学技术主动创制染色体变异具有截然不同的切入点和目标。这种方式是基于预定的设计和目标,有针对性地引发染色体重排、删除或插入特定基因的现象,以实现所需的性状变异(图 2C)。在作物遗传育种中,植物染色体重排的应用还涉及到对染色体结构的精准设计。通过基因编辑技术,尤其是CRISPR-Cas9系统,育种者可以有针对性地调整染色体的特定区域,实现对性状的定向改良。这种精准设计的优势在于不仅可以避免不需要的基因改变,还能够更有效地实现目标性状的引入或改变,为作物育种提供更高的灵活性和精确性。例如,利用CRISPRCas9技术对水稻重组关键基因HEI10的转录调控区域进行编辑,实现对遗传重组的正向和负向调控,为加速新品种的培育提供重要理论和技术支撑[37]。借助远缘杂交的方法引进外源染色体,也可导致小麦自身染色体的缺失和易位。例如,小麦和黑麦之间发生1BL/1RS易位,为小麦品种选育创制了新材料[62]。
|
| A:常规育种依赖自然选择与人工选择驯化野生物种;B:从头驯化对某些驯化相关基因进行基因编辑;C:系统性染色体重排靶向基因组范围内的重复区域,引发大规模的染色体变异;不同颜色的圆圈示意不同的遗传变异类型 A: Conventional breeding relies on natural selection and artificial selection to domesticate wild species; B: De novo domestication involves gene editing of certain domestication-related genes; C: Systematic chromosomal rearrangement targets repeat regions within the genome, inducing large-scale chromosomal variation; Different colored circles represent different types of genetic mutations 图 2 提高作物遗传多样性的策略比较 Fig. 2 Comparison of strategies to enhance genetic diversity in crops |
4.2 改良农作物的重要性状和环境适应性
4.2.1 改良作物产量性状 作物产量性状在我国农业领域至关重要,直接影响着粮食供给和农业经济发展。基因多效性普遍存在,完全敲除某个基因可能同时产生其他性状,相比之下,染色体重排技术,如拷贝数变异和重复序列等,通过调控基因表达的剂量,更加精准且灵活地调节基因表达水平,从而产生更有利的性状。水稻穗发育基因FZP具有阻止腋芽分生组织形成并建立花分生组织的作用,与水稻产量密切相关,该基因上游存在18 bp的转录沉默子,通过自然群体分析结合分子标记辅助育种技术,筛选到转录沉默子拷贝数变异的品种,可使水稻增产15%[63]。Alonge等[64]利用高通量的PromethION平台对100个不同的番茄基因组进行了测序,每个基因组鉴定出25 000 ~ 45 000个结构变异(Structure variations,SVs),其中包括1个83 kb的染色体串联重复,可提高果实产量(表 1)。
4.2.2 改良作物抗性性状 作为世界一半以上人口的主粮,水稻受稻瘟病、稻曲病、白叶枯病等危害,严重影响其产量和品质,然而高抗的水稻品种往往产量受损。rbl1基因可提高水稻对稻瘟病和白叶枯病的抗性,通过设计靶向rbl1的多重靶点,筛选到12 bp缺失、但稳产高抗的水稻品种[65]。在麦类作物中,小麦和簇毛麦的6VS/6AL发生易位,为小麦增添了抗白粉病的新基因[66-67]。Li等[68]利用TALEN系统靶向突变MLO基因,从后代中筛选到Tamlo-R32突变体,该突变体除了mlo突变之外,还缺失了MLO-B1邻近304 kb的染色体片段,创制了既抗白粉病又高产的小麦种质(表 1)。
4.2.3 改良作物营养品质 通过调控与作物营养品质相关的基因表达、失活有害代谢产物基因等方式,有望实现对作物营养品质的精准调控和改良。玉米27-kDa γ- 醇溶蛋白编码基因γ27在不同玉米自交系间存在拷贝数变异,含2个γ27拷贝的等位位点(Standard allele,S)可通过基因重排变成一个拷贝位点(Rearranged allele,R),其上游调控基因O2突变后(o2)醇溶蛋白含量大幅度下降,富含赖氨酸的其他蛋白互补性上调,在营养品质方面得以提高,然而o2是粉质表型,无法直接应用于产业。通过创制的UniformMu转座子突变体库筛选到携带三拷贝的γ27材料,通过靶向醇溶蛋白基因的RNAi转基因中导入该材料后,后代品系富含赖氨酸、胚乳硬质,提高了该品系在产业应用的前景[69]。棉花种子富含油脂和蛋白质,但由于腺体中的代谢物棉酚具有毒性而不能食用。Li等[47]利用棉花耐高温,结合CRISPR/LbCpf1(LbCas12a)介导的基因组编辑系统,创制了纯合、无棉酚的棉花材料,为棉花分子育种提供了新的种质资源(表 1)。
4.2.4 改良作物环境适应性 除了改良产量、抗病等性状,植物染色体重排还能够促进植物对环境的适应性和提高生态系统的稳定性。例如微生态选择压驱动适应性进化和生殖隔离的形成,同域情况下野生二粒小麦群体在受到强辐射时会采用不同的适应性策略,从而分离出SFS1和SFS2两个群体。SFS2群体通过早花规避生育后期高温、强辐射、干旱造成的非生物胁迫,而SFS1群体则在强辐射诱导下发生DNA断裂及染色体重新融合,产生晚花、耐强辐射性状,最终导致亚群间的生殖隔离[70](表 1)。
4.2.5 染色体重排技术的其他应用 在新型抗除草剂水稻研发方面,提高水稻中PPO1(原卟啉原IX氧化酶,Protoporphyrinogen IX oxidase)和HPPD(羟基苯基丙酮酸双加氧酶,Hydroxyphenyl pyruvate dioxygenase)的表达量能够提高水稻对相应除草剂的抗性,通过CRISPRCas9系统实现片段倒位和片段重复,分别对PPO1和HPPD基因表达实现了敲高调控,使水稻植株表现出抗除草剂性状[71]。微同源介导的末端连接(Microhomology-mediated end joining,MMEJ)是另一种DNA修复机制,需要基于DSB附近4~25 bp微同源序列(MHS)之间的重组,利用这种机制可有效促进基因组的大片段删除[72]。Tan等[73]利用微同源介导的末端连接体系实现了转基因水稻植株中抗性基因(潮霉素抗性基因)HPT的大片段删除。通过在微同源位点附近设计双sgRNAs,Zhu等[74]也在番茄中实现了大片段删除。LYU等[75]发现基于CRIPSR/Cas12i3的iMAGE系统在水稻中产生的基因组大片段易位效率显著高于CRISPR/Cas9。该技术有望在靶基因上游有强自动子时,被用于大片段删除,以增强靶基因表达的效果。Sun等[76]研究开发了PrimeRoot(Prime editing-mediated Recombination of opportune targets)技术,通过系统整合优化的引导编辑工具和位点特异性重组酶系统,实现水稻中长达11.1 kb大片段DNA的高效精准定点插入,为研究更加复杂的性状和植物合成生物学的应用提供工具(表 1)。
5 总结与展望植物染色体重排技术作为一种强大的工具,在植物遗传育种和基因组研究领域具有广阔的应用前景。通过操纵植物基因组的结构,可以引入新的遗传变异,从而改善作物的产量、抗性和适应性。植物染色体重排技术还有助于解析植物基因组的结构与功能,为基因定位和功能鉴定提供重要手段。此外,植物染色体重排技术可以促进作物的适应性进化、打破连锁效应,从而创造新的基因组合,提高作物遗传的多样性和复杂性。这将有助于培育强适应性、抗性和高产性的新品种,从而提升作物的产量和质量,为粮食安全和农业可持续发展作出贡献。
植物染色体重排技术在推动作物遗传改良和基因组研究方面取得了显著进展,然而,其应用也伴随着一系列风险,需要进行细致的评估,具体包括:(1)染色体重排技术的广泛应用可能引起作物表型的非正常化。尽管该技术可以引入有益的遗传变异,但随之而来的未知基因组变异可能导致某些不良表型,进而损害或抑制植物的正常生长和发育。因此,在实际应用中,必须充分考虑和评估由染色体重排引发不良表型的可能性。(2)生物安全性评估是植物染色体重排技术应用的一个重要环节。在推动作物遗传改良的同时,我们必须确保该技术不会对生态系统和人类健康造成潜在威胁。因此,未来的研究需要加强生物安全性评估,全面了解染色体重排技术对环境和生态系统的长期影响。(3)传统的杂交育种和理化方法创制染色体重排的概率相对较低。虽然这些方法在基因组改良方面取得了成功,但其精确性和效率相对较低。与之相比,现代生命科学技术,如基因编辑,提供了更为主动、高效的手段来创制染色体重排。然而,基因编辑创制染色体重排的具体机制和规律尚不清楚,需要进一步深入研究。(4)未来需要解决植物染色体重排技术在精确控制过程中的挑战。尽管现代技术使得全基因组范围内的系统性染色体重排成为可能,但仍面临着精准定位和重排染色体片段的技术复杂性。为了克服这一挑战,需要开发更为精准的工具和策略,确保染色体重排的精准性和可控性。
通过持续改进技术手段、加强监管和推动国际合作,我们有望解锁植物染色体重排技术更广泛的潜力,为全球农业的可持续发展提供更多创新的可能性。这将为培育更具适应性、抗性和高产性的新品种,解决全球粮食安全和环境可持续发展等重大挑战提供实质性的方案。
| [1] |
CHEN R, DENG Y, DING Y, GUO J, QIU J, WANG B, WANG C, XIE Y, ZHANG Z, CHEN J, CHEN L, CHU C, HE G, HE Z, HUANG X, XING Y, YANG S, XIE D, LIU Y, LI J. Rice functional genomics: Decades' efforts and roads ahead[J]. Science China Life Sciences, 2022, 65(1): 33-92. DOI:10.1007/s11427-021-2024-0 |
| [2] |
NAVARRO A, BARTON N H. Chromosomal speciation and molecular divergence—accelerated evolution in rearranged chromosomes[J]. Science, 2003, 300(5617): 321-324. DOI:10.1126/science.1080600 |
| [3] |
RIESEBERG L H. Chromosomal rearrangements and speciation[J]. Trends in Ecology & Evolution, 2001, 16(7): 351-358. DOI:10.1016/s0169-5347(01)02187-5 |
| [4] |
STEWART N B, ROGERS R L. Chromosomal rearrangements as a source of new gene formation in Drosophila yakuba[J]. PLoS Genetics, 2019, 15(9): e1008314. DOI:10.1371/journal.pgen.1008314 |
| [5] |
YANG N, WU S, YAN J. Structural variation in complex genome: Detection, integration and function[J]. Science China Life Sciences, 2019, 62(8): 1098-1100. DOI:10.1007/s11427-019-9664-4 |
| [6] |
LYE Z N, PURUGGANAN M D. Copy number variation in domestication[J]. Trends in Plant Science, 2019, 24(4): 352-365. DOI:10.1016/j.tplants.2019.01.003 |
| [7] |
ZHANG X, CHEN X, LIANG P, TANG H. Cataloging plant genome structural variations[J]. Current Issues in Molecular Biology, 2018, 27: 181-194. DOI:10.21775/cimb.027.181 |
| [8] |
GABUR I, CHAWLA H S, SNOWDON R J, PARKIN IA P. Connecting genome structural variation with complex traits in crop plants[J]. Theoretical and Applied Genetics, 2019, 132(3): 733-750. DOI:10.1007/s00122-018-3233-0 |
| [9] |
BLANC G, BARAKAT A, GUYOT R, COOKE R, DELSENY M. Extensive duplication and reshuffling in the Arabidopsis genome[J]. The Plant Cell, 2000, 12(7): 1093-1101. DOI:10.1105/tpc.12.7.1093 |
| [10] |
LIVINGSTONE K, RIESEBERG L. Chromosomal evolution and speciation: A recombination-based approach[J]. New Phytologist, 2004, 161(1): 107-112. DOI:10.1046/j.1469-8137.2003.00942.x |
| [11] |
FANG Z, PYHÄJÄRVI T, WEBER A L, DAWE R K., GLAUBITZ J C, GONZÁLEZ J D J S, ROSS-IBARRA C, DOEBLEY J, MORRELL P L, ROSS-IBARRA J. Megabase-scale inversion polymorphism in the wild ancestor of maize[J]. Genetics, 2012, 191(3): 883-894. DOI:10.1534/genetics.112.138578 |
| [12] |
SCHUBERT I, VU G T H. Genome stability and evolution: Attempting a holistic view[J]. Trends in Plant Science, 2016, 21(9): 749-757. DOI:10.1016/j.tplants.2016.06.003 |
| [13] |
QIN P, LU H, DU H, WANG H, CHEN W, CHEN Z, HE Q, OU S, ZHANG H, LI X, LI X, LI Y, LIAO Y, GAO Q, TU B, YUAN H, MA B, WANG Y, QIAN Y, FAN S, LI W, WANG J, HE M, YIN J, LI T, JIANG N, CHEN X, LIANG C, LI S. Pan-genome analysis of 33 genetically diverse rice accessions reveals hidden genomic variations[J]. Cell, 2021, 184(13): 3542-3558. DOI:10.1016/j.cell.2021.04.046 |
| [14] |
PUCHTA H. The repair of double-strand breaks in plants: Mechanisms and consequences for genome evolution[J]. Journal of Experimental Botany, 2005, 56(409): 1-14. DOI:10.1093/jxb/eri025 |
| [15] |
TAAGEN E, BOGDANOVE A J, SORRELLS M E. Counting on crossovers: Controlled recombination for plant breeding[J]. Trends in Plant Science, 2020, 25(5): 455-465. DOI:10.1016/j.tplants.2019.12.017 |
| [16] |
WANG Y, COPENHAVER G P. Meiotic recombination: Mixing it up in plants[J]. Annual Review of Plant Biology, 2018, 69(1): 577-609. DOI:10.1146/annurev-arplant-042817-040431 |
| [17] |
李进. 水稻穗长QTL qPL-3的精细定位[D]. 扬州: 扬州大学, 2020. DOI: 10.27441/d.cnki.gyzdu.2020.000108. LI J. Fine-mapping of QTL qPL-3 for rice panicle length[D]. Yangzhou: Yangzhou University, 2020. DOI: 10.27441/d.cnki.gyzdu.2020.000108. |
| [18] |
杨旭东, 赵芳明, 罗洪发, 赵福胜, 潘清洁, 张治海, 查仁明. 基于染色体片段代换系的水稻穗部性状QTL定位[J]. 山地农业生物学报, 2020, 39(5): 78-81. DOI:10.15958/j.cnki.sdnyswxb.2020.05.011 YANG X D, ZHAO F M, LUO H F, ZHAO F S, PAN Q J, ZHANG Z H, ZHA R M. QTL mapping of panicle traits based on rice chromosome substitution lines[J]. Journal of Mountain Agriculture and Biology, 2020, 39(5): 78-81. DOI:10.15958/j.cnki.sdnyswxb.2020.05.011 |
| [19] |
张治海. 水稻染色体片段代换系Z745的鉴定及产量相关性状QTL定位[D]. 贵阳: 贵州大学, 2020. DOI: 10.27047/d.cnki.ggudu.2020.000680. ZHANG Z H. Identif ication of rice chromosome seg ment substitution line Z745 and QTL mapping of yield-related traits[D]. Guiyang: Guizhou University, 2020. DOI: 10.27047/d.cnki.ggudu.2020.000680. |
| [20] |
敬樊, 王亮明, 武军, 杜万里, 王秀娟, 苏佳妮, 昝凯, 赵继新, 陈新宏. 利用甲基磺酸乙酯(EMS) 诱导小麦- 华山新麦草染色体易位的研究[J]. 农业生物技术学报, 2015, 23(5): 561-570. JING F, WANG L M, WU J, DU W L, WANG X J, SU J N, ZAN K, ZHAO J X, CHEN X H. Study on inducing chromosome translocation of wheat (Triticum aestivum)-Psathyrostachys huashanica using ethylmethylsulfone (EMS)[J]. Journal of Agricultural Biotechnology, 2015, 23(5): 561-570. |
| [21] |
BIE T D, CAO Y P, CHEN P D. Mass production of intergeneric chromosomal translocations through pollen irradiation of Triticum durum-Haynaldia villosa amphiploid[J]. Journal of Integrative Plant Biology, 2007, 49(11): 1619-162. DOI:10.1111/j.1774-7909.2007.00578.x |
| [22] |
SHI J, TIAN Z, LAI J, HUANG X. Plant pan-genomics and its applications[J]. Molecular Plant, 2023, 16(1): 168-186. DOI:10.1016/j.molp.2022.12.009 |
| [23] |
ZHANG J, PETERSON T. Transposition of reversed Ac element ends generates chromosome rearrangements in maize[J]. Genetics, 2004, 167(4): 1929-1937. DOI:10.1534/genetics.103.026229 |
| [24] |
WANG M, LI J, QI Z, LONG Y, PEI L, HUANG X, GROVER C E, DU X, XIA C, WANG P, LIU Z, YOU J, TIAN X, MA Y, WANG R, CHEN X, HE X, FANG D D, SUN Y, TU L, JIN S, ZHU L, WENDEL J F, ZHANG X. Genomic innovation and regulatory rewiring during evolution of the cotton genus Gossypium[J]. Nature Genetics, 2022, 54(12): 1959-1971. DOI:10.1038/s41588-022-01237-2 |
| [25] |
KIM S, PARK J, YEOM S I, KIM Y M, SEO E, KIM K T, KIM M S, LEE J M, CHEONG K, SHIN H S, KIM S B, HAN K, LEE J, PARK M, LEE H A, LEE H Y, LEE Y, OH S, LEE J H, CHOI E, CHOI E, LEE S E, JEON J, KIM H, CHOI G, SONG H, LEE J, LEE S C, KWON J K, LEE H Y, KOO N, HONG Y, KIM R W, KANG W H, HUH J H, KANG B C, YANG T J, LEE Y H, BENNETZEN J L, CHOI D. New reference genome sequences of hot pepper reveal the massive evolution of plant disease-resistance genes by retroduplication[J]. Genome Biology, 2017, 18(1): 210. DOI:10.1186/s13059-017-1341-9 |
| [26] |
KRISHNASWAMY L, ZHANG J, PETERSON T. Reversed end Ds element: A novel tool for chromosome engineering in Arabidopsis[J]. Plant Molecular Biology, 2008, 68(4): 399-411. DOI:10.1007/s11103-008-9377-6 |
| [27] |
YU C, HAN F, ZHANG J, BIRCHLER J, PETERSON T. A transgenic system for generation of transposon Ac/Ds-induced chromosome rearrangements in rice[J]. Theoretical and Applied Genetics, 2012, 125(7): 1449-1462. DOI:10.1007/s00122-012-1925-4 |
| [28] |
QIN M, BAYLEY C, STOCKTON T, OW D W. Cre recombinasemediated site-specific recombination between plant chromosomes[J]. Proceedings of the National Academy of Sciences of the United States of America, 1994, 91(5): 1706-1710. DOI:10.1073/pnas.91.5.1706 |
| [29] |
MEDBERRY S L, DALE E, QIN M, OW D W. Intra-chromosomal rearrangements generated by Cre-lox site-specific recombination[J]. Nucleic Acids Research, 1995, 23(3): 485-490. DOI:10.1093/nar/23.3.485 |
| [30] |
OSBORNE B I, WIRTZ U, BAKER B. A system for insertional mutagenesis and chromosomal rearrangement using the Ds transposon and Cre-lox[J]. The Plant Journal, 1995, 7(4): 687-701. DOI:10.1046/j.1365-313X.1995.7040687.x |
| [31] |
ZHANG S, RAINA S, LI H, LI J, DEC E, MA H, HUANG H, FEDOROFF N V. Resources for targeted insertional and deletional mutagenesis in Arabidopsis[J]. Plant Molecular Biology, 2003, 53(1): 133-150. DOI:10.1023/B:PLAN.0000009271.08420.d9 |
| [32] |
QI Y, LI X, ZHANG Y, STARKER C G, BALTES N J, ZHANG F, SANDER J D, REYON D, JOUNG J K, VOYTAS D F. Targeted deletion and inversion of tandemly arrayed genes in Arabidopsis thaliana using zinc finger nucleases[J]. G3 (Bethesda), 2013, 3(10): 1707-1715. DOI:10.1534/g3.113.006270 |
| [33] |
PETOLINO J F, WORDEN A, CURLEE K, CONNELL J, STRANGE MOYNAHAN T L, LARSEN C, RUSSELL S. Zinc finger nucleasemediated transgene deletion[J]. Plant Molecular Biology, 2010, 73(6): 617-628. DOI:10.1007/s11103-010-9641-4 |
| [34] |
SHAN Q, WANG Y, CHEN K, LIANG Z, LI J, ZHANG Y, ZHANG K, LIU J, VOYTAS D F, ZHENG X, ZHANG Y, GAO C. Rapid and efficient gene modification in rice and using TALENs[J]. Molecular Plant, 2013, 6(4): 1365-1368. DOI:10.1093/mp/sss162 |
| [35] |
PACHER M, SCHMIDT-PUCHTA W, PUCHTA H. Two unlinked double-strand breaks can induce reciprocal exchanges in plant genomes via homologous recombination and nonhomologous end joining[J]. Genetics, 2007, 175(1): 21-29. DOI:10.1534/genetics.106.065185 |
| [36] |
江桐欣, 李晓琳, 朱庆锋, 张琪, 张爱霞, 刘勤坚, 于洋, 刘文华. 基于同源重组的CRISPR精准基因编辑技术及其在农作物育种中的应用[J]. 广东农业科学, 2022, 49(11): 119-127. DOI:10.16768/j.issn.1004-874X.2022.11.013 JIANG T X, LI X L, ZHU Q F, ZHANG Q, ZHANG A X, LIU Q J, YU Y, LIU W H. Homologous recombination-based CRISPR precision gene editing technology and its application in crop breeding[J]. Guangdong Agricultural Sciences, 2022, 49(11): 119-127. DOI:10.16768/j.issn.1004-874X.2022.11.013 |
| [37] |
WEI X, LIU Q, SUN T, JIAO X, LIU C, HUA Y, CHEN X, WANG K. Manipulation of genetic recombination by editing the transcriptional regulatory regions of a meiotic gene in hybrid rice[J]. Plant Communications, 2023, 4(2): 100474. DOI:10.1016/j.xplc.2022.100474 |
| [38] |
SCHMIDT C, SCHINDELE P, PUCHTA H. From gene editing to genome engineering: Restructuring plant chromosomes via CRISPR/Cas[J]. aBIOTECH, 2020(1): 21-31. DOI:10.1007/s42994-019-00002-0 |
| [39] |
李文龙, 栾鑫, 张强, 余宁, 冯晓敏, 刘志霞. 基于CRISPR/Cas9基因编辑技术的水稻定向改良研究进展[J]. 广东农业科学, 2022, 49(9): 114-124. DOI:10.16768/j.issn.1004-874X.2022.09.012 LI W L, LUAN X, ZHANG Q, YU N, FENG X M, LIU Z X. Target improvement of rice based on CRISPR/Cas9 gene editing technology[J]. Guangdong Agricultural Sciences, 2022, 49(9): 114-124. DOI:10.16768/j.issn.1004-874X.2022.09.012 |
| [40] |
任代胜, 刘浩, 乔保建. 基于CRISPR/Cas9基因编辑技术靶向敲除OsFAD2创制高油酸水稻突变体[J]. 广东农业科学, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 REN D S, LIU H, QIAO B J. Creation of high oleic acid rice mutant by targeted knockout of OsFAD2 via CRISPR/Cas9 genome editing technology[J]. Guangdong Agricultural Sciences, 2021, 48(11): 1-7. DOI:10.16768/j.issn.1004-874X.2021.11.001 |
| [41] |
LIU J, WANG F Z, LI C, LI Y, LI J F. Hidden prevalence of deletioninversion bi-alleles in CRISPR-mediated deletions of tandemly arrayed genes in plants[J]. Nature Communications, 2023, 14(1): 6787. DOI:10.1038/s41467-023-42490-1 |
| [42] |
SCHMIDT C, PACHER M, PUCHTA H. Efficient induction of heritable inversions in plant genomes using the CRISPR/Cas system[J]. The Plant Journal, 2019, 98(4): 577-589. DOI:10.1111/tpj.14322 |
| [43] |
RÖNSPIES M, SCHINDELE P, WETZEL R, PUCHTA H. CRISPR– Cas9-mediated chromosome engineering in Arabidopsis thaliana[J]. Nature Protocols, 2022, 17(5): 1332-1358. DOI:10.1038/s41596-022-00686-7 |
| [44] |
SCHWARTZ C, LENDERTS B, FEIGENBUTZ L, BARONE P, LLACA V, FENGLER K, SVITASHEV S. CRISPR–Cas9-mediated 75.5-Mb inversion in maize[J]. Nature Plants, 2020, 6(12): 1427-1431. DOI:10.1038/s41477-020-00817-6 |
| [45] |
CAI Y, CHEN L, SUN S, WU C, YAO W, JIANG B, HAN T, HOU W. CRISPR/Cas9-mediated deletion of large genomic fragments in soybean[J]. International Journal of Molecular Sciences, 2018, 19(12): 3835-3835. DOI:10.3390/ijms19123835 |
| [46] |
LI B, RUI H, LI Y, WANG Q, ALARIQI M, QIN L, SUN L, DING X, WANG F, ZOU J, WANG Y, YUAN D, ZHANG X, JIN S. Robust CRISPR/Cpf1 (Cas12a)-mediated genome editing in allotetraploid cotton (Gossypium hirsutum)[J]. Plant Biotechnology Journal, 2019, 17(10): 1862-1864. DOI:10.1111/pbi.13147 |
| [47] |
LI B, LIANG S, ALARIQI M, WANG F, WANG G, WANG Q, XU Z, YU L, NAEEM ZAFAR M, SUN L, SI H, YUAN D, GUO W, WANG Y, LINDSEY K, ZHANG X, JIN S. The application of temperature sensitivity CRISPR/LbCpf1 (LbCas12a) mediated genome editing in allotetraploid cotton (G. hirsutum) and creation of nontransgenic, gossypol-free cotton[J]. Plant Biotechnology Journal, 2021, 19(2): 221-223. DOI:10.1111/pbi.13470 |
| [48] |
WANG Q, ALARIQI M, WANG F, LI B, DING X, RUI H, LI Y, XU Z, QIN L, SUN L, LI J, ZOU J, LINDSEY K, ZHANG X, JIN S. The application of a heat-inducible CRISPR/Cas12b (C2c1) genome editing system in tetraploid cotton (G. hirsutum) plants[J]. Plant Biotechnology Journal, 2020, 18(12): 2436-2443. DOI:10.1111/pbi.13417 |
| [49] |
LI Y, HUANG B, CHEN J, HUANG L, XU J, WANG Y, CUI G, ZHAO H, XIN B, SONG W, ZHU J K, LAI J. Targeted large fragment deletion in plants using paired crRNAs with type I CRISPR system[J]. Plant Biotechnology Journal, 2023, 21(11): 2196-2208. DOI:10.1111/pbi.14122 |
| [50] |
LIU Y, MA G, GAO Z, LI J, WANG J, ZHU X, MA R, YANG J, ZHOU Y, HU K, ZHANG Y, GUO Y. Global chromosome rearrangement induced by CRISPR-Cas9 reshapes the genome and transcriptome of human cells[J]. Nucleic Acids Research, 2022, 50(6): 3456-3474. DOI:10.1093/nar/gkac153 |
| [51] |
JHA T B. Karyotype diversity in cultivated and wild Indian rice through EMA-based chromosome analysis[J]. Journal of Genetics, 2021, 100: 81. DOI:10.1007/s12041-021-01332-z |
| [52] |
周燕, 高华, 高自旸, 高述民, 胡青. 染色体原位杂交技术与植物显微技术实验探索[J]. 生物学通报, 2011, 46(2): 43-45. ZHOU Y, GAO H, GAO Z Y, GAO S M, HU Q. Experimental exploration of chromosome in situ hybridization and plant microscopy[J]. Bulletin of Biology, 2011, 46(2): 43-45. |
| [53] |
BRAZ G T, DO VALE MARTINS L, ZHANG T, ALBERT P S, BIRCHLER J A, JIANG J. A universal chromosome identification system for maize and wild Zea species[J]. Chromosome Research, 2020, 28(2): 183-194. DOI:10.1007/s10577-020-09630-5 |
| [54] |
DE OLIVEIRA BUSTAMANTE F, DO NASCIMENTO T H, MONTENEGRO C, DIAS S, DO VALE MARTINS L, BRAZ G T, BENKO-ISEPPON A M, JIANG J, PEDROSA-HARAND A, BRASILEIRO-VIDAL A C. Oligo-fish barcode in beans: A new chromosome identification system[J]. Theoretical and Applied Genetics, 2021, 134(11): 1-12. DOI:10.1007/s00122-021-03921-z |
| [55] |
佘朝文. 几种植物与模式植物基因组的分子细胞遗传学比较分析[D]. 武汉: 武汉大学, 2006. SHE C W. Molecular cytogenetic comparisons between model plant and several plant genomes[D]. Wuhan: Wuhan University, 2006. |
| [56] |
JAYAKODI M, PADMARASU S, HABERER G, BONTHALA V S, GUNDLACH H, MONAT C, LUX T, KAMAL N, LANG D, HIMMELBACH A, ENS J, ZHANG X Q, ANGESSA T T, ZHOU G, TAN C, HILL C, WANG P, SCHREIBER M, BOSTON L B, PLOTT C, JENKINS J, GUO Y, FIEBIG A, BUDAK H, XU D, ZHANG J, WANG C, GRIMWOOD J, SCHMUTZ J, GUO G, ZHANG G, MOCHIDA K, HIRAYAMA T, SATO K, CHALMERS K J, LANGRIDGE P, WAUGH R, POZNIAK C J, SCHOLZ U, MAYER K F X, SPANNAGL M, LI C, MASCHER M, STEIN N. The barley pan-genome reveals the hidden legacy of mutation breeding[J]. Nature, 2020, 588(7837): 284-289. DOI:10.1038/s41586-020-2947-8 |
| [57] |
HU H, SCHEBEN A, WANG J, LI F, LI C D, EDWARDS D, ZHAO J. Unravelling inversions: Technological advances, challenges, and potential impact on crop breeding[J]. Plant Biotechnology Journal, 2023. DOI:10.1111/pbi.14224 |
| [58] |
ZHANG F, XUE H, DONG X, LI M, ZHENG X, LI Z, XU J, WANG W, WEI C. Long-read sequencing of 111 rice genomes reveals significantly larger pan-genomes[J]. Genome Research, 2022, 32(5): 853-863. DOI:10.1101/gr.276015.121 |
| [59] |
SHANG L, LI X, HE H, YUAN Q, SONG Y, WEI Z, LIN H, HU M, ZHAO F, ZHANG C, LI Y, GAO H, WANG T, LIU X, ZHANG H, ZHANG Y, CAO S, YU X, ZHANG B, ZHANG Y, TAN Y, QIN M, AI C, YANG Y, ZHANG B, HU Z, WANG H, LYU Y, WANG Y, MA J, WANG Q, LU H, WU Z, LIU S, SUN Z, ZHANG H, GUO L, LI Z, ZHOU Y, LI J, ZHU Z, XIONG G, RUAN J, QIAN Q. A super pangenomic landscape of rice[J]. Cell Research, 2022, 32(10): 878-896. DOI:10.1038/s41422-022-00685-z |
| [60] |
YU H, LIN T, MENG X, DU H, ZHANG J, LIU G, CHEN M, JING Y, KOU L, LI X, GAO Q, LIANG Y, LIU X, FAN Z, LIANG Y, CHENG Z, CHEN M, TIAN Z, WANG Y, CHU C, ZUO J, WAN J, QIAN Q, HAN B, ZUCCOLO A, WING R A, GAO C, LIANG C, LI J. A route to de novo domestication of wild allotetraploid rice[J]. Cell, 2021, 184(5): 1156-1170. DOI:10.1016/j.cell.2021.01.013 |
| [61] |
YU H, LI J. Breeding future crops to feed the world through de novo domestication[J]. Nature Communications, 2022, 13(1): 1171. DOI:10.1038/s41467-022-28732-8 |
| [62] |
GRAYBOSCH R A, PETERSON C J, HANSEN L E, MATTERN P J. Relationships between protein solubility characteristics, 1BL/1RS, high molecular weight glutenin composition, and end-use quality in winter wheat germ plasm[J]. Cereal Chemistry, 1990, 67(4): 342-349. |
| [63] |
BAI X, HUANG Y, HU Y, LIU H, ZHANG B, SMACZNIAK C, HU G, HAN Z, XING Y. Duplication of an upstream silencer of FZP increases grain yield in rice[J]. Nature Plants, 2017, 3(11): 885-893. DOI:10.1038/s41477-017-0042-4 |
| [64] |
ALONGE M, WANG X, BENOIT M, SOYK S, PEREIRA L, ZHANG L, SURESH H, RAMAKRISHNAN S, MAUMUS F, CIREN D, LEVY Y, HAREL T H, SHALEV-SCHLOSSER G, AMSELLEM Z, RAZIFARD H, CAICEDO AL, TIEMAN D M, KLEE H, KIRSCHE M, AGANEZOV S, RANALLO-BENAVIDEZ T R, LEMMON Z H, KIM J, ROBITAILLE G, KRAMER M, GOODWIN S, MCCOMBIE W R, HUTTON S, VAN ECK J, GILLIS J, ESHED Y, SEDLAZECK F J, VAN DER KNAAP E, SCHATZ M C, LIPPMAN Z B. Major impacts of widespread structural variation on gene expression and crop improvement in tomato[J]. Cell, 2020, 182(1): 145-161. DOI:10.1016/j.cell.2020.05.021 |
| [65] |
SHA G, SUN P, KONG X, HAN X, SUN Q, FOUILLEN L, ZHAO J, LI Y, YANG L, WANG Y, GONG Q, ZHOU Y, ZHOU W, JAIN R, GAO J, HUANG R, CHEN X, ZHENG L, ZHANG W, QIN Z, ZHOU Q, ZENG Q, XIE K, XU J, CHIU T Y, GUO L, MORTIMER J C, BOUTTÉ Y, LI Q, KANG Z, RONALD P C, LI G. Genome editing of a rice CDPDAG synthase confers multipathogen resistance[J]. Nature, 2023, 618(7967): 1017-1023. DOI:10.1038/s41586-023-06205-2 |
| [66] |
CHEN P D, QI L L, ZHOU B, ZHANG S Z, LIU D J. Development and molecular cytogenetic analysis of wheat-Haynaldia villosa 6VS/6AL translocation lines specifying resistance to powdery mildew[J]. Theoretical and Applied Genetics, 1995, 91(6): 1125-1128. DOI:10.1007/BF00223930 |
| [67] |
WANG B, MENG T, XIAO B, YU T, YUE T, JIN Y, MA P. Fighting wheat powdery mildew: From genes to fields[J]. Theoretical and Applied Genetics, 2023, 136(9): 196. DOI:10.1007/s00122-023-04445-4 |
| [68] |
LI S, LIN D, ZHANG Y, DENG M, CHEN Y, LYU B, LI B, LEI Y, WANG Y, ZHAO L, LIANG Y, LIU J, CHEN K, LIU Z, XIAO J, QIU J L, GAO C. Genome-edited powdery mildew resistance in wheat without growth penalties[J]. Nature, 2022, 602(7897): 455-460. DOI:10.1038/s41586-022-04395-9 |
| [69] |
LIU H, HUANG Y, LI X, WANG H, DING Y, KANG C, SUN M, LI F, WANG J, DENG Y, YANG X, HUANG X, GAO X, YUAN L, AN D, WANG W, HOLDING D R, WU Y. High frequency DNA rearrangement at qγ27 creates a novel allele for quality protein maize breeding[J]. Communications Biology, 2019, 2(1): 460. DOI:10.1038/s42003-019-0711-0 |
| [70] |
WANG H, YIN H, JIAO C, FANG X, WANG G, LI G, NI F, LI P, SU P, GE W, LYU Z, XU S, YANG Y, HAO Y, CHENG X, ZHAO J, LIU C, XU F, MA X, SUN S, ZHAO Y, BAO Y, LIU C, ZHANG J, PAVLICEK T, LI A, YANG Z, NEVO E, KONG L. Sympatric speciation of wild emmer wheat driven by ecology and chromosomal rearrangements[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(11): 5955-5963. DOI:10.1073/pnas.1920415117 |
| [71] |
LU Y, WANG J, CHEN B, MO S, LIAN L, LUO Y, DING D, DING Y, CAO Q, LI Y, LI Y, LIU G, HOU Q, CHENG T, WEI J, ZHANG Y, CHEN G, SONG C, HU Q, SUN S, FAN G, WANG Y, LIU Z, SONG B, ZHU J K, LI H, JIANG L. A donor-DNA-free CRISPR/Cas-based approach to gene knock-up in rice[J]. Nature Plants, 2021, 7(11): 1445-1452. DOI:10.1038/s41477-021-01019-4 |
| [72] |
DECOTTIGNIES A. Microhomology-mediated end joining in fission yeast is repressed by pku70 and relies on genes involved in homologous recombination[J]. Genetics, 2007, 176(3): 1403-1415. DOI:10.1534/genetics.107.071621 |
| [73] |
TAN J, ZHAO Y, WANG B, HAO Y, WANG Y, LI Y, LUO W, ZONG W, LI G, CHEN S, MA K, XIE X, CHEN L, LIU Y G, ZHU Q. Efficient CRISPR/Cas9-based plant genomic fragment deletions by microhomology-mediated end joining[J]. Plant Biotechnology Journal, 2020, 18(11): 2161-2163. DOI:10.1111/pbi.13390 |
| [74] |
ZHU G, ZHANG L, MA L, LIU Q, WANG K, LI J, QU G, ZHU B, FU D, LUO Y, ZHU H. Efficient large fragment deletion in plants: Double pairs of sgRNAs are better than dual sgRNAs[J]. Horticulture Research, 2023, 10(10): uhad168. DOI:10.1093/hr/uhad168 |
| [75] |
LYU P, SU F, CHEN F, YAN C, XIA D, SUN H, LI S, DUAN Z, MA C, ZHANG H, WANG M, NIU X, ZHU J K, ZHANG J. Genome editing in rice using CRISPR/Cas12i3[J]. Plant Biotechnology Journal, 2023, 22(2): 379-385. DOI:10.1111/pbi.14192 |
| [76] |
SUN C, LEI Y, LI B, GAO Q, LI Y, CAO W, YANG C, LI H, WANG Z, LI Y, WANG Y, LIU J, ZHAO K T, GAO C. Precise integration of large DNA sequences in plant genomes using PrimeRoot editors[J]. Nature Biotechnology, 2023, 42(2): 316-327. DOI:10.1038/s41587-023-01769-w |
| [77] |
KIM H, KIM S-T, RYU J, KANG B C, KIM J S, KIM S G. CRISPR/Cpf1-mediated DNA-free plant genome editing[J]. Nature Communications, 2017, 8(1): 14406. DOI:10.1038/ncomms14406 |
| [78] |
GAO J, WANG G, MA S, XIE X, WU X, ZHANG X, WU Y, ZHAO P, XIA Q. CRISPR/Cas9-mediated targeted mutagenesis in Nicotiana tabacum[J]. Plant Molecular Biology, 2015, 87(1): 99-110. DOI:10.1007/s11103-014-0263-0 |
(责任编辑 马春敏)
 2024, Vol. 51
2024, Vol. 51