文章信息
基金项目
- 广东省自然科学基金(2019A1515010820);广州市科技计划项目(201904010183)
作者简介
- 廖冰(1998—),女,在读硕士生,研究方向为水稻抗旱性机理,E-mail:519480030@qq.com.
通讯作者
- 何平(1967—),男,博士,副教授,研究方向为水稻抗旱性机理,E-mail:phe@scau.edu.cn.
文章历史
- 收稿日期:2024-01-05
2. 华南农业大学农学院,广东 广州 510642
2. College of Agriculture, South China Agricultural University, Guangzhou 510642, China
【研究意义】水稻是广东最主要的粮食作物,年种植面积大且年产量占比粮食作物总产量高达76%,广东省是我国传统的水稻主产区,2021年广东省水稻总产量1 104.4万t,占全国总产量的5.19%,但在全球气候变化的背景下,干旱频率、强度和持续时间都在显著增加,导致水稻产量下降,高昂的经济和社会成本让人们越来越关注干旱[1-3]。干旱是主要的非生物胁迫因素之一,植物的干旱胁迫应答是一个复杂的生理生化过程[4-5]。【前人研究进展】野生稻是研究水稻抗逆性的天然基因库,具有复杂的遗传背景和丰富的遗传多样性,在长期的自然进化过程中其可不断地适应外界环境、抵抗多种逆境[6]。广东也是我国野生稻的主要分布省份之一,保存有丰富多样的稻种资源[7]。前人对野生稻遗传进行了大量研究,周少霞[8]以对西东乡野生稻为供体构建的渗入系进行30% PEG胁迫研究,共定位到11个与抗旱相关的的QTL,其中在3条染色体上发现4个QTLs的加性效应值为正。胡标林等[9]选用R974//R974/ 东乡野生稻回交重组自交系及其亲本等66份水稻材料,进行苗期抗旱性鉴定及其综合指标筛选,结果BIL群体苗期抗旱性的遗传主要由2个独立基因+ 多基因控制。王黎明等[10]利用30对SSR标记对来自不同地区的36份普通野生稻和34份栽培稻进行遗传多样性研究,共检测出121个等位变异,发现野生稻较栽培稻具有更加丰富的遗传多样性。【本研究切入点】许多学者通过构建单片段代换系(Singlesegment substitution lines,SSSLs)群体材料对水稻优良性状QTL进行鉴定及分析,SSSLs和其他定位的作图群体相比,排除了背景干扰,具有单一性等优点,是进行野生稻基因精细定位、克隆、等位性分析及育种的优良材料。【拟解决的关键问题】南方野生稻和展颖野生稻属于AA染色体组型野生稻,是野生稻家族中的重要成员,本研究利用以展颖野生稻和南方野生稻为供体、‘华粳籼74’为受体构建的单片段代换系(SSSLs)为供试材料[11],将SSSLs培养至三叶一心期后利用20% PEG-6000模拟干旱胁迫,进行4个耐旱性状的测定,并根据代换片段信息进行QTL鉴定[12],可为SSSLs苗期耐旱QTLs精细定位与克隆研究奠定基础。
1 材料与方法 1.1 供试材料试验于2022—2023年在华南农业大学生命科学学院实验室进行。供试材料为以展颖野生稻和南方野生稻为供体、‘华粳籼74’(HJX74)为受体构建的93份SSSLs[11],以HJX74为对照,其中4份SSSLs(编号以X开头)供体为展颖野生稻,89份SSSLs(编号以M开头)供体为南方野生稻。
1.2 SSSLs培养与处理参考黄秀艳等[13]的培养方法,每份SSSL经打破休眠、消毒处理后,将种子放入培养皿(直径9 cm)进行催芽,在培养皿上标记材料的编号信息;将培养皿放入光照培养箱(28 ℃,12 h光照,光强18 000 Lux;12 h黑暗)催芽,在催芽过程中需每天更换蒸馏水,待幼苗生长至3~4 cm后,将其插入96孔板中,每间隔1个孔插入1株幼苗,每个96孔板插45株幼苗,放入黑色托盘(32 cm×23.5 cm×5 cm)中培养,每个托盘放入10个96孔板和2 L培养液,幼苗采用1/4、1/2和全营养液的浓度梯度培养。
将幼苗培养至三叶一心期,处理组以20% PEG-6000模拟干旱胁迫,对照组不加干旱胁迫剂培养,共处理5 d,期间每天补充蒸馏水至2 L。每处理挑选长势相同的幼苗10株,测定苗高和根长;然后将幼苗装入信封,105 ℃杀青1 h,再80 ℃烘干至恒质量,称量其根干质量与苗干质量。以各性状的相对值作为评价指标,包括相对根长(Relative root length,RRL)、相对苗高(Relative seedling height,RSH)、相对根干质量(Relative root dry weight,RRDW)、相对苗干质量(Relative seedling dry weight,RSDW)。
1.3 数据计算与统计分析1.3.1 隶属函数值计算 结合测定的4个耐旱指标,并参考黄秀艳等[13]利用隶属函数值法进行SSSLs耐旱性综合评价。计算公式如下:
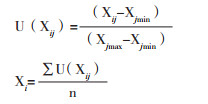
|
式中,U(Xij)表示i品系j指标的隶属函数度,Xij表示i品系j指标的测定值,Xjmax、Xjmin分别表示最大值和最小值,n为测定指标个数,Xi表示i材料n个测定指标的平均隶属函数值(Mean membership function values,MMFV)。
1.3.2 QTL鉴定 QTL的命名参考Mccouch[14]的方法。QTL的加性效应(Additive effect,A)和表型贡献率(Phenotypic contribution rate,P)计算参考Eshed等[15]的方法,计算公式如下:
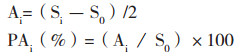
|
式中S0为HJX74的表型值,Si为SSSL的表型值。
1.3.3 数据处理 采用IBM SPSS 26.0、GraphPad Prism和Mapchart 2.3软件进行数据统计分析及作图,利用相关性分析和方差分析(ANOVA)、Duncan’s多重比较法检测HJX74与SSSLs之间的表型差异。
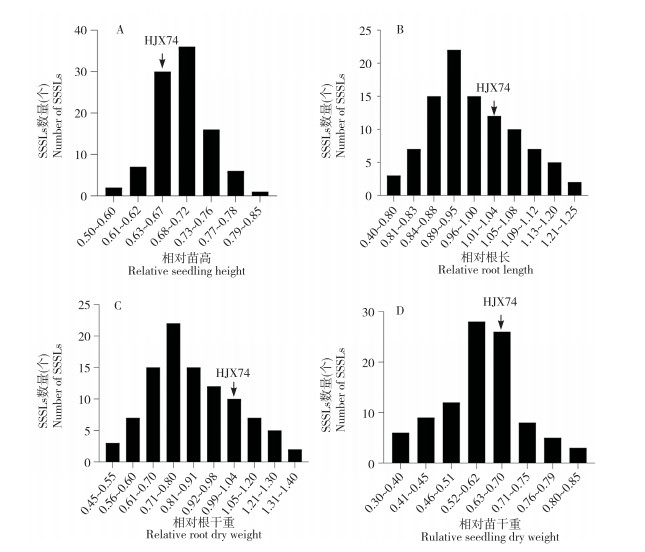
2 结果与分析 2.1 93份SSSLs苗期耐旱指标相对值的变化20% PEG-6000处理条件下,93份SSSLs苗期4个耐旱指标的相对值频次分布均呈正态分布(图 1),各指标相对值在SSSLs间存在差异,其中相对苗高的分布范围为0.50~0.85;相对根长的分布范围为0.40~1.25;相对根干质量的分布范围为0.45~1.40;相对苗干质量的分布范围为0.30~0.85。
|
| 图中箭头指向分别表示6个指标中HJX74处于的分布范围 Arrows in the figure indicate the distribution ranges of HJX74 of six indicators 图 1 93份SSSLs苗期耐旱指标相对值频次分布 Fig. 1 Frequency distribution of drought tolerance indexes in 93 SSSLs |
2.2 93份SSSLs苗期耐旱指标相对值与平均隶属函数相关性分析
在20% PEG-6000处理条件下,对HJX74和93个SSSLs的4个耐旱指标与平均隶属函数值进行相关性分析,结果(表 1)显示RSH与RSDW呈极显著正相关(P<0.01),表明幼苗生长与苗的干物质积累有关;RRDW与RSDW呈极显著正相关(P<0.01),表明幼苗的生长受干物质积累的影响;各指标间与平均隶属函数值均呈极显著正相关,表明4个指标可作为SSSLs苗期耐旱性鉴定和评价指标。
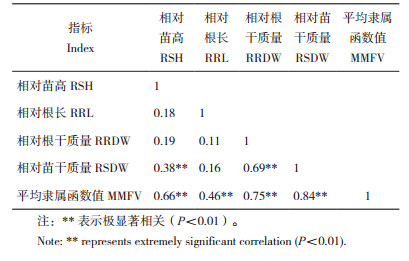
|
2.3 隶属函数法综合指标分析
通过公式计算出HJX74和SSSLs数据指标的隶属函数值,93份SSSLs表现出不同的耐旱性(表 2),根据模糊隶属函数法原理,MMFV越大表示该材料的耐旱性越强[13]。M124的平均隶属函数值为0.71,在所有SSSLs中耐旱性最强,而平均隶属函数值最小的材料为M99(0.20),其抗旱性最弱。HJX74平均隶属函数值为0.54,高于HJX74的SSSLs有25个,平均隶属函数值大于0.60的SSSLs有14个,表明这14个SSSLs的耐旱能力较强。

|
2.4 耐旱性SSSLs筛选及QTL鉴定
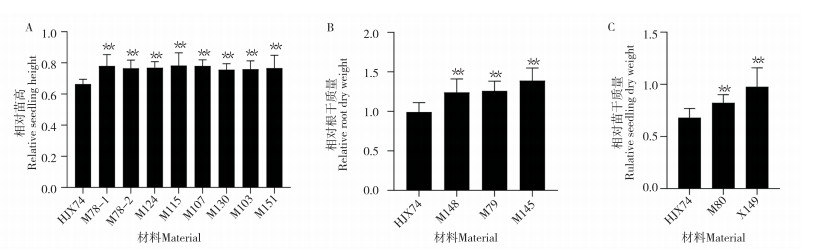
2.4.1 耐旱性SSSLs QTL鉴定材料筛选 在P<0.01水平下进行Duncan’s比较93份SSSLs材料和HJX74目的性状的差异显著性。若某个指标的相对值显著大于HJX74,则认为该材料代换片段上携带相应的耐旱性QTL。结果(图 2)显示,93份SSSLs相对苗高、相对根干质量和相对苗干质量存在显著差异,而相对根长无显著差异,因此耐旱QTL鉴定材料的筛选指标可选择相对苗高、相对根干质量和相对苗干质量。以相对苗高为性状指标时,M78-1、M78-2、M124、M115、M107、M130、M103和M151,这8个SSSLs的相对苗高均显著大于HJX74,表明这8个SSSLs的耐旱性较强;以相对根干质量为性状指标时,M148、M79和M145这3个SSSLs也显著大于HJX74,表明上述SSSLs的耐旱性较强;以相对苗干质量为性状指标时,M80和X149 SSSLs的地上部干质量显著大于HJX74,表明二者的耐旱性较强。93份SSLs的平均隶属函数值分布范围在0.20~0.71,其中M124平均隶属函数值最大、排序最高,达0.71,表明M124具有较强的耐旱性,且在相对苗高指标鉴定到与相对苗高有关的QTL。
|
| **表示差异显著(P<0.01) ** represents significant difference (P < 0.01) 图 2 20% PEG-6000处理条件下耐旱SSSLs中的相对苗高(A)、相对根干质量(B)和相对苗干质量(C)QTLs Fig. 2 QTLs of relative seedling height (A), relative root dry weight (B) and relative seedling dry weight (C) in drought-tolerant SSSLs under 20% PEG-6000 treatment |
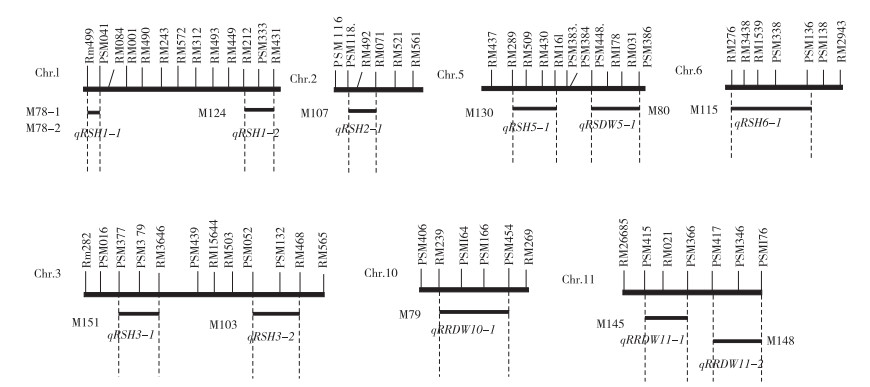
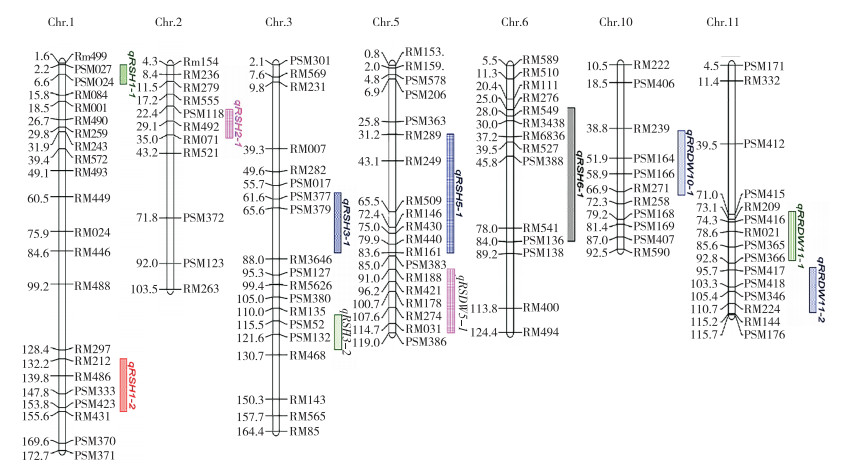
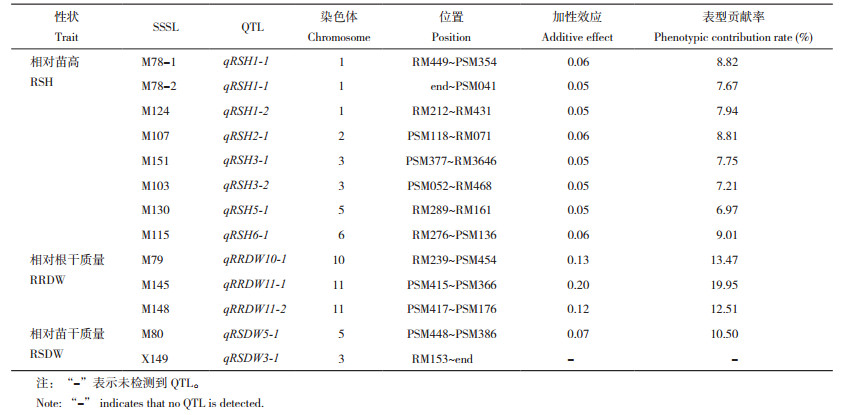
2.4.2 相对苗高QTL鉴定及其遗传效应 根据SSSLs QTL鉴定原理[16],如图 3、图 4和表 3所示,以相对苗高进行QTL鉴定,M78-1与M78-2材料的单片段在1号染色体上存在重叠片段end~PSM041,认为该区间可能存在一个共同的QTL,为qRSH1-1;从QTL的加性效应来看,M78-1与M78-2的QTL加性效应分别为0.06和0.05,表型贡献率分别为8.82% 和7.67%,表明M78-1与M78-2的代换片段上携带有来自南方野生稻与相对苗高相关的QTL。此外,M124和M107分别在1号和2号染色体上携带有来自南方野生稻与相对苗高相关的QTL,为qRSH1-2和qRSH2-1,加性效应为0.05和0.06,表型贡献率为7.94% 和8.81%;M151和M103在3号染色体上携带相对苗高相关QTL,为qRSH3-1和qRSH3-2,加性效应均为0.05,表型贡献率为7.75% 和7.21%;M130在5号染色体上携带相对苗高相关QTL,为qRSH5-1,加性效应为0.05,表型贡献率为6.97%;M115在6号染色体上携带相对苗高相关QTL,为qRSH6-1,加性效应为0.06,表型贡献率为9.01%;在相对苗高鉴定到的QTLs表型贡献率均为正值,表明上述SSSLs上携带来自供体野生稻的代换片段。
|
| 图 3 耐旱SSSLs中QTLs的替换图谱 Fig. 3 Substitution mapping of QTLs in drought-tolerant SSSLs |
|
| 图 4 耐旱QTLs在染色体上的分布 Fig. 4 Chromosomal distribution of identified drought-tolerant QTLs |
|
2.4.3 相对根干质量QTL鉴定及其遗传效应 2以相对根干质量作为耐旱指标进行QTL鉴定(图 3、图 4、表 3)发现,M79在10号染色体上携带相对根干质量相关QTL,为qRRDW10-1,加性效应为0.13,表型贡献率为13.47%;M145和M148在11号染色体上携带相对根干质量相关QTL,为qRRDW11-1和qRRDW11-2,加性效应为0.20和0.12,表型贡献率为19.95% 和12.51%。
2.4.4 相对苗干质量QTL鉴定及其遗传效应 以相对苗干质量作为耐旱指标进行QTL鉴定发现,M80在5号染色体上携带相对苗干质量相关QTL,为qRSDW5-1(图 3和图 4),加性效应为0.07,表型贡献率为10.50%(表 3)。
3 讨论干旱是影响水稻产量的重要限制因素之一,筛选耐旱水稻种质资源是实现旱作水稻生产的首要任务和前提[17]。本研究在20% PEG-6000胁迫条件下,通过单因素方差分析得出相对苗高、相对根干质量和相对苗干质量作为SSSLs苗期耐旱性QTL鉴定的指标,从这3个指标筛选出来的SSSLs加性效应几乎均为正值,表明SSSLs携带来自供体野生稻相关的等位基因,抵御干旱胁迫的能力较强,幼苗生长及干物质积累受干旱影响较小。此外,观察到所有SSSLs的苗高随着干旱胁迫时间延长而表现出生长延缓的现象,此现象可能是植物为避免蒸腾作用引起的水分损失而采取的策略。
水稻的抗旱性机制是由多基因控制的数量性状,在水稻不同生长发育时期调控干旱的基因发挥作用不同,但只有在特定时期发挥作用且具有效应的QTL才会被检测出来[18-19]。本研究在3个指标中检测到相关的QTL共11个,其中调控相对苗高7个、相对根干质量3个、相对苗干质量1个,主要分布在1、2、3、5、6、10和11号染色体上。关于水稻苗期相关性状QTL定位有较多报道,王娇[20]以IAPAR-9/ 秋光构建的自交系群体为材料,在3个指标相对值检测到6个与干旱有关的QTL,其中在5号染色体RM7118-RM430区间检测到1个与相对苗高有关的QTL,该区间包含在本研究不同相对苗高QTL qRSH5-1的RM289~RM161区间内。岳兵[21]和姜雪等[22]均在同一区间(RM459~RM161)定位到相关性状的QTL,前者在该区间定位为产量相关性状,后者性状为苗高,其研究的区间包含在本研究中相对苗高QTL qRSH5-1的RM289-RM161区间内,表明5号染色体上该区间存在与苗高及产量相关的QTL;此外,姜雪等[22]在1号染色体的RM472~RM104区间检测到1个苗高QTL,该区间包含在本研究中相对苗高QTL qRSH1-2的RM212~RM431区间,表明该区间存在与苗高有关的耐旱QTL。姜雪[23]对重组自交系群体进行苗期耐旱QTL定位,在胁迫处理条件下共检测到6个与可溶性蛋白相关的QTL,其中在第11号染色体RM021~RM206和RM206~RM144区间检测到2个QTL,qSP-11-1和qSP-11-2,该区间在本研究鉴定的相对根干质量QTL qRRDW11-1和qRRDW11-2的PSM415~PSM366和PSM417~PSM176区间内,表明水稻11号染色体上该区间存在与渗透调节有关的耐旱QTL。Baisakh等[24]对温室干旱条件下的水稻产量进行QTL鉴定,在5号染色体上发现与每穗籽粒数相关的QTL qGN5.1与本研究检测到的相对苗高QTL qRSH5-1在RM289~RM161区间内,说明5号染色体上存在与产量和苗高有关的QTL。Yue等[25]在5号染色体的RM507~RM13区间鉴定到1个相对生物量QTL qRbm5,包含在本研究检测到的相对苗高QTL qRSW5-1的PSM448~PSM386区间内。此外,在11号染色体的RM287~RM229检测到多个性状的QTL,包括最大根长和长根率等性状,该区间在本研究检测到的相对根干质量的QTL qRRDW11-1的PSM415~PSM366区间内,说明11号染色体上存在与根部有关的QTL。Sabar等[26]利用418个水稻组成的F2群体进行QTL鉴定,探究群体抗旱性和产量相关的QTL,采用复合区间作图法共检测到21个QTL,其中在1号染色体的RM212~RM265区间检测到1个苗高的QTL qHt1,本研究在1号染色体RM212~RM431区间检测到相对苗高的QTL qRSH1-2,qHt1在本研究区间内,说明1号染色体上存在与相对苗高相关的QTL;在2号染色体的RM110~RM174鉴定到1个与杆长有关的QTL qCmL2,与本研究检测到的相对苗高QTL qRSH2-1的PSM118~RM492区间有重叠的部分。
本研究在相对苗高QTL qRSH1-1的位置找到了2个已克隆的耐旱基因,分别是OsNAC6和OsPP2C09。OsNAC6 cDNA全长1 423 bp,包含有3个外显子,编码1个由205氨基酸组成的转录调控因子,芯片分析结果表明许多受非生物和生物胁迫诱导表达的基因在过量表达OsNAC6的植株中表达量都显示上调。OsNAC6不仅在植物适应非生物胁迫中发挥作用,在整合非生物和生物胁迫信号中也发挥作用,过量表达OsNAC6的转基因水稻对干旱胁迫的耐受性增强[27-28]。OsPP2C09蛋白与脱落酸(ABA)信号的核心成分相互作用,并在体外显示PP2C磷酸酶活性。OsPP2C09通过ABA信号对植株生长有正向调节作用,但对抗旱性有负调节作用[29-30]。OsPP2C09也可以通过激活DRE启动子,激活1条不依赖于ABA的信号通路,正向调控干旱[31]。上述结果表明本研究鉴定到的耐旱SSSLs携带苗期耐旱QTL,可为后续发掘水稻相关耐旱基因奠定基础。
4 结论本研究以平均隶属函数综合反应指标评价水稻苗期耐旱程度,筛选出耐旱能力较强(隶属函数值>0.60)的SSSLs有14个(M124、M123、M145、M157、M80、M130、M78-2、M82、M122、M101、M79、M139、M78-1、M90);根据QTL鉴定原理在相对苗高、相对根干质量和相对苗干质量这3个指标上共鉴定到11个QTL(qRSH1-1、qRSH1-2、qRSH2-1、qRSH3-1、qRSH3-2、qRSH5-1、qRSH6-1、qRRDW10-1、qRRDW11-1、qRRDW11-2、qRSDW5-1),均表现为正向加性效应和表型贡献率,其中有4个QTL与前人研究鉴定的部分区间存在重合片段,且在SSSLs M124的相对苗高qRSH1-2找到2个与耐旱相关的基因;7个QTL发现新的耐旱定位区间。以上SSSLs携带苗期耐旱QTL,可作为发掘耐旱基因的重要材料。
| [1] |
ZHAO W X, CHOU J M, LI J N, YUAN X, LI Y M, HAO Y D. Impacts of extreme climate events on future rice yields in global major rice producing regions[J]. International Journal of Environmental Research and Public Health, 2022, 19(8): 4437. DOI:10.3390/ijerph19084437 |
| [2] |
RAHMAN B, ZHANG J H. Trends in rice research: 2030 and beyond[J]. Food and Energy Security, 2023, 12(2): 390. DOI:10.1002/fes3.390 |
| [3] |
陈树嘉, 罗克波, 林桂芬, 陈达刚, 刘传光, 周新桥, 郭洁, 陈可. 2004—2022年广东审定水稻品种优质化与产量、抗性协同改良育种进展[J]. 广东农业科学, 2023, 50(12): 73-82. DOI:10.16768/j.issn.1004-874X.2023.12.007 CHEN S J, LUO K B, LIN G F, CHEN D G, LIU C G, ZHOU X Q, GUO J, CHEN K. Progress in collaborative improvement of high quality, yield, and disease resistance breeding of approved rice varieties in Guangdong from 2004 to 2022[J]. Guangdong Agricultural Sciences, 2023, 50(12): 73-82. DOI:10.16768/j.issn.1004-874X.2023.12.007 |
| [4] |
包秋娟. 干旱胁迫下棉花转录组分析[D]. 乌鲁木齐: 新疆大学, 2018. BAO Q J. The transcriptome analysis of cotton under drought stress[D]. Urumqi: Xinjiang University, 2018. |
| [5] |
SHIVHARE R, ASIFA M H, LATA C. Comparative transcriptome analysis reveals the genes and pathways involved in terminal drought tolerance in pearl millet[J]. Plant Molecular Biology, 2010, 103(6): 639-652. DOI:10.1007/s11103-020-01015-w |
| [6] |
钟代彬, 罗利军, 应存山. 野生稻有利基因转移研究进展[J]. 中国水稻科学, 2000(2): 40-43. DOI:10.16819/j.1001-7216.2000.02.008 ZHONG D B, LUO L J, YING C S. Advances on transferring elite gene from wild rice species into cultivated rice[J]. Chinese Journal of Rice Science, 2000(2): 40-43. DOI:10.16819/j.1001-7216.2000.02.008 |
| [7] |
潘大建, 范芝兰, 邹建运. 广东野生稻种质资源保护与育种利用[J]. 广东农业科学, 2022, 49(9): 92-104. DOI:10.16768/j.issn.1004-874X.2022.09.010 PAN D J, FAN Z L, ZOU J Y. Conservation and breeding utilization of wild rice germplasm resources in Guangdong Province[J]. Guangdong Agricultural Sciences, 2022, 49(9): 92-104. DOI:10.16768/j.issn.1004-874X.2022.09.010 |
| [8] |
周少霞. 江西东乡普通野生稻抗旱渗入系的构建及抗旱基因定位[D]. 北京: 中国农业大学, 2005. ZHOU S X. Development of drought tolernce introgression lines of common wild rice (Oryza rufipogon Griff. ) from Dongxiang in Jiangxi Province and QTL mapping of drought tolerance[D]. Beijing: China Agricultural University, 2005. |
| [9] |
胡标林, 扬平, 万勇, 李霞, 罗世友, 罗向东, 谢建坤. 东乡野生稻BILs群体苗期抗旱性综合评价及其遗传分析[J]. 植物遗传资源学报, 2013, 14(2): 249-256. DOI:10.13430/j.cnki.jpgr.2013.02.022 HU B L, YANG P, WAN Y, LI X, LUO S Y, LUO X D, XIE J K. Comprehensive assessment of drought resistance of BILs ppulation derived from Dongxiang wild rice (Oryza rufipogon Griff.) at seedling stage and its genetic analysis[J]. Journal of Plant Genetic Resources, 2013, 14(2): 249-256. DOI:10.13430/j.cnki.jpgr.2013.02.022 |
| [10] |
王黎明, 李战胜, 高旭华, 沈雪峰, 方越, 陈勇. 杂草稻、栽培稻及野生稻的遗传多样性比较[J]. 华中农业大学学报, 2012, 31(3): 275-280. DOI:10.13300/j.cnki.hnlkxb.2012.03.015 WANG L M, LI Z S, GAO X H, SHEN X F, FANG Y, CHEN Y. Genetic diversities among weedy rices, cultivated rices and wild rices[J]. Journal of Huazhong Agricultural University, 2012, 31(3): 275-280. DOI:10.13300/j.cnki.hnlkxb.2012.03.015 |
| [11] |
ZHAO H W, SUN L L, XIONG T Y, WANG Z Q, LIAO Y, ZOU T, ZHENG M M, ZHANG Z, PAN X P, HE N, ZHANG G Q, ZHU H T, LIU Z Q, HE P, FU X L. Genetic characterization of the chromosome single-segment substitution lines of O. glumaepatula and O. barthii and identification of QTLs for yield-related traits[J]. Molecular Breeding, 2019, 39(4): 51. DOI:10.1007/s11032-019-0960-0 |
| [12] |
CHURCHILL G A, DOERGE R W. Empirical threshold values for quantitative trait mapping[J]. Genetics, 1994, 138(3): 963-971. DOI:10.1093/genetics/138.3.963 |
| [13] |
黄秀艳, 栗俊波, 廖冰, 傅雪琳, 何平. 野生稻单片段代换系芽期耐旱性评价及QTLs鉴定[J]. 干旱地区农业研究, 2023, 41(4): 32-40. DOI:10.7606/j.issn.1000-7601.2023.04.04 HUANG X Y, LI J B, LIAO B, FU X L, HE P. Evaluation of drought tolerance and identification of QTLs in single-segment substitution lines of wild rice at bud stage.[J]. Agricultural Research in the Arid Areas, 2023, 41(4): 32-40. DOI:10.7606/j.issn.1000-7601.2023.04.04 |
| [14] |
MCCOUCH S R. Gene nomenclature system for rice[J]. Rice, 2008, 1(1): 72-84. DOI:10.1007/s12284-008-9004-9 |
| [15] |
ESHED Y, ZAMIR D. An introgression line population of Lycopersicon pennellii in the cultivated tomato enables the identification and fine mapping of yield-associated QTL[J]. Genetics, 1995, 141(3): 1147-1162. DOI:10.1093/genetics/141.3.1147 |
| [16] |
王璋强, 李蕊芯, 陆怡, 杨帆, 刘田田, 何平, 张鸿兴, 熊毅, 傅雪琳. 展颖野生稻单片段代换系芽期耐Cd材料筛选及QTL鉴定[J]. 华南农业大学学报, 2020, 41(1): 69-77. DOI:10.7671/j.issn.1001-411X.201905023 WANG Z Q, LI R X, LU Y, YANG F, LIU T T, HE P, ZHANG H X, XIONG Y, FU X L. Cd-tolerant germplasm screen and QTL identification of single-segment substitution lines of Oryza glumaepatula at the germination period[J]. Journal of South China Agricultural University, 2020, 41(1): 69-77. DOI:10.7671/j.issn.1001-411X.201905023 |
| [17] |
李双香, 钱亦璐, 章瑾诗, 张维林, 王涛, 杨玲. 耐旱水稻种质的筛选鉴定[J]. 浙江师范大学学报(自然科学版), 2023, 46(2): 196-200. DOI:10.16218/j.issn.1001-5051.2023.020 LI S X, QIAN Y L, ZHANG J S, ZHANG W L, WANG T, YANG L. Screening and identification of drought-tolerance rice germplasm[J]. Journal of Zhejiang Normal University (Natural Sciences), 2023, 46(2): 196-200. DOI:10.16218/j.issn.1001-5051.2023.020 |
| [18] |
PATIL S, SRIVIDHYA A, VEERAGHATTAPU R, DRBORAH D A K, KADAMBARI G M, NAGIREDDY R, SIDDIQ E A, VEMIREDDY L. Molecular dissection of a genomic region governing root traits associated with drought tolerance employing a combinatorial approach of QTL mapping and RNA-seq in rice[J]. Plant Molecular Biology Reporter, 2017, 35(4): 457-468. DOI:10.1007/s11105-017-1037-z |
| [19] |
RASHEED A, HASSAN M U, AAMER M, BATOOL M, SHENG F, WU Z M, LI H J. A critical review on the improvement of drought stress tolerance in rice (Oryza sativa L.)[J]. Notulae Botanicae Horti Agrobotanici Cluj-Napoca, 2020, 48(4): 1756-1788. DOI:10.15835/48412128 |
| [20] |
王娇. 水稻抗旱性相关性状的QTL定位分析[D]. 银川: 宁夏大学, 2015. WANG J. QTL mapping analysis for rice drought resistant related traits[D]. Yinchuan: Ningxia University, 2015. |
| [21] |
岳兵. 水稻后期抗旱性遗传基础研究[D]. 武汉: 华中农业大学, 2005. YUE B. Study on the genetic basis of drought resistance at reproductive stage in rice[D]. Wuhan: Huazhong Agricultural University, 2005. |
| [22] |
姜雪, 马孝松, 罗利军, 刘鸿艳. 水稻苗期模拟干旱胁迫条件下表型性状QTL定位[J]. 作物杂志, 2016(5): 31-37. DOI:10.16035/j.issn.1001-7283.2016.05.006 JIANG X, MA X S, LUO L J, LIU H Y. QTL mapping of phenotypic traits under drought stress simulated by PEG-6000 in rice seedlings[J]. Crops, 2016(5): 31-37. DOI:10.16035/j.issn.1001-7283.2016.05.006 |
| [23] |
姜雪. 水稻苗期耐旱性基因位点的发掘[D]. 武汉: 华中农业大学, 2015. JIANG X. The exploration of drought tolerance gene loci in rice seedlings[D]. Wuhan: Huazhong Agricultural University, 2015. |
| [24] |
BAISAKH N, YABES J, GUTIERREZ A, MANGU V, MA P Y, FAMOSO A, PEREIRA A. Genetic mapping identifies consistent quantitative trait loci for yield traits of rice under greenhouse drought conditions[J]. Genes, 2020, 11(1): 62. DOI:10.3390/genes11010062 |
| [25] |
YUE B, XUE W Y, XIONG L Z, YU X Q, LUO L J, GUI K H, JIN D M, XING Y Z, ZHANG Q F. Genetic basis of drought resistance at reproductive stage in rice: Separation of drought tolerance from drought avoidance[J]. Genetics, 2006, 172(2): 1213-1228. DOI:10.1534/genetics.105.045062 |
| [26] |
SABAR M, SHABIR G, SHAH S M, ASLAM K, NAVEED S A, ARIF M. Identification and mapping of QTLs associated with drought tolerance traits in rice by a cross between super basmati and IR55419-04[J]. Breeding Science, 2019, 69(1): 169-178. DOI:10.1270/jsbbs.18068 |
| [27] |
OHNISHI T, SUGAHARA S, YAMADA T, KIKUCHI K, YOSHIBA Y, HIRANO H Y, TSUTSUMI N. OsNAC6, a member of the NAC gene family, is induced by various stresses in rice[J]. Genes & Genetic Systems, 2005, 80(2): 135-139. DOI:10.1266/ggs.80.135 |
| [28] |
NAKASHIMA K, TRAN L S P, VAN NGUYEN D, FUJITA M, MARUYAMA K, TODAKA D, ITO Y, HAYASHI N, SHINOZAKI K, YAMAGUCHI-SHINOZAKI K. Functional analysis of a NAC-type transcription factor OsNAC6 involved in abiotic and biotic stress-responsive gene expression in rice[J]. The Plant Journal, 2007, 51(4): 617-630. DOI:10.1111/j.1365-313X.2007.03168.x |
| [29] |
MIAO J, LI X F, LI X B, TAN W C, YOU A Q, WU S J, TAO Y J, CHEN C, WANG J, ZHANG D P, GONG Z Y, Yi C D, YANG Z F, GU M C, LIANG G H, ZHOU Y. OsPP2C09, a negative regulatory factor in abscisic acid signaling, plays an essential role in balancing plant growth and drought tolerance in rice[J]. New Phytologist, 2020, 227(5): 1417-1433. DOI:10.1111/nph.16670 |
| [30] |
KIM S, PARK S I, KWON H, CHO M H, KIM B G, CHUNG J H, NAM M H, SONG J S, KIM KH, YOON I S. The rice abscisic acid-responsive RING finger E3 ligase OsRF1 targets OsPP2C09 for degradation and confers drought and salinity tolerance in rice[J]. Frontiers in Plant Science, 2022, 12: 797940. DOI:10.3389/fpls.2021.797940 |
| [31] |
MIN M K, KIM R, HONG W J, JUNG K H, LEE K Y, KIM B G. OsPP2C09 is a bifunctional regulator in both ABA-dependent and independent abiotic stress signaling pathways[J]. International Journal of Molecular Sciences, 2021, 22(1): 393. DOI:10.3390/ijms22010393 |
(责任编辑 陈丽娥)
 2024, Vol. 51
2024, Vol. 51