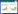文章信息
基金项目
- 江西省双千人才-自然科学领军人才长期项目(jxsq2023101038);江西省急需紧缺海外人才项目(20223BCJ25027);九江市基地和人才计划重点领域科技创新团队项目(S2022TDJS029)
作者简介
- 范思庆(1995—),男,硕士,研究实习员,研究方向为植物功能基因的鉴定及挖掘,E-mail:sqfan0310@163.com.
通讯作者
- 程春松(1987—),男,博士,副研究员,研究方向为中药资源与遗传学,E-mail:chengcs@lsbg.cn.
文章历史
- 收稿日期:2023-10-16
【研究意义】生长素是最早被发现的植物激素,在植物生长发育的多个阶段(如胚胎形成、顶端优势、生根、组织器官分化以及应激响应等)均发挥着至关重要的作用[1-4]。生长素的信号传导途径涉及3类蛋白质,即生长素响应因子(Auxin response factor,ARF)、AUX/IAA(Auxin/indole-3-acetic acid)和SCF(SKP1/Cullin1/F-box蛋白)复合物。其中,ARF被认为是调控生长素响应基因的核心蛋白[5-7]。ARF能够与生长素应答元件(Auxin response element, AuxRE)特异性结合,从而促进或抑制生长素响应基因的表达[8-9]。白木香〔Aquilaria sinensis(Lour.)Gilg〕是我国广泛栽培的香料树木,属于瑞香科(Thymelaeaceae)沉香属(Aquilaria),又被称作土沉香、女儿香等[10-12]。白木香的香气源于其含树脂的香心材,具有镇痛、镇静、抗炎、抗菌和神经保护等作用[13]。然而,香心材中的沉香物质属于白木香的次生代谢产物,白木香只有在受到昆虫啃食、雷击或其他生物与非生物胁迫的刺激时才会形成香心材。白木香的人工诱导结香技术已有多项研究报道,主要分为物理、化学和生物3种诱导方法[14-15]。物理方法包括打针、钻孔、割伤、断干、半断干、火烫和剥皮等传统手段,物理方法虽然可能会影响白木香的无性繁殖过程,但由于其简单、快捷、成本低等特点,是最常用的诱导方法;化学诱导法是在白木香伤口处涂抹甲酸、氯化铁、乙烯利等化学成分诱导结香,存在化学药品残留的风险;生物诱导法则通过各种微生物和昆虫刺激树体,以达到结香目的,但不同生物体处理得到的沉香品质差异较大。选择易结香的白木香品系作为人工诱导的白木香,是提高沉香产量的重要因素。易结香白木香品系是从普通白木香中筛选易于产香的优良树木,利用嫁接等无性繁殖方式获得的,其香气生成速度快且产量高。目前,关于白木香香气生成的完整机制尚无定论,国内外研究主要集中在人工诱导方法[16-17]、沉香成分分析[18-20]、沉香形成机理假说的验证[10, 21]、沉香物质合成代谢途径以及形成过程的信号调控机制等领域[22-23]。至今尚未有关于白木香ARF转录因子家族的研究报道,尤其是该转录因子家族成员是否参与了白木香的结香过程。【前人研究进展】ARF蛋白通常由2个关键结构域组成,即与靶基因结合的B3结构域和ARF结构域[24-25]。ARF基因家族普遍存在于多个植物物种中,但不同物种的ARF基因家族成员有一定差异。最早的ARF基因研究来自拟南芥(Arabidopsis thaliana),目前已确认该物种存在23个ARF家族成员[26]。随着下一代测序技术(Next generation sequencing,NGS)的不断进步,ARF基因在常见植物和作物中的功能研究不断深入,如水稻(Oryza sativa)[27-28]、玉米(Zea mays)[29-30]、大麦(Hordeum vulgare)[31]和番茄(Solanum lycopersicum)[32]等。【本研究切入点】基于已公开的白木香基因组[33]和转录组数据[34],利用生物信息学的方法鉴定白木香ARF基因,并从多角度深入分析理化性质、系统进化、基因结构、保守基序、启动子顺式作用元件、基因复制和共线性等。此外,通过RNA-seq分析技术,探索ARF基因在白木香结香过程中可能参与的潜在机制。【拟解决的关键问题】本研究通过生物信息学分析等手段鉴定白木香的ARF转录因子家族成员,并深入研究白木香ARF基因在机械损伤诱导结香过程中的功能,旨在为解析白木香结香机制提供理论支持,并为进一步研究提供理论基础。
1 材料与方法 1.1 白木香ARF基因家族成员的鉴定及理化性质分析下载白木香的基因组序列和基因结构注释文件[33],并利用TBtools[35]工具获取白木香ARF蛋白质序列。从TAIR网站(https://www.arabidopsis.org/)搜寻拟南芥AtARF基因家族的蛋白序列作为参考序列,并将其与白木香ARF蛋白质序列进行比对,去除冗余结果。将获得的白木香ARF蛋白质序列在NCBI的UniProtKb/Swiss-Prot(https://www.uniprot.org/)数据库进行blastp比对[36],将人工矫正blastp后蛋白序列作为AsARF基因家族成员,在Expasy(https://www.expasy.org/)网站预测白木香ARF蛋白的理化性质[37]。
1.2 白木香ARF基因家族成员进化分析利用MEGA-X[38]将白木香ARF蛋白序列与拟南芥、水稻的ARF蛋白序列进行多序列比对,采用最大似然法(ML)构建白木香、拟南芥和水稻的ARF家族系统进化树,其中bootstrap值设置1 000,氨基酸替换模型的选择依据AIC、BIC最优结果,本研究最优模型为JTT+G(Jones-Taylor-Thornton + Gamma Distributed)。
1.3 白木香ARF基因结构、保守基序及启动子区域顺式作用元件分析利用TBtools工具绘制AsARF基因结构图,在MEME网站(http://meme-suite.org/)预测ARF蛋白保守基序[39-40];以AsARF基因上游区域2 000 bp序列作为启动子,利用PlantCARE网站[41](https://bioinformatics.psb.ugent.be/webtools/plantcare/html/)对其进行顺式作用元件分析,最后利用R语言进行数据可视化。
1.4 白木香ARF基因染色体定位及共线性分析根据白木香基因组序列及基因结构注释文件,利用TBtools绘制AsARF基因在染色体上的位置;使用MCScanX进行共线性分析,并对共线性结果可视化。
1.5 白木香ARF基因家族的RNA-seq分析下载有关白木香伤害应激转录组数据[34](NCBI, BioProject PRJNA761258),经质控过滤、与白木香基因组比对、定量后,计算ARF转录因子在不同样品中的TPM值,以log2(TPM+1) 作为ARF基因表达水平,然后Z-score进行标准化后,最后利用R语言绘制AsARF基因表达热图。利用DESeq2对AsARF基因进行差异分析。
2 结果与分析 2.1 AsARF基因家族成员鉴定使用已报道的GigaDB数据库中23个拟南芥AtARF基因家族成员的蛋白质序列作为索引,在与白木香的蛋白序列进行双向blastp比对后,获取潜在的AsARF成员。基于保守结构域,对潜在成员进一步筛选,最终获得21个AsARF基因,临时命名为AsARF1~AsARF21(表 1)。21个AsARF基因的蛋白序列长度范围为580~1 124个氨基酸,平均长度约787个氨基酸;相对分子质量的范围介于63.27~124.6 KD;21个候选蛋白均属于亲水性蛋白(GRAY<0),其中酸性蛋白(pI<7)占90%。
2.2 AsARF蛋白进化关系分析
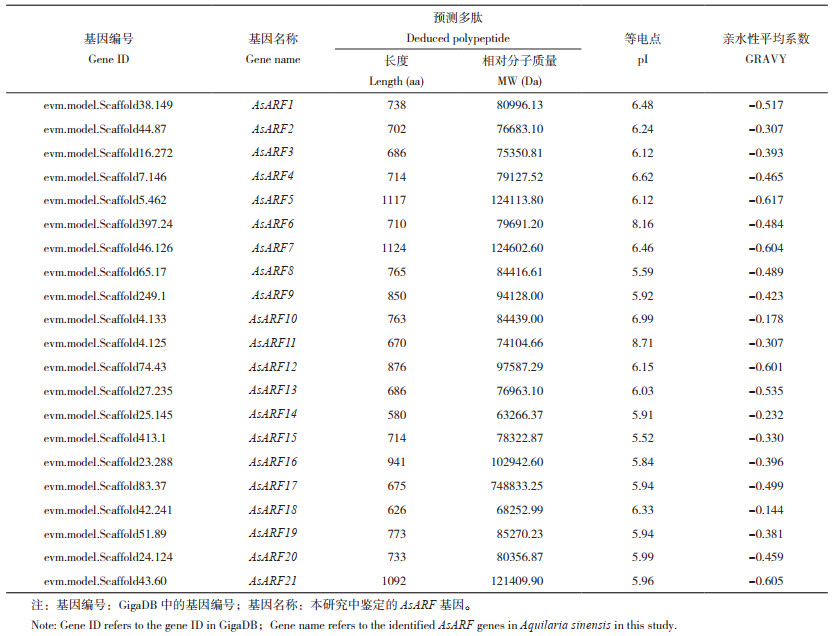
为研究AsARF基因家族成员之间的进化关系,利用最大似然法(ML)构建了拟南芥AtARF、水稻OsARF和白木香AsARF基因家族的系统发育进化树(图 1)。根据进化树的分支,将白木香、拟南芥和水稻ARF蛋白分成6个类群(Ⅰ~Ⅵ),分别包含35、11、25、15、2和4个成员。这些不同分类群的蛋白可能在功能上存在一定差异。除了Ⅵ类群,其余5个类群中都有相关的AsARF成员分布,Ⅰ类群包含5个成员(AsARF13、AsARF4、AsARF17、AsARF12、AsARF6),Ⅱ类群包含3个成员(AsARF19、AsARF1、AsARF20),Ⅲ类群包含7个成员(AsARF8、AsARF9、AsARF15、AsARF16、AsARF7、AsARF5、AsARF21),Ⅳ类群包含5个成员(AsARF11、AsARF10、AsARF2、AsARF18、AsARF3),而Ⅴ类群仅有1个成员(AsARF14)。从进化树的分支来看,在Ⅱ和Ⅵ类群中,AsARF与AtARF的亲缘关系非常近;在Ⅰ、Ⅲ和Ⅳ类群中,AsARF和AtARF的亲缘关系较近;而5个类群中的AsARF与OsARF的亲缘关系都较远。
|
| 图 1 ARF家族成员系统发育进化树 Fig. 1 Phylogenetic tree of ARF family members |
2.3 AsARF基因结构及保守基序分析
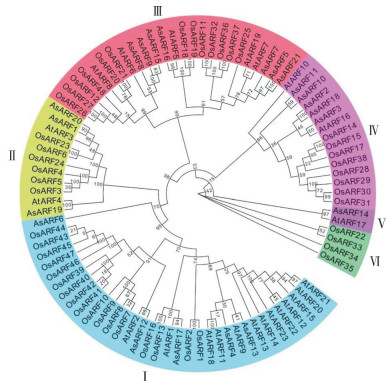
通过多重序列比较构建了21个AsARF蛋白序列进化树,并将基因结构进行了亚族比较(类群Ⅰ~Ⅴ)。基因结构分析表明,21个AsARF基因均含有外显子和内含子。不同AsARF基因所含外显子的数量存在很大差异,类群Ⅰ~Ⅲ成员的外显子数量分别为14~16、10~12、12~16个,Class Ⅳ成员的外显子数量差异较大,范围在3~19个之间,而类群Ⅴ成员仅1个,且外显子数量也最少(2个)。进一步对AsARF蛋白进行保守基序(Conserved motif)分析,共鉴定了10个Motif。其中,AsARF10含有5个基序,AsARF15含有7个基序,AsARF1、AsARF8、AsARF18、AsARF20含有8个基序,AsARF11和AsARF14含有9个基序,而其余的蛋白均含有10个保守基序(图 2)。
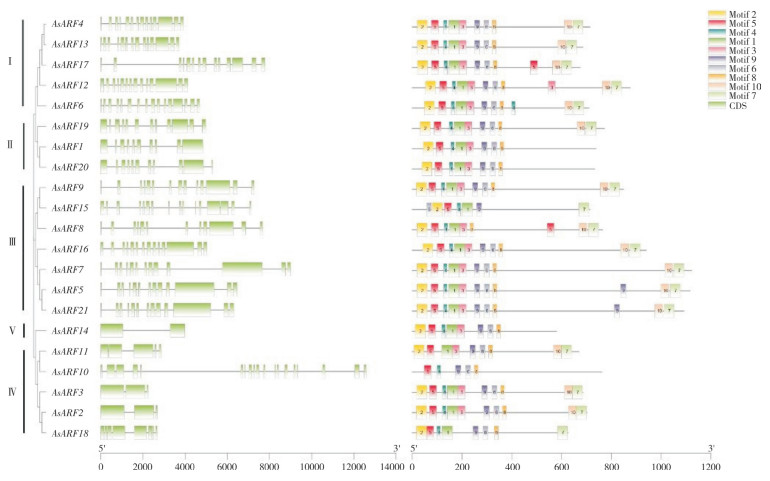
|
| 图 2 AsARF基因结构及保守基序 Fig. 2 Gene structures and conserved motif of AsARF |
2.4 AsARF启动子区域顺式作用元件分析
为探究AsARF的潜在调控机制,对其上游启动子区域的2 000 bp进行顺式作用元件分析,发现AsARF的启动子区域包含多种类型的响应元件,包括脱落酸响应元件(ABRE)、厌氧和缺氧诱导响应元件(ARE、GC-motif)、光响应元件(AE-box、GT1-motif、Sp1、I-box、G-box、Box4等)、赤霉素响应元件(GARE-motif、P-box、TATC-box)和茉莉酸甲酯响应元件(TGACG-motif、CGTCA-motif)等(图 3)。AsARF成员之间存在不同类型和数量的顺式作用元件,表明AsARF成员可能具有不同的生物学功能。
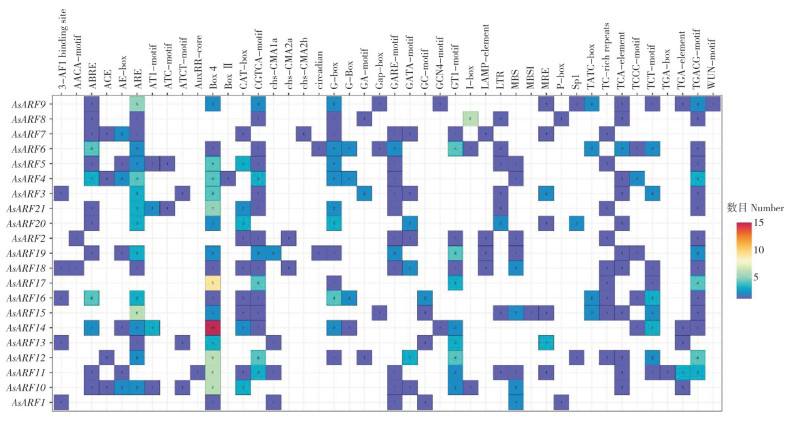
|
| 图 3 AsARF启动子区域顺式作用元件分析 Fig. 3 Analysis of cis-regulatory elements in the promoter region of AsARF |
2.5 AsARF在染色体的定位及共线性分析
将鉴定到的AsARF基因定位到染色体上,并观察结果(图 4)。除6、7号染色体外,其余6条染色体上都有AsARF基因分布。2、7号染色体上各有2个基因,1、8号染色体上各有3个基因,而3号染色体有5个基因。分布基因最多的为4号染色体(6个)。以氨基酸一致性>80%和基因比对覆盖率>90%为标准进行筛选,确定了分布在同一条染色体上的2个复制基因对(AsARF2/AsARF18、AsARF5/AsARF21)。尽管AsARF2和AsARF18看起来距离很近,但实际相隔≥ 1.4 Mb,表明在AsARF基因的扩增中并没有发生串联复制事件,且所有复制基因均为片段复制。2个复制基因对的Ka/Ks比率均<1(表 2),表明其主要在纯化选择下进化。共线性分析结果(图 5)显示,白木香与拟南芥基因组存在较多的共线性模块,但只有3对ARF直系同源基因。相比之下,白木香与水稻之间的共线性模块较少,且两者之间无ARF直系同源基因,表明白木香ARF基因更接近于拟南芥,而与水稻的进化距离较远。
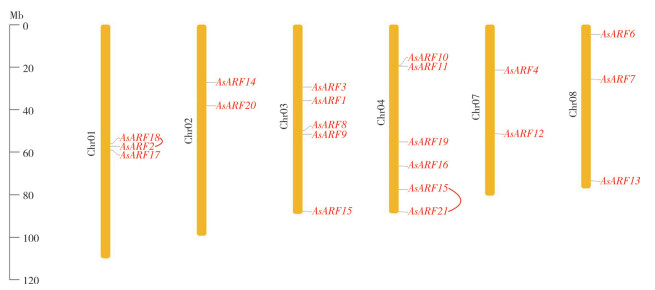
|
| AsARF基因的重复同源对用红线相连 Duplicated paralogous pairs of AsARF genes are connected with red lines 图 4 AsARF基因的染色体分布和片段复制事件 Fig. 4 Chromosomal distribution and segmental duplication events of AsARF genes |
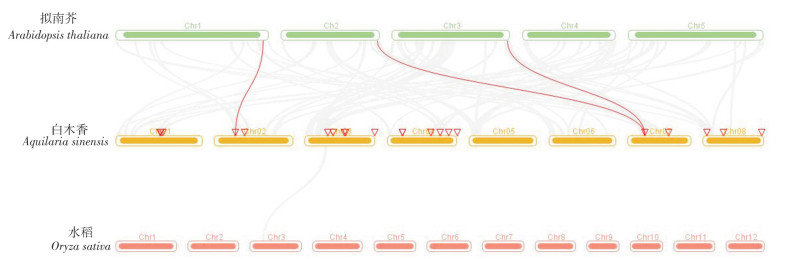
|
| 背景中的灰线表示白木香和其他植物基因组中的共线模块,而红线则表示共线ARF基因对 Gray lines in the background indicate collinear blocks within Aquilaria sinensis and other plant genomes, while red lines highlight collinear ARF gene pairs 图 5 拟南芥、水稻和白木香的ARF基因共线性分析 Fig. 5 Collinearity analysis on the ARF genes of Arabidopsis thaliana, Oryza sativa and Aquilaria sinensis |
2.6 AsARF基因在机械损伤下的表达分析
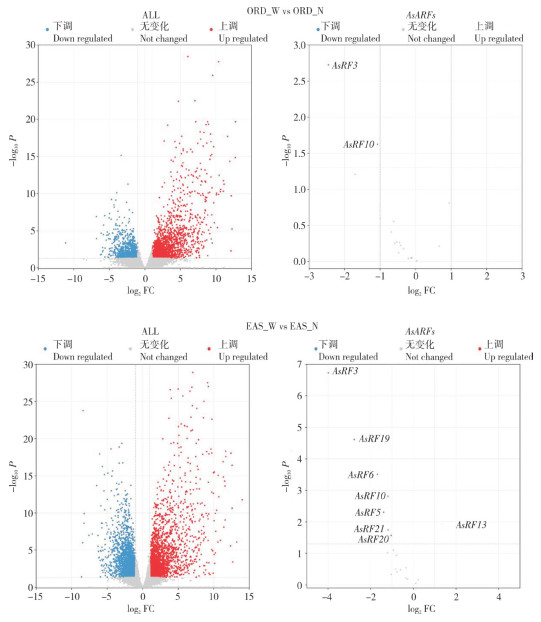
利用Zhang等[41]在NCBI SRA数据库上传的转录组数据对普通白木香和易结香白木香2种品系在机械损伤因素下的AsARF家族基因表达进行分析(图 6、图 7)。未经机械损伤的白木香,其大部分AsARF的表达量比机械损伤下的高。在普通白木香中,仅AsARF3和AsARF10在机械损伤条件下表达显著下降。而在易结香白木香中,AsARF3、AsARF10、AsARF5、AsARF6、AsARF19、AsARF20和AsARF21的表达水平在机械损伤下均显著降低,同时AsARF13的表达量显著上升。推测AsARF5、AsARF6、AsARF13、AsARF19、AsARF20和AsARF21可能参与了易结香白木香品系中结香过程的调控。
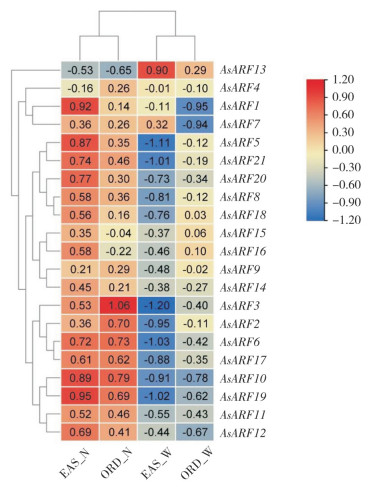
|
| EAS_N:未处理的易结香白木香;EAS_W:机械损伤方式处理的易结香白木香;ORD_N:未处理的普通白木香;ORD_W:机械损伤方式处理的普通白木香 EAS_N: Easier-induced A. sinensis with normal treatment; EAS_W: Easier induced A. sinensis with mechanical damage treatment; ORD_N: Ordinary A. sinensis with normal treatment; ORD_W: Ordinary A. sinensis with mechanical damage treatment 图 6 白木香中ARF基因家族的表达谱 Fig. 6 Expression profile of ARF gene family in Aquilaria sinensis |
|
| 图 7 利用RNASeq进行白木香ARF基因差异表达分析 Fig. 7 Differential expression analysis of ARF genes in Aquilaria sinensis by using RNA-seq |
3 讨论
随着下一代测序和生物信息技术的迅速发展,功能基因挖掘和定位已成为研究热点,并为相关基因家族的研究提供了极大便利。目前,鉴定植物基因家族主要有全基因组水平和转录组水平鉴定这2种方法。全基因组水平的鉴定方法已应用于许多物种,如拟南芥[26]、大豆[42]、杨树[43]、铁皮石斛[44]等。而基于转录组水平的鉴定方法已应用于玉米[45]、剑麻[46]、百香果[47]、荔枝[48]等物种。ARF基因家族已经被证明参与植物的生理生化进程,包括花果发育、器官形态形成、激素调节和环境应激反应等[49-51]。本研究通过全基因组水平的鉴定方法,鉴定出21个AsARF基因,系统进化树分析将这21个AsARF的蛋白分为5个类群(Ⅰ~Ⅴ),其中每个类群都包含了拟南芥和水稻的ARF蛋白。然而,根据ML树分支上的自展值,白木香在进化关系上更接近于拟南芥而不是水稻。此外,通过对白木香、拟南芥和水稻ARF家族基因的共线性分析,也得出了相同结论。
基因结构和基序可以为基因家族的进化关系提供重要依据[52-53]。在基因扩张、重排和功能多样化方面,串联复制、片段复制和全基因组复制起着重要作用[54]。已有研究报道大麦HvARF家族基因通过片段复制进行扩增[55],蒺藜苜蓿MtARF家族基因通过串联复制和片段复制进行扩增[56],而苹果MdARF家族基因的扩增方式为串联复制、片段复制和全基因组复制3种方式[57]。本研究发现,同一分支中的AsARF基因具有相似的基因结构和基序,AsARF基因家族通过片段复制方式进行扩张,而不存在其他扩增方式。为判断AsARF家族基因是否受到环境压力选择的影响,对共线性分析得到的2对片段重复基因的Ka/Ks值进行计算分析。一般来说,Ka/Ks > 1表示基因发生非同义突变后作为优势基因保留下来,受到正向选择的影响;而Ka/Ks<1表示基因发生非同义突变后作为劣势基因被淘汰,受到纯化选择的影响[58-59]。本研究发现,2对重复基因的Ka/Ks均<1,表明环境压力对这些基因的影响产生了纯化选择效应,说明该家族在系统进化过程中具有较强的保守性。
在调节植物生长发育和逆境胁迫方面,启动子的顺式作用元件起着重要的作用。当白木香受到机械损伤胁迫时,其生长素含量降低、光合机构如光系统Ⅱ(PS Ⅱ)受到伤害、光化学活性降低,从而导致光抑制[60]。本研究发现,AsARF3、AsARF5、AsARF6、AsARF10、AsARF19、AsARF20、AsARF21基因的启动子序列中含有大量的光响应元件,这可能是这些基因表达量显著下降的原因。机械损伤作为一种刺激因子,引发了白木香的防御反应,导致白木香产生次生代谢产物,并与细胞的其他组分组合形成导管填充物,使次生木质部导管堵塞,最终形成沉香来抵御进一步伤害。关于白木香结香过程的防御反应已有大量研究,但对与其生长相关的内源激素生长素的生理特性研究较少。白木香结香过程是一个防御与生长的动态平衡过程。植物的防御反应依赖于多种信号通路的复杂相互作用,每种激素启动一个特定的信号途径,这些不同的激素途径在一个复杂的网络中相互协同、拮抗和作用[61]。易结香的白木香在受到机械损伤时,防御反应被激活,同时负责植物生长的生长素含量也降低,对应的生长素响应因子的表达量也下降。相比之下,AsARF13的表达量上升,意味着白木香的防御反应占主导地位。而普通白木香在受到机械损伤时,只有AsARF3和AsARF10的表达量下降,说明防御反应相对生长来说没有占主导地位,这也是导致其结香速度慢、产量低的原因之一。
4 结论本研究鉴定了21个AsARF基因,可分为5个类群,同一类群的基因具有相似的基因结构和蛋白保守基序,而不同类群的AsARF基因在外显子和内含子的数量上存在显著差异;21个基因的蛋白序列平均长度约787个氨基酸且均属于亲水性蛋白;在顺式作用元件分析中发现AsARF基因启动子区域含有大量与光、厌氧和赤霉素等响应元件有关的序列;在基因复制和共线性分析中可知,片段复制事件对于AsARF基因家族的进化和扩张起着重要作用,与水稻ARF基因相比,白木香和拟南芥的ARF基因更为密切相关;此外,通过RNA-seq分析发现AsARF5、AsARF6、AsARF19、AsARF20和AsARF21基因参与白木香结香过程中防御与生长的动态平衡。
| [1] |
YAMAUCHI T, TANAKA A, INAHASHI H, NISHIZAWA N K, TSUTSUMI N, INUKAI Y, NAKAZONO M. Fine control of aerenchyma and lateral root development through AUX/IAA-and ARF-dependent auxin signaling[J]. Proceedings of the National Academy of Sciences, 2019, 116(41): 20770-20775. DOI:10.1073/pnas.1907181116 |
| [2] |
MO Z H, ZHANG Y, HU L J, ZHAI M, XUAN J P. Genome-wide identification and expression analysis of auxin response factor (ARF) gene family in pecan indicates its possible roles during graft union formation[J]. Scientia Horticulturae, 2023, 322: 112401. DOI:10.1016/J.SCIENTA.2023.112401 |
| [3] |
QI Y N, WANG L M, LI W J, DANG Z, XIE Y P, ZHAO W, ZHAO L R, LI W, YANG C X, XU C M, ZHANG J P. Genome-wide identification and expression analysis of auxin response factor gene family in Linum usitatissimum[J]. International Journal of Molecular Sciences, 2023, 24(13): 11006. DOI:10.3390/IJMS241311006 |
| [4] |
MEI M, AI W F, LIU L, XU X, LU X J. Genome-wide identification of the auxin response factor (ARF) gene family in Magnolia sieboldii and functional analysis of MsARF5[J]. Frontiers in Plant Science, 2022, 13: 958816. DOI:10.3389/FPLS.2022.958816 |
| [5] |
TIWARI S B, HAGEN G, GUILFOYLE T. The roles of auxin response factor domains in auxin-responsive transcription[J]. The Plant Cell, 2003, 15: 533-543. DOI:10.1105/tpc.008417 |
| [6] |
罗永坚, 葛蓉, 刘军, 李清. 小豆AUX/IAA基因家族全基因组鉴定及表达分析[J]. 广东农业科学, 2022, 49(11): 170-182. DOI:10.16768/j.issn.1004-874X.2022.11.018 LUO Y J, GE R, LIU J, LI Q. Genome-wide identification and expression analysis of AUX/IAA gene family members in Vigna angularis[J]. Guangdong Agricultural Sciences, 2022, 49(11): 170-182. DOI:10.16768/j.issn.1004-874X.2022.11.018 |
| [7] |
GARCIA-GONZALEZ J, KEBRLOVA S, SEMERAK M, LACEK J, KOTANNAL B I, PETRASEK J, SCHWARZEROVA K. Arp2/3 complex is required for auxin-driven cell expansion through regulation of auxin transporter homeostasis[J]. Frontiers in Plant Science, 2020, 11: 486. DOI:10.3389/fpls.2020.00486 |
| [8] |
HAGEN G, GUILFOYLE T. Auxin-responsive gene expression: Genes, promoters and regulatory factors[J]. Plant Molecular Biology, 2002, 49: 373-385. DOI:10.1023/A:1015207114117 |
| [9] |
FREIRE-RIOS A, TANAK A K, CRESPO I, VAN D W E, SIZENTSOVA Y, LEVITSKY V, LINDHOUD S, FONTANA M, HOHLBEIN J, BOER D R, MIRONOVA V, WEIJERS D. Architecture of DNA elements mediating ARF transcription factor binding and auxin-responsive gene expression in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2020, 117(39): 24557-24566. DOI:10.1073/pnas.2009554117 |
| [10] |
MA Y M, RAN J D, LI G Q, WANG M C, YANG C M, WEN X, GENG X, ZHANG L P, LI Y, ZHANG Z. Revealing the roles of the JAZ family in defense signaling and the agarwood formation process in Aquilaria sinensis[J]. International Journal of Molecular Sciences, 2023, 24(12): 9872. DOI:10.3390/IJMS24129872 |
| [11] |
XU J R, DU R Y, CHEN J H. RNA-sequencing reveals the involvement of sesquiterpene biosynthesis genes and transcription factors during an early response to mechanical wounding of Aquilaria sinensis[J]. Genes, 2023, 14(2): 464. DOI:10.3390/GENES14020464 |
| [12] |
Editorial Committee of Flora of China. Flora of China[DB]. Beijing: Science Press, 2018.
|
| [13] |
LIU Y Y, WEI J H, GAO Z H, ZHANG Z, LYU J C. A review of quality assessment and grading for agarwood[J]. Chinese Herbal Medicines, 2017, 9(1): 22-30. DOI:10.1016/S1674-638417.60072-8 |
| [14] |
张立宝, 袁瑞玲, 曹达虎, 陈鹏. 白木香结香机理及人工结香技术[J/OL]. 林业科技通讯, [2024-05-24]. DOI: 10.13456/j.cnki.lykt.2023.05.15.0002. ZHANG L B, YUAN R L, CAO D H, CHEN P. Mechanism of agarwood fragrance and artificial fragrance conjugation technology[J/OL]. Forest Science and Technology, [2024-05-24]. DOI: 10.13456/j.cnki.lykt.2023.05.15.0002. |
| [15] |
林婉婷, 刘亮锋, 李波, 黄曦, 梁玉龙. 沉香品质分级及关联因素研究进展[J]. 中国现代中药, 2023, 25(10): 2246-2254. DOI:10.13313/j.issn.1673-4890.20221128005 LIN W T, LIU L F, LI B, HUANG X, LIANG Y L. Progress of research on quality grading and correlation factors of agarwood[J]. Modern Chinese Medicine, 2023, 25(10): 2246-2254. DOI:10.13313/j.issn.1673-4890.20221128005 |
| [16] |
ABDUL K F A, AZIZAN K A, OTHMAN R. Transcriptome of Aquilaria malaccensis containing agarwood formed naturally and induced artificially[J]. BMC Research Notes, 2021, 14(1): 117-121. DOI:10.1186/S13104-021-05532-9 |
| [17] |
邓加艾, 戴好富, 王宇光, 陈慧琴, 谭志琼, 梅文莉. 沉香样品中曲霉属真菌菌株HNWSW-20的分离鉴定及其次生代谢产物的研究[J]. 热带作物学报, 2018, 39(8): 1618-1624. DOI:10.3969/j.issn.1000-2561.2018.08.022 DENG J A, DAI H F, WANG Y G, CHEN H Q, TAN Z Q, MEI W L. Isolation and identification of the Fungus aspergillus sp. HNWSW-20 from Chinese agarwood and its secondary metabolites[J]. Chinese Journal of Tropical Crops, 2018, 39(8): 1618-1624. DOI:10.3969/j.issn.1000-2561.2018.08.022 |
| [18] |
LI W, CHEN H Q, WANG H, MEI W L, DAI H F. Natural products in agarwood and Aquilaria plants: Chemistry, biological activities and biosynthesis[J]. Natural Product Reports, 2021, 38(3): 528-565. DOI:10.1039/d0np00042f |
| [19] |
王军, 梅文莉, 王昊, 王雅丽, 段瑞军, 黄圣卓, 戴好富. 马来西亚产小果沉香所结沉香的GC-MS分析[J]. 热带作物学报, 2021, 42(8): 2377-2386. DOI:10.3969/j.issn.1000-2561.2021.08.034 WA NG J, M E I W L, WA NG H, WA NG Y L, DUA N R J, HUANG S Z, DAI H F. GC-MS analysis of agar wood from Aquilaria microcarpa in Malaysia[J]. Chinese Journal of Tropical Crops, 2021, 42(8): 2377-2386. DOI:10.3969/j.issn.1000-2561.2021.08.034 |
| [20] |
ZHUANG Z L, WU S H, DENG S M, ZHU P J, ZHOU X, CHEN X Y, ZHANG W M, GAO X X. A study on the quality evaluation of agarwood of Aquilaria sinensis and Aquilaria malaccensis induced by different inducers based on gray correlation degree and TOPSIS[J]. Pharmacognosy Magazine, 2022, 79(18): 713-728. DOI:10.4103/PM.PM_340_21 |
| [21] |
ZHANG Y, MENG H, LYU F F, FAN X H, LIU P W, HE X, HUANG Y, CHEN B, YANG Y, WEI J H. Temporal characteristics of agarwood formation in Aquilaria sinensis after applying whole-tree agarwoodinducing technique[J]. Chinese Herbal Medicines, 2023, 15(1): 37-44. DOI:10.1016/J.CHMED.2022.07.003 |
| [22] |
XU Y H, LIAO Y C, ZHANG Z, LIU J, SUN P W, GAO Z H, SUI C, WEI J H. Jasmonic acid is a crucial signal transducer in heat shock induced sesquiterpene formation in Aquilaria sinensis[J]. Scientifi c Reports, 2016, 6(1): 1-9. DOI:10.1038/srep21843 |
| [23] |
LIU J, XU Y H, ZHANG Z, WEI J H. Hydrogen peroxide promotes programmed cell death and salicylic acid accumulation during the induced production of sesquiterpenes in cultured cell suspensions of Aquilaria sinensis[J]. Functional Plant Biology, 2014, 42(4): 337-346. DOI:10.1071/FP14189 |
| [24] |
LI S B, XIE Z Z, HU C G, ZHANG J Z. A review of auxin response factors (ARFs) in plants[J]. Frontiers in Plant Science, 2016, 7: 47. DOI:10.3389/fpls.2016.00047 |
| [25] |
XU S Z, SUN M Y, YAO J L, LIU X X, XUE Y S, YANG G Y, ZHU R X, JIANG W T, WANG R Z, XUE C, MAO Z Q, WU J. Auxin inhibits lignin and cellulose biosynthesis in stone cells of pear fruit via the PbrARF13-PbrNSC-PbrMYB132 transcriptional regulatory cascade[J]. Plant Biotechnology Journal, 2023, 21(7): 1408-1425. DOI:10.1111/PBI.14046 |
| [26] |
OKUSHIMA Y, OVERVOORDE P J, ARIMA K, ALONSO J M, CHAN A, CHANG C, ECKER J R, HUGHES B, LUI A, NGUYEN D, ONODERA C, QUACH H, SMITH A, YU G, THEOLOGIS A. Functional genomic analysis of the auxin response factor gene family members in Arabidopsis thaliana: Unique and overlapping functions of ARF7 and ARF19[J]. The Plant Cell, 2005, 17(2): 444-463. DOI:10.1105/tpc.104.028316 |
| [27] |
MA F Y, ZHANG F, ZHU Y, LAN D Y, YAN P W, WANG Y, HU Z J, ZHANG X W, HU J, NIU F, LIU M Y, HE S C, CUI J H, YUAN X Y, YAN Y, WU S J, CAO L M, BIAN H W, YANG J S, LI Z K, LUO X J. Auxin signaling module OsSK41-OsIAA10-OsARF regulates grain yield traits in rice[J]. Journal of Integrative Plant Biology, 2023, 65(7): 1753-1766. DOI:10.1111/JIPB.13484 |
| [28] |
BI JAYALAXMI M. Genomic architecture of promoters and transcriptional regulation of candidate genes in rice involved in tolerance to anaerobic germination[J]. Current Plant Biology, 2022, 29: 100236. DOI:10.1016/J.CPB.2022.100236 |
| [29] |
李文兰, 李文才, 孙琦, 于彦丽, 赵勐, 鲁守平, 李艳娇, 孟昭东. 玉米生长素响应因子家族基因的表达模式分析[J]. 作物学报, 2021, 47(6): 1138-1148. DOI:10.3724/SP.J.1006.2021.03043 LI W L, LI W C, SUN Q, YU Y L, ZHAO M, LU S P, LI Y J, MENG Z D. A study of expression pattern of auxin response factor family genes in maize (Zea mays L.)[J]. Acta Agronomica Sinica, 2021, 47(6): 1138-1148. DOI:10.3724/SP.J.1006.2021.03043 |
| [30] |
SINGH G S, MANISH T. Frontline warrior microRNA167: A battle of survival[J]. International Journal of Plant Biology, 2022, 13(4): 598-600. DOI:10.3390/IJPB13040047 |
| [31] |
TOMBULOGLU H. Genome-wide analysis of the auxin response factors (ARF) gene family in barley (Hordeum vulgare L.)[J]. Journal of Plant Biochemistry and Biotechnology, 2019, 28: 14-24. DOI:10.1007/s13562-018-0458-6 |
| [32] |
GAMBHIR P, SINGH V, PARIDA A, RAGHUVANSHI U, KUMAR R, SHARMA A K. Ethylene response factor ERF.D7 activates auxin response factor 2 paralogs to regulate tomato fruit ripening[J]. Plant Physiology, 2022, 190(4): 2775-2796. DOI:10.1093/PLPHYS/KIAC441 |
| [33] |
DING X P, MEI W L, LIN Q, WANG H, WANG J, PENG S Q, LI H L, ZHU J H, LI W, WANG P, CHEN H Q, DONG W H, GUO D, CAI C H, HUANG S Z, CUI P, DAI H F. Genome sequence of the agarwood tree Aquilaria sinensis (Lour.) Spreng: The first chromosome-level draft genome in the Thymelaeceae family[J]. Gigascience, 2020, 9(3): 13-18. DOI:10.1093/gigascience/giaa013 |
| [34] |
ZHANG P, LI X F, CUI Z Y, XU D P. Morphological, physiological, biochemical and molecular analyses reveal wounding-induced agarwood formation mechanism in two types of Aquilaria sinensis (Lour.) Spreng[J]. Idustrial Crops and Products, 2022, 178(2): 114603. DOI:10.1016/j.indcrop.2022.114603 |
| [35] |
CHEN C J, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y H, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [36] |
谢炳春, 黄俊霖, 温松森, 李涛, 李颖, 徐小万, 徐晓美, 吴智明, 周衡. 中国辣椒BCAT基因家族鉴定、表达分析及克隆[J]. 广东农业科学, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 XIE B C, HUANG J L, WEN S S, LI T, LI Y, XU X W, XU X M, WU Z M, ZHOU H. Identification, expression analysis and cloning of Capsicum chinense BCAT gene family[J]. Guangdong Agricultural Sciences, 2022, 49(12): 34-43. DOI:10.16768/j.issn.1004-874X.2022.12.004 |
| [37] |
WILKINS M R, GASTEIGER E, BAIROCH A, SANCHEZ J C, WILLIAMS K L, APPEL R D, HOCHSTRASSER D F. Protein identification and analysis tools in the ExPASy server[J]. Methods Molecular Biology, 1999, 112: 531-552. DOI:10.1385/1-59259-584-7:531 |
| [38] |
KUMAR S, STECHER G, LI M, KNYAZ C, TAMURA K, MEGA X. Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547. DOI:10.1093/molbev/msy096 |
| [39] |
谭建彬, 徐进, 师沛琼, 周鸿凯. 小菜蛾清道夫受体基因家族鉴定与生物信息学分析[J]. 广东农业科学, 2023, 50(5): 94-102. DOI:10.16768/j.issn.1004-874X.2023.05.011 TAN J B, XU J, SHI P Q, ZHOU H K. Genome-wide identification and bioinformatics analysis of scavenger receptor gene family in Plutella xylostella[J]. Guangdong Agricultural Sciences, 2023, 50(5): 94-102. DOI:10.16768/j.issn.1004-874X.2023.05.011 |
| [40] |
BAILEY T L, BODEN M, BUSKE F A, FRITH M, GRANT C E, CLEMENTI L, REN J, LI W W, NOBLE W S. MEME SUITE: Tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37: 202-208. DOI:10.1093/nar/gkp335 |
| [41] |
LESCOT M, DÉHAIS P, THIJS G, MARCHAL K, MOREAU Y, VAN DE PEER Y, ROUZÉ P, ROMBAUTS S. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327. DOI:10.1093/nar/30.1.325 |
| [42] |
HA C V, LE D T, NISHIYAMA R, WATANABE Y, SULIEMAN S, TRAN U T, MOCHIDA K, DONG N V, YAMAGUCHI-SHINOZAKI K, SHINOZAKI K, TRAN L S. The auxin response factor transcription factor family in soybean: Genome-wide identification and expression analyses during development and water stress[J]. DNA Research, 2013, 20(5): 511-524. DOI:10.1093/dnares/dst027 |
| [43] |
KALLURI U C, DIFAZIO S P, BRUNNER A M, TUSKAN G A. Genome-wide analysis of Aux/IAA and ARF gene families in Populus trichocarpa[J]. BMC Plant Biology, 2007, 7: 1-14. DOI:10.1186/1471-2229-7-59 |
| [44] |
CHEN Z H, YUAN Y, FU D, SHEN C J, YANG Y J. Identification and expression profiling of the auxin response factors in Dendrobium officinale under abiotic stresses[J]. International Journal of Molecular Sciences, 2017, 18(5): 927. DOI:10.3390/ijms18050927 |
| [45] |
DU H, FENG B R, YANG S S, HUANG Y B, TANG Y X. The R2R3-MYB transcription factor gene family in maize[J]. PloS One, 2012, 7: e37463. DOI:10.1371/journal.pone.0037463 |
| [46] |
鹿志伟, 侯晓婉, 杨子平, 张燕梅, 李俊峰, 周文钊. 基于转录组的剑麻ARF基因家族的鉴定及表达分析[J]. 热带作物学报, 2019, 40(10): 1972-1978. DOI:10.3969/j.issn.1000-2561.2019.10.09 LU Z W, HOU X W, YANG Z P, ZHANG Y M, LI J F, ZHOU W Z. Transcriptome-wide identification and expression profiling of the auxin response factor (ARF) gene family in Agave.hybrid No.11648[J]. Chinese Journal of Tropical Crops, 2019, 40(10): 1972-1978. DOI:10.3969/j.issn.1000-2561.2019.10.09 |
| [47] |
张小雪, 韦晓霞, 陈发兴. 基于转录组的百香果ARF基因家族的鉴定及表达分析[J]. 福建农林大学学报(自然科学版), 2021, 50(2): 236-243. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2021.02.013 ZHANG X X, WEI X X, CHEN F X. Transcriptome data-based identification and expression profiling of the auxin response factor (ARF) gene family in Passiflora edulia Sims.[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition), 2021, 50(2): 236-243. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2021.02.013 |
| [48] |
董晨, 魏永赞, 王弋, 郑雪文, 李伟才. 基于转录组的荔枝ARF基因家族的鉴定及表达分析[J]. 热带作物学报, 2017, 38(8): 1485-1491. DOI:10.3969/j.issn.1000-2561.2017.08.018 DONG C, WEI Y Z, WANG Y, ZHENG X W, LI W C. Transcriptomewide identification and expression profiling of the auxin response factor (ARF) gene family in Litchi chinensis Sonn.[J]. Chinese Journal of Tropical Crops, 2017, 38(8): 1485-1491. DOI:10.3969/j.issn.1000-2561.2017.08.018 |
| [49] |
DHARMASIRI N, ESTELLE M. Auxin signaling and regulated protein degradation[J]. Trends in Plant Science, 2004, 9(6): 302-308. DOI:10.1016/j.tplants.2004.04.003 |
| [50] |
LI Y L, GAO Z H, SONG J, WANG W X, SHI T. Auxin response factor (ARF) and its functions in plant growth and development[J]. Plant Physiology Journal, 2017, 53(10): 1842-1858. DOI:10.13592/j.cnki.ppj.2017.0271 |
| [51] |
ABEL S, THEOLOGIS A. Early genes and auxin action[J]. Plant Physiology, 1996, 111(1): 9. DOI:10.1104/pp.111.1.9 |
| [52] |
BOUDET N, AUBOURG S, TOFFANO-NIOCHE C, KREIS M, LECHARNY A. Evolution of intron/exon structure of DEAD helicase family genes in Arabidopsis, Caenorhabditis and Drosophila[J]. Genome Research, 2001, 11: 2101-2114. DOI:10.1101/gr.200801 |
| [53] |
BABENKO V N, ROGOZIN I B, MEKHEDOV S L, KOONIN E V. Prevalence of intron gain over intron loss in the evolution of paralogous gene families[J]. Nucleic Acids Research, 2004, 32: 3724-3733. DOI:10.1093/nar/gkh686 |
| [54] |
FLAGEL L E, WENDEL J F. Gene duplication and evolutionary novelty in plants[J]. New Phytologist, 2009, 183(3): 557-564. DOI:10.1111/j.1469-8137.2009.02923.x |
| [55] |
郭宝健, 李赢, 袁泽宸, 吕超, 张新忠, 许如根. 大麦ARF基因家族的全基因组分析[J]. 麦类作物学报, 2016, 36(11): 1426-1432. DOI:10.7606/j.issn.1009-1041.2016.11.03 GUO B J, LI Y, YUAN Z C, LYU C, ZHANG X Z, XU R G. Genomewide analysis of auxin response factor (ARF) family in barley[J]. Journal of Triticeae Crops, 2016, 36(11): 1426-1432. DOI:10.7606/j.issn.1009-1041.2016.11.03 |
| [56] |
SHEN C J, YUE R Q, SUN T, ZHANG L, XU L Q, TIE S G, WANG H Z, YANG Y J. Genome-wide identification and expression analysis of auxin response factor gene family in Medicago truncatula[J]. Frontiers in Plant Science, 2015, 6: 73. DOI:10.3389/fpls.2015.00073 |
| [57] |
LI H F, RAN K, HE P, RAN K, WANG H B, CHANG Y S, SUN Q R, CHEN L L, LI L G. Genome-wide identification and expression analysis of auxin response factor (ARF) gene gamily in apple[J]. Plant Physiology Journal, 2015, 51(7): 1045-1054. DOI:10.13592/j.cnki.ppj.2015.0173 |
| [58] |
YANG Z, BIELAWSKI J P. Statistical methods for detecting molecular adaptation[J]. Trends in Ecology and Evolution, 2000, 15(12): 496-503. DOI:10.1016/s0169-5347(00)01994-7 |
| [59] |
HURST L D. The Ka/Ks ratio: Diagnosing the form of sequence evolution[J]. Trends in Genetics, 2002, 18(9): 486. DOI:10.1016/s0168-9525(02)02722-1 |
| [60] |
张鹏, 薛世玉, 李小飞, 徐大平, 崔之益. 白木香响应真菌侵染与机械损伤胁迫的生理机制[J]. 林业科学研究, 2022, 35(3): 1-8. DOI:10.13275/j.cnki.lykxyj.2022.03.006 ZHANG P, XUE S Y, LI X F, XU D P, CUI Z Y. Physiological mechanisms in response to fungal infestation and mechanical injury stress in Aquilaria sinensis (Lour.) Gilg[J]. Forest Research, 2022, 35(3): 1-8. DOI:10.13275/j.cnki.lykxyj.2022.03.006 |
| [61] |
AERTS N, PEREIRA MENDES M, VAN WEES S C M. Multiple levels of crosstalk in hormone networks regulating plant defense[J]. The Plant Journal, 2021, 105(2): 489-504. DOI:10.1111/tpj.15124 |
(责任编辑 陈丽娥)
 2024, Vol. 51
2024, Vol. 51