文章信息
基金项目
- 中国热带农业科学院基本科研业务费专项资金(1630102017002)
作者简介
- 徐磊(1984—),男,硕士,助理研究员,研究方向为热带旱作遗传改良,E-mail:xulei00100@163.com.
通讯作者
- 刘洋(1980—),男,博士,副研究员,研究方向为热带旱作遗传改良,E-mail:lyfull@163.com.
文章历史
- 收稿日期:2020-10-27
【研究意义】高粱(Sorghum bicolor)是禾本科高粱属1年生草本植物,不仅是世界上重要的粮食和能源作物,也是优质的饲料作物。高粱具有耐旱、耐涝、耐盐碱等多种特性[1],其基因组较小(约750 Mb),具有丰富的遗传多样性,其优良的基因资源在作物改良中具有广阔的应用前景[2]。转录因子又称反式作用因子,对转录过程起诱导或抑制作用[3]。WRKY转录因子作为植物中一类重要转录因子,广泛参与植物生长发育、响应激素诱导和生物及非生物胁迫等生物学进程[4],开展高粱WRKY基因耐旱性表达特征分析具有重要意义,可为高粱WRKY基因的功能研究奠定基础。【前人研究进展】 WRKY基因家族作为高等植物中备受关注的基因家族之一,自第一个WRKY转录因子基因从甘薯中克隆以来[5],随着许多物种基因组测序工作完成,越来越多的WRKY基因从基因组水平上鉴定出来,已在青稞[6]、中粒咖啡[7]、大麦[8]和杨树[9]基因组分别鉴定了41、49、98、122个WRKY基因。WRKY转录因子包含1~2个保守WRKY结构域,结构域由约60个氨基酸序列组成,其保守的7个核心氨基酸序列为“WRKYGQK”。根据WRKY保守结构域数目和锌指结构类型,可将WRKY转录因子分为组Ⅰ、组Ⅱ和组Ⅲ三大类,其中组Ⅱ又分为5个亚类(Ⅱ a~ Ⅱ e)。组Ⅰ WRKY转录因子包含2个WRKY结构域,其锌指结构的氨基酸类型为C2H2,组Ⅱ和组Ⅲ转录因子只含有1个WRKY结构域,锌指类型分别为C2H2、C2HC型[10]。据报道,WRKY转录因子参与了植物生长发育的许多进程,如开花[11]、次级代谢物合成[12]和衰老[13]等。也有较多研究表明,WRKY转录因子响应各种压力胁迫,如玉米ZmWRKY62-like基因响应盐和干旱胁迫[14];小麦TaWRKY33受盐胁迫诱导表达,TaWRKY33转基因拟南芥比对照具有更好的耐盐性[15];甜高粱SSWRKY28、SSWRKY76基因的表达结果表明,它们可能在应答干旱胁迫时发挥一定作用[16];高粱SbWRKY30基因通过影响拟南芥和水稻的根系结构来提高对干旱胁迫的耐受性,拟南芥和水稻的转基因植株在干旱胁迫后脯氨酸含量、SOD、POD和CAT酶活性高于野生型植株,而MDA含量低于野生型植株[17]。【本研究切入点】前人研究结果表明,组Ⅲ WRKY成员广泛参与植物生长发育进程、生物和非生物胁迫应答反应[18]。赵兴奎等[19]已从高粱基因组鉴定了16个组Ⅲ WRKY成员,但没有研究其响应逆境胁迫的表达谱。【拟解决的关键问题】本研究以高粱品种BTx623为试材,采用PEG6000溶液模拟干旱胁迫[20-21],研究高粱组Ⅲ WRKY成员响应干旱胁迫的表达谱,为挖掘耐旱候选基因提供参考。
1 材料与方法 1.1 试验材料供试高粱品种为BTx623,由广东省旱作节水农业工程技术研究中心保存,该材料有参考基因组序列,为中等耐旱性材料[22]。高粱组Ⅲ WRKY基因信息来自参考文献[19],基因序列下载自Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)。
主要试剂:TransZol Plant植物总RNA纯化试剂盒、Trans2K® Plus DNA Marker、TransStart® Top Green qPCR SuperMix,购自北京全式金生物技术有限公司;ThermoScientific反转录试剂盒K1622,购自Thermo Fermentas公司。
1.2 试验方法1.2.1 干旱胁迫处理 供试高粱种子用1%(W/ W)NaClO溶液浸泡10 min,然后用灭菌的蒸馏水冲洗干净,采用0.5×Hoagland营养液水培,植株在植物光照培养箱中培养,生长条件为28 ℃、12 h光照、12 h黑暗,光照强度为9 600 lx。营养液每2 d更换1次,待植株生长至3叶期,用含20%(W/W)PEG6000的营养液水培,模拟干旱胁迫处理,于处理后0、1、3、6、12 h收集整株植物,每个样本混合6株植物,每个时间点取3个生物学重复样本。
1.2.2 SbWRKY基因的表达谱分析 从NCBI数据库(https://www.ncbi.nlm.nih.gov)下载有关干旱胁迫的高粱28K芯片表达数据(GSE48205),利用GeneSpring GX 11.5软件(安捷伦科技有限公司)进行分析,获得探针的相对表达倍数,计算log2(fold change)值,然后根据探针对应的基因编号,整理基因的相对表达值。
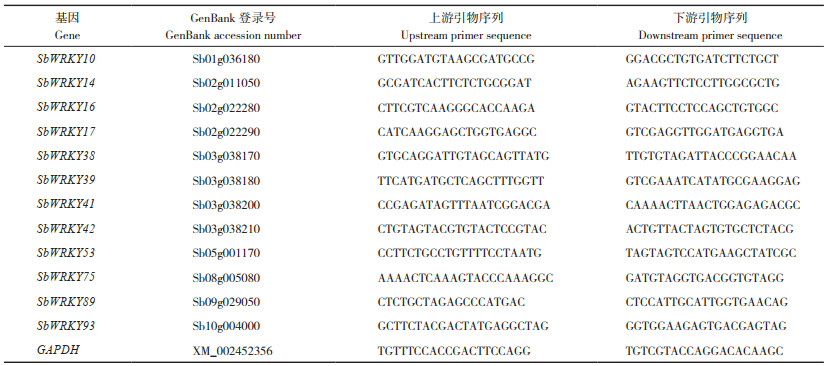
1.2.3 总RNA提取、cDNA合成及荧光定量表达分析 采用植物总RNA纯化试剂盒提取植株总RNA,对总RNA用1.5% 琼脂糖凝胶电泳检测RNA完整性;以总RNA为模板,使用反转录试剂盒获得cDNA,所用Marker为Trans2K® Plus DNA Marker。以cDNA为模板使用qRT-PCR Mix按照操作说明配制反应体系,在LightCycler480 Ⅱ实时荧光定量PCR仪上进行反应,内参基因选用高粱GAPDH基因。PCR反应体系(10 μL):cDNA 1 μL,2×Mix 5 μL,上游引物、下游引物(表 1)各0.25 μL,补ddH2O至10 μL;PCR扩增程序:95 ℃ 3 min;95 ℃ 10 s,60 ℃ 35 s,35个循环。
|
1.2.4 qRT-PCR结果统计分析 按照2-Δ ΔCT法计算基因相对表达值[23]。使用EXCEL的student’s T检验进行差异显著性分析。
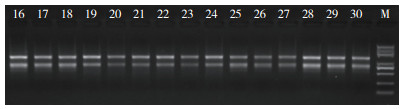
2 结果与分析 2.1 总RNA完整性检测为保证反转录和qRT-PCR试验顺利开展,本研究对提取的15个总RNA样本进行琼脂糖凝胶电泳检测,其完整性较好(图 1),可用于后续试验。
|
| 图 1 15个RNA样本的琼脂糖凝胶电泳检测 Fig. 1 Detection of 15 RNA samples by agarose gel electrophoresis Trans2KR Plus DNA Marker;16~18:0 h样本;19~21:1 h样本;22~24:3 h样本;25~27:6 h样本;28~30:12 h样本 Trans2KR Plus DNA Marker, 16-18: 0 h samples, 19-21: 1 h samples, 22-24: 3 h samples, 25-27: 6 h samples, 28-30: 12 h samples |
2.2 SbWRKY的基因芯片分析结果
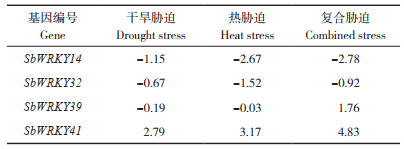
为研究高粱响应干旱胁迫的表达特征,从NCBI数据库下载的高粱28K芯片(GSE48205)包含对照、干旱胁迫、热胁迫和复合胁迫(干旱胁迫和热胁迫共同处理)共4组表达值,将表达值导入GeneSpring GX 11.5软件,经数据过滤、标准化处理、重要性分析和差异分析等流程,最后筛选得到4个组Ⅲ SbWRKY基因的差异表达倍数值(表 2)。由结果可知,SbWRKY14、SbWRKY32下调表达;SbWRKY39在干旱、热胁迫时下调表达,但在复合胁迫时上调表达;SbWRKY41在3种胁迫处理时均上调表达。
|
2.3 SbWRKY基因的qRT-PCR分析结果
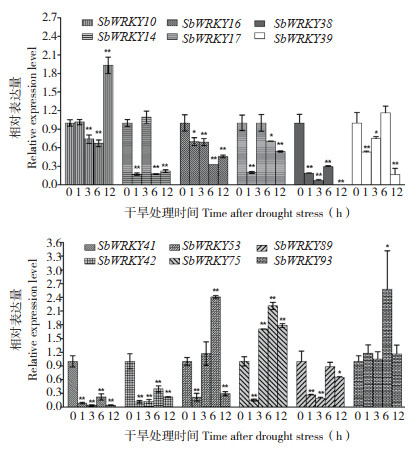
为研究组Ⅲ SbWRKY基因响应干旱胁迫的表达谱,采用PEG6000模拟干旱胁迫于不同时间点取样,通过qRT-PCR分析基因处理与对照之间的相对表达值,在16个组Ⅲ成员中成功获得了12个基因的表达谱(图 2);各基因在干旱胁迫处理后不同时间点与对照(0 h)相比,SbWRKY10在处理后3、6 h下调表达,而在处理后12 h上调表达;SbWRKY14在处理后1、6、12 h下调表达;SbWRKY16、SbWRKY38、SbWRKY41和SbWRKY42在处理后1、3、6、12 h均下调表达;SbWRKY17在处理后1、6、12 h下调表达;SbWRKY39和SbWRKY89在处理后1、3、12 h下调表达;SbWRKY53在处理后1、12 h下调表达,而在6 h上调表达;SbWRKY75在处理后1 h下调表达,而在3、6、12 h上调表达;SbWRKY93在处理后6 h上调表达。
|
| 图 2 干旱胁迫下12个SbWRKY基因的表达谱 Fig. 2 Expression profiles of 12 SbWRKY genes under drought stress |
3 讨论
WRKY转录因子在响应各种非生物胁迫方面已有相关报道,WRKY转录因子在植物对干旱胁迫的响应中起重要作用[24]。在拟南芥中过表达TaWRKY1和TaWRKY33激活了几个与逆境相关的下游基因,提高了拟南芥在各种胁迫下的萌发率,促进了根系的生长[25]。在烟草和梨中过表达PbrWRKY53增强了对干旱胁迫的耐受性。与野生型相比,转基因植株的活性氧生成相对更少、抗氧化酶活性和代谢产物更高。此外,在转基因烟草中过量表达PbrWRKY53导致PbrNCED1的表达水平提高;敲除PbrWRKY53可下调PbrNCED1的表达丰度,同时降低其耐旱性[26]。水稻组Ⅲ WRKY转录因子OsWRKY11在干旱和高温胁迫时诱导表达,以热激蛋白HSP101基因作启动子驱动OsWRKY11表达时,转基因植株耐热性和耐旱性增强,叶片萎蔫变慢,离体叶片失水较慢;结果表明OsWRKY11基因在应对高温和干旱胁迫反应中起重要作用,可能有助于提高植物的抗逆性[27]。
为了解SbWRKY基因在干旱胁迫调控中所起作用,本研究利用公共数据库基因芯片表达谱数据,分析高粱组Ⅲ WRKY基因在干旱胁迫、热胁迫和复合胁迫条件下的表达模式,2个基因(SbWRKY14和SbWRKY32)在3种处理条件下均下调表达,1个基因(SbWRKY41)在3种处理条件下上调表达。基因的表达趋势通常反映其相应的功能,在雷蒙德棉中,干旱胁迫处理后,有16个基因(其中包含3个组Ⅲ成员)诱导表达,15个基因表达量减少[28];本研究通过qRTPCR技术检测出12个基因的表达谱,其中4个基因(SbWRKY10、SbWRKY53、SbWRKY75和SbWRKY93)在1个或多个时间点诱导表达,而4个基因(SbWRKY16、SbWRKY38、SbWRKY41和SbWRKY42)在所有时间点均抑制表达。这些结果表明,不同的SbWRKY基因在应对干旱胁迫时可能起不同的调控作用。SbWRKY41的qRT-PCR检测结果与基因芯片检测的表达谱呈相反表达趋势,可能是由于两个研究采用的材料、干旱处理方法不同所致:本研究所用试材为BTx623,以20% PEG6000模拟干旱胁迫;而芯片试验所用试材为R16,采用自然干旱胁迫处理[29]。
此外,WRKY转录因子广泛参与了植物生长发育、各种生物及非生物胁迫进程,如在水稻中已验证了16个OsWRKY转录因子的功能,结果表现为Ⅰ类参与抗病进程,Ⅱ b类具有抗病和抗逆功能,Ⅱ d类调控植物的营养生长,而Ⅲ类具有调控营养生长和抗逆等功能[30]。本研究只开展了Ⅲ类基因在干旱胁迫条件下的表达谱分析,这些成员还可能响应低温、高温和高盐等胁迫反应并发挥一定作用。为深入了解这些基因的功能,后续还需克隆这些基因,通过转基因实验验证其生物学功能。
4 结论高粱WRKY转录因子基因在应对逆境胁迫中起重要调控作用,本研究在前人研究基础上挑选16个高粱组Ⅲ WRKY转录因子基因开展进一步研究。通过基因芯片分析结果表明,SbWRKY14和SbWRKY32在3种处理条件下全部下调表达,表明这2个WRKY基因在干旱胁迫、热胁迫和复合胁迫下具有相似功能;SbWRKY41在3种胁迫条件下诱导表达。为进一步分析该组成员的表达谱,以20% PEG6000模拟干旱胁迫,在0、1、3、6、12 h取样,采用qRT-PCR检测出该组12个基因的表达谱;SbWRKY10、SbWRKY53、SbWRKY75和SbWRKY93在1个或多个时间点诱导表达,而SbWRKY16、SbWRKY38、SbWRKY41和SbWRKY42在所有时间点均抑制表达。通过这两种分析方法,筛选到诱导和抑制表达基因,这为我们今后深入了解SbWRKY基因在干旱胁迫中的调控功能提供参考,也为我们克隆耐旱相关基因提供候选基因。
| [1] |
王芳, 高秋, 王杰, 马金星, 孙娟. 利用SSR标记分析高粱属种质资源的遗传多样性[J]. 草业学报, 2016, 25(5): 125-133. WANG F, GAO Q, WANG J, MA J X, SUN J. Analysis of genetic diversity in sorghum germplasm collections using SSR markers[J]. Acta Prataculturae Sinica, 2016, 25(5): 125-133. |
| [2] |
DUAN Y H, ZOU J Q, LU F. Drought resistance mechanism of sorghum and ensuring seedling technique[J]. Rain Fed Crops, 2009, 29(1): 25-27. |
| [3] |
YANG Z R, WANG X C, LI X M. Advance on the study of transcription fators in higher plants[J]. Hereditas(Beijing), 2004, 26(3): 403-408. DOI:10.1088/1009-0630/6/5/011 |
| [4] |
LI K, ZOU C J. Research Progress in WRKY transcription factors in plants[J]. Plant Physiology Journal, 2014, 50(9): 1329-1335. |
| [5] |
ISHIGURO S, NAKAMURA K. Characterization of a cDNA encoding a novel DNA-binding protein, SPF1, that recognizes SP8 sequences in the 5'upstream regions of genes coding for sporamin and β-amylase from sweet potato[J]. Molecular Genetics and Genomics, 1994, 244(6): 563-571. DOI:10.1007/BF00282746 |
| [6] |
蒋礼玲, 陈丽华, 尼玛扎西, 马晓岗, 黄胜雄, 李高原. 基于转录组测序数据的青稞WRKY基因家族的生物信息学分析[J/OL]. 分子植物育种, 2020. https://kns.cnki.net/kcms/detail/46.1068.S.20200814.1346.004.html.2020-08-14. JIANG L L, CHEN L H, NI M Z X, MA X G, HUANG S X, LI G Y. Bioinformatics analyses of WRKY family members in Hordeum vulgare L. var. nudum based on RNA-seq data[J]. Molecular Plant Breeding, 2020. https://kns.cnki.net/kcms/detail/46.1068.S.20200814.1346.004.html.2020-08-14. |
| [7] |
付春, 唐雪, 杨瑶君, 江纳. 中粒咖啡W R K Y基因家族的生物信息学分析[J/OL]. 广西植物, 2020. https://knscnki.net/kcms/detail/45.1134.Q.20200622.1515.002.html.2020-06-23. DOI:10.11931/guihaia.gxzw202003075. FU C, TANG X, YANG Y J, JIANG N. Bioinformatics analysis of WRKY gene family in Coffee canephora[J/OL]. Guihaia, 2020. https://kns.cnki.net/kcms/detail/45.1134.Q.20200622.1515.002.html. 2020-06-23.DOI:10.11931/guihaia.gxzw202003075. |
| [8] |
毛成志, 王蕾, 孔豆豆, 王寒冬, 张怀刚, 沈裕虎, 徐金青. 大麦全基因组WRKY基因家族生物信息学分析[J]. 分子植物育种, 2019, 17(23): 7623-7633. DOI:10.13271/j.mpb.017.007623 MAO C Z, WANG L, KONG D D, WANG H D, ZHANG H G, SHEN Y H, XU J Q. Bioinformatics analysis of WRKY gene family in barley genome[J]. Molecular Plant Breeding, 2019, 17(23): 7623-7633. DOI:10.13271/j.mpb.017.007623 |
| [9] |
周静, 曾玫艳, 安新民. 杨树WRKY基因家族鉴定及其干旱胁迫响应模式分析[J]. 中国细胞生物学学报, 2019, 41(11): 2160-2173. DOI:10.11844/cjcb.2019.11.0012.cjcb.2019.11.0012 ZHOU J, ZENG M Y, AN X M. Identification of Populus trichocarpa WRKY gene family and its' response to drought stress[J]. Chinese Journal of Cell Biology, 2019, 41(11): 2160-2173. DOI:10.11844/cjcb.2019.11.0012.cjcb.2019.11.0012 |
| [10] |
AGARWAL P, REDDY M P, CHIKARA J. WRKY: its structure, evolutionary relationship, DNA-binding selectivity, role in stress tolerance and development of plants[J]. Molecular Biology Reports, 2011, 38(6): 3883-3896. DOI:10.1007/s11033-010-0504-5 |
| [11] |
ZHANG L P, CHEN L G, YU D Q. Transcription factor WRKY75 interacts with DELLA proteins to affect f lowering[J]. Plant Physiology, 2018, 176(1): 790-803. DOI:10.1104/pp.17.00657 |
| [12] |
DUAN S W, WANG J J, GAO C H, JIN C Y, LI D, PENG D S, DU G M, LI Y Q, CHEN M X. Functional characterization of a heterologously expressed Brassica napus WRKY41-1 transcription factor in regulating anthocyanin biosynthesis in Arabidopsis thaliana[J]. Plant Science, 2018, 268: 47-53. DOI:10.1016/j.plantsci.2017.12.010 |
| [13] |
GU L J, Dou L L, Guo Y N, Wang H T, Li L B, Wang C C, Ma L, Wei H L, Yu S X. The WRKY transcription factor GhWRKY27 coordinates the senescence regulatory pathway in upland cotton(Gossypium hirsutum L. )[J]. BMC Plant Biology, 2019, 19(1): 116. DOI:10.1186/s12870-019-1688-z |
| [14] |
决登伟, 桑雪莲, 舒波, 刘丽琴, 王一承, 石胜友. 玉米WRKY转录因子非生物胁迫的表达分析[J]. 广东农业科学, 2017, 44(1): 15-22. DOI:10.16768/j.issn.1004-874X.2017.01.003 JUE D W, SANG X L, SU B, LIU L Q, WANG Y C, SHI S Y. Expression analysis of maize WRKY transcription factor genes under abiotic stress[J]. Guangdong Agricultural Sciences, 2017, 44(1): 15-22. DOI:10.16768/j.issn.1004-874X.2017.01.003 |
| [15] |
张惠媛, 刘永伟, 杨军峰, 张双喜, 于太飞, 陈隽, 陈明, 周永斌, 马有志, 徐兆师, 付金东. 小麦转录因子基因TaWRKY33的耐盐性分析[J]. 中国农业科学, 2018, 51(24): 4591-4602. DOI:10.3864/j.issn.0578-1752.2018.24.001 ZHANG H Y, LIU Y W, YANG J F, ZHANG S X, YU T F, CHEN J, CHEN M, ZHOU Y B, MA Y Z, XU Z S, FU J D. Identification and analysis of salt tolerance of wheat transcription factor TaWRKY33 protein[J]. Scientia Agricultura Sinica, 2018, 51(24): 4591-4602. DOI:10.3864/j.issn.0578-1752.2018.24.001 |
| [16] |
万庆. 甜高粱中SSWRKY28、SSWRKY76基因的克隆和功能分析[D]. 开封: 河南大学, 2018. WAN Q. Cloning and Expression analysis of SSWRKY28 and SSWRKY76 genes in Sorghum dochna[D]. KAIFENG: Henan University, 2018. |
| [17] |
YANG Z, CHI X Y, GUO F F, JIN X Y, SUN B. SbWRKY30 enhances the drought tolerance of plants and regulates a drought stress-responsive gene, SbRD19, in sorghum[J]. Journal of Plant Physiology, 2020(246/247): 246-247. DOI:10.1016/j.jplph.2020.153142 |
| [18] |
DOU L L, GUO Y N, ONDATI E, PANG C Y, WEI H L, SONG M Z, FAN S L. Identification and expression analysis of group Ⅲ WRKY transcription factors in cotton[J]. Journal of Integrative Agriculture, 2016, 15(11): 2469-2480. DOI:10.1016/S2095-3119(15)61306-5 |
| [19] |
赵兴奎, 范昕琦, 聂萌恩, 王海燕, 柳青山, 段永红. 高粱WRKY家族成员鉴定及生物信息学分析[J]. 分子植物育种, 2020, 18. DOI:10.13271/j.mpb.018.004170 ZHAO X K, FAN X Q, NIE M A, WANG H Y, LIU Q S, DUAN Y H. Identification and bioinformatics analysis of WRKY family in sorghum[J]. Molecular Plant Breeding, 2020(13): 18-4181. DOI:10.13271/j.mpb.018.004170 |
| [20] |
高雪, 尼玛扎西, 刘国一, 谭海运. PEG模拟干旱胁迫对白菜型春油菜芽期生长特性的影响[J]. 广东农业科学, 2020, 47(7): 9-17. DOI:10.16768/j.issn.1004-874X.2020.07.002 GAO X, NI M Z X, LIU G Y, TAN H Y. Effects of PEG simulation drought stress on growth characteristics of spring rapeseed(Brassica campestris L.)at bud stage[J]. Guangdong Agricultural Sciences, 2020, 47(7): 9-17. DOI:10.16768/j.issn.1004-874X.2020.07.002 |
| [21] |
袁玉婷. PEG模拟干旱胁迫西藏甘蓝型春油菜芽期根系特征及抗旱性研究[J]. 广东农业科学, 2020, 47(7): 18-25. DOI:10.16768/j.issn.1004-874X.2020.07.003 YUAN Y T. Root characteristics and drought resistance of Tibetan spring rapeseed(Brassica napus L.)at bud stage under PEG simulation drought stress[J]. Guangdong Agricultural Sciences, 2020, 47(7): 18-25. DOI:10.16768/j.issn.1004-874X.2020.07.003 |
| [22] |
MAHESHWARI P, MUMMARI D, PALAKOLANU S R, TEJASWI U N, NAGARAJU M, RAJASHEKER G, JAWAHAR G, JALAJA N, RATHNAGIRI P, KISHORID P B K. Genome-wide identification and expression profile analysis of nuclear factor Y family genes in Sorghum bicolor L.(Moench)[J]. PLOS ONE, 2019, 14(9): e0222203. DOI:10.1371/journal.pone.0222203 |
| [23] |
PFAFFL M W. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Research, 2001, 29(9): e45. DOI:10.1093/nar/29.9.e45 |
| [24] |
GUO Y, PING W J, CHEN J T, ZHU L Y, HUANG Y Q. Meta-analysis of the effects of overexpression of WRKY transcription factors on plant responses to drought stress[J]. BMC Genetics, 2019, 20(1): 1-14. DOI:10.1186/s12863-019-0766-4 |
| [25] |
HE G H, XU J Y, WANG Y X, LIU J M, LI P S, CHEN M, MA Y Z, XU Z S. Drought-responsive WRKY transcription factor genes TaWRKY1 and TaWRKY33 from wheat confer drought and /or heat resistance in Arabidopsis[J]. BMC Plant Biology, 2016, 16(1): 116-131. DOI:10.1186/s12870-016-0806-4 |
| [26] |
LIU Y, YANG T Y, LIN Z K, GU B J, XI NG, C H, ZHAO L Y, DONG H Z, GAO J Z, XIE Z H, ZHANG S L, HUANG X S. A WRKY transcription factor PbrWRKY53 from Pyrus betulaefolia is involved in drought tolerance and As accumulation[J]. Plant Biotechnology Journal, 2019, 17(9): 1770-1787. DOI:10.1111/pbi.13099 |
| [27] |
WU X L, SHIROTO Y, KISHITANI S, ITO Y, TORIYAMA K. Enhanced heat and drought tolerance in transgenic rice seedlings overexpressing OsWRKY11 under the control of HSP101 promoter[J]. Plant Cell Reports, 2009, 28(1): 21-30. DOI:10.1007/s00299-008-0614-x |
| [28] |
CAI C P, NIU E L, DU H, ZHAO L, FENG Y, GUO W Z. Genome-wide analysis of the WRKY transcription factor gene family in Gossypium raimondii and the expression of orthologs in cultivated tetraploid cotton[J]. The Crop Journal, 2014, 2(2/3): 87-101. DOI:10.1016/j.cj.2014.03.001 |
| [29] |
JOHNSON SM, LIM F L, FINKLER A, FROMM H, SLABAS A R, KNIGHT M R. Transcriptomic analysis of Sorghum bicolor responding to combined heat and drought stress[J]. BMC Genomics, 2014, 15: 456. DOI:10.1186/1471-2164-15-456 |
| [30] |
郑超, 郑二松, 王栩鸣, 李冬月, 杨勇, 余初浪, 周洁, 严成其, 陈剑平. 水稻WRKY转录调控因子研究进展[J]. 生物技术通报, 2018, 29(2): 286-294. DOI:10.3969/j.issn.1009-0002.2018.02.026 ZHENG C, ZHENG E S, WANG X M, LI D Y, YANG Y, YU C L, ZHOU J, YAN C Q, CHEN J P. Research progress on rice WRKY transcription factors[J]. Letters in Biotechnology, 2018, 29(2): 286-294. DOI:10.3969/j.issn.1009-0002.2018.02.026 |
 2021, Vol. 48
2021, Vol. 48




