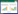文章信息
基金项目
- 广东省科技计划公益研究与能力建设专项(2018B030320007)
作者简介
- 黄建睿(1997—),女,在读硕士生,研究方向为植物资源保护与利用,E-mail:jrhuang2021@163.com.
通讯作者
- 缪绅裕(1965—),男,博士,教授,研究方向为植物多样性及其保护,E-mail:miaoshy@gzhu.edu.cn.
文章历史
- 收稿日期:2022-06-15
2. 深圳市中国科学院仙湖植物园,广东 深圳 518004
2. Fairy Lake Botanical Garden, Shenzhen & Chinese Academy of Sciences, Shenzhen 518004, China
叶绿体是质体家族中的一种细胞器,含有丰富的叶绿素,是绿色植物进行能量转化和光合作用的主要场所,赋予植物在地球生态环境中充当生产者的角色。此外,叶绿体是许多生化过程的基本场所,如氨基酸、核苷酸、脂肪酸、植物激素、维生素的合成以及硫和氮的同化[1]。叶绿体中具有半自主性的细胞器,自身拥有相对独立的遗传物质,即叶绿体基因组DNA(cpDNA),其结构一般为双链环状的DNA分子,少数为线形,是仅次于核基因组的第二大基因组[2]。与核基因组相比,叶绿体基因组因其相对稳定的基因组结构、基因内容和基因序列,已被证明是用于遗传多样性评估的DNA条形码的重要数据来源,被广泛应用于植物系统学研究[3]。叶绿体基因组按功能可分为遗传系统基因、光合系统基因、合成系统基因、功能未知基因4类[4]。叶绿体基因组由于拷贝数高、母系遗传、基因结构和排列保守的特点,是研究近缘物种系统发育关系的有力工具[5-7]。在被子植物中,叶绿体的进化速率也极其缓慢[8]。植物叶绿体基因组为系统发育、DNA条形码和种群间生物地理学提供了宝贵的资源。基因组学研究在基因组序列的遗传组成、结构、组织、功能多样性中,发挥了重要作用,尤其在系统发育研究中,能够直观地体现出植物间的进化关系[9-10]。
紫薇属(Lagerstroemia)隶属于千屈菜科(Lythraceae),全世界约有60种紫薇属植物,目前已培育出500多个品种[11]。我国现有紫薇属植物21种,其中大花紫薇(Lagerstroemia speciosa)、南洋紫薇(L. siamica)、棱萼紫薇(L. turbinate)从东南亚引入[12]。紫薇属植物多为落叶或常绿灌木或乔木,树干多光滑,木材坚硬、耐腐,可作家具木材、建筑等使用,分布于亚洲东部、东南部、南部的热带、亚热带等地区[13]。紫薇(Lagerstroemia indica L.)原产于中国,至少有1 500年的种植历史,随后被引种到美国南部,采取杂交育种、诱变育种等多种培育方式,开始了紫薇属植物在国外的育种历程[14]。大多数紫薇属植物具有大而美丽的圆锥花序,花期通常在夏季和秋季持续约3个月或更久。此外,紫薇属植物具有一定的药用价值,根和枝叶入药可用于治疗过敏反应,具有止痒功效,而花和叶入药有清热解毒、利尿的效果,其叶子还可通过吸收烟雾和灰尘来净化空气,因而紫薇属植物作为观赏、药用兼环保的优良花木,在园艺和园林应用中具有重要价值[15-17]。
本文以叶绿体基因组的研究现状为背景,归纳紫薇属植物叶绿体基因组的结构特征,总结了紫薇属叶绿体基因组在DNA条形码、简单重复序列以及系统发育中的应用,并分析了已完成序列测定的22种植物的系统发育关系,旨在进一步归纳总结紫薇属叶绿体基因组的研究现状以及应用前景,为紫薇属植物种质资源鉴定、分类和系统发育分析等方面的进一步研究提供理论铺垫,同时为紫薇属其他物种的叶绿体基因组研究以及物种进化和亲缘关系分析等研究提供数据基础。
1 叶绿体基因组研究概况 1.1 叶绿体全基因组序列测定1986年,植物叶绿体基因组的全序列测定最早在烟草(Nicotiana tabacum)中发表[18]。近年来,由于测序技术的不断发展,测序成本逐渐降低,利用快速发展的第二代测序技术,越来越多物种的叶绿体基因组先后被测序用于系统发育研究,美国国家生物技术中心(The National Center for Biotechnology Information,NCBI)中关于叶绿体全基因组的数据不断被增加充实。
最初,基因组测序主要使用以Sanger测序为核心的测序技术,该法需要分离纯化叶绿体基因组DNA或者构建全基因组细菌人工染色体(Bacterial artificial chromosome,BAC)文库,再利用含有物种叶绿体DNA片段的载体进行测序,过程复杂且难度大,测序耗时长、成本高,因此未被大范围使用。直到新一代高通量测序的出现,极大地扩大了测序通量,缩短了测序时长,为大规模叶绿体基因组测序提供了可能[19-21]。随着测序成本的显著降低,对整个叶绿体基因组进行测序变得更为便捷。目前,NCBI数据库已获得超过900个陆地植物完整的叶绿体基因组[22]。此外,在叶绿体全基因组中开发了众多组装软件,如GetOrganelle[23]、FastPlast[24]、NOVOPlasty[25]、ORG. Asm[26]、chloroExtractor [27]、IOGA [28]、Chloroplast assembly protocol[29]等。
1.2 叶绿体基因组结构研究叶绿体全基因组序列由于其相对稳定的基因组结构、基因内容和基因序列,已被广泛接受为在分子进化方面有价值的数据来源。叶绿体基因组具有高度保守的环状DNA结构,少数为线形,是仅次于核基因组的第2大基因组[30]。叶绿体基因组结构通常为高度保守的四分体结构,通常由1个大单拷贝区(Large Single Copy,LSC)、1个小单拷贝区(Small Single Copy,SSC)和2个反向重复区(Inverted Repeats,IRs)组成。LSC区长约81~90 kbp,SSC区范围在18~20 kbp,2个反向重复区大小介于20~30 kbp[31]。虽然叶绿体基因组的结构和大小高度保守,但IR/SC边界区域的扩张和收缩常被认为是造成高等植物叶绿体基因组长度差异的主要原因[32]。大多数被子植物的叶绿体基因组大小在107~218 kbp之间,由大约120个基因组成,分别编码rRNA、tRNA和蛋白质[33]。虽然叶绿体基因组结构高度保守,但也会发生基因缺失现象,且可能存在突变热点[34-35],这为紫薇属叶绿体基因组的相关研究奠定了基础。
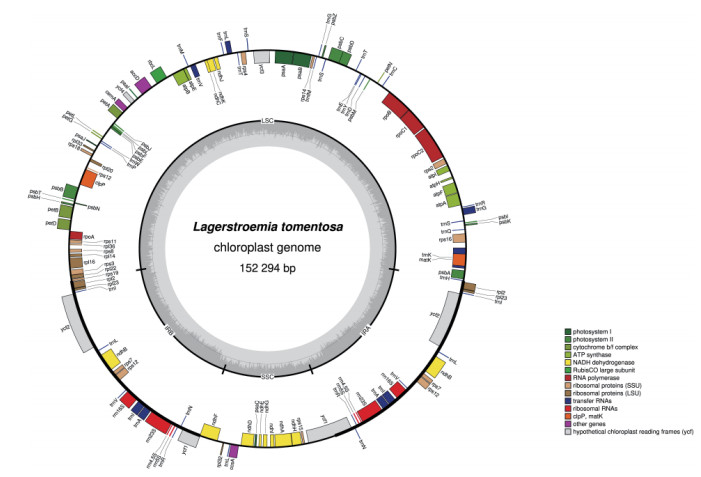
2 紫薇属植物叶绿体基因组结构研究现状 2.1 紫薇属植物叶绿体基因组测序随着基因工程技术的发展,高通量技术的进步降低了测序成本,极大地促进了基因组和系统发育研究的进步。越来越多物种的叶绿体基因组序列被测序,这对紫薇属植物叶绿体基因组的比较研究提供了一定的分子基础,有助于提升对其叶绿体基因组应用价值的评价。目前,NCBI显示已完成22种紫薇属植物的叶绿体全基因组测序。表 1为目前已完成叶绿体全基因组测序的紫薇属植物和5个近缘属的相关信息。

|
2.2 紫薇属植物叶绿体基因组结构特征
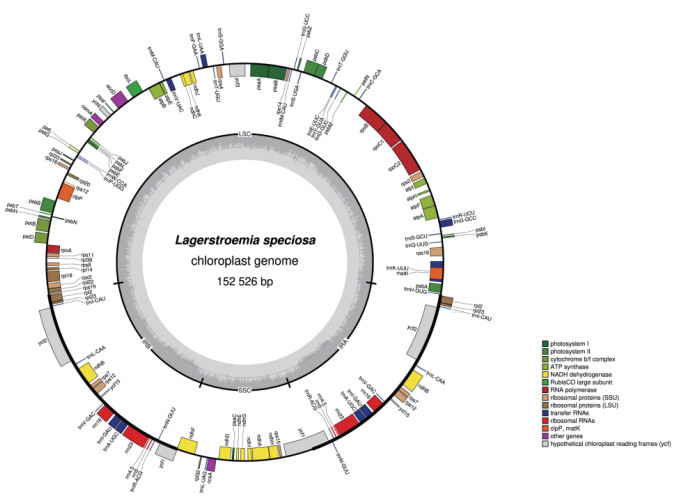
从NCBI数据库下载紫薇(L. indica)和绒毛紫薇(L. tomentosa)的叶绿体基因组序列,登录号分别为NC_030484、MT019851,使用OGDRAW在线软件生成叶绿体基因组图谱。由图 1和图 2可知,紫薇属植物的叶绿体基因组呈闭合环状结构,为高度保守的四分体结构,其叶绿体基因组通常也由4部分组成,分别为1个大单拷贝区(LSC)、1个小单拷贝区(SSC)和2个反向重复区(IRs),其中2个反向重复区域的序列相同,但方向相反,通常命名为IRa、IRb,该区域会在紫薇属叶绿体基因组进化过程中延伸或者缩小;LSC和SSC区的变异大于IR区,非编码区的分化程度大于编码区[36]。由表 2可知,22种紫薇属植物的叶绿体基因组大小约为150 kbp,最大的L. venusta长度为152 521 bp,最小的L. guilinensis长度为151 968 bp,其中LSC区长度为83~84 kbp,SSC区长度约为16 kbp,IR区约为25 kbp,叶绿体基因组中嘌呤(GC)含量为37.6%~37.7%。紫薇属植物的叶绿体基因组的基因数量大多为112个,其中包括78个蛋白编码基因、30个tRNA基因、4个rRNA基因,但L. balansae的叶绿体基因组含有130个基因,包括85个蛋白编码基因、37个tRNA基因、8个rRNA基因。叶绿体基因组虽然在基因结构上高度保守,但IR/SC边界区域的扩张和收缩引起的IR/SC连接位置的变化,通常被认为是造成高等植物叶绿体基因组长度变异的主要机制[37-38]。Zheng等[39]发现13种紫薇在IR/SC交界区表现出相似的特征,而Xu等[40]在3个新测序的紫薇叶绿体基因组中观察到rpl2内含子缺失,紫薇rpl2内含子缺失的发生被认为是千屈菜科中重要的进化事件之一。
|
| 环内基因顺时针转录,环外基因逆时针转录;不同颜色代表不同基因类型,环内灰色区域为GC含量 Intra-loop genes are transcribed clockwise, and extra-loop genes are transcribed counterclockwise. Different colors represent different gene types, and the gray area in the ring is GC content 图 1 大花紫薇叶绿体基因组物理图谱 Fig. 1 Gene map of the choloroplast genome of Lagerstroemia speciosa |
|
| 环内基因顺时针转录,环外基因逆时针转录;不同颜色代表不同基因类型,环内灰色区域为GC含量 Intra-loop genes were transcribed clockwise, and extra-loop genes were transcribed counterclockwise. Different colors represent different gene types, and the gray area in the ring is GC content 图 2 绒毛紫薇叶绿体基因组物理图谱 Fig. 2 Gene map of the choloroplast genome of Lagerstroemia tomentosa |
|
2.3 22种紫薇属植物的系统发育关系
为了确定紫薇属植物的系统发育关系,根据表 1中由NCBI下载的FASTA序列,使用软件PhyloSuite,采用贝叶斯法(Bayesian inference,BI)构建22种紫薇属植物的系统发育树,另设置5个近源外群Duabanga grandiflora、Oenothera biennis、O. argillicola、Ludwigia octovalvis、Erodium carvifolium。由图 3可知,L. calyculata、L. loudonii、L. sp.2、L. tomentosa、L. sp. 3、L. floribunda、L. balansae、L. intermedia、L. siamica、L. speciosa、L. venusta、L. anhuiensis、L. glabra、L. caudata、L. excelsa、L. indica、L. guilinensis、L. sp. 4、L. limii、L. subcostata、L. fauriei、L. villosa共22种紫薇属植物单独为一支,为单系群,具有较大的支持率。

|
| 图 3 基于27个物种的叶绿体全基因组用贝叶斯法(Bayes)构建的系统发育树 Fig. 3 Phylogenetic tree based on the 27 species chloroplast genome sequences with Bayes' method |
3 紫薇属植物叶绿体基因组的应用研究进展 3.1 DNA条形码研究
DNA条形码(DNA barcode)是指生物体内能够代表该物种的、标准的、有足够变异的、易扩增且相对较短的DNA片段。该技术是利用生物体DNA中一个或几个保守片段对物种进行快速准确鉴定的新兴生物技术。叶绿体上的DNA序列片段(如matK、rbcL、trnH-psbA、rpoC1、rpoB、accD、ycf5等)在植物DNA条形码被广泛应用。Xu等[40]从6种紫薇属植物的叶绿体基因组中选择12个相对较高的变异区(trnK-rps 16、trnStrnG、trnG-trnR-atpa、trnE-trnT、rbcLaccd、psbL-psbF-psbE、trnP-psaJ-rpl33、rrn16-trni、ccsa、ndhG-ndhI、rps15-ycf1和ycf1)作为cp DNA标记,推测它们在物种和品种水平上经历了更快的核苷酸替换,可以作为分子标记应用于紫薇属植物的系统发育分析和植物鉴定;Zheng等[39]从13种紫薇属植物中选择识别度最高的7个基因片段(ndhF、ycf1、trnK-rps16、psbKPSBI、trnR-ucu-atpa、rpl32-trnL和rrn16-trni)作为DNA条形码,结果发现这7个条形码能有效鉴别13个种。Dong等[41]比较了20种紫薇属植物中的4个高变量标记和国际DNA通用条形码(rbcL、matK、psbA-trnH),发现4个高变量标记的进化速率是DNA通用条形码的2.5倍。马丽[43]针对国际DNA条形码对紫薇属植物的鉴定进行研究,结果发现trnH-psbA在13个紫薇属物种中变异最大,但是鉴定成功率仅为38.46%;MatK和rbcL变异太小不适合用作紫薇属DNA条形码;而组合片段rbcL+trnH-psbA和mat K+rbc L+trn H-psb A鉴定能力高,可用作紫薇属特异性条形码;构建NJ树对15个高变片段进行评估,发现其中5个片段petA-psbJ、ndh F-rpl32、ndhGndhI、trnS-trnG和trnR-atpA的鉴定能力最强,因此建议将它们作为紫薇属的候选DNA条形码。前人从紫薇属叶绿体基因组中开发的DNA标记能弥补国际通用条形码在紫薇属中的鉴定缺陷,对紫薇种质资源的种间鉴定、定向育种以及系统发育关系提供了强有力的分子手段。
3.2 SSR研究简单重复序列(Simple sequence repeat,SSR)是由1~6个核苷酸组成的简单重复的串联序列,SSR在基因组的不同位置不同分布,长度一般在200 bp以下,通常有6种核苷酸类型,即单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸以及六核苷酸,且SSR在真核和原核生物中广泛分布[44-45]。SSR分子标记具有在基因中覆盖率高、重复次数多、多态性丰富、稳定性好等优点,能够检测出品种亲缘关系之间的细小差异,现已经广泛应用于品种间亲缘关系及遗传多样性的研究[46]。
随着生物学技术的发展,分子标记已成为紫薇属植物在遗传多样性及亲缘关系分析等研究的重要手段。例如,王献等[47]设计并优化了20个紫薇和南紫薇的AFLP银染色反应系统,该技术还应用于分析30个紫薇品种和2个近缘种的亲缘关系;顾翠花[48]在王献等[47]的基础上针对紫薇建立AFLP实验反应体系,筛选出可用于分析紫薇种质资源的引物,并用于评价13个紫薇群体的亲缘关系;徐静静等[49]利用ISSR技术,基于4个紫薇群体和48个不同花色的紫薇品种,分析了紫薇属品种的花色遗传多样性;Wang等[50]利用78个SSR标记分析评价了51个紫薇品种和5个屋久岛紫薇品种的遗传多样性,验证了现有品种与已鉴定但未开发种质资源的品种之间的关系。这些研究在紫薇品种鉴定、分类、遗传多样性评价等方面取得了一定进展。
叶绿体基因组中的简单重复序列在种内水平上可能是高度可变的,因此经常被用作群体遗传学和进化研究中的遗传标记。Gu等[36]分析了22种千屈菜科植物的SSR位点,其中包含14种紫薇属植物,研究发现每个种均有211~332个SSR,长度包含8~16个碱基,共发现单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸6种SSR,以单核苷酸的重复序列最常见,数量在123~212个,其中,紫薇属植物中只有L. siamica和L. intermedia存在六核苷酸。SSR位于31个编码基因和57个基因间隔区中,结果表明叶绿体基因组变异较大的SSR可用于近缘物种的鉴定和系统发育研究[35]。Xu等[40]分析了6种紫薇属植物的叶绿体基因组中的SSR,发现SSR的长度在10~15个碱基之间,对6个紫薇基因组序列的比较分析表明,共检测到5类SSR(单核苷酸、二核苷酸、三核苷酸、四核苷酸和五核苷酸)重复。Zheng等[39]通过比较13种紫薇属植物的叶绿体基因组中SSR的分布和数量,发现了从单核苷酸到六核苷酸的SSR,前5种SSR分别存在于紫薇属13种植物中,六核苷酸重复序列也仅存在于Lagerstroemia siamica和L. intermedia的叶绿体基因组中,发现SSR序列分布在31个基因编码区和57个基因间隔区,这与Gu在22种千屈菜科的14种紫薇属植物的SSR分布研究结果一致,SSR在叶绿体基因组中分布不均的现象特征,有助于SSR分子标记技术应用到种下层面的系统发育分析。马丽[43]研究了13种紫薇的66个SSR位点,选择了7个SSR数量最高的基因,发现同源SSR没有显着差异,这也从侧面说明了SSR的多态性。在紫薇属物种中鉴定SSR位点为多态性SSR,为紫薇属SSR标记的开发提供参考序列。
3.3 系统发育关系研究系统发育也称为系统发展,它是指某一个类群的形成和发展过程。通过建立系统发育树,能更直观地分析类群的亲缘关系。Xu等[40]使用最大简约(MP)、最大似然(ML)和贝叶斯推断(BI)方法,基于4个叶绿体基因组全序列、编码区、非编码区和12个高变区,在高Bootstrap支持下完全区分了所有6个紫薇属分类群,获得了较大的支持率。Zheng等[39]基于32种物种(其中包括13种紫薇)的66个共享蛋白质编码基因构建的系统发育树,揭示了紫薇属内物种的系统发育关系及其在桃金娘目中的系统发育位置。紫薇属植物的系统发育,包括更具代表性的物种和大量的分子标记,对于了解紫薇属植物的进化史、新品种的选育和紫薇种质资源的保护至关重要[50]。
4 结语与展望紫薇属植物具有花期长、花色艳丽且抗污能力强的特点,是我国夏季重要的园林观赏植物。本文在分析叶绿体基因组各结构中,由于L.loudonii、L. sp. 2 WD-2021、L.sp. 3 WD-2021、L. sp. 4 WD-2021的参考文献未发布,暂只对19种紫薇属植物的叶绿体基因组结构进行概述,且已进行叶绿体基因组测序的物种仅占已有物种的1/3,因此对紫薇属植物叶绿体基因组结构的概括不够系统。进一步完成未进行叶绿体基因序列的测定,有利于紫薇属内亲缘关系以及属内基因组的比较研究。
相比利用基因片段作为DNA标记,简短的片段无法准确评估物种在系统发育树中的位置,因此亟需通过完整的叶绿体基因组序列判定物种亲缘关系,利用完整的叶绿体基因组序列进行基因组比较研究更具有说服力[51-53]。新测序的紫薇属植物叶绿体全基因组将利于提高对紫薇属植物叶绿体基因组的认识,并有助于对该物种开展资源保护工作。通过比较完整的叶绿体基因组,提高对叶绿体基因组进化、物种鉴定和系统发育关系的理解,同时有助于提升对叶绿体基因组应用价值的评价,为紫薇属植物后续的种质资源鉴定、分类和系统发育分析等方面的进一步研究提供理论铺垫。
| [1] |
DANIELL H, LIN C S, YU M, CHANG W J. Chloroplast genomes: Diversity, evolution, and applications in genetic engineering[J]. Genome Biology, 2016, 17(1): 134. DOI:10.1186/s13059-016-1004-2 |
| [2] |
徐贝. 巴山松及其近缘种的叶绿体基因组比较分析[D]. 西安: 西北大学, 2021. DOI: 10.27405/d.cnki.gxbdu.2021.002063. XU B. Comparative analysis of chloroplast genome of Pinus pinus and its related species[D]. Xi'an: Northwest University, 2021. DOI: 10.27405/d.cnki.gxbdu.2021.002063. |
| [3] |
付涛, 王志龙, 钱萍仙, 李文, 袁冬明, 严春风. 高等植物DNA条形码最新研究进展及其应用[J]. 核农学报, 2016, 30(5): 887-896. DOI:10.11869/j.issn.100-8551.2016.05.0887 FU T, WANG Z L, QIAN P X, LI W, YUAN D M, YAN C F. Recent advances and applications of DNA barcoding in higher plants[J]. Journal of Nuclear Agricultural Sciences, 2016, 30(5): 887-896. DOI:10.11869/j.issn.100-8551.2016.05.0887 |
| [4] |
BOCK R, KNOOP V. Genomics of chloroplasts and mitochondria[J]. Advances in Photosynthesis & Respiration, 2012, 35(3): 377-377. DOI:10.1007/987-94-007-2920-9 |
| [5] |
DONG W, XU C, WU P, CHENG T, YU J, ZHOU S, HONG D Y. Resolving thesystematic positions of enigmatic taxa: Manipulating the chloroplast genome data of Saxifragales[J]. Molecular Phylogenetics and Evolution, 2018, 126: 321-330. DOI:10.1016/j.ympev.2018.04.033 |
| [6] |
DONG W, XU C, LI W, XIE X, LU Y, LIU Y, JIN X, SUO Z. Phylogenetic resolution in juglans based on complete chloroplast genomes and nuclear DNA sequences[J]. Frontiers in Plant Science, 2017, 8: 1148. DOI:10.3389/fpls.2017.01148 |
| [7] |
DONG W, XU C, WEN J, ZHOU S. Evolutionary directions of single nucleotide substitutions and structural mutations in the chloroplast genomes of the family Calycanthaceae[J]. BMC Evolutionary Biology, 2020, 20(1): 96. DOI:10.1186/s12862-020-01661-0 |
| [8] |
焦凯丽, 郑凯欣, 朱宇佳, 王慧中, 冯尚国. 茄科植物叶绿体基因组研究进展[J]. 杭州师范大学学报(自然科学版), 2019, 18(2): 160-167. DOI:10.3969/j.issn.1674-232X.2019.02.009 JIAO K L, ZHENG K X, ZHU Y J, WANG H Z, FENG S G. Advances in chloroplast genome research of Solanaceae plants[J]. Journal of Hangzhou Normal University (Natural Science Edition), 2019, 18(2): 160-167. DOI:10.3969/j.issn.1674-232X.2019.02.009 |
| [9] |
CAI J, MA P F, LI H T, LI D Z. Complete plastid genome sequencing of four Tilia species (Malvaceae): A comparative analysis and phylogenetic implications[J]. PLoS One, 2015, 10: e0142705. DOI:10.1371/journal.pone.0142705.eCollection2015 |
| [10] |
张闻婷, 焦萌, 王继华. 药用植物基因组学研究进展[J]. 广东农业科学, 2021, 48(12): 138-150. DOI:10.16768/j.issn.1004-874X.2021.12.016 ZHANG W T, JIAO M, WANG J H. Advances in genomics of medicinal plants[J]. Guangdong Agricultural Sciences, 2021, 48(12): 138-150. DOI:10.16768/j.issn.1004-874X.2021.12.016 |
| [11] |
CAI M, PAN H T, WANG X F, HE D, WANG X Y, WANG X J, ZHANG Q X. Development of novel microsatellites in Lagerstroemia indica and DNA fingerprinting in Chinese Lagerstroemia cultivars[J]. Scientia Horticulturae, 2011, 131: 88-94. DOI:10.1016/j.scienta.2011.09.031 |
| [12] |
王金凤, 柳新红, 陈卓梅. 紫薇属植物育种研究进展[J]. 园艺学报, 2013, 40(9): 1795-1804. DOI:10.16420/j.issn.0513-353x.2013.09.016 WANG J F, LIU X H, CHEN Z M. Research progress in breeding of Lagerstroemia plant[J]. Acta Horticulturae Sinica, 2013, 40(9): 1795-1804. DOI:10.16420/j.issn.0513-353x.2013.09.016 |
| [13] |
李振芳, 彭婵, 黄国伟, 马林江, 柯尊发, 杨彦伶. 紫薇属植物染色体倍性鉴定研究进展[J]. 湖北林业科技, 2021, 50(2): 14-17, 33. DOI:10.12677/BR.2021.103042 LI Z F, PENG C, HUANG G W, MA L J, KE Z F, YANG Y L. Research progress on chromosome ploidy identification of Lagerstroemia[J]. Hubei Forestry Science and Technology, 2021, 50(2): 14-17, 33. DOI:10.12677/BR.2021.103042 |
| [14] |
汪磊, 吴笛. 紫薇属植物育种研究进展[J]. 花卉, 2017(18): 5-6. WANG L, WU D. Research progress of Lagerstroemia breeding[J]. Flowers, 2017(18): 5-6. |
| [15] |
黄建民, 侯伯鑫, 索志立. 邵阳市紫薇品种调查研究Ⅲ[J]. 农学学报, 2013, 3(5): 34-41. HUANG J M, HOU B X, SUO Z L. Study on the Lagerstroemia indica cultivars in Shaoyang City Ⅲ[J]. Journal of Agriculture, 2013, 3(5): 34-41. |
| [16] |
漆淑华, 吴大刚, 马云保, 吴少华, 梅文莉, 罗晓东. 桂林紫薇化学成分研究[J]. 中草药, 2002, 10(33): 879-880. QI S H, WU D G, MA Y B, WU S H, MEI W L, LUO X D. Study on chemical constituents of Lagerstroemia guilinensis[J]. Chinese Traditiongal and Herbal Drugs, 2002, 10(33): 879-880. |
| [17] |
牟少华, 刘庆华, 王奎玲. 紫薇研究进展[J]. 莱阳农学院学报, 2002(4): 276-278. DOI:10.3969/j.issn.1674-148X.2002.04.011 MOU S H, LIU Q H, WANG K L. Research progress of Lagerstroemia[J]. Journal of Qingdao Agricultural University (Natural Science), 2002(4): 276-278. DOI:10.3969/j.issn.1674-148X.2002.04.011 |
| [18] |
SHINOZAKI K, OHME M, TANAKA M, WAKASUGI T, HAYASHIDA N, MATSUBAYASHI T, ZAITA N, CHUNWONGSE J, OBOKATA J, YAMAGUCHI-SHINOZAKI K, OHTO C, TORAZAWA K, MENG BY, SUGITA M, DENO H, KAMOGASHIRA T, YAMADA K, KUSUDA J, TAKAIWA F, KATO A, TOHDOH N, SHIMADA H, SUGIURA M. The complete nucleotide sequence of the tobacco chloroplast genome: its gene organization and expression[J]. Plant Molecular Biology Reporter, 1986, 5(9): 2043-2049. DOI:10.1002/j.1460-2075.1986.tb04464.x |
| [19] |
刘振波. DNA测序技术比较[J]. 生物学通报, 2012, 47(7): 14-17. LIU Z B. Comparison of DNA sequencing te chnologies[J]. Bulletin of Biology, 2012, 47(7): 14-17. |
| [20] |
李妍, 徐兴祥. 高通量测序技术的研究进展[J]. 中国医学工程, 2019, 27(3): 32-37. DOI:10.19338/j.issn.1672-2019.2019.03.009 LI Y, XU X X. Research progress of high throughput sequencing technology[J]. China Medical Engineering, 2019, 27(3): 32-37. DOI:10.19338/j.issn.1672-2019.2019.03.009 |
| [21] |
詹晓娟, 姚登举, 朱怀球. 高通量DNA测序数据的生物信息学方法[J]. 大数据, 2016, 2(2): 76-87. DOI:10.11959/j.issn.2096-0271.2016021 ZHAN X J, YAO D J, ZHU H Q. Bioinformatics methods for high throughput DNA sequencing data[J]. Big Data Research, 2016, 2(2): 76-87. DOI:10.11959/j.issn.2096-0271.2016021 |
| [22] |
WU Z Q, TEMBROCK L R. Two complete chloroplast genomes of white campion (Silene latifolia) from male and female individuals[J]. Mitochondrial DNA, 2015, 1736: 1-2. DOI:10.3109/19401736.2015.1126829 |
| [23] |
JIN J J, YU W B, YANG J B, SONG Y, CLAUDE W DEPAMPHILIS, YI T S, LI D Z. GetOrganelle: A fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome Biology, 2020, 21(1): 241. DOI:10.1186/s13059-020-02154-5 |
| [24] |
MCKAIN M, AFINIT M. Fast-Plast: Fast-Plast V. 1.2.6[M]. Zenodo (2017).
|
| [25] |
NICOLAS D, PATRICK M, GUILLAUME S. NOVOPlasty: de novo assembly of organelle genomes from whole genome data[J]. Nucleic Acids Research, 2016, 45(4): 4. DOI:10.1093/nar/gkw955 |
| [26] |
COISSAC E, HOLLINGSWORTH P M, SÉBASTIEN L, PIERRE T. From barcodes to genomes: Extending the concept of DNA barcoding[J]. Molecular Ecology, 2016, 25(7): 1423-1428. DOI:10.1111/mec.13549 |
| [27] |
ANKENBRAND M J, PFAFF S, TERHOEVEN N, QUREISCHI M, GÜNDEL M, WEIB C, HACKL C, FÖRSTER F. chloroExtractor: Extraction and assemb ly of the chloroplast genome from whole genome shotgun data[J]. The Journal of Open Source Software, 2018, 3(21): 464. DOI:10.21105/joss.00464 |
| [28] |
FREEK T B, LEI D, YU J Y, SETAREH M, WEI Z, SARA V D K, BARBARA G, MATHIJS N, MARTIJN S, DAVID E, ALQUEZAR P, RENS H. Herbarium genomics: plastome sequence assembly from a range of herbarium specimens using an Iterative Organelle Genome Assembly pipeline[J]. Biological Journal of the Linnean Society, 2016(1): 1. DOI:10.1007/s10531-011-0082-4 |
| [29] |
RUBÉN S, CANTALAPIEDRA C P, DIANA L, SEAN P, JOHN P, PILAR B. Comparative plastome genomics and phylogenomics of Brachypodium: Flowering time signatures, introgression and recombination in recently diverged ecotypes[J]. N ew Phytologist, 2017, 218(4). DOI:10.1111/nph.14926 |
| [30] |
周会, 荆胜利, 李刚, 张磊, 覃瑞, 刘虹. 叶绿体基因组分析在植物系统发育中的应用[J]. 植物学研究, 2014, 3(1): 1-9. DOI:10.12677/br.2014.31001 ZHOU H, JING S L, LI G, ZHANG L, QIN R, LIU H. The applications of chloroplast genome analysis in plant system development[J]. Botanical Research, 2014, 3(1): 1-9. DOI:10.12677/br.2014.31001 |
| [31] |
ASAF S, KHAN A L, KHAN M A, WAQAS M, KANG S M, YUN B W, LEE I J. Chloroplast genomes of Arabidopsis halleri ssp.[J]. Scien tifi c Reports, 2017, 77: 556. DOI:10.1038/s41598-017-07891-5 |
| [32] |
KAUR S, PANESAR P S, BERA M-B, KAUR V. Simple sequence repeat markers in genetic divergence and marker-assisted selection of rice cultivars: A review[J]. Critical Reviews in Food Science and Nutrition, 2015, 55: 41-49. DOI:10.1080/10408398.2011.646363 |
| [33] |
DANIELL H, LIN C S, YU M, CHANG W J. Chloroplast genomes: diversity evolution and applications in genetic engineering[J]. Genome Biology, 2016, 17: 134. DOI:10.1186/s13059-016-1004-2 |
| [34] |
CAI Z, GUISINGER M, KIM H G, RUCK E, BLAZIER J C, MCMURTRY V, KUEHL J V, BOORE J, JANSEN R K. Extensive reorganization of the plastid genome of Trifolium subterraneum (Fabaceae) is associated with numerous repeated sequences and novel DNA in sertions[J]. Journal of Molecular Evolution, 2008, 67(6): 696-704. DOI:10.1007/s00239-008-9180-7 |
| [35] |
RÖSCHENBLECK J, WICKE S, WEINL S, KUDLA J, MÜLLER K F. Genus-wide screening reveals four distinct ty pes of structural plastid genome organization in Pelargonium (Geraniaceae)[J]. Genome Biology Evolutionary, 2017, 9(1): 64-76. DOI:10.1093/gbe/evw271 |
| [36] |
GU C, MA L, WU Z, CHEN K, WANG Y. Comparative analyses of chloroplast genomes from 22 Ly thraceae species: Inferences for phylogenetic relationships and genome evolu tion within Myrtales[J]. BMC Plant Biology, 2019, 19(1): 281. DOI:10.1186/s12870-019-1870-3 |
| [37] |
KIM K J, LEE H L. Wide spread occurrence of small inversions in the chloroplast ge nomes of land plants[J]. Molecular Cell, 2005, 19: 104-113. DOI:10.1007/s11103-019-00913 |
| [38] |
DONG W P, XU C, LI D L, JIN X B, LU Q, SUO Z L. Comparative analysis of the complete chloroplast genome seq uences in Haloxylon psammophytic species (Amaranthaceae)[J]. Peer Journal, 2016, 4: e2699. DOI:10.7717/peerj.2699 |
| [39] |
ZHENG G, WEI L, MA L, WU Z, GU C, CHEN K. Comparative analyses of chloroplast genomes from 13 Lagerstroemia (Lythraceae) species: Identification of highly divergent regions and inference of phylogenetic relationships[J]. Plant Molecular Biology, 2020, 102(6): 659-676. DOI:10.1007/s11103-020-00972-6 |
| [40] |
XU C, DONG W, LI W, LU Y, XIE X, JIN X, SHI J, HE K, SUO Z. Comparative analysis of six Lagerstroemia complete chloroplast genomes[J]. Frontiers in Plant Science, 2017, 8: 15. DOI:10.3389/fpls.2017.00015 |
| [41] |
DONG W, XU C, LIU Y, SHI J, LI W, SUO Z. Chloroplast phylogenomics and divergence times of Lagerstroemia (Lythraceae)[J]. BMC Genomics, 2021, 22(1): 434. DOI:10.1186/s12864-021-07769-x |
| [42] |
QIN B, SUN K, HUANG X. The complete chloroplast genome of Lagerstroemia balansae, an endanger ed species of genus Lagerstroemia native to China[J]. Mitochondrial DNA B: Resour, 2021, 6(2): 684-685. DOI:10.1080/23802359.2021.1882352 |
| [43] |
马丽. 紫薇属叶绿体比较基因组学及DNA条形码研究[D]. 杭州: 浙江农林大学, 2020. DOI: 10.27756/d.cnki.gzjlx.2020.000069. MA L. Comparative genomics and DNA barcoding of Myrtle chloroplast[D]. Hangzhou: Zhejiang Agriculture and Forestry University, 2020. DOI: 10.27756/d.cnki.gzjlx.2020.000069. |
| [44] |
TAUTZ D, RENZ M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes[J]. Nucleic Acids Research, 1984, 12(10): 4127-4138. DOI:10.1093/nar/12.10.4127 |
| [45] |
EDWARDS A, CIVITELLO A, HAMMOND H A, CASKEY C T. DNA typing and genetic mapping with trimeric and tetrameric tandem repeats[J]. The American Journal of Human Genetics, 1991, 49(4): 746. DOI:10.1007/BF01224774 |
| [46] |
冷嘉文, 乔中全, 唐丽, 王晓明, 陈前欣, 邵雯雯. 基于SSR分子标记的紫薇遗传多样性分析[J]. 湖南生态科学学报, 2021, 8(4): 1-7. DOI:10.3969/j.issn.2095-7300.2021.04.001 LENG J W, QIAO Z Q, TANG L, WANG X M, CHEN Q X, SHAO W W. Geneti c diversity analysis of Lagerstroemia based on SSR markers[J]. Journal of Hunan Ecological Science, 2021, 8(4): 1-7. DOI:10.3969/j.issn.2095-7300.2021.04.001 |
| [47] |
王献, 柴永生, 职永普. 紫薇叶片DNA的提取及AFLP反应体系的建立[J]. 河南农业大学学报, 2004(2): 189-192, 230. DOI:10.16445/j.cnki.1000-2340.2004.02.016 WANG X, CHAI Y S, ZHI Y P. Extraction of DNA from Crape Myrtle leave s and establishment of AFLP reaction system[J]. Journal of Henan Agricultural University, 2004(2): 189-192, 230. DOI:10.16445/j.cnki.1000-2340.2004.02.016 |
| [48] |
顾翠花. 中国紫薇属种质资源及紫薇、南紫薇核心种质构建[D]. 北京: 北京林业大学, 2008. GU C H. Studies on Lagerstroemia germplasm and e stablishment of core collection in L. indica and L. subcostata[D]. Beijing: Beijing Forestry University, 2008. |
| [49] |
徐静静, 王立新, 郁建锋. 不同花色紫薇的ISSR分析[J]. 常熟理工学院学报, 2010, 24(4): 13-17. DOI:10.16101/j.cnki.cn32-1749/z.2010.04.018 XU J J, WANG L X, YU J F. ISSR analysis of the different colorflowered Lagerstroemia indica[J]. Journal of Changshu Institute of Technology, 2010, 24(4): 13-17. DOI:10.16101/j.cnki.cn32-1749/z.2010.04.018 |
| [50] |
WANG X, WADL P A, POUNDERS C, TRIGIANO R N, CABRERA R I, SCHEFFLER B E, POOLER M, RINEHART T A. Evaluation of genetic diversity and pedigree within Crapemyrtle cultivars using simple sequence repeat markers[J]. Journal of the Ameri can Society for Horticultural Science, 2011, 136(2): 116-128. DOI:10.21273/JASHS.136.2.116 |
| [51] |
邓演文, 刘晓洲, 卓定龙, 曾凤. 肖蒲桃叶绿体基因组结构特征及系统发育关系[J]. 广东农业科学, 2021, 48(6): 38-46. DOI:10.16768/j.issn.1004-874X.2021.06.006 DENG Y W, LIU X Z, ZHUO D L, ZENG F. Structural characteristics and phylogenetic relationship of ch loroplast genome of Xiaopu Peach[J]. Guangdong Agricultural Sciences, 2021, 48(6): 38-46. DOI:10.16768/j.issn.1004-874X.2021.06.006 |
| [52] |
付瑜华, 贺尔奇, 雷石富, 李向勇, 张正学, 卢加举. 2种甘蔗病害胁迫果蔗NBS类抗病基因同源序列的表达分析[J]. 广东农业科学, 2017, 44(2): 118-123, 2. DOI:10.16768/j.issn.1004-874X.2017.02.018 FU Y H, HE E Q, LEI S F, LI X Y, ZHANG Z X, LU J J. Expression analysis of NBS resistance gene analogs from chewing cane under two sugarcane diseases stress[J]. Guangdong Agricultural Sciences, 2017, 44(2): 118-123, 2. DOI:10.16768/j.issn.1004-874X.2017.02.018 |
| [53] |
时涛, 李超萍, 蔡吉苗, 李博勋, 黄贵修. 一个预测的木薯衰老相关基因的获得及其功能分析[J]. 广东农业科学, 2017, 44(2): 6-11. DOI:10.16768/j.issn.1004-874X.2017.02.002 SHI T, LI C P, CAI J M, LI B X, HUANG G X. Obtaining and function analysis of predicted senescence-associated gene from cassava[J]. Guangdong Agricultural Sciences, 2017, 44(2): 6-11. DOI:10.16768/j.issn.1004-874X.2017.02.002 |
(责任编辑 邹移光)
 2022, Vol. 49
2022, Vol. 49