文章信息
基金项目
- 广州市科技计划项目(202102021008);广东省农业科学院院长基金(202021)
作者简介
- 罗永坚(1996—),男,在读硕士生,研究方向为种子生物学,E-mail:851022933@qq.com; 李清,博士,助理研究员,毕业于中国农业大学,2018年入职广东省农业科学院农业生物基因研究中心,主要从事水稻种子活力调控机制研究与种质资源鉴评。先后主持广州市科技计划项目和广东省农业科学院院长基金等科研项目,参与国家自然科学基金、广东省自然科学基金、广东省重点研发专项等多个项目。在《Plant Journal》《Plant Physiology》《Journal of Experimental Botany》等期刊发表科技论文多篇.
通讯作者
- 李清(1989—),女,博士,助理研究员,研究方向为农作物种质资源的基因挖掘,E-mail:411066120@qq.com.
文章历史
- 收稿日期:2022-08-30
【研究意义】生长素是一种普遍存在的激素,在多种植物发育过程中起着重要作用,如细胞分裂、胚胎发生、维管束伸展、侧根起始、叶片伸展、开花、果实成熟、顶端优势、向下生长和抗逆性[1]。生长素并不直接参与这些调节作用,而是通过的生长素信号的转导的受体,如AUX/IAA(auxin/indole-3-acetic acid)、ARF(auxin response factor)、GH3(Gretchen hagen 3)和SAUR(small auxin-up RNA)[2-3]。其中,AUX/IAA蛋白作为生长素信号通路的重要组成部分,该蛋白主要存在细胞质和细胞核中且存在时间较短[4-5],通过释放生长素反应因子(Auxin Response Factors)参与生长素信号通路调控下游大量的基因表达[6–8]。小豆(Vigna angularis),又名红豆、红小豆,豆科(Leguminosae)蝶形花亚科(Papilionoideae)豇豆属(Vigna)的一种1年生直立或缠绕草本作物[9],已有12 000年的栽培历史[10]。在当今大力发展优质、高效农业的时代背景下,小豆已成为首选作物之一。但在实际生产中,作物常常会因不可避免的环境胁迫而减产,甚至绝收[11]。而AUX/IAA基因家族在植物的生长发育和植物逆境胁迫应答过程中都起着重要作用,目前已在多种植物中开展了相关研究,但在小豆中AUX/IAA基因家族还尚未进行系统研究和报道。【前人研究进展】Aux/IAA蛋白一般具有4个典型的保守结构域(Domain Ⅰ、Ⅱ、Ⅲ、Ⅳ),该基因家族中也包括缺失一个或两个结构域的蛋白质[7]。其中,Domain Ⅰ是由一个亮氨酸重复基序(“LxLxL”)组成,充当乙烯应答因子(ERF)与EAR基序结合的阻遏域[12–14],结构域Ⅱ是AUX/IAA不稳定的关键成分,存在与降解子高度保守序列(GWPPV),能被泛素酶体蛋白(TIP1)所识别并降解[15–17]。结构域Ⅲ和Ⅳ中C末端结构域上存在介导AUX/IAA蛋白和生长素反应因子同源或异源二聚化[7, 18-19]。自从AUX/IAA基因首次从大豆中被鉴定出来[20],目前,已从多个植物的全基因组中鉴定出AUX/IAA基因家族,其中拟南芥(Arabidopsis thaliana)[21]、毛果杨(Populus trichocarpa Torr)[22]、番茄(Lycopersicon esculentum)[23]、高粱(Sorghum bicolor)[24]、葡萄(Vitis vinifera)[25]、水稻(Oryza sativa)[26]和玉米(Zea mays)[27]。据相关报道,AUX/IAA基因家族的成员在植物的生长发育过程中具有不同的生物学功能,对植物的性状起着重要的调控作用[28]。如:在白花蛇舌草(Hedyotis diffusa)中,HcIAA2和HcIAA4在花香形成过程中起着重要作用[29]。紫花苜蓿(Medicago sativa)的MtIAA6和MtIAA7具有根特异性表达,MtIAA9在花中的表达水平较高[30]。在中华龙竹(Dendrocalamus barbatus)中,DsIAA3、DsIAA4、DsIAA15和DsIAA20可能是调控新梢发育的重要基因[31]。VvIAA4[32]、VvIAA9[33]、SlIAA15[34]、SlIAA17[35]是果实结实过程的关键调节因子。OsIAA5[36]和OsIAA6[37]与水稻的耐旱性具有关联性。在拟南芥中,AtIAA5、AtIAA6和AtIAA9阻遏蛋白通过特异性调控脂肪族芥子油苷的表达来影响植物的耐旱性[38]。【本研究切入点】基于已公布的小豆基因组和转录组数据,利用生物信息学方法鉴定小豆中的AUX/IAA基因,并从理化性质、系统进化、基因结构、蛋白质互作分析、各组织的基因表达水平等进行分析。【拟解决的关键问题】本研究为后续阐明AUX/IAA基因的相关功能研究提供一定的试验基础和理论依据。
1 材料与方法 1.1 家族成员鉴定及蛋白质理化性质分析从Esembl Plant数据库(http://plants.ensembl.org/index.html)下载小豆全部蛋白质序列,从Pfam数据库(http://pfam.xfam.org/)中下载AUX/IAA保守结构域的HMM模型,利用HMMER3.0软件和本地BLASTP程序设置阈值E-value值≤ e-10鉴定含有AUX/IAA蛋白结构域的序列,通过SMARTG1,CDD[39-40]在线软件预测候选蛋白质序列。利用Expasy[41]分析小豆AUX/IAA成员的蛋白质序列长度、分子量及等电点。利用Euk-mP-Loc2.0进行亚细胞定位。利用prabi(https://npsa-prabi.ibcp.fr/)在线分析软件进行二级结构分析
1.2 系统进化树构建AUX/IAA氨基酸序列在MEGA X软件采用邻近法(Neighbor-joining)构建系统进化树,其中Bootstrap设置为1000,构建系统发育进化树,用iTOL(https://itol.embl.de/itol.cgi)对进化树进行分类和注释。
1.3 保守基序和基因结构分析利用在线软件MEME(https://meme-suite.org/meme/doc/meme.html)对小豆AUX/IAA蛋白保守基序,其中利用在线网站CSDS2.0(https://www.csds2.0.in/)对小豆AUX/IAA基因家族成员进行结构分析,通过Tbtools软件对小豆AUX/IAA蛋白保守基序和保守结构域结合其系统进化树进行可视化分析。
1.4 加倍复制分析及Ka、Ks计算使用Linux版本的MCScan-X[42]软件,分别对小豆基因组内共线性进行分析,并利用TBtools[43]软件和绘图程序将结果可视化,将共线性基因计算Ka/Ks值。
1.5 顺式作用元件分析提取AUX/IAA家族基因上游1 500 bp长度的序列作为启动子序列,提交到Plant CARE[44](http://bioinformatics.psb.ugent.be/)数据库,对小豆AUX/IAA家族基因进行上游顺式作用元件分析,预测转录因子可识别并特异性结合的顺式作用元件。
1.6 蛋白-蛋白互作网络及GO功能注释基于在线分析软件STRING(https://string-db.org/),将小豆AUX/IAA基因家族氨基酸序列与模式植物拟南芥库作为参考进行比对,对AUX/IAA基因的蛋白-蛋白互作信息进行评估和预测。1.7基因表达模式分析在NCBI的SRA数据库下载8个小豆组织(叶、根、胚轴、花、根瘤、顶端、幼荚、子叶)以及0.9 mmol/L NaCl幼苗盐胁迫的RNA-seq数据(PRJDB3778, PRJNA576763)[45],计算表达量FPKM,绘制表达图谱。
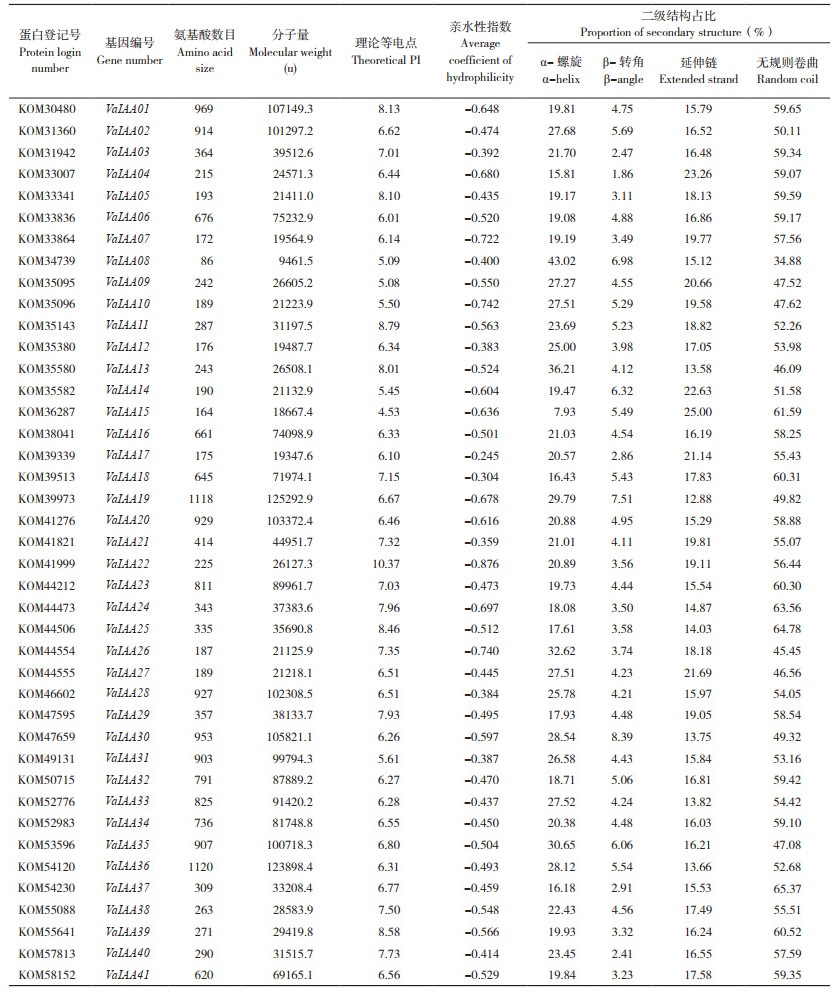
2 结果与分析 2.1 基因家族成员鉴定及理化性质分析从小豆全基因组中筛选出41个完整和非重复冗余的AUX/IAA蛋白序列,根据基因在染色体位置上进行排序,分析AUX/IAA基因的理化性质。结果(表 1)表明,小豆AUX/IAA基因编码的多肽为86~1 120个氨基酸残基,相对分子质量为9 461.5~125 292.9 u,等电点为4.53~10.37。在蛋白亲水性方面41个VaIAA蛋白亲水性系数均小于1,为亲水性蛋白,亚细胞定位显示均为定位为细胞核,与其响应生长素转导位置相同。预测蛋白的二级结构,α-螺旋占比为7.93%~43.02%,β-转角占比为1.86%~8.39%,延伸链占比为12.88%~25.00%,无规则卷曲占比为34.88%~65.37%,从结果来看,VaIAA蛋白以无规则卷曲和a-螺旋为主体结构,β-转角及延伸链为次要结构。
|
2.2 AUX/IAA蛋白的系统进化
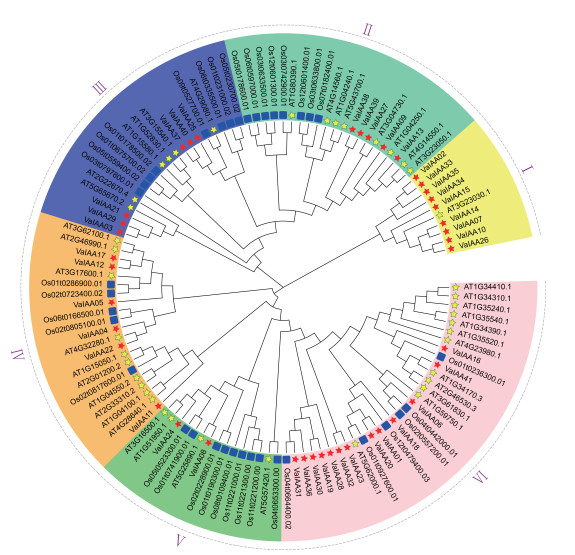
将本研究鉴定出的41个VaIAAs基因与拟南芥和水稻AUX/IAA基因构建系统发育树,共分为6个类别(Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ、Ⅵ),Ⅰ类包含10个IAA基因(9个VaIAAs、1个AtIAAs),Ⅱ类包含22个IAA基因(8个AtIAAs、6个OsIAAs、8个VaIAAs),Ⅲ类包含20个IAA基因(6个AtIAAs、8个OsIAAs、6个VaIAAs),Ⅳ类包含21个IAA基因(10个AtIAAs、5个OsIAAs、6个VaIAAs),Ⅴ类包含15个IAA基因(4个AtIAAs、9个OsIAAs、2个VaIAAs),Ⅵ类包含31个IAA基因(12个AtIAAs、6个OsIAAs、13个VaIAAs)。进化关系中呈现7对姐妹对,其余为拟南芥与水稻;旁系同源蛋白占75.86%(22对),小豆10对、拟南芥11对、水稻11对,其余全为直系同源蛋白(图 1)。
|
| 图 1 小豆AUX/IAA基因家族进化树 Fig. 1 Phylogenetic tree of AUX/IAA gene family in Vigna angularis |
2.3 蛋白基序及基因结构分析
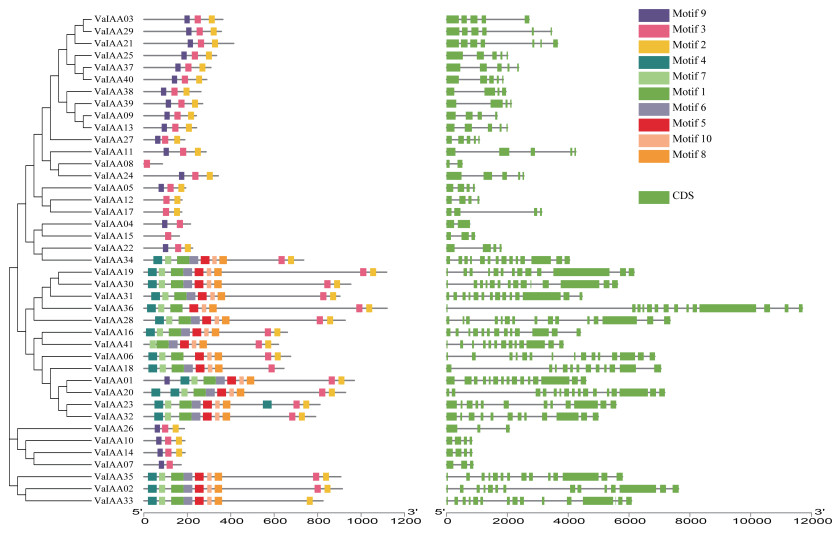
为探究VaIAAs家族的保守性,在可信范围(e-value ≤ e-5)内,VaIAA家族成员编码蛋白共含有10种motif,分别命名为motif 1~motif 10。结果(图 2)显示,共有41个成员具有4个完整的典型保守结构域(保守基序):Domain Ⅰ(motif 3)、Domain Ⅱ(motif 9)、Domain Ⅲ(motif 10)、Domain Ⅳ(motif 2)。其中Domain Ⅰ(motif 3)在所有AUX/IAA基因中均鉴定到,有4个成员(VaIAA 08/04/015/07)发生了Domain Ⅳ(motif 9)缺失;而Domain Ⅱ(motif 9)、Domain Ⅲ(motif 10)只在部分基因中鉴定得到。而基因的结构在决定基因的表达和功能中起着重要因素,而且基因结构多样性是许多基因家族进化的主要动力。因此,对小豆AUX/IAA基因家族结构进行分析。结果(图 2)表明,多数VaAUX/IAA包含4个内含子,即多具有5个外显子和4个内含子的典型基因结构模式,相同的基因结构和保守基序的基因在聚类在一起提高了系统发育树的准确性。
|
| 图 2 小豆AUX/IAA家族基因结构及保守基序 Fig. 2 Gene structure and conserved motifs of AUX/IAA family in Vigna angularis |
2.4 小豆AUX/IAA家族基因的染色体定位、种内共线性和选择压力分析
通过染色体定位,结果(图 3)表明41条VaIAA基因不均匀地分布在9条染色体上,其中2号染色体上分布最多,有9个VaIAA成员;共线性分析结果(图 3)显示,41个VaIAA家族成员存在13对共线性关系,所有基因均为片段复制。通过计算基因对的Ka/Ks值,发现Ka/Ks值均小于1,说明VaIAA基因在进化过程中可能经历受到纯化选择压力的影响。

|
| 图 3 小豆AUX/IAA家族基因染色体定位及共线性分析 Fig. 3 Chromosome and collinearity analysis of AUX/IAA gene family in Vigna angularis |
2.5 小豆AUX/IAA转录因子顺式作用元件分析
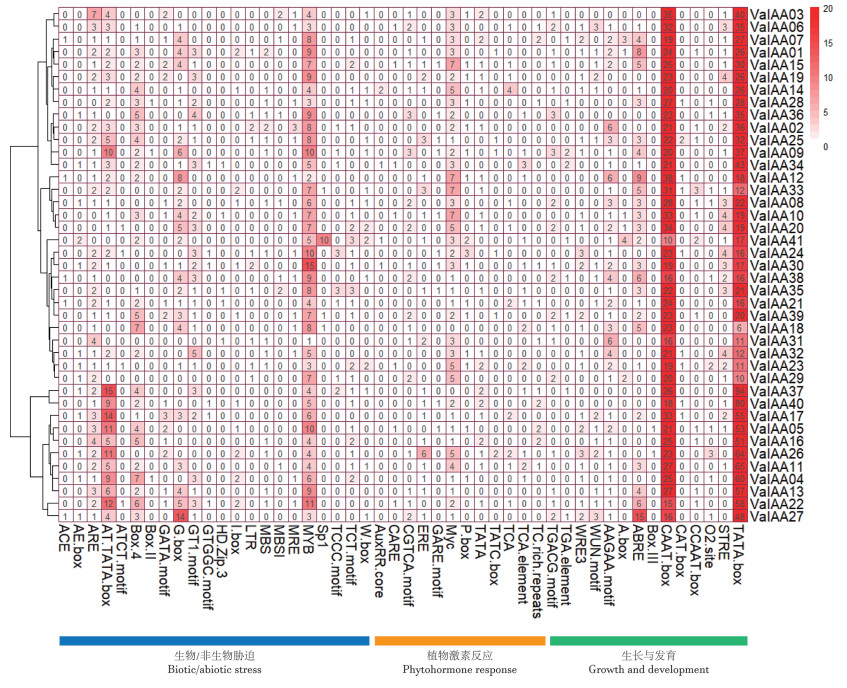
通过plantCARE对VaIAAs基因上游1.5 kb进行启动子顺式作用元件分析,结果(图 4)发现共有51种顺式作用元件,对VaIAAs基因顺式作用元件分类可分为响应生物/非生物胁迫、植物激素、生长发育的顺式作用元件。其中参与响应生物/非生物胁迫顺式作用元件最多,包括最常识别到的ATTATA.bos、G-box和sp1基序,还发现富集在厌氧诱导响应元件ARE和植物抗逆相关的MYB元件。植物激素响应元件共12种,包括赤霉素响应元件(P-box)、茉莉酸响应元件(TGAGG-motif)和水杨酸响应元件(TCA-element)和乙烯响应元件(ERE)。在参与植物生长发育顺式元件共有14个,其中VaIAA基因富集在TATA.box、CAAT.box、ABRE,其中TATA-box基序在预测元件中数目最多。顺式作用元件分析结果表明,VaIAA基因存在不同的反应基序,不同的VaIAA成员在基序上富集情况和作用模式不尽相同,说明VaIAA基因在植物生长及逆境胁迫响应中发挥重要作用。
|
| 图 4 小豆AUX/IAA家族响应激素顺式作用元件 Fig. 4 Cis-acting elements in response to hormones of AUX/IAA family in Vigna angularis |
2.6 蛋白-蛋白互作网络及GO功能富集分析
为研究VaIAAs家族蛋白间的相互作用,以拟南芥为参考对象构建VaIAAs蛋白与其他蛋白的调控网络。预测结果(图 5)表明,整个互作网络中共有25个节点,节点间存在227组蛋白互作关系,所有VaIAAs均与ARF蛋白产生互作关系,其中存在一些IAA之间存在蛋白互作关系,15个VaIAA基因与AXR3(生长素诱导阻遏因子),20个VaIAAs基因与MP(转录因子B3家族蛋白),暗示VaIAAs蛋白可能受多种蛋白调控。
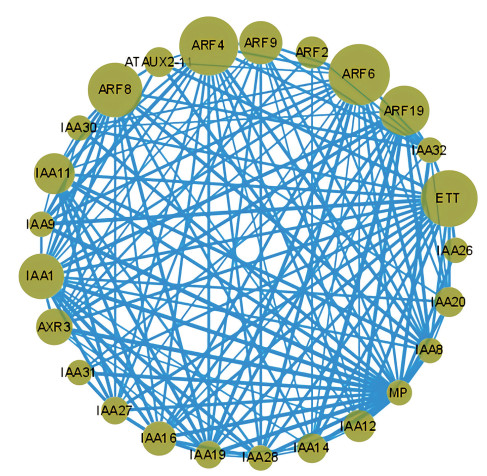
|
| 图 5 小豆VaIAAs家族蛋白互作网络 Fig. 5 Protein interaction network of VaIAAs family in Vigna angularis |
为更好理解VaIAAs的功能,将所有VaIAAs转录因子进行基因本体注释(Gene ontology),结果见图 6,GO功能注释主要富集在生物过程(Biological Process)、细胞组分(Cellular Component)和分子功能(Molecular Function)3大类。其中,生物进程富集的基因通路最多,分子功能次之,细胞成分最少,在生物过程分类中,所有主要富集在生长素信号激活通路和转录调控功能,细胞组成分类中AUX/IAA基因主要参与细胞和细胞核的组成,在分子功能中主要富集在DNA结合和DNA转录因子活性。
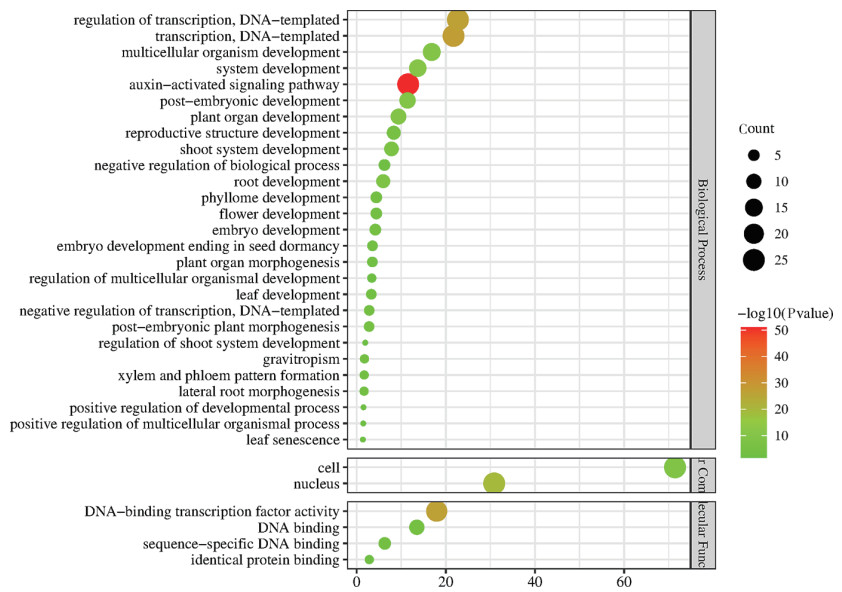
|
| 图 6 小豆VaIAAs家族基因GO富集分析 Fig. 6 GO enrichment analysis of VaIAAs gene family in Vigna angularis |
2.7 小豆不同组织基因表达分析
VaIAAs基因的组织特异性表达模式与生物学功能密切相关,利用小豆在叶、根、胚轴和花等不同组织的RNA-seq数据对41个VaIAAs成员在不同组织中的转录水平进行分析,结果(图 7A)表明有10个基因在各组织表达量较低(FPKM < 10),包括7个成员(VaI AA08/17/18/05/15/23/12/31/32/22)。大多数AUX/IAA基因在茎和花组织上表达较高,在幼荚和子叶这两个组织中VaIAAs表达水平相对于其他组织低。还发现VaIAA08/17/22/15/12在组织间的表达差异最小,表明这些基因的调控可能主要发生在翻译后水平。而VaIAA39/29在多个组中均表达较高,可这能参与小豆整个生长过程。此外还发现VaAUX/IAA基因在不同组织的优先表达,表明这些基因可能在组织特异性转录因子下发挥作用,参与小豆特定组织的发育进程。基于NaCl胁迫下RNA-seq数据比较18个DlIAAs的表达水平,结果(图 7B)显示有10个VaIAAs(VaI AA12/20/40/14/03/26/32/01/07/31)表达显著上调,推测这10个成员可能在盐胁迫下起关键的作用。
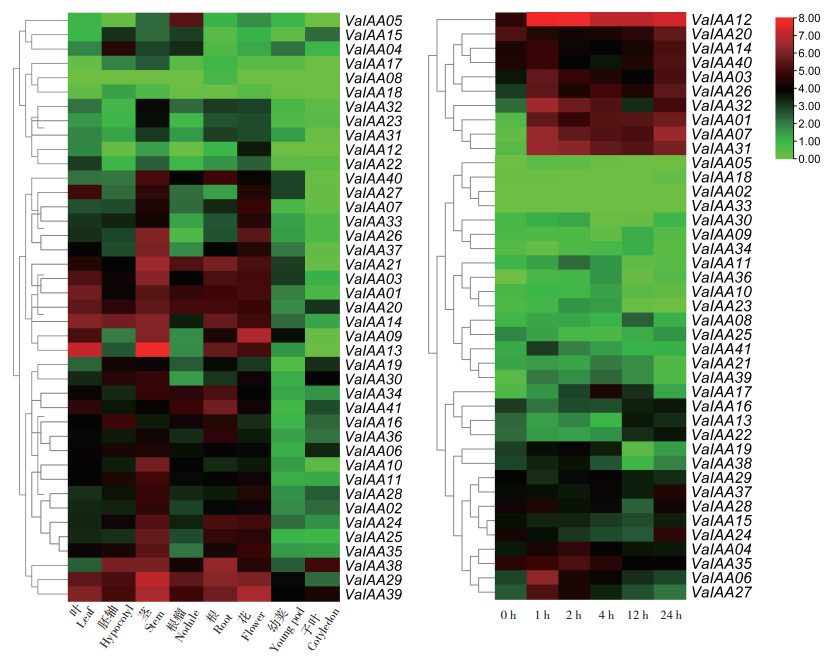
|
| A:小豆不同组织中VaIAAs基因的表达;B:小豆在盐胁迫下的表达模式 A: Expression of VaIAAs gene in different tissues of Vigna angularis; B: Expression pattern of Vigna angularis under NaCl stress 图 7 小豆VaIAAs家族基因在RNA-seq的表达 Fig. 7 Expression of VaIAAs gene family of Vigna angularis in different RNA-seq databases |
3 讨论
植物在生长过程中受到多种激素的影响,生长素是植物生长发育过程中的关键信号分子。作为最早发现的植物激素,其生理作用广泛,影响细胞的分裂、扩大和分化。AUX/IAA蛋白在植物生长素信号转导通路中被认为与ARFs结合,在没有生长素的情况下阻止生长素反应基因的激活,以调控植物的生长[46]。研究AUX/IAA的功能和表达图谱有助于揭示生长素是如何调控植物体的生长发育和响应特定时空上的环境变化。随着高通量测序技术的发展,已经对许多植物的全基因组进行了测序,并在多种植物中发现了大量的AUX/IAA基因。小豆的全基因组公布使得在基因组水平上鉴定分析AUX/IAA基因家族成为可能。在本研究中,在小豆全基因组中共鉴定出41个AUX/IAA基因家族,而在前人的研究中如:拟南芥鉴定出29个成员[21],毛果杨中鉴定出35个成员[22],番茄[23]、高粱[24]、葡萄[25]中均包含26个成员,水稻[26]和玉米[27]中鉴定出31个成员,这表明不同植物物种之间AUX/IAA基因家族的数量存在很大差异。
利用NJ法将41个VaIAAs基因与双子叶植物拟南芥和单子叶植物水稻AUX/IAA基因进行系统发育分析。结果分为6类,其中VaIAAs主要集中在Ⅰ类和Ⅵ类(58.53%),因此,推断聚类在一起的IAAs基因结构或功能可能为高度相同且具有保守性。在进化关系中呈现7对姐妹对,其中3对姐妹对是小豆与拟南芥,其余均为拟南芥与水稻形成的姐妹对,这可能是因为小豆和拟南芥都为双子叶植物VaIAAs与AtIAAs在进化关系上更近,而相近的蛋白序列在植物生理上具有相似功能。基因家族扩张的主要动力是片段复制和串联复制[47],VaIAAs家族中10对旁系同源蛋白,均无串联复制事件,由小豆基因组内共线性分析,发现VaIAAs基因家族中共有13对复制基因,且Ka/Ks均小于1,说明片段复制为VaIAAs基因家族进化扩张的主要动力,这可能与其基因组在进化过程中经历了多次全基因组复制事件有关[48]。
基因的表达往往受到启动子区域顺式作用元件的调控,本研究鉴定了3类直接参与生长素信号通路的顺式元件。鉴于AUX/IAA蛋白作为光信号和生长素反应之间的连接物,在VaIAAs基因的启动子区域中观察到12种对光信号有反应的顺式元件。此外,生长素还可以在植物防御反应中发挥调节作用[49]。在VaIAAs基因启动子序列中还发现了4种与病原菌抗性相关的顺式元件。此外,在VaIAA基因的启动子区域还发现了与非生物胁迫适应相关的顺式元件,如DRE(脱水反应元件)、LTRE(低温反应元件)。VaIAAs基因启动子中存在不同的顺式元件说明生长素信号转导可以与其他代谢途径相互作用。生长素和非生物胁迫信号通路之间的串扰已经在水稻中得到证实[50]。毫无疑问,VaIAAs基因启动子中顺式元件的多样性也与AUX/IAA蛋白在植物生命中起着关键作用有关。
植物的基因在不同组织中的特异性表达在不同发育阶段调控中起着重要作用,基因表达调控导致植物对生长、发育、非生物和生物胁迫产生响应[51]。RNA-seq显示几乎所有VaIAAs基因均有表达,但在不同发育阶段,其表达水平有较大差异。许多变量可能导致Aux/IAA基因表达的差异,包括组织特异性生长素受体、细胞类型依赖性、游离生长素浓度的差异调节、生长素依赖性转录的不同模式和转录后调节等[52]。而值得注意的是,在本研究发现VaIAA41基因在小豆根瘤组织的基因中表达水平较高。豆科植物与根瘤菌共生固氮是植物氮素营养的一个重要来源[53],因此根瘤的发育对节约氮肥具有重要作用,有研究表明IAA的积累对根瘤菌的形成具有正向效应[54],推测该基因可能在小豆根瘤组织或者在根的发育和生长过程中发挥重要作用。而在0.9 mmol/L NaCl胁迫处理的RNA-seq数据中,有10个VaIAA(VaIAA12/20/40/14/03/26/32/01/07/31)在NaCl处理1 h后表达量显著上调,是对照组的7倍,此外对这10个基因顺式作用元件检索发现含有多个MYB和ARF作用元件。有研究表明,Aux/IAA通过与植株生长素反应因子ARF(auxin response factor)结合抑制了生长素诱导基因的表达,在生长素所调控的对盐胁迫的响应过程中起着核心作用[55], 表明这些基因可能直接或间接响应调控小豆的抗盐能力。
4 结论本研究以小豆基因组数据为基础,鉴定并分析了小豆中AUX/IAA转录因子家族成员的基因结构、理化性质、保守基序、系统发育关系、染色体定位、共线性关系和顺式作用元件。分析结果表明,从小豆全基因组中共鉴定出41个AUX/IAA基因,系统进化将其分为6个亚族(Ⅰ、Ⅱ、Ⅲ、Ⅳ、Ⅴ和Ⅵ)。基因定位显示,41个AUX/IAA基因不均匀地分布在9条染色体上。基因结构分析表明,AUX/IAA中外显子的数目从1 ~ 17个不等,同一亚族内的成员具有相似的基因结构和基序组成。AUX/IAA转录因子氨基酸数目为86~1 120个;蛋白质分子量为9 461.5~123 898.4 u;等电点在4.53~10.37,AUX/IAA基因都定位在细胞核上。顺式作用元件分析表明,大多数AUX/IAA基因含有1个或多个顺式作用元件,表明AUX/IAA基因可能响应不同的非生物胁迫。不同条件下的表达模式分析结果为进一步研究AUX/IAA基因在小豆生长发育和非生物胁迫响应中潜在的功能提供有价值的信息,进而促进AUX/IAA基因在小豆抗逆分子育种中的应用。
| [1] |
JIN J H, ZHANG H X, TAN J Y, YAN M J, LI D W, KHAN A, GONG Z H. A new ethylene-responsive factor CaPTI1 gene of pepper (Capsicum annuum L.) involved in the regulation of defense response to Phytophthora capsici[J]. Frontiers in Plant Science, 2016, 8: 6-1217. DOI:10.3389/fpls.2015.01217 |
| [2] |
SHEN C, YUE R, YANG Y, ZHANG L, SUN T, XU L, TIE S, WANG H. Genome-wide identification and expression profiling analysis of the Aux/IAA gene family in Medicago truncatula during the early phase of Sinorhizobium meliloti infection[J]. PLoS ONE, 2014, 9(9): e107495. DOI:10.1371/journal.pone.0107495 |
| [3] |
SHEN C, YUE R, SUN T, ZHANG L, XU L, TIE S, WANG H, YANG Y. Genome-wide identification and expression analysis of auxin response factor gene family in Medicago truncatula[J]. Frontiers in Plant Science, 2015, 22(19): 10494. DOI:10.3389/fpls.2015.00073 |
| [4] |
ARASE F, NISHITANI H, EGUSA M, NISHIMOTO N, SAKURAI S, SAKAMOTO N, KAMINAKA H. IAA8 involved in lateral root formation interacts with the TIR1 auxin receptor and ARF transcription factors in Arabidopsis[J]. PLoS ONE, 2012, 7(8): e43414. DOI:10.1371/journal.pone.0043414 |
| [5] |
ABEL S, OELLER P W, THEOLOGIS, A. Early auxin-induced genes encode short-lived nuclear proteins[J]. Proceedings of the National Academy of Sciences, 1994, 91(1): 326-330. DOI:10.1073/pnas.91.1.326 |
| [6] |
DHARMASIRI S, ESTELLE M. The role of regulated protein degradation in auxin response[J]. Plant Molecular Biology, 2002, 49(3/4): 401-408. DOI:10.1023/A:1015203013208 |
| [7] |
HAGEN G, GUILFOYLE T. Auxin-responsive gene expression: Genes, promoters and regulatory factors[J]. Plant Molecular Biology, 2002, 49(3/4): 373-385. DOI:10.1023/A:1015207114117 |
| [8] |
李晓燕, 陈雪梅, 盖颖. 杨树不同组织中ARF表达与生长素相关性研究[J]. 广东农业科学, 2013, 40(6): 134-137, 238. DOI:10.16768/j.issn.1004-874X.2013.06.058 LI X Y, CHEN X M, GAI Y. Correlation between ARF expression and auxin in different tissues of poplar[J]. Guangdong Agricultural Sciences, 2013, 40(6): 134-137, 238. DOI:10.16768/j.issn.1004-874X.2013.06.058 |
| [9] |
LIU L, BESTEL S, SHI J, SONG Y, CHEN X. Paleolithic human exploitation of plant foods during the last glacial maximum in North China[J]. Proceedings of the National Academy of Sciences, 2013, 110(14): 5380-5385. DOI:10.1073/pnas.1217864110 |
| [10] |
TOMOOKA N, VAUGHAN D A, MOSS H, MAXTED N. Genetic Resources//The Asian Vigna[M]. Springer Netherlands: Dordrecht, 2002: 29-55.
|
| [11] |
梁辉, 严诗琦, 李海云. 我国城镇化与耕地资源的时空耦合关系研究——基于2001—2016年的数据[J]. 湖北经济学院学报, 2019, 17(4): 79-89. LIANG H, YAN S Q, LI H Y. Study on the spatial-temporal coupling relationship between urbanization and cultivated land resources in China: Based on the data from 2001 to 2016[J]. Journal of Hubei University of Economics, 2019, 17(4): 79-89. |
| [12] |
TIWARI S B, HAGEN G, GUILFOYLE T J. Aux/IAA proteins contain a potent transcriptional repression domain[J]. The Plant Cell, 2004, 16(2): 533-543. DOI:10.1105/tpc.017384 |
| [13] |
SZEMENYEI H, HANNON M, LONG J A. TOPLESS mediates auxin-dependent transcriptional repression during Arabidopsis embryogenesis[J]. Science (New York), 2008, 319(5868): 1384-1386. DOI:10.1126/science.1151461 |
| [14] |
曹凌燕, 陈亚娟, 王宏芝, 张杰伟, 丁莉萍, 魏建华. 毛白杨AP2/ERF类转录因子PtoSHN的克隆及分析[J]. 广东农业科学, 2014, 41(17): 140-144. DOI:10.16768/j.issn.1004-874X.2014.17.036 CAO L Y, CHEN Y J, WANG H Z, ZHANG J W, DING L P, WEI J H. Cloning and analysis of AP2/ERF class transcription factor PtoSHN from Populus tomentosis[J]. Guangdong Agricultural Sciences, 2014, 41(17): 140-144. DOI:10.16768/j.issn.1004-874X.2014.17.036 |
| [15] |
QUINT M, GRAY W M. Auxin signaling[J]. Current Opinion in Plant Biology, 2006, 9(5): 448-453. DOI:10.1016/j.pbi.2006.07.006 |
| [16] |
KEPINSKI S, LEYSER O. The Arabidopsis F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435(7041): 446-451. DOI:10.1038/nature03542 |
| [17] |
DHARMASIRI N, DHARMASIRI S, ESTELLE M. The F-box protein TIR1 is an auxin receptor[J]. Nature, 2005, 435(7041): 441-445. DOI:10.1038/nature03543 |
| [18] |
REMINGTON D L, VISION T J, GUILFOYLE T J, REED J W. Contrasting modes of diversification in the Aux/IAA and ARF gene families[J]. Plant Physiology, 2004, 135(3): 1738-1752. DOI:10.1104/pp.104.039669 |
| [19] |
OKUSHIMA Y, OVERVOORDE P J, ARIMA K, ALONSO J M, CHAN A, CHANG C, ECKER J R, HUGHES B, LUI A, NGUYEN D, ONODERA C, QUACH H, SMITH A, YU G, THEOLOGIS A. Functional genomic analysis of the AUXIN response factor gene family members in Arabidopsis thaliana: Unique and overlapping functions of ARF7 and ARF19[J]. The Plant Cell, 2005, 17(2): 444-463. DOI:10.1105/tpc.104.028316 |
| [20] |
WALKER J C, KEY J L. Isolation of cloned cDNAs to auxin-responsive poly(A)+RNAs of elongating soybean hypocotyl[J]. Proceedings of the National Academy of Sciences, 1982, 79(23): 7185-7189. DOI:10.1073/pnas.79.23.7185 |
| [21] |
OVERVOORDE P J, OKUSHIMA Y, ALONSO J M, CHAN A, CHANG C, ECKER J R, HUGHES B, LIU A, ONODERA C, QUACH H, SMITH A, YU G, THEOLOGIS A. Functional genomic analysis of the AUXIN/INDOLE-3-ACETIC ACID gene family members in Arabidopsis thaliana[W]. The Plant Cell, 2005, 17(12): 3282-3300. DOI: 10.1105/tpc.105.036723.
|
| [22] |
KALLURI U C, DIFAZIO S P, BRUNNER A M, TUSKAN G A. Genome-wide analysis of Aux/IAA and ARF gene families in Populus trichocarpa[J]. BMC Plant Biology, 2007, 7(1): 59. DOI:10.1186/1471-2229-7-59 |
| [23] |
WU J, PENG Z, LIU S, HE Y, CHENG L, KONG F, WANG J, LU G. Genome-wide analysis of Aux/IAA gene family in Solanaceae species using tomato as a model[J]. Molecular Genetics and Genomics, 2012, 287(4): 295-311. DOI:10.1007/s00438-012-0675-y |
| [24] |
WANG S, BAI Y, SHEN C, WU Y, ZHANG S, JIANG D, GUILFOYLE T J, CHEN M, QI Y. Auxin-related gene families in abiotic stress response in Sorghum bicolor[J]. Functional & Integrative Genomics, 2010, 10(4): 533-546. DOI:10.1007/s10142-010-0174-3 |
| [25] |
AKIR B, KILI KAYA O, OLCAY A C. Genome-wide analysis of Aux/IAA genes in Vitis vinifera: cloning and expression profiling of a grape Aux/IAA gene in response to phytohormone and abiotic stresses[J]. Acta Physiologiae Plantarum, 2012. DOI:10.1007/s11738-012-1079-7 |
| [26] |
JAIN M, KAUR N, GARG R, THAKUR J K, TYAGI A K, KHURANA J P. Structure and expression analysis of early auxin-responsive Aux/IAA gene family in rice (Oryza sativa)[J]. Functional & Integrative Genomics, 2006, 6(1): 47-59. DOI:10.1007/s10142-005-0005-0 |
| [27] |
WANG Y, DENG D, BIAN Y, LV Y, XIE Q. Genome-wide analysis of primary auxin-responsive Aux/IAA gene family in maize (Zea mays L.)[J]. Molecular Biology Reports, 2010, 37(8): 3991-4001. DOI:10.1007/s11033-010-0058-6 |
| [28] |
DIAZ M, LAWRENCE C. An update on the role of translesion synthesis DNA polymerases in Ig hypermutation[J]. Trends in Immunology, 2005, 26(4): 215-220. DOI:10.1016/j.it.2005.02.008 |
| [29] |
KE Y, ABBAS F, ZHOU Y, YU R, YUE Y, LI X, YU Y, FAN Y. Genome-wide analysis and characterization of the Aux/IAA family genes related to floral scent formation in Hedychium coronarium[J]. International Journal of Molecular Sciences, 2019, 20(13): 3235. DOI:10.3390/ijms20133235 |
| [30] |
PAUL P, DHANDAPANI V, RAMENENI J J, LI X, SIVANANDHAN G, CHOI S R, PANG W, IM S, LIM Y P. Genome-wide analysis and characterization of Aux/IAA family genes in Brassica rapa[J]. PLOS ONE, 2016, 11(4): e0151522. DOI:10.1371/journal.pone.0151522 |
| [31] |
CHEN L, ZHENG X, GUO X, CUI Y, YANG H. The roles of Aux/IAA gene family in development of Dendrocalamus sinicus (Poaceae: Bambusoideae) inferred by comprehensive analysis and expression profiling[J]. Molecular Biology Reports, 2019, 46(2): 1625-1634. DOI:10.1007/s11033-019-04611-2 |
| [32] |
AKIR B, KILI KAYA O, OLCAY A C. Genome-wide analysis of Aux/IAA genes in Vitis vinifera: cloning and expression profiling of a grape Aux/IAA gene in response to phytohormone and abiotic stresses[J]. Acta Physiologiae Plantarum, 2012. |
| [33] |
JUNG C J, HUR Y Y, YU H J, NOH J H, PARK K S, LEE H J. Gibberellin application at pre-bloom in grapevines down-regulates the expressions of VvIAA9 and VvARF7, negative regulators of fruit set initiation, during parthenocarpic fruit development[J]. PLoS ONE, 2014, 9(4): e95634. DOI:10.1371/journal.pone.0095634 |
| [34] |
WANG S, BAI Y, SHEN C, WU Y, ZHANG S, JIANG D, GUILFOYLE T J, CHEN M, QI Y. Auxin-related gene families in abiotic stress response in Sorghum bicolor[J]. Functional & Integrative Genomics, 2010, 10(4): 533-546. |
| [35] |
SU L, BASSA C, AUDRAN C, MILA I, CHENICLET C, CHEVALIER C, BOUZAYEN M, ROUSTAN J P, CHERVIN C. The auxin Sl-IAA17 transcriptional repressor controls fruit size Via the regulation of endoreduplication-related cell expansion[J]. Plant and Cell Physiology, 2014, 55(11): 1969-1976. DOI:10.1093/pcp/pcu124 |
| [36] |
PELEG Z, REGUERA M, TUMIMBANG E, WALIA H, BLUMWALD E. Cytokinin-mediated source/sink modifications improve drought tolerance and increase grain yield in rice under water-stress: Cytokinin-mediated drought tolerance in rice[J]. Plant Biotechnology Journal, 2011, 9(7): 747-758. DOI:10.1111/j.1467-7652.2010.00584.x |
| [37] |
HU B, JIN J, GUO A Y, ZHANG H, LUO J, GAO G. GSDS 2.0: An upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296-1297. DOI:10.1093/bioinformatics/btu817 |
| [38] |
YADAV B, JOGAWAT A, RAHMAN M S, NARAYAN O P. Secondary metabolites in the drought stress tolerance of crop plants: A review[J]. Gene Reports, 2021, 23: 101040. DOI:10.1016/j.genrep.2021.101040 |
| [39] |
MARCHLER-BAUER A, BO Y, HAN L, HE J, LANCZYCKI C J, LU S, CHITSAZ F, DERBYSHIRE M K, GEER R C, GONZALES N R, GWADZ M, HURWITZ D I, LU F, MARCHLER G H, SONG J S, THANKI N, WANG Z, YAMASHITA R A, ZHANG D, ZHENG C, GEER L Y, BRYANT S H. CDD/SPARCLE: functional classification of proteins via subfamily domain architectures[J]. Nucleic Acids Research, 2017, 45(D1): 200-203. DOI:10.1093/nar/gkw1129 |
| [40] |
FINN R D. Pfam: Clans, web tools and services[J]. Nucleic Acids Research, 2006, 34(90001): 247-251. DOI:10.1093/nar/gkj149 |
| [41] |
ARTIMO P, JONNALAGEDDA M, ARNOLD K, BARATIN D, CSARDI G, DE CASTRO E, DUVAUD S, FLEGEL V, FORTIER A, GASTEIGER E, GROSDIDIER A, HERNANDEZ C, IOANNIDIS V, KUZNETSOV D, LIECHTI R, MORETTI S, MOSTAGUIR K, REDASCHI N, ROSSIER G, XENARIOS I, STOCKINGER H. ExPASy: SIB bioinformatics resource portal[J]. Nucleic Acids Research, 2012, 40(W1): 597-603. DOI:10.1093/nar/gks400 |
| [42] |
WANG Y, TANG H, DEBARRY J D, TAN X, LI J, WANG X, LEE T H, JIN H, MARLER B, GUO H, KISSINGER J C, PATERSON A H. MCScanX: A toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research, 2012, 40(7): e49. DOI:10.1093/nar/gkr1293 |
| [43] |
CHEN C, CHEN H, ZHANG Y, THOMAS H R, FRANK M H, HE Y, XIA R. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194-1202. DOI:10.1016/j.molp.2020.06.009 |
| [44] |
LESCOT M. PlantCARE, a database of plant cis-acting regulatory elements and a portal to tools for in silico analysis of promoter sequences[J]. Nucleic Acids Research, 2002, 30(1): 325-327. DOI:10.1093/nar/30.1.325 |
| [45] |
SAYERS E W, BECK J, BOLTON E E, BOUREXIS D, BRISTER J R, CANESE K, COMEAU D C, FUNK K, KIM S, KLIMKE W, MARCHLER-BAUER A, LANDRUM M, LATHROP S, LU Z, MADDEN T L, O'LEARY N, PHAN L, RANGWALA S H, SCHNEIDER V A, SKRIPCHENKO Y, WANG J, YE J, TRAWICK B W, PRUITT K D, SHERRY S T. Database resources of the National Center for Biotechnology Information[J]. Nucleic Acids Research, 2021, 49(D1): 10-17. DOI:10.1093/nar/gkaa892 |
| [46] |
LUO J, ZHOU J J, ZHANG J Z. Aux/IAA Gene Family in plants: Molecular structure, regulation, and function[J]. International Journal of Molecular Sciences, 2018, 19(1): 259. DOI:10.3390/ijms19010259 |
| [47] |
SASABE, M. Phosphorylation of NtMAP65-1 by a MAP kinase down-regulates its activity of microtubule bundling and stimulates progression of cytokinesis of tobacco cells[J]. Genes & Development, 2006, 20(8): 1004. |
| [48] |
SCHMUTZ J, CANNON S B, SCHLUETER J, MA J, MITROS T, NELSON W, HYTEN D L, SONG Q, THELEN J J, CHENG J, XU D, HELLSTEN U, MAY G D, YU Y, SAKURAI T, UMEZAWA T, BHATTACHARYYA M K, SANDHU D, VALLIYODAN B, LINDQUIST E, PETO M, GRANT D, SHU S, GOODSTEIN D, BARRY K, FUTRELL-GRIGGS M, ABERNATHY B, DU J, TIAN Z, ZHU L, GILL N, JOSHI T, LIBAULT M, SETHURAMAN A, ZHANG X C, SHINOZAKI K, NGUYEN H T, WING R A, CREGAN P, SPECHT J, GRIMWOOD J, ROKHSAR D, STACEY G, SHOEMAKER R C, JACKSON S A. Genome sequence of the palaeopolyploid soybean[J]. Nature, 2010, 463(7278): 178-183. DOI:10.1038/nature08670 |
| [49] |
KAZAN K, MANNERS J M. Linking development to defense: auxin in plant–pathogen interactions[J]. Trends in Plant Science, 2009, 14(7): 373-382. DOI:10.1016/j.tplants.2009.04.005 |
| [50] |
JAIN M, KHURANA J P. Transcript profiling reveals diverse roles of auxin-responsive genes during reproductive development and abiotic stress in rice[J]. FEBS Journal, 2009, 276(11): 3148-3162. DOI:10.1111/j.1742-4658.2009.07033.x |
| [51] |
SONAWANE A R, PLATIG J, FAGNY M, CHEN C Y, PAULSON J N, LOPES-RAMOS C M, DEMEO D L, QUACKENBUSH J, GLASS K, KUIJJER M L. Understanding tissue-specific gene regulation[J]. Cell Reports, 2017, 21(4): 1077-1088. DOI:10.1016/j.celrep.2017.10.001 |
| [52] |
ZHANG Q, CHEN W, SUN L, ZHAO F, HUANG B, YANG W, TAO Y, WANG J, YUAN Z, FAN G, XING Z, HAN C, PAN H, ZHONG X, SHI W, LIANG X, DU D, SUN F, XU Z, HAO R, LV T, LV Y, ZHENG Z, SUN M, LUO L, CAI M, GAO Y, WANG J, YIN Y, XU X, CHENG T, WANG J. The genome of Prunus mume[J]. Nature Communications, 2012, 3: 1318. DOI:10.1038/ncomms2290 |
| [53] |
MASSON-BOIVIN C, SACHS J L. Symbiotic nitrogen fixation by rhizobia—the roots of a success story[J]. Current Opinion in Plant Biology, 2018, 44: 7-15. DOI:10.1016/j.pbi.2017.12.001 |
| [54] |
GHOSH P K, SAHA P, MAYILRAJ S, MAITI T K. Role of IAA metabolizing enzymes on production of IAA in root, nodule of Cajanus cajan and its PGP Rhizobium sp[J]. Biocatalysis and Agricultural Biotechnology, 2013, 2(3): 234-239. DOI:10.1016/j.bcab.2013.04.002 |
| [55] |
LUO J, ZHOU J J, ZHANG J Z. Aux/IAA gene family in plants: Molecular structure, regulation, and function[J]. International Journal of Molecular Sciences, 2018, 19(1): e259. DOI:10.3390/ijms19010259 |
(责任编辑 杨贤智)
 2022, Vol. 49
2022, Vol. 49




