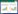文章信息
基金项目
- 河北省现代农业产业技术体系奶牛创新团队建设项目(HBCT2018120205);河北省重点研发项目(20326603D);广东省科学院人才专项(2016GDASRC-0205);北京动物园圈养野生动物技术北京市重点实验室开放课题(ZDK202105)
作者简介
- 贾仲昕(1996—),男,在读硕士生,研究方向为功能性微生物开发,E-mail:786525150@qq.com.
通讯作者
- 秦建华(1964—),男,博士,教授,研究方向为动物疫病防控技术,E-mail:qjhqqq@126.com.
文章历史
- 收稿日期:2022-03-04
2. 广东省科学院动物研究所 / 广东省动物保护与资源利用重点实验室 / 广东省野生动物保护与利用公共实验室,广东 广州 510260
2. Institute of Zoology, Guangdong Academy of Science / Guangdong Key Laboratory of Animal Conservation and Resource Utilization / Guangdong Public Laboratory of Wild Animal Conservation and Utilization, Guangzhou 510260, China
【研究意义】2020年农业农村部全面禁止除中药外一切促生长类药物饲料添加剂的使用,标志着我国畜牧生产和养殖业“无抗时代”的到来。而蛋白酶一方面可以提高动物对蛋白质的消化并促进对养分的吸收,另一方面还可以有效减少进入后肠中的未消化蛋白数量,减少肠道菌群失衡的可能,从而保障动物肠道健康,因此蛋白酶成为抗生素促生长剂替代品的选择之一[1-2]。目前市场上的饲用蛋白酶主要由芽孢杆菌产生,因此探究影响芽孢杆菌产蛋白酶能力的重要基因显得尤为重要。【前人研究进展】芽孢杆菌产生的细胞外蛋白酶被认为参与细菌对营养物质的获取过程。研究发现,枯草芽孢杆菌(Bacillus subtilis)可以产生至少8种独特的胞外或细胞壁相关蛋白酶[3],包括枯草杆菌蛋白酶AprE、中性金属蛋白酶NprE、丝氨酸蛋白酶Epr和Vpr、杆状肽酶F(Bpr)、中性蛋白酶B(NprB)、细胞壁相关蛋白酶WprA和金属蛋白酶Mpr。Yang等[4]研究表明,蛋白酶基因缺失的唯一表型效应是蛋白酶活性的丧失。目前,芽孢杆菌蛋白酶基因的相关报道多集中于研究蛋白酶基因的调控机制以及高产蛋白酶突变株的诱导。Liu等[5]发现degU和spo0A以及kinA的上调部分导致B. pumilus突变株SCU11碱性蛋白酶的高产量。Zhou等[6]研究发现,在地衣芽孢杆菌(B. licheniformis)中反应调节因子Spo0A可能直接参与aprE的转录与激活。Zolfaghari等[7]通过点突变等系统生物学方法,使aprE基因的表达水平和枯草杆菌蛋白酶E(Subtilisin E)的稳定性得到提高。【本研究切入点】随着测序技术的发展,细菌全基因组信息可以更加容易获得。通过比较不同菌种的基因组信息并结合细菌的相关表型,可以深入分析造成细菌表型差异的重要基因[8]。此外,芽孢杆菌全基因组注释信息的完善也为利用比较基因组学分析影响芽孢杆菌产蛋白酶能力的重要基因提供了研究基础。【拟解决的关键问题】本研究对15株芽孢杆菌进行全基因组测序,通过检测15株芽孢杆菌的产蛋白酶能力,利用比较基因组学,挖掘影响芽孢杆菌产蛋白酶能力的重要基因,分析芽孢杆菌基因组中胞外蛋白酶基因与产蛋白酶能力的关联性,为产蛋白酶芽孢杆菌的开发提供依据。
1 材料与方法 1.1 试验材料供试15株菌株来源于广东省科学院动物研究所,具体如下:
枯草芽孢杆菌(B. subtiliis)N1282-4at、N1108-5at、N1303-2Ay,贝莱斯芽孢杆菌(B. velezensis)C1,蜡状芽孢杆菌(B. cereus)N435-1,高地芽孢杆菌(B. altitudinis)N435-3,巨大芽孢杆菌(B. megaterium)N447-3、B1,假真菌样芽孢杆菌(B. pseudomycoides)N451-2,海洋芽孢杆菌(B. oceanisediminis)N497-ZHONG,阿耶波多氏芽孢杆菌(B. aryabhattai)B2,副短短芽孢杆菌(Brevibacillus parabrevis)B3,芽孢杆菌(Bacillus sp.)N470-1、N447-1、A1。
培养基配方参照文献[9-10]并稍作优化。蛋白酶发酵培养基配方(g/L):酪蛋白10.00、KH2PO4 0.360、NaH2PO4·2H2O 0.360、MgSO4·7H2O 0.500、FeSO4·7H2O 0.002、CaCl2·2H2O 0.480、NaCl 5.00。筛选培养基配方(g/L):酪蛋白10.0、牛肉膏粉3.0、Na2HPO4 2.0、NaCl 5.0、琼脂15.0、溴麝香草酚蓝0.05。MHB培养基配方(g/L):酪蛋白水解物17.5、牛肉浸粉5.0、淀粉1.5。NA培养基配方(g/L):蛋白胨10、牛肉膏粉3.0、NaCl 5.0、琼脂17.0。
1.2 芽孢杆菌产蛋白酶能力检测1.2.1 产蛋白酶菌株初筛 取出保藏的细菌冻存液接种于NA培养基上,35 ℃培养24 h,挑取单菌落接种至MHB培养基中,32 ℃、160 r/min振荡培养24 h。将菌液梯度稀释,吸取100 μL涂布于酪蛋白琼脂上,35 ℃培养48 h。用游标卡尺测量水解圈直径(H)和菌落直径(C)并计算H与C的比值H/C。
1.2.2 蛋白酶活性测定 挑取初筛时酪蛋白琼脂上的单菌落,接种至MHB培养基中,32 ℃、160 r/ min振荡培养24 h后,将菌液按2%(V/V)接种至发酵培养基中,32 ℃、160 r/min振荡培养24 h,调整菌液浓度OD600为0.500,9 000 r/min离心15 min,上清液即为粗酶液。蛋白酶活性测定参考GB/T 23527-2009福林酚法并做适当修改。测定前先将粗酶液与酪蛋白溶液于40 ℃水浴预热5 min。
(1)试验组:取1 mL粗酶液加入1 mL 10 g/L酪蛋白溶液,40 ℃水浴10 min。之后立即加入3 mL 0.4 mol/L三氯乙酸溶液终止反应。静置10 min后12 000 r/min离心10 min。吸取上清液1 mL,加入5.0 mL 0.4 mol/L碳酸钠溶液和1 mL福林试剂使用溶液,40 ℃水浴显色20 min,用紫外分光光度计在680 nm处测定样品的OD680。
(2)对照组:先加三氯乙酸使蛋白酶失活再加酪蛋白,其他操作同上。
计算酶活性(X):
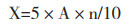
|
式中,X为样品酶活性(U/mL),A为由样品测得的净OD值,查标准曲线所得相对应的酪氨酸含量,n为酶液的稀释倍数,10代表反应10 min,5代表 5 mL反应液。
酶活性定义:在40 ℃下,每分钟内酪蛋白被水解产生1 μg酪氨酸为1个蛋白酶活性单位(U/mL)。
1.3 芽孢杆菌全基因组测序将15株菌株送至北京百迈客生物科技有限公司,基于Nanopore测序技术平台进行全基因组测序。试验流程[11-12]按照ONT公司提供的标准执行,包括样品质量检测、文库构建、文库质量检测和文库测序等流程。利用预测得到的基因序列与COG、KEGG、Swiss-Prot、TrEMBL、Nr等功能数据库进行BLAST比对,得到基因功能注释结果。
1.4 芽孢杆菌蛋白酶基因分析查找芽孢杆菌中主要报道的8种特征化细胞外或细胞壁相关蛋白酶[13](AprE、NprE、Bpr、Epr、Vp、NprB、WprA、Mpr),以及从细菌全基因组数据注释结果中发现的丝氨酸蛋白酶(Serine protease,AprX[14-15])的相关基因数据,包括菌株基本信息,蛋白酶基因的种类、数量和转录方向,蛋白酶基因间的相对距离等。利用IBS线上作图工具(http://ibs.biocuckoo.org)进行绘图。从全基因组数据结果中获取菌株的16S rDNA序列,结合NCBI数据库中已有的数据,利用MEGA X软件绘制系统发育树。
1.5 菌株蛋白酶基因的mRNA水平检测按照菌株产蛋白酶的相关表型,选取菌株B. altitudinis N435-3、B. oceanisediminis N497-ZHONG、B. velezensis C1、B. subtiliis N1282-4at进行蛋白酶基因的mRNA水平检测。根据细菌相应蛋白酶基因序列,由生工生物工程(上海)股份有限公司设计与合成菌株引物(表 1)。提取上述菌株在不同培养基(NB培养基和发酵培养基)条件下的总RNA,以总RNA为模板反转录为cDNA,以cDNA为模板进行qPCR,16S rDNA为内参基因。
试验数据采用SPSS 19.0软件进行单因素方差分析(One-way ANOVA)。
2 结果与分析 2.1 Bacillus sp. A1和B. velezensis C1产蛋白酶能力较强在选取的15株菌株中,有6株菌株(Bacillus sp. A1、B. velezensis C1、B. altitudinis N435-3、B. subtiliis N1282-4at、B. subtiliis N1108-5at、B. subtiliis N1303-2Ay)在接种培养48 h,菌株透明圈直径与菌落直径的比值(H/C)显著高于其他9株菌株。Bacillus sp. A1和B. velezensis C1的H/C值最高,其次为B. subtiliis N1282-4at、B. subtiliis N1108-5at、B. subtiliis N1303-2Ay和B. altitudinis N435-3(图 1)。由于H/C值在一定程度上反映了细菌产蛋白酶能力[16],因此上述6株菌株的产蛋白酶能力相对较强。
|
| 柱上小写英文字母不同者表示差异显著 Different lowercase letters above the column indicate significant differences 图 1 产蛋白酶菌株初筛的H/C值 Fig. 1 H/C value of primary screening of protease production strains |
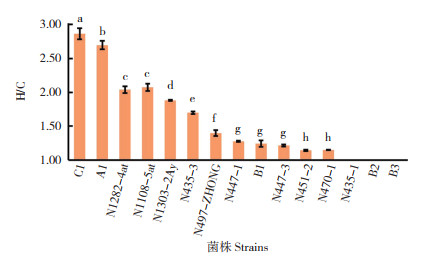
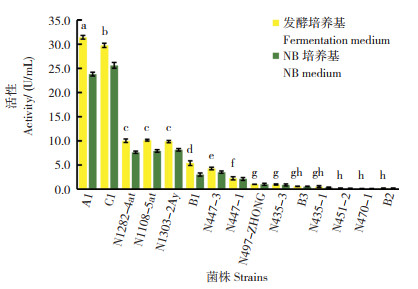
由图 2可知,15株菌株中蛋白酶活性最高的2株菌株分别为Bacillus sp. A1(31.40±0.40 U/mL)和B. velezensis C1(29.75±0.43 U/mL),蛋白酶活性均达到30 U/mL,显著高于其他13株菌株;其次为B. subtiliis N1282-4at、B. subtiliis N1108-5at、B. subtiliis N1303-2Ay等3株菌株,蛋白酶活性在7.40~8.57 U/mL之间,三者差异不显著;B. megaterium N447-3和B. megaterium B1,蛋白酶活性仅为5 U/mL左右;其他8株菌株的蛋白酶活性均小于2 U/mL。菌株在不同培养基条件下酶活性不同,使用发酵培养基(底物酪蛋白为唯一氮源)后,15株菌株的中性蛋白酶活性普遍有所提升,但菌株间蛋白酶活性的相对大小并未发生改变。
|
| 柱上小写英文字母不同者表示差异显著 Different lowercase letters above the column indicate significant differences 图 2 菌株在不同培养基条件下的蛋白酶活性检测结果 Fig. 2 Detection results of protease activity of strains under different medium conditions |
2.2 不同菌种携带不同的胞外蛋白酶相关基因组合
通过对菌株全基因组上蛋白酶相关基因的筛查与统计,芽孢杆菌属细菌携带有8种胞外蛋白酶相关基因,包括aprE、aprX、nprE、epr、vpr、nprB、wprA和mpr。对上述基因的种类、转录方向、序列长度和染色体组中相对位置进行分析,可将其分为7种基因型(G1~G7,图 3)。3株B. subtiliis菌株N1282-4at、N1108-5at、N1303-2Ay属于G1型,特点是染色体组上携带有上述8种胞外蛋白酶基因。Bacillus sp. A1、B. velezensis C1属于G2型,染色体组上携带aprX、vpr、epr、mpr。B. altitudinis N435-3属于G3型,相比G1型缺少mpr、nprB和nprE。B. oceanisediminis N497-ZHONG属于G4型,染色体组上携带aprX、vpr、nprB。B. megaterium N447-3、B. megaterium B1、B. aryabhattai B2属于G5型,染色体组上仅携带wprA、vpr,但同时存在2个vpr。G6型可分为3个亚型G6.1、G6.2和G6.3,其中B. cereus N435-1属于G6.1型,菌株染色体组上携带vpr、nprB和mpr;Bacillus N447-1属于G6.2型,相比G6.1型缺少mpr;而Bacillus N470-1则属于G6.3型,染色体组上仅携带vpr。B. pseudomycoides N451-2属于G7型,染色体组=上仅携带nprB。菌株Brevibacillus parabrevis B3的染色体上则没有携带上述8种基因。
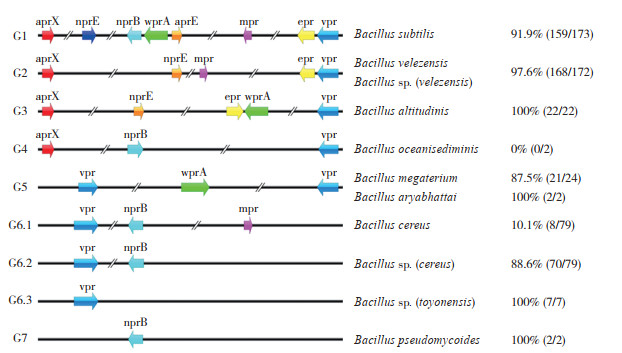
|
| 图 3 芽孢杆菌属菌株蛋白酶的基因型分类结果 Fig. 3 Genotype classification results of Protease in Bacillus strains |
通过与NCBI数据库现有芽孢杆菌属细菌的全基因组数据(截至2021年11月21日)进行比对,结果(图 3)发现,在NCBI数据库有173株B. subtiliis具有全基因数据,其中159株符合G1型基因分布(91.9%,159/173);B. velezensis有97.6%(168/172)符合G2型;B. altitudinis 100%(22/22)符合G3型;没有B. oceanisediminis符合G4型,考虑到NCBI数据库中仅有2株B. oceanisediminis的全基因组数据,因此存在较大偶然性;B. megaterium有87.5%(21/24)、B. aryabhattai有100%(2/2)符合G5型;而B. cereus中仅有10.1%(8/79)符合G6.1型,更多的是88.9%(70/79)符合G6.2型,B. toyonensis 100%(7/7)符合G6.3型;B. pseudomycoides 100%(2/2)符合G7型。
2.3 细菌蛋白酶基因分型与其进化关系存在关联绘制基于细菌16S rDNA序列的系统发育树(图 4),15株菌株(红色)可分为Bacillus和Brevibacillus两个属,其中14株属于Bacillus,1株属于Brevibacillus。Bacillus属可分为3个分支,其中G1、G2、G3型所属菌株的亲缘关系接近为一个分支(绿色),G4、G5型所属菌株的亲缘关系接近为一个分支(黄色),G6、G7型所属菌株的亲缘关系接近为一个分支(蓝色)。Brevibacillus parabrevi B3作为一个单独的分支出现,表明其与其他细菌物种的亲缘关系较远。通过与细菌的蛋白酶基因分型(图 3)进行比对可发现,菌株与枯草芽孢杆菌亲缘关系越近,其染色体上携带的胞外蛋白酶基因种类越齐全。
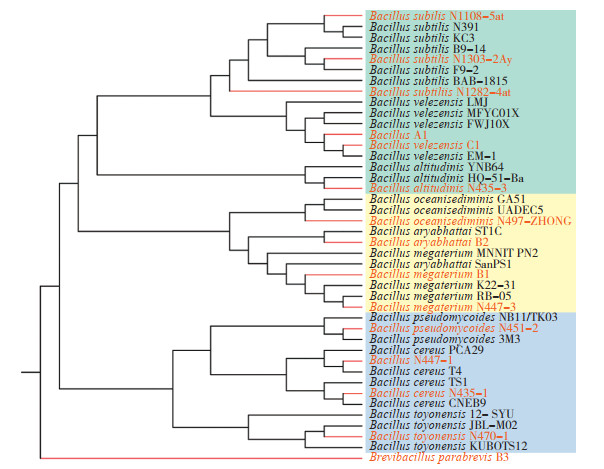
|
| 图 4 基于细菌16S rDNA基因序列的系统发育树 Fig. 4 Phylogenetic tree based on 16S rDNA sequences of strains |
2.4 芽孢杆菌蛋白酶基因分析
结合菌株的基因型分型结果(图 3)与产蛋白酶能力检测结果(图 2)发现,mpr、epr、vpr、nprB和wprA等5个蛋白酶基因在产蛋白酶能力高和产酶能力低的菌株中均有携带情况。nprE基因仅有G1型菌株携带,但其产酶能力低于不携带nprE的G2型。存在aprX和aprE的G1型和G2型菌株,其H/C值和蛋白酶活性也均高于其他菌株,而也携带aprX和aprE的G3型和携带aprX的G4型菌株虽然其蛋白酶活性较低,但在酪蛋白琼脂上的H/C也是高于除G1型和G2型菌株外的其他菌株。G1~G4型菌株的共同特征是携带aprX或aprE基因,因此本研究将影响菌株产蛋白酶能力的重要基因初步确定为aprE和aprX。
2.5 不同菌种间蛋白酶基因aprX表达差异显著为了探究影响菌株产蛋白酶能力的重要基因是否为aprX和aprE,本研究检测了B. velezensis C1、B. subtiliis N1282-4at、B. altitudinis N435-3、B. oceanisediminis N497-ZHONG在不同培养基(NB培养基和发酵培养基)条件下aprX、aprE的mRNA水平。在NB培养基(图 5A,以B. subtiliis N1282-4at作为对照),B. velezensis C1的aprX mRNA相对水平显著高于其他3株菌株,为B. subtiliis N1282-4at的2.7倍左右;B. altitudinis N435-3则仅为N1282-4at的60%;而B. oceanisediminis N497-ZHONG则无表达或相比B. subtiliis N1282-4at表达量过小。在发酵培养基(图 5C),相比NB培养基,B. velezensis C1的aprX mRNA相对水平上升为B. subtiliis N1282-4at的3.6倍左右;B. altitudinis N435-3也有所升高,为B. subtiliis N1282-4at的80%;而B. oceanisediminis N497-ZHONG仍无表达或相比B. subtiliis N1282-4at表达量过小。由图 5B、图 5D可见,发酵培养基中B. altitudinis N435-3的aprE相对表达水平为B. subtiliis N1282-4at的约1.2倍,差异显著,其余差异均不显著。根据结果推测,aprX是影响芽孢杆菌产蛋白酶能力的重要基因。
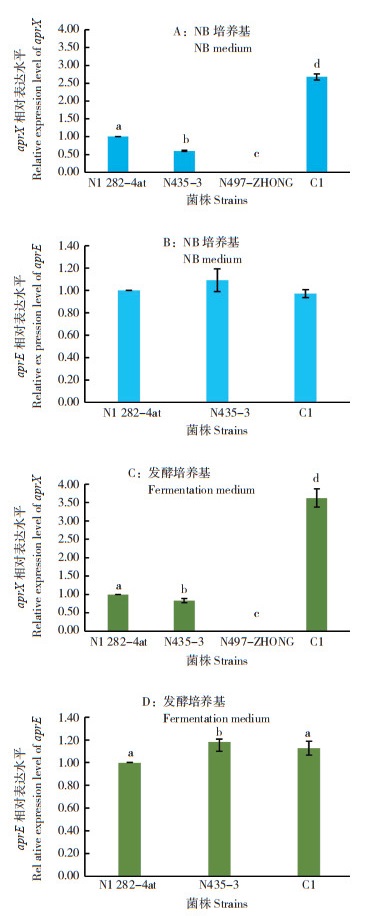
|
| 柱上小写英文字母不同者表示差异显著 Different lowercase letters above the column indicate significant differences 图 5 不同培养基菌株aprX和aprE的相对表达水平 Fig. 5 Relative expression levels of aprX and aprE under different medium conditions |
3 讨论
本研究通过对15株菌株进行全基因组测序,获得了菌株的全基因组信息,并根据菌株染色体上携带的蛋白酶基因数量、种类、转录方向将其分为7种基因型,并进行比较基因组学分析。在15株菌株中仅有G1型的B. subtiliis拥有最全的8种蛋白酶基因,其他菌株都缺乏多种蛋白酶基因。通过对NCBI数据库中已有菌株全基因组信息的比对,发现不同来源的相同菌种绝大多数归为同一基因型,可以认为在芽孢杆菌属不同来源的相同菌种间,蛋白酶基因携带情况保守。
有研究指出,枯草芽孢杆菌的主要胞外蛋白酶分别是aprE编码的枯草杆菌蛋白酶和nprE编码的中性金属蛋白酶[17-18]。但本研究结果显示,仅G3型菌株B. altitudinis N435-3的aprE mRNA水平在发酵培养基条件下是G1型菌株B. subtiliis N1282-4at的约1.2倍,其他情况差异不显著。而nprE仅G1型菌株携带,但缺乏nprE的G2型菌株的产蛋白酶能力高于G1型菌株。可能是因为G1型菌株的nprE表达水平过低使NprE的活性没有表现出来,同时aprE表达水平与G2和G3型细菌处于同一水平,而G2型的aprX表达水平却显著高于G1型菌株所致。
芽孢杆菌属菌株通常在指数生长期后期开始大量产生胞外蛋白酶,这使得细菌可以高效利用外界的营养物质,从而确保其正常的生长发育[19]。控制芽孢杆菌胞外蛋白酶表达的调节途径非常复杂,如AbrB、SinR和ScoC等调节因子可以抑制芽孢杆菌蛋白酶基因的表达,但Spo0A可以抑制AbrB和SinR的活性从而促进蛋白酶的产生[19]。此外,aprE和nprE还受到全局转录调节因子CodY的强大且直接的负反馈调节,但由于CodY同时也抑制ScoC[20],因此CodY除了是aprE和nprE的直接负调节因子外,还通过抑制scoC的高水平表达充当相同基因的间接正调节因子。由于CodY和ScoC都是aprE和nprE的强阻遏物,并且CodY会在含有过量氨基酸的培养基中具有活性,但随着氨基酸耗尽会失去活性[21]。考虑到G1型和G2型菌株的进化关系接近,这或许可以解释为何G1型细菌的产蛋白酶能力低于G2型细菌。本研究在细菌生长的后期阶段检测了细菌产蛋白酶的能力和基因的表达,而此时营养物质的消耗导致CodY的部分失活,从而减轻了对ScoC的抑制,使ScoC水平升高,持续抑制aprE和nprE的表达。因此结合荧光定量PCR结果,aprE和nprE由于CodY的失活导致对ScoC抑制的减弱,从而受到ScoC的强抑制,使细菌aprE和nprE的mRNA水平降低。因此除aprE和nprE外,aprX是影响芽孢杆菌属细菌产胞外蛋白酶能力的又一重要基因。
蛋白酶是一种诱导酶,其在细菌内部的生物合成受到多种因素诱导(如蛋白酶底物和底物类似物),同时也会受到细菌生存环境中易利用氮源(如氨基酸和铵盐等)的阻遏[22-23]。因此,当外界环境中存在大量营养物质时,细菌只会产生少量蛋白酶来维持必要的生理活动,只有在外界环境缺乏易利用氮源时,细菌才会大量产生蛋白酶,去获取环境中其他难利用的大分子蛋白质。这也解释了为何细菌在发酵培养基(唯一氮源为酪蛋白)条件下aprX和aprE的表达水平比在营养肉汤培养基条件下高。
4 结论本研究对15株芽孢杆菌进行全基因组测序获得了菌株的全基因组信息,并根据菌株携带的蛋白酶基因情况进行了基因分型,通过对NCBI数据库中已有菌株全基因组信息的比对,发现芽孢杆菌不同来源的相同菌种间蛋白酶基因携带情况保守。通过构建进化树并与基因分型结果进行比对,发现芽孢杆菌属菌株与枯草芽孢杆菌亲缘关系越近,携带的胞外蛋白酶基因种类越齐全。通过对15株芽孢杆菌进行产蛋白酶能力检测,并进行比较基因组学分析和qRT-PCR验证,发现aprX是影响芽孢杆菌属细菌产蛋白酶能力的重要基因。
| [1] |
王何中. 养殖业抗生素使用现状及应对措施[J]. 现代农业科技, 2019(22): 164-166. WANG H Z. Current situation and countermeasures of antibiotic used in aquaculture industry[J]. Modern Agricultural Science and Technology, 2019(22): 164-166. |
| [2] |
伍楚妍, 黄晓冰, 刘少君, 李洋, 谢为天, 徐春厚. 海洋源芽孢杆菌的分离鉴定及其消化酶代谢产物的测定[J]. 广东农业科学, 2021, 48(7): 137-144. DOI:10.16768/j.issn.1004-874X.2021.07.017 WU C Y, HUANG X B, LIU S J, LI Y, XIE W T, XU C H. Isolation and identification of marine Bacillus and determination of its digestive enzyme metabolites[J]. Guangdong Agricultural Sciences, 2021, 48(7): 137-144. DOI:10.16768/j.issn.1004-874X.2021.07.017 |
| [3] |
CHATTERJEE J, GIRI S, MAITY S, SINHA A, RANJAN A, RAJSHEKHAR, GUPTA S. Production and characterization of thermostable alkaline protease of Bacillus subtilis (ATCC 6633) from optimized solid-state fermentation[J]. Biotechnology and Applied Biochemistry, 2015, 62(5): 709-718. DOI:10.1002/bab.1309 |
| [4] |
YANG M Y, FERRARI E, HENNER D J. Cloning of the neutral protease gene of Bacillus subtilis and the use of the cloned gene to create an in vitro-derived deletion mutation[J]. Journal of Bacteriology, 1984, 160(1): 15-21. DOI:10.1128/jb.160.1.15-21.1984 |
| [5] |
LIU Y C, HAN L L, CHEN T Y, LU Y B, FENG H. Characterization of a protease hyper-productive mutant of Bacillus pumilus by comparative genomic and transcriptomic analysis[J]. Current Microbiology, 2020, 77(11): 3612-3622. DOI:10.1007/s00284-020-02154-5 |
| [6] |
ZHOU C, ZHOU H, FANG H, JI Y, WANG H, LIU F, ZHANG H, LU F. Spo0A can efficiently enhance the expression of the alkaline protease gene aprE in Bacillus licheniformis by specifically binding to its regulatory region[J]. International Journal of Biological Macromolecules, 2020, 159: 444-454. DOI:10.1016/j.ijbiomac.2020.05.035 |
| [7] |
ZOLFAGHARI EMAMEH R, KAZOKAITĖ J, YAKHCHALI B. Bioinformatics analysis of extracellular subtilisin E from Bacillus subtilis[J]. Journal of Biomolecular Structure & Dynamics, 2021, 4: 1-8. DOI:10.1080/07391102.2021.1894979 |
| [8] |
曾倩倩, 刘岩, 朱墨, 邱宗波. 基于转录组测序挖掘小麦叶片抗旱相关基因[J]. 广东农业科学, 2022, 49(2): 1-8. DOI:10.16768/j.issn.1004-874X.2022.02.001 ZENG Q Q, LIU Y, ZHU M, QIU Z B. Screening of candidate genes resistant to drought stress in wheat leaves based on transcriptome sequencing[J]. Guangdong Agricultural Sciences, 2022, 49(2): 1-8. DOI:10.16768/j.issn.1004-874X.2022.02.001 |
| [9] |
万文结, 薛芷筠, 张泽文, 李晓华, 程国军, 何冬兰. 一株产碱性蛋白酶菌株的筛选鉴定及酶学特性研究[J]. 微生物学报, 2017, 57(5): 737-747. DOI:10.13343/j.cnki.wsxb.20160419 WAN W J, XUE Z J, ZHANG Z W, LI X H, CHENG G J, HE D L. Isolation and identification of an alkaline protease producing strain and study on enzymatic properties[J]. Acta Microbiologica Sinica, 2017, 57(5): 737-747. DOI:10.13343/j.cnki.wsxb.20160419 |
| [10] |
柯野, 伍嘉慧, 曾松荣, 周威. 一株产蛋白酶菌株的筛选鉴定及酶学性质分析[J]. 广东农业科学, 2015, 42(2): 137-141, 193. DOI:10.16768/j.issn.1004-874X.2015.02.002 KE Y, WU H J, ZENG S R, ZHOU W. Screening and identification of a strain producing proteases and analysis on its enzymatic properties[J]. Guangdong Agricultural Sciences, 2015, 42(2): 137-141, 193. DOI:10.16768/j.issn.1004-874X.2015.02.002 |
| [11] |
DEAMER D, AKESON M, BRANTON D. Three decades of Nanopore sequencing[J]. Nature Biotechnology, 2016, 34(5): 518-524. DOI:10.1038/nbt.3423 |
| [12] |
MAGI A, SEMERARO R, MINGRINO A, GIUSTI B, D'AURIZIO R. Nanopore sequencing data analysis: state of the art, applications and challenges[J]. Briefings Bioinformatics, 2018, 19(6): 1256-1272. DOI:10.1093/bib/bbx062 |
| [13] |
BARBIERI G, VOIGT B, ALBRECHT D, HECKER M, ALBERTINI A M, SONENSHEIN A L, FERRARI E, BELITSKY B R. CodY regulates expression of the Bacillus subtilis extracellular proteases Vpr and Mpr[J]. Journal of Bacteriology, 2015, 197(8): 1423-1432. DOI:10.1128/JB.02588-14 |
| [14] |
VALBUZZI A, FERRARI E, ALBERTINI A M. A novel member of the subtilisin-like protease family from Bacillus subtilis[J]. Microbiology (Reading, England), 1999, 145(11): 3121-3127. DOI:10.1099/00221287-145-11-3121 |
| [15] |
ZHANG C, BIJL E, SVENSSON B, HETTINGA K. The extracellular protease aprx from Pseudomonas and its spoilage potential for uht milk: a review[J]. Comprehensive Reviews in Food Science and Food Safety, 2019, 18(4): 834-852. DOI:10.1111/1541-4337.12452 |
| [16] |
刘晓艳, 封健, 韩傲, 高宁, 连莲, 国立东. 高产中/碱性蛋白酶的枯草芽孢杆菌W1菌株的筛选及条件优化[J]. 现代食品科技, 2020, 36(4): 157-163. DOI:10.13982/j.mfst.1673-9078.2020.4.021 LIU X Y, FENG J, HAN A, GAO N, LIAN L, GUO L D. Isolation and optimization of Bacillus subtilis W1 strain with high neutral/alkaline protease activity[J]. Modern Food Science and Technology, 2020, 36(4): 157-163. DOI:10.13982/j.mfst.1673-9078.2020.4.021 |
| [17] |
KAWAMURA F, DOI R H. Construction of a Bacillus subtilis double mutant deficient in extracellular alkaline and neutral proteases[J]. Journal of Bacteriology, 1984, 160(1): 442-444. DOI:10.1128/jb.160.1.442-444.1984 |
| [18] |
CONTESINI FJ, MELO RR, SATO HH. An overview of Bacillus proteases: from production to application[J]. Critical Reviews in Biotechnology, 2018, 38(3): 321-334. DOI:10.1080/07388551.2017.1354354 |
| [19] |
TOYMENTSEVA A A, MASCHER T, SHARIPOVA M R. Regulatory characteristics of Bacillus pumilus protease promoters[J]. Current Microbiology, 2017, 74(5): 550-559. DOI:10.1007/s00284-017-1212-3 |
| [20] |
BARBIERI G, ALBERTINI A M, FERRARI E, SONENSHEIN A L, BELITSKY B R. Interplay of CodY and ScoC in the regulation of major extracellular protease genes of Bacillus subtilis[J]. Journal of Bacteriology, 2016, 198(6): 907-920. DOI:10.1128/JB.00894-15 |
| [21] |
SLACK F J, SERROR P, JOYCE E, SONENSHEIN A L. A gene required for nutritional repression of the Bacillus subtilis dipeptide permease operon[J]. Molecular Microbiology, 1995, 15(4): 689-702. DOI:10.1111/j.1365-2958.1995.tb02378.x |
| [22] |
SUN Y, QIAN Y, ZHANG J, WANG Y, LI X, ZHANG W, WANG L, LIU H, ZHONG Y. Extracellular protease production regulated by nitrogen and carbon sources in Trichoderma reesei[J]. Journal of Basic Microbiology, 2021, 61(2): 122-132. DOI:10.1002/jobm.202000566 |
| [23] |
ELLEUCH J, HADJ KACEM F, BEN AMOR F, HADRICH B, MICHAUD P, FENDRI I, ABDELKAFI S. Extracellular neutral protease from Arthrospira platensis: Production, optimization and partial characterization[J]. International Journal of Biological Macromolecules, 2021, 167: 1491-1498. DOI:10.1016/j.ijbiomac.2020.11.102 |
(责任编辑 崔建勋)
 2022, Vol. 49
2022, Vol. 49