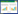文章信息
基金项目
- 广东省林业科技创新项目(2018KJCX025)
作者简介
- 李落叶(1974—),女,博士,助理研究员,研究方向为植物保护,E-mail:liluoye822@163.com.
通讯作者
- 柯碧英(1976—),女,硕士,副教授,研究方向为森林培育,E-mail:22919020@qq.com.
文章历史
- 收稿日期:2022-03-24
【研究意义】厚朴(Magnolia officinalis) 是我国特有的珍贵药用植物,主产于四川、湖北、浙江,在江西、湖南、广东、广西、贵州、陕西、甘肃等地也有分布。厚朴主要以干皮、根皮、枝皮入药,具有燥湿消痰、下气除满的作用,主治湿滞伤中、脘痞吐泻、食积气滞、腹胀便秘、痰饮喘咳等,是200多种中成药的主要配方原料[1],市场需求旺盛。由于厚朴野生资源消耗殆尽,当前药材市场上的厚朴主要来源于人工药源林,药材质量因种源、生境及树龄等不同而参差不齐,药材品质难以得到保证。据报道,厚朴药材有效成分含量与地理种源显著相关,不同种源的药效成分含量差异大[2],其有效成分含量主要由遗传因素决定[3]。因此,鉴定厚朴种源,选育、推广优良的厚朴品种是保证药材质量的基础。【前人研究进展】随着分子生物学技术的发展,分子标记技术已被广泛用于重要性状基因标记定位、种源或种子纯度鉴定、遗传多样性分析、品种指纹图谱绘制、分子标记辅助育种等方面[4-6]。中药材方面,SSR、RAPD、RFLP等标记技术已经用于甄别中药材真伪、正品与替代品,用于中药材遗传多样性鉴定、产地鉴别、年限鉴别、品种与杂交种质纯度鉴定以及分子标记辅助育种等方面[7-8]。有关厚朴分子标记的应用已有报道,比如利用AFLP进行厚朴遗传背景分析,利用RAPD鉴别厚朴伪品,利用SRAP标记构建厚朴种质资源指纹图谱[9],但这些标记技术存在步骤繁琐、技术要求高,或稳定性和重复性差等问题。近几年开发了稳定性好、操作相对简便的厚朴SSR标记[10-11],但是可用的SSR标记数量太少。【本研究切入点】总的来说,厚朴分子标记研究不多,可利用的分子标记少,难以满足需要。TRAP是一种基于已知cDNA或EST序列信息的PCR标记,是以已知cDNA或EST序列设计一条固定引物,和另一条随机引物配对使用,进行PCR反应后在不同品种或个体间表现出不同带型的过程,是控制表型差异的目标基因区多态性的表现。TRAP标记具有简单、高效、多态性高、重复性好等优点[12]。目前,对于厚朴的遗传背景了解很少,更没有全基因组序列信息可用,但杨旭等对厚朴根皮、茎皮和叶的转录组进行了测序分析,并公布了相关cDNA序列信息[10],这为开发厚朴TRAP标记提供了方便。【拟解决的关键问题】本研究首次利用厚朴cDNA序列设计开发厚朴TRAP标记,并用TRAP标记联合厚朴SSR标记,对广东乐昌龙山林场保存的厚朴进行遗传多样分析,构建了它们之间的亲缘关系树状图,为后续开展厚朴优株鉴别、优良品种选育奠定基础,为厚朴种质资源的保护和利用提供支持,有利于促进厚朴产业健康发展。
1 材料与方法 1.1 试验材料厚朴叶片于2020年10月采自广东乐昌龙山林场,种源来自江西省各地,共采集了14株厚朴树的无病嫩叶,包括L1、L8、L11、L12、L14、L15、L19等7株母树和Ls1、Ls8、Ls11、Ls12、Ls14、Ls15、Ls19等7株子代,叶片用液氮速冻后保存于-80 ℃冰箱备用。
1.2 厚朴基因组DNA提取利用离心柱型植物基因组DNA提取试剂盒(北京鼎国昌盛生物技术有限责任公司)提取厚朴基因组DNA。剪取约100 mg叶片,在液氮中快速研磨成粉状后,转移至1.5 mL灭菌离心管中,然后按照试剂盒说明书进行提取DNA,最后溶于100 μL TE buffer中。
用核酸定量仪(Thermo ScientificTM NanoDropTM One) 检测各样品DNA的浓度和质量,根据测定结果,用TE将各样品DNA的浓度调至50 ng/μL,-20 ℃冰箱保存备用。
1.3 厚朴种群遗传多样性分析1.3.1 TRAP标记分析 (1) 固定引物设计:从公共数据库GenBank下载厚朴cDNA序列,用cDNA序列在NCBI网站(National Center for Biotechnology Information Search database, https://www.ncbi.nlm.nih.gov)进行Primer BLAST搜索,主要参数设置:长度18~20 bp,退火温度50~60 ℃。每段cDNA合成1条固定引物。
(2) 随机引物设计:与SRAP引物相同,针对启动子或内含子中富含AT核心区的任意序列设计,本研究采用SRAP随机引物em1~em5,引物序列如下[13]:
em1: 5' GACTGCGTACGAATTAAT 3'
em2: 5' GACTGCGTACGAATTTGC 3'
em3: 5' GACTGCGTACGAATTGAC 3'
em4: 5' GACTGCGTACGAATTTGA 3'
em5: 5' GACTGCGTACGAATTAAC 3'
所有引物由上海捷瑞生物工程有限公司合成。
(3) TRAP标记扩增条件:反应体系共20 μL,包括10 μL的2×Ex Taq Premix(Takara公司生产),1 μL固定引物(10 μmol/L),0.2 μL随机引物(10 μmol/L),1 μL厚朴基因组DNA(50 ng/μL)和7.8 μLddH2O。
PCR反应程序参考冯俊彦等[14]的方法略有改动:94 ℃预变性4 min;94 ℃变性45 s、40 ℃退火45 s、72 ℃延伸1 min,5个循环;94 ℃变性45 s、52 ℃退火45 s、72 ℃延伸1 min,35个循环;72 ℃延伸7 min。PCR仪为BIO-RAD S1000 Thermal Cycler。
(4) PCR产物电泳检测:采用聚丙烯酰胺凝胶电泳。每个扩增产物加入5.0 µL上样缓冲液(北京鼎国昌盛生物技术有限责任公司),混匀;用微量进样器取3.0 µL在3% 聚丙烯酰胺凝胶上电泳,分子量标记为100 bp Ladder marker(北京鼎国昌盛生物技术有限责任公司)。以1 × TBE为电泳缓冲液,100 V电压电泳60 min。DYY- Ⅲ型垂直电泳系统为北京市六一仪器厂产品。电泳结束后,取出凝胶在蒸馏水中漂洗1 min,重复2次,再转入0.1% AgNO3溶液中染色10 min,然后用蒸馏水漂洗2次,最后用显影液适度着色后转入蒸馏水中。
1.3.2 SSR标记分析 (1) 引物:参考麦静[11]的研究,选择多态性较好的来自厚朴及其近缘种共13对SSR引物,引物序列信息见表 1。
(2) SSR标记扩增条件:反应体系共25 μL,包括12.5 μL的2×Ex Taq Premix(Takara公司生产),1 μL正向引物(10 μmol/L),1 μL反向引物(10 μmol/L),1 μL厚朴基因组DNA(50 ng/μL) 和9.5 μL ddH2O。
PCR反应程序为:94 ℃预变性4 min;94 ℃变性30 s、55 ℃退火30 s、72 ℃延伸1 min,35个循环;最后72 ℃延伸10 min,4 ℃保存。
PCR仪为BIO-RAD S1000 Thermal Cycler。
(3) PCR产物电泳检测:采用聚丙烯酰胺凝胶电泳,具体方法与1.3.1(4)的检测方法相同。
1.4 数据分析采用二进位制记录TRAP标记和SSR标记的扩增产物中1 kb以内清晰的电泳条带,在相同迁移率处有带的记为1,无带的记为0,按照NTSYSpc2.10e软件要求的格式,将结果记录在Excel表中,利用NTSYSpc2.10e进行统计及聚类分析。
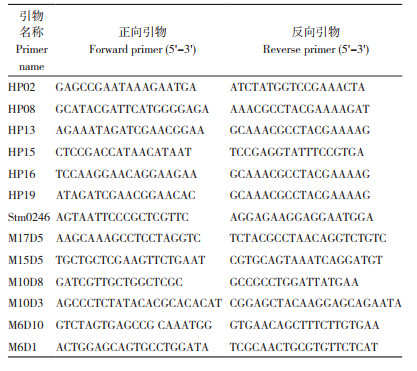
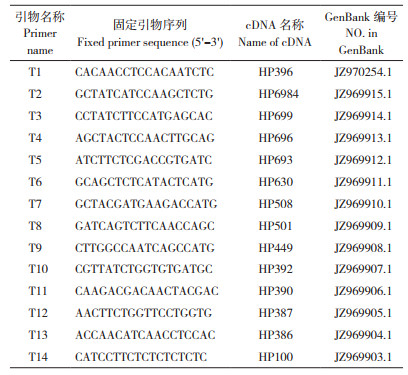
2 结果与分析 2.1 TRAP引物开发根据厚朴cDNA序列信息,共设计了14条固定引物(表 2),分别与em1~dm5共5条随机引物组对,获得70对引物。经过初筛后,获得T1-em2、T3-em2、T4-em1、T6-em3、T6-em4、T11-em5和T12-em5共7对扩增条带清晰、多态性好的引物,用于厚朴材料遗传多样性分析(图 1)。
|
|
A: 引物对T4 + em1;B:引物对T6+em4; 1~14代表 14个厚朴材料,依次为L1、Ls1、L8、Ls8、L11、Ls11、L12、Ls12、L14、Ls14、L15、Ls15、L19、Ls19 A: Primer pair“T4 + em1”; B: Primer pair“T4 + em1”; 1-14 represent the fourteen Magnolia officinalis: L1, Ls1, L8, Ls8, L11, Ls11, L12, Ls12, L14, Ls14, L15, Ls15, L19 and Ls19 图 1 部分TRAP标记对14个厚朴材料基因组DNA的扩增产物电泳结果 Fig. 1 Electrophoretogram of PCR amplification products of genomic DNA of 14 Magnolia officinalis materials from partial TRAP molecular markers |
2.2 SSR标记分析
参加测试的13对SSR引物在所有被检测的厚朴材料中都能扩增出清晰条带,其中2对引物(Stm0246和M10D8)表现出多态性(图 2),可用于本研究厚朴材料遗传分析。
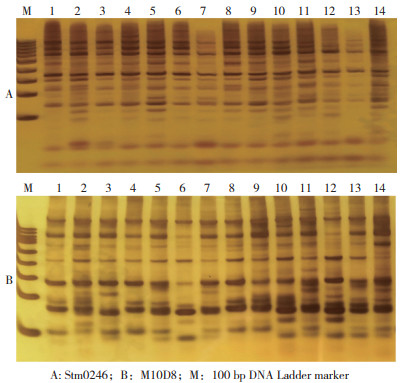
|
| 1~14代表14个厚朴材料,依次为L1、Ls1、L8、Ls8、L11、Ls11、L12、Ls12、L14、Ls14、L15、Ls15、L19、Ls19 1-14 represent the fourteen Magnolia officinalis : L1, Ls1, L8, Ls8, L11, Ls11, L12, Ls12, L14, Ls14, L15, Ls15, L19 and Ls19 图 2 SSR引物对14个厚朴材料基因组DNA的扩增产物电泳结果 Fig. 2 Electrophoretogram of PCR amplification products of genomic DNA of 14 Magnolia officinalis materials from SSR molecular markers |
2.3 广东乐昌龙山林场厚朴遗传多样性分析
本研究设计并筛选出扩增条带清晰、多态性良好的7对TRAP引物和2对SSR引物,分别扩增厚朴单株基因组DNA,扩增产物的电泳条带经判读后转化为数据记录在Excel表中,用NTSYSpc2.10e软件进行聚类分析,构建了龙山林场厚朴资源的亲缘关系树状图。结果(图 3)显示,龙山林场厚朴资源遗传相似系数在0.75~0.88之间,遗传相似系数为0.75时,厚朴母株被分成两大类,第一类包括L1、L8、L12、L14,第二类包括L11、L15、L19。母株与对应子代在聚类图上并未表现出相对更高的遗传相似性,亲子之间甚至相距较远,如L14与LS14存在较大遗传差异,这可能与厚朴的繁殖特性有关。厚朴自交不结实,种子基本都是杂交种[15],且后代在亲缘关系上更偏向父本[11],群体内基因交流频繁,因此较好地保留了厚朴的遗传多样性,这对厚朴良种选育有利。

|
| 图 3 基于TRAP和SSR标记分析的14个厚朴材料的亲缘关系树状图 Fig. 3 Phylogenetic tree of 14 Magnolia officinalis plants based on TRAP and SSR molecular marker |
3 讨论
本研究利用公共数据库GenBank中厚朴cDNA序列设计了TRAP引物,分别与SRAP随机引物配对,共开发出扩增产物电泳条带清晰、带型丰富、多态性好的厚朴TRAP标记7个,并从已知的厚朴SSR标记中筛选到2个有用的SSR标记。利用这9个分子标记分析了广东乐昌龙山林场厚朴资源的亲缘关系,明确了各单株之间的相似系数。开发的新标记将为后续进行厚朴种植资源鉴定、优株鉴别、分子标记辅助选择提供工具,为优良品种选育奠定基础。
选育、培植优良厚朴品种是保证厚朴药材质量的基础。在新品种选育方面,由于厚朴生长周期长,从定植到采收需要15年以上[16],利用药效成分、生长特性、产量等性状筛选优良种质时间周期太长、效率太低。因此,迫切需要改进鉴定方法,加快良种选育进程。分子标记辅助育种在很多植物上已取得成功并应用成熟[17-19],但厚朴分子标记研究相对滞后,虽然有将RAPD[3]、AFLP[20]和SSR[10]标记用于厚朴遗传多样性、药材道地性分析,但可供选择使用的标记数量非常有限。
在探讨种源的亲缘关系或进行种质资源鉴定时,开发TRAP标记是方便、经济、有效的办法。如刘嘉霖等[21]成功开发TRAP标记对黄淮海和南方大豆品种进行遗传结构和多样性分析,明确了大豆品种分布与地域无明显相关;索相敏等[22]开发TRAP标记分析了苹果属种间遗传多样性。TRAP标记是控制表型差异的目标基因区多态性的表现[20],利用厚朴cDNA序列开发的TRAP标记将更加适合用于对我们所关注的表型进行鉴别和多样性分析。本研究率先开发出厚朴TRAP标记,并利用其对厚朴资源进行了遗传多样性分析,结果发现母树与子代并未成对出现在亲缘关系树状图中,母子遗传相似系数不高,这与“厚朴自交不结实,种子基本都是杂交种[15] ”、“后代在亲缘关系上更偏向父本[11] ”等观点一致。
在SSR引物筛选过程中,发现对浙江、湖南等地厚朴材料表现良好多态性的13对SSR引物[11]在本研究材料中多态性比较低,只有其中的2对SSR引物表现出多态性。说明来自不同地区的厚朴资源遗传背景差异较大,用一个地区厚朴材料开发出的分子标记可能不适合其他地区的厚朴资源,同时也表明不同地理种源的厚朴可能具有不同的遗传背景和品质特征,这对厚朴优良品种选育有利,也对厚朴种质资源保护具有指导意义。
4 结论本研究设计、开发了7个厚朴新型TRAP标记,并筛选到2个厚朴SSR标记。利用新开发的TRAP分子标记和SSR标记分析了广东乐昌龙山林场厚朴资源的遗传多样性,构建了厚朴资源的亲缘关系树状图,各单株之间的遗传相似系数在0.75~0.88之间,母株与对应子代在聚类图上并未表现出更高的遗传相似性,为厚朴子代是杂交种且遗传上更偏向父本提供了佐证,进一步证明厚朴种群内基因交流频繁,遗传多样性丰富,为厚朴良种选育提供理论依据。新型分子标记的不断开发利用,必将促进厚朴优株鉴别、优良品种选育、分子标记辅助选择等研究工作的更快发展。
| [1] |
杨旭, 杨志玲, 谭美, 程小燕, 曾平生, 谭梓峰, 李公荣. 不同地理种源9年生厚朴的药材质量及早期选择[J]. 福建农林科技大学学报(自然科学版), 2020, 49(1): 74-79. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2020.01.013 YANG X, YANG Z L, TAN M, CHENG X Y, ZENG P S, TAN Z F, LI G R. Quality and early selection of 9 year-old Houpoea officinalis from different geographical provenances[J]. Journal of Fujian Agriculture and forestry University (Natural Science Edition), 2020, 49(1): 74-79. DOI:10.13323/j.cnki.j.fafu(nat.sci.).2020.01.013 |
| [2] |
孔玲. 厚朴良种选择及其RAPD种源鉴别研究[D]. 福州: 福建农林大学, 2010. KONG L. Study on variety section of Magnolia officinalis and its identification of provenance of RAPD[D]. Fuzhou: Fujian Agriculture and forestry University, 2010. |
| [3] |
郭宝林, 吴勐, 斯金平, 李家实, 肖培根. 厚朴DNA分子标记的研究--正品的RAPD研究[J]. 药学学报, 2001, 36(5): 386-389. DOI:10.16438/j.0513-4870.2001.05.017 GUO B L, WU M, SI J P, LI J S, XIAO P G. Research on DNA molecular marker of Magnolia officinalis-RAPD study on certified species[J]. Acta Pharrnaceutiea Sinica, 2001, 36(5): 386-389. DOI:10.16438/j.0513-4870.2001.05.017 |
| [4] |
张小康, 张雪丽, 熊秋芳. 萝卜叶缘裂刻性状分子标记的开发与应用[J]. 广东农业科学, 2019, 46(7): 17-22. DOI:10.16768/j.issn.1004-874X.2019.07.003 ZHANG X K, ZHANG X L, XIONG Q F. Development and application of molecular markers for radish leaf edge cracking traits[J]. Guangdong Agricultural Sciences, 2019, 46(7): 17-22. DOI:10.16768/j.issn.1004-874X.2019.07.003 |
| [5] |
王玉杰, 冷春旭, 孙中义, 王珣, 赵曦, 李晓娟, 赵伟, 吴立成. 分子标记技术在农作物种子检测中的应用[J]. 中国种业, 2022(3): 38-40. DOI:10.19462/j.cnki.1671-895x.2022.03.035 WANG Y J, LENG C X, SUN Z Y, WANG X, ZHAO X, LI X J, ZHAO W, WU L C. Application of molecular marker technology in crop seed detection[J]. China Seed Industry, 2022(3): 38-40. DOI:10.19462/j.cnki.1671-895x.2022.03.035 |
| [6] |
刘斌. 紧跟世界科技发展前沿, 水稻分子育种初见成效-- 广东省农业科学院水稻分子育种进展[J]. 广东农业科学, 2020, 47(12): 12-23. DOI:10.16768/j.issn.1004-874X.2020.12.002 LIU B. Following the frontier of scientific and technological development, significant progress has been made in molecular rice breeding ─ A brief introduction to the work in molecular rice breeding of rice research institute of Guangdong Academy of Agricultural Sciences[J]. Guangdong Agricultural Sciences, 2020, 47(12): 12-23. DOI:10.16768/j.issn.1004-874X.2020.12.002 |
| [7] |
谢薇. 中药材鉴定运用分子标记技术的实践探寻[J]. 光明中医, 2021, 36(19): 3262-3264. DOI:10.3969/j.issn.1003-8914.2021.19.019 XIE W. Application of molecular tracer technique in identification of traditional Chinese medicine[J]. Guangming Journal of Chinese Medicine, 2021, 36(19): 3262-3264. DOI:10.3969/j.issn.1003-8914.2021.19.019 |
| [8] |
钱润, 周骏辉, 杨健, 黄璐琦, 袁媛. 中药材分子标记辅助育种技术研究进展[J]. 中国中药杂志, 2020, 45(20): 4812-4818. DOI:10.19540/j.cnki.cjcmm.20200710.602 QIAN R, ZHOU J H, YANG J, HUANG L Q, YUAN Y. Study progress on molecular marker-assisted breeding of Chinese medicinal materials[J]. China Journal of Chinese Materia Medica, 2020, 45(20): 4812-4818. DOI:10.19540/j.cnki.cjcmm.20200710.602 |
| [9] |
翁剑, 陈宝钢, 李书平, 郑郁善. 厚朴分子标记技术应用现状及建议[J]. 湖北林业科技, 2013, 42(5): 45-48, 80. DOI:10.3969/j.issn.1004-3020.2013.05.013 WENG J, CHEN B M, LI S P, ZHEGN Y S. The application status and suggestion of molecular marker technology in Magnolia officinalis[J]. Hubei Forestry Science Technology, 2013, 42(5): 45-48, 80. DOI:10.3969/j.issn.1004-3020.2013.05.013 |
| [10] |
杨旭, 杨志玲, 谭美, 程小燕. 厚朴转录组特征分析及EST-SSR标记的开发[J]. 核农学报, 2019, 33(7): 1318-1329. DOI: 10.11869/j.issn.100-8551.2019.07.1318. YANG X, YANG Z L, CHENG X Y. Characteristics analysis of Houpoea officinalis transcription and development of EST-SSR markers[J]. Journal of Nuclear Agricultural Sciences, 2019, 33(7): 1318-1329. DOI: 10.11869/j.issn.100-8551.2019.07.1318. |
| [11] |
麦静. 厚朴杂交亲本及子代遗传多样性分析[D]. 北京: 中国林业科学研究院, 2015. MAI J. Genetic diversity analysis of hybrid progeny and hybrid parents of Magnolia officinalis[D]. Beijing: Chinese Academy of Forestry, 2015. |
| [12] |
柳李旺, 龚义勤, 黄浩, 朱献文. 新型分子标记SRAP与TRAP及其应用[J]. 遗传, 2004, 26(5): 777-781. DOI:10.16288/j.yczz.2004.05.039 LIU L W, GONG Y Q, HUANG H, ZHU X W. Novel molecular marker systems-SRAP and TRAP and their application[J]. Hereditas, 2004, 26(5): 777-781. DOI:10.16288/j.yczz.2004.05.039 |
| [13] |
LI G Y, QUIROS C F. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics, 2001, 103(2): 455-461. DOI:10.1007/s001220100570 |
| [14] |
冯俊彦, 蒲志刚, 李明, 张聪, 阎文昭, 王勇, 邢小军, 刘东阳. 9份烟草品种(系)及其航天诱变后代的TRAP分析[J]. 西南农业学报, 2017, 30(2): 273-279. DOI:10.16213/j.cnki.scjas.2017.2.006 FENG J Y, PU Z G, LI M, ZHAGN C, YAN W Z, WANG Y, XING X J, LIU D Y. Analysis of 9 tobacco varieties (lines) and their space mutation progenies with TRAP markers[J]. Southwest China Journal of Agricultural Sciences, 2017, 30(2): 273-279. DOI:10.16213/j.cnki.scjas.2017.2.006 |
| [15] |
王洁, 杨志玲, 杨旭, 檀国印, 何正松. 野生厚朴花粉萌发及花粉管生长过程观察[J]. 生态与农村环境学报, 2013, 29(1): 53-57. DOI:10.3969/j.issn.1673-4831.2013.01.009 WANG J, YANG Z L, YANG X, TAN G Y, HE Z S. Pollen germination and pollen tube growth of wild Magnolia officinalis as observed[J]. Journal of Ecology and Rural Environment, 2013, 29(1): 53-57. DOI:10.3969/j.issn.1673-4831.2013.01.009 |
| [16] |
马建烈. 厚朴栽培及采收加工技术[J]. 特种经济动植物, 2016, 19(3): 34-36. DOI:10.3969/j.issn.1001-4713.2016.03.023 MA J L. Cultivation, harvesting and processing techniques of Magnolia officinalis[J]. Special Economic Animal and Plant, 2016, 19(3): 34-36. DOI:10.3969/j.issn.1001-4713.2016.03.023 |
| [17] |
张信童, 何永军, 刘杨, 陈火英. 茄果皮着色光敏性dCAPS标记开发与验证[J]. 南京农业大学学报, 2021, 44(4): 637-645. DOI:10.7685/jnau.202009022 ZHANG X T, HE Y J, LIU Y, CHEN H Y. Development and validation of molecular marker for photosensitivity of anthocyanin production in eggplant fruit[J]. Journal of Nanjing Agricultural University, 2021, 44(4): 637-645. DOI:10.7685/jnau.202009022 |
| [18] |
韩英鹏, 杨振红. 大豆抗病性和分子标记及分子辅助育种研究进展[J]. 大豆科技, 2021(5): 18-25. DOI:10.3969/j.issn.1674-3547.2021.05.005 HAN Y P, YANG Z H. Review of soybean disease resistance, molecu la r ma rker s a nd molecu la r-a ssisted breeding[J]. Soybean Science and Technology, 2021(5): 18-25. DOI:10.3969/j.issn.1674-3547.2021.05.005 |
| [19] |
莫璋红, 梁俊, 谢金兰, 林丽, 李长宁. 基于分子标记的甘蔗遗传育种研究综述[J]. 南方农业, 2021, 15(20): 233-234. DOI:10.19415/j.cnki.1673-890x.2021.20.106 MO Z H, LIANG J, XIE J L, LIN L, LI C N. Review on sugarcane genetics and breeding based on molecular markers[J]. South China Agriculture, 2021, 15(20): 233-234. DOI:10.19415/j.cnki.1673-890x.2021.20.106 |
| [20] |
蒋燕锋, 斯金平, 黄华宏, 程龙军. 厚朴AFLP分子标记体系的建立[J]. 浙江林学院学报, 2010, 27(2): 304-309. DOI:10.3969/j.issn.2095-0756.2010.02.024 JIANG Y F, SI J P, HUANG H H, CHENG L J. An AFLP molecular labeling technique for Magnolia officinalis[J]. Journal of Zhejiang Forestry College, 2010, 27(2): 304-309. DOI:10.3969/j.issn.2095-0756.2010.02.024 |
| [21] |
刘嘉霖, 陈琪, 谢慧敏, 罗火林, 杨柏云, 熊冬金. 黄淮海和南方大豆育成品种TRAP标记的遗传结构研究[J]. 大豆科学, 2020, 39(3): 341-351. DOI:10.11861/j.issn.1000-9841.2020.03.0341 LIU J L, CHEN Q, XIE H M, LUO H L, YANG B Y, XIONG D J. Genetic structure and diversity of soybean cultivars released from Huang Huai-Hai and southern China based on TRAP marker[J]. Soybean Science, 2020, 39(3): 341-351. DOI:10.11861/j.issn.1000-9841.2020.03.0341 |
| [22] |
索相敏, 郝婕, 冯建忠, 鄢新民, 王献革, 李学营. 基于TRAP标记的苹果属种间遗传多样性分析[J]. 果树学报, 2019, 36(12): 1609-1618. DOI:10.13925/j.cnki.gsxb.20190211 SOU X M, HAO J, FENG J Z, YAN X M, WANG X G, LI X Y. Analysis of genetic diversity of Malus species resources by TRAP markers[J]. Journal of Fruit Science, 2019, 36(12): 1609-1618. DOI:10.13925/j.cnki.gsxb.20190211 |
(责任编辑 杨贤智)
 2022, Vol. 49
2022, Vol. 49