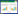文章信息
基金项目
- 国家自然科学基金(32072047);广东省自然科学基金(2020A1515011051);广州市科技计划项目(201804020078);广东省科技计划国际合作项目(2021A0505030048);广东省农业科学院院长基金(202016)
作者简介
- 杨梯丰,博士,副研究员,硕士生导师,广东省农业科学院水稻研究所分子育种研究室副主任。主要从事水稻耐冷和耐热等重要农艺性状相关基因的定位、克隆以及分子育种等研究。先后主持国家自然科学基金项目2项,广东省自然科学基金1项,广东省科技计划项目2项;先后参与国家科技部973计划前期研究项目、国家自然科学基金国际合作重点项目、广东省科技计划重点项目、广州市科学研究重点项目等20多项。利用单片段代换系等遗传材料,采用全基因组关联分析等分析工具标记定位了40多个水稻耐冷QTL、10多个水稻耐热QTL,精细定位和克隆2个QTL,利用这些优良QTL和广东省主推水稻品种开展分子标记辅助育种,创制了一批耐冷抗病适于直播的优良水稻品系。研究结果分别在Plant Biotechnology Journal、Rice、Molecular Breeding、The Crop Journal、《分子植物育种》等期刊发表,已发表科技论文20多篇,申报专利5项,获授权发明专利2项,申报品种权5项; 主要从事水稻功能基因组学、生物信息学和分子育种相关研究。先后主持广东省自然科学基金重点项目、广东省科技计划国际合作项目、广州市科技计划项目、广东省重点研发专项子课题等省部级科研项目10多项。主要针对水稻产业亟需的优异农艺性状,利用广泛多样性种质资源进行功能基因克隆和分子机制研究;成功克隆早期生长速度、苗期耐冷、重金属低积累、抗黑条矮缩病等水稻重要农艺性状功能基因,剖析水稻早期生长和厌氧发芽等直播相关性状的分子机制。开展水稻比较基因组学和生物信息学研究,完成国际稻种资源大规模测序和泛基因组构建研究工作。共发表SCI论文20多篇,其中以第一作者或通信作者在Plant Biotechnology Journal、Rice、The Crop Journal、Journal of Environmental Sciences等国际知名杂志发表论文10多篇;以第一发明人获授权发明专利2项,软件著作权2项;作为副主编参与编写农业农村部“十三五”规划教材《基因工程原理与实验》.
通讯作者
- 赵均良(1979—),男,博士,副研究员,研究方向为水稻分子遗传,E-mail:otest@163.com.
文章历史
- 收稿日期:2021-08-16
【研究意义】随着我国经济快速发展,农村主要劳动力向城市转移,劳动力短缺已成为水稻生产的突出问题。采取轻简化、机械化的生产方式,是解决这一问题的根本出路。直播是一种轻简、高效的栽培模式,近年来发展迅猛。在双季稻地区,强的低温发芽力是保证早季直播水稻形成整齐、健壮秧苗的关键因素之一。但目前许多主栽水稻品种低温发芽力低[1],已经成为限制直播稻发展的关键因素。选育耐低温发芽力强的水稻品种是推动水稻直播发展的关键。但水稻低温发芽力是多基因控制的数量性状[2],直接选择的效率很低,应用常规遗传育种技术难以进行有效育种。在水稻中挖掘低温发芽力优异基因并加以应用,将是水稻低温发芽力育种取得突破的关键。近年来,分子标记辅助选择已经被广泛应用到水稻育种中[3-8]。相比基因的连锁标记,位于基因内的分子标记可以有效避免遗传累赘,使目标基因的选择更高效、准确。因此,基因内分子标记的应用可提高选择的准确性,从而加快分子育种进程。
【前人研究进展】分子标记技术的发展为水稻低温发芽力的遗传基础解析提供了一种有效工具。自分子标记技术发展以来,国内外对水稻低温发芽力QTL的标记定位已有一些报道。国内学者最早以典型的籼粳交(窄叶青8号/京系17)的DH群体为材料,用15 ℃低温处理种子16 d,鉴定到2个低温发芽力QTL(qLTG9和qLTG4),其贡献率分别为11.1% 和12.6%[9]。随着分子标记技术的成熟,利用双亲遗传作图群体对水稻低温发芽力的遗传解析逐渐增多[10-28]。近年来,全基因组关联分析(GWAS)作为一种高效的水稻QTL鉴定方法,也被应用于水稻低温发芽力的定位中[29-36]。目前,通过双亲作图群体标记定位的水稻低温发芽力QTL已超过110个,通过GWAS分析检测到与低温发芽力相关的位点多达174个,这些QTL分布在水稻12条染色体上,其中2个已被克隆,分别是qLTG3-1[37]和OsSAP16[38]。
qLTG3-1是最早被克隆的低温发芽力QTL,其功能基因Os03g0103300编码一个由184个氨基酸组成的功能未知蛋白。在品系“Hayamasari”中该基因编码区71 bp的缺失是导致低温发芽力出现差异的原因[37]。通过对62份日本水稻核心种质中1 735 bp的qLTG3-1区域测序比较,发现在编码区有1个非同义替换(T/A)以及3个插入缺失(18 bp和9 bp缺失,9 bp或36 bp插入),共有10种等位基因,但在62份水稻种质中未发现71 bp缺失的变异类型[39]。最近,Shim等[40]通过对2个有低温发芽力差异的品种进行序列分析发现,在qLTG3-1基因的编码区存在3处可导致氨基酸变异的差异(核苷酸替换变异T/A、CGG/TTC和18 bp缺失),并针对这3处变异设计了引物对98份亚洲栽培稻的基因型进行分析,根据这3处编码区的变异以及71 bp的序列缺失,将该基因划分为5种单倍型。
【本研究切入点】Shim等[40]发现18 bp缺失(GGCGGTGGCGGTGGCGGT)以及CGG/TTC变异与低温发芽力显著相关,带有18 bp缺失的qLTG3-1有较强的低温发芽力。本项目组在对单片段代换系文库亲本的低温发芽力评价时发现,南洋占的低温发芽力显著强于华粳籼74[41];通过比较这两份品种的基因组重测序信息发现,南洋占带有在编码区+127位无18 bp缺失的qLTG3-1,而华粳籼74带有在编码区+127位有18 bp缺失的qLTG3-1。阐明“18 bp缺失能否标示qLTG3-1的低温发芽力强弱”这个问题,将为在水稻中利用qLTG3-1开展低温发芽力的分子育种提供借鉴。【拟解决的关键问题】虽然Shim等[40]根据测序结果,设计了扩增qLTG3-1中编码区+127位18 bp差异的引物,但是用这对引物对试验材料进行扩增,出现了3种扩增带型,序列分析显示第3种带型是因为其所用的5个籼稻品种既含有18 bp缺失,又增加了36 bp插入,而36 bp插入可能是籼稻中的一个特殊变异。可见,该引物无法特异区分该区域的18 bp变异。因此,需要设计特异扩增这18 bp变异的分子标记来进行基因型和低温发芽力表型对应关系的分析。本研究设计了可以特异检测qLTG3-1中这18 bp变异的分子标记Gltg3-1,并对该标记在华南籼稻的检测效果进行了评价。
1 材料与方法 1.1 试验材料1.1.1 单片段代换系及亲本 供体亲本南洋占、受体亲本华粳籼74(籼稻)以及含有南洋占qLTG3-1的单片段代换系1037由广东省植物分子育种重点实验室提供[42]。单片段代换系是通过多代回交以及分子标记辅助选择创制的一种特殊遗传材料。它与受体亲本之间只存在1个已知代换片段的差异,所有单片段代换系与受体亲本之间可遗传的表型差异都与这个代换片段相关[43]。单片段代换系1037带有来源于南洋占的第3染色体的代换片段,代换区间为:短臂末端--RM14259-RM14312-RM14380- RM14427-- RM175(0~3.8 Mb)。南洋占、华粳籼74的qLTG3-1部分序列差异见图 1。
|
| 图 1 南洋占、华粳籼74的qLTG3-1部分序列差异 Fig. 1 Partial sequence variations of qLTG3-1 in Nanyangzhan and Huajingxian74 *:序列变异位点 *: Sequence variation site |
1.1.2 籼稻品种/系 以111份国内籼稻品种/系为研究材料(表 1),2019年晚季种植于广东省农业科学院大丰试验基地,7月23日播种,8月8日移栽,每个品种/系种植4行,每行8株,株行距为19. 8 cm× 19.8 cm,采用常规栽培和水肥管理。种子成熟后,按株系收获种子,晒干,室温存储3个月后用于低温发芽力评鉴。
1.2 试验方法
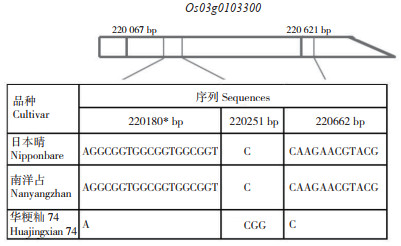
1.2.1 低温发芽力评价 参照Yang等[36]的方法,略作修改。每个水稻品种/系取240粒饱满的种子置恒温箱内,49 ℃处理96 h以打破休眠。用3% 次氯酸钠消毒20 min,蒸馏水冲洗干净,每80粒种子排列于1个铺有滤纸的9.0 cm培养皿内,加入12 mL蒸馏水,置于15 ℃生长箱中进行低温发芽试验,设置3个重复。以芽长或根长≥ 1 mm为萌发标准,在萌发10 d时检测种子的发芽率,然后调整温度为26 ℃,4 d后再次调查各品种/ 系的发芽率,以低温发芽力作为种子低温发芽的指标。
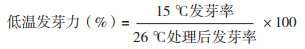
|
1.2.2 总DNA提取 DNA的微量抽提采用TPS法[44]。具体过程如下:于分蘖盛期取上部长约2~4 cm的叶片放入2 mL离心管中,加入钢珠及800 μL TPS(100 mmol/L Tris-HCl、10 mmol/L EDTA、1 mol/L KCL)抽提液,用研磨仪震荡研磨,然后将其放入75 ℃水浴锅中水浴20 min,12 000 r/min离心10 min,取上清液约500 μL于1.5 mL离心管中,加入等体积的冰乙醇至DNA析出,12 000 r/min离心10 min,去上清,取沉淀,晾干,加灭菌双蒸水200 μL至沉淀溶解。
1.2.3 引物设计 针对qLTG3-1的18 bp缺失,从RAP-DB(https://rapdb.dna.affrc.go.jp/)下载以日本晴为参考基因组的Os03g0103300序列。找到目标变异位点,截取上下游各250 bp序列,用Primer Premier 5软件设计引物,并通过NCBI(https://blast.ncbi.nlm.nih.gov/Blast.cgi)的Primerblast进行引物特异性检测,选取特异性好的引物。
1.2.4 PCR扩增和电泳检测 PCR扩增参照Yang等[6]的方法。15 μL反应体系中包含0.15 μmol/L引物、200 μmol/L dNTP、1×PCR反应缓冲液(50 mmol/L KCl,10 mmol/L Tris-HCl、pH8.3,1.5 mmol/L MgCl2,0.01%明胶)、50~100 ng DNA模板、1U Taq酶。PCR反应在9700型DNA扩增仪中进行,反应程序为:94 ℃预变性5 min;94 ℃ 30 s、55 ℃ 30 s、72 ℃ 40 s,35次循环;72 ℃延伸8 min。扩增产物用8.0%聚丙烯酰胺凝胶电泳分离,经Goldview染色后用凝胶成像系统(美国Bio-rad公司)检测带型。
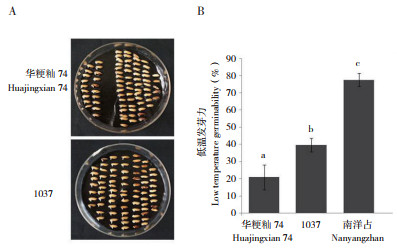
2 结果与分析 2.1 不同qLTG3-1等位基因水稻的低温发芽力比较为检验南洋占带有的qLTG3-1能否提升水稻的低温发芽力,从单片段代换系库中选取以华粳籼74为遗传背景,对携带来源于南洋占qLTG3- 1的单片段代换系1037(代换片段为第3染色体短臂端0~3.8 Mb区间)以及南洋占进行低温发芽力评价。结果(图 2)表明,单片段代换系1037的低温发芽力显著高于华粳籼74,而南洋占的低温发芽力显著高于1037。可见,来源于南洋占的qLTG3-1能显著提高华粳籼74的低温发芽力。
|
| 图 2 来源于南洋占的qLTG3-1对水稻低温发芽力的影响 Fig. 2 Effect of qLTG3-1 from Nanyangzhan on low temperature germinability in rice 多重比较采用Duncan函数,柱状图上不同小写字母表示差异显著 Duncan function is used for multiple comparisons, and different lowercase letters on the bar chart represent significant differences |
2.2 qLTG3-1基因内分子标记的开发
从RAP-DB网站下载日本晴的Os03g0103300序列信息。根据文献[39-40],在缺失18 bp序列(GGCGGTGGCGGTGGCGGT)的位置两侧,设计出特异性较好的可扩增该区域的分子标记Gltg3-1和Gltg3-2(表 2)。预测对没有18 bp缺失的序列,分别可扩增出147、247 bp的片段(R带型);而对存在18 bp缺失的序列,则扩增出129、229 bp的片段(D带型)。
2.3 分子标记的筛选
利用Gltg3-1和Gltg3-2引物,随机选取30份籼稻(材料编号1~30,表 1)进行扩增。Gltg3-1扩增出两种带型,即存在和不存在18 bp缺失的带型;而Gltg3-2扩增出4种带型(图 3)。这可能是由于Gltg3-2扩增区间较大,在扩增区间内还存在其他序列变异[39]。可见,Gltg3-1能特异区分qLTG3-1的18 bp差异。
2.4 Gltg3-1标记对华南籼稻qLTG3-1的检测效果
利用分子标记Gltg3-1对111份籼稻DNA进行扩增分型,结果显示,51份含有18 bp缺失(D带型),53份无18 bp缺失(R带型),7份为杂合型(图 4A)。
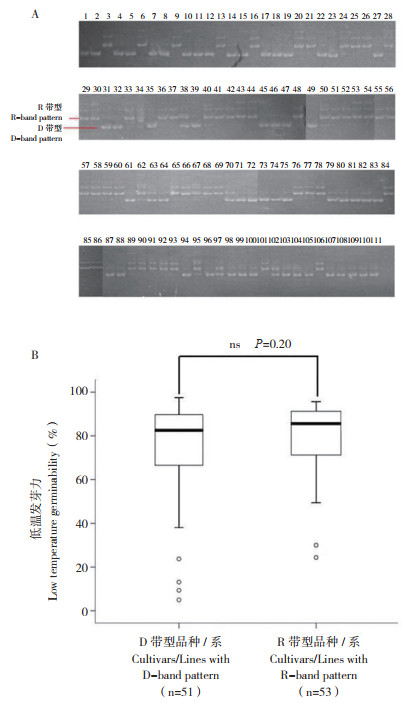
|
| 图 4 Gltg3-1标记的扩增带型(A)及不同扩增带型品种/系的低温发芽力(B) Fig. 4 Band patterns amplified by molecular marker Gltg3-1 and low temperature germinability of varieties/lines with different band patterns 1~111为表 1试验材料编号;ns表示差异不显著 1-111 are Codes of experimental materials of Table 1; ns represents that the difference is not significant |
对qLTG3-1纯合的品种/系进行低温发芽力测定,在51份D带型的品种/系中,4份发芽力低于30.0%,其余47份品种/系的发芽力为38.1%~97.5%,平均值为74.0%;在53份R带型的品种/系中,2份发芽力低于30%,其余51份品种/系的发芽力为49.4%~95.7%,平均值为79.0%;但两组低温发芽力差异不显著(图 4B)。结果表明,该分子标记能区分存在18 bp缺失和无18 bp缺失的qLTG3-1,但是这种带型差异在华南籼稻中与低温发芽力强弱无对应关系。
3 讨论qLTG3-1是控制水稻的低温发芽力的QTL[37];在Hwaseong/IRGC105491构建的近等基因系群体中也发现qLTG3-1控制水稻的低温发芽力,而且与QTL qLTG1无显著互作,带有这两个QTL的株系低温发芽力显著高于只带有单个QTL的株系[40]。在本研究中,带有来源于南洋占的qLTG3-1能显著提高华粳籼74的低温发芽力。可见,qLTG3-1是1个控制水稻低温发芽力的重要QTL。
在找到功能基因后确定其功能位点将大大提升该基因在分子育种中的利用效率。已有的等位基因分析表明,qLTG3-1存在多种复等位基因[39-40]。在本研究中,通过比较南洋占和华粳籼74的qLTG3-1发现,在编码区有18 bp缺失以及2 bp插入,但是没有检测到其他变异位点。可见,qLTG3-1的序列变异丰富,存在多种类型的等位基因。在这些序列变异中,+127位的18 bp缺失在多个研究试验中都存在,可见这个变异在水稻种质中广泛存在。
为了探究这18 bp变异是否与低温发芽力表型显著相关,本研究设计分子标记Gltg3-1特异扩增该位点,通过扩增带型将品种/系分组进行低温发芽力差异分析,结果发现两组的低温发芽力无显著差异,即qLTG3-1的18 bp变异并不能标示低温发芽力的强弱。这一结果与Shim等[40]的结论不一致,可能的主要原因:一是因为Shim等进行低温发芽力表型鉴定的材料是韩国的核心稻种,大部分为粳稻,所有出现18 bp缺失带型的都是籼稻(含2份aus稻),共27份;而无缺失带型的都是粳稻,共42份;而本研究所用的111份试材均是华南稻区的籼稻,扩增带型显示华南籼稻中也广泛存在这18 bp变异,其中51份为缺失带型,53份为无缺失带型。基于有限的试验材料得到的结果有一定局限性。二是由于水稻的低温发芽力是多基因控制的复杂性状,还有一些重要的低温发芽力QTL在发挥作用。从文献报道的QTL定位结果来看,通过作图群体定位到大量的低温发芽力QTL,绝大多数QTL都位于不同的染色体区间[9-35]。可见,在不同遗传材料中,往往存在多个控制低温发芽力的QTL,QTL位置一般并不相同,单个QTL的效应比较小。在本研究中,单片段代换系1037的低温发芽力显著高于华粳籼74,而南洋占的低温发芽力显著高于1037。可见,在南洋占中也存在多个控制低温发芽力的QTL。
在Shim等[40]研究中,来源O.rufi pogon缺失了18 bp的qLTG3-1等位基因能显著增强低温发芽力;在本研究中,来源于南洋占带有无缺失的qLTG3-1能显著增强低温发芽力。这进一步证实,qLTG3-1的18 bp变异无法区分低温发芽力的高低。因此,在水稻低温发芽力改良中,需要对qLTG3-1等位基因的效应进行评价,获得增强水稻低温发芽力的等位基因用于品种改良。在本研究中,鉴定到来源于南洋占的qLTG3-1能显著提高华粳籼74的低温发芽力,该等位基因可用于提高水稻低温发芽力的改良。
分子标记辅助选择育种是利用与目标QTL紧密连锁或者来自目标基因内部的分子标记,对杂交后代的基因型进行准确判别,进而辅助选择目标性状。它不受外界环境的干扰,能显著提高育种效率,已成为水稻遗传改良的重要工具。开发简便、经济和实用的分子标记是高效开展水稻分子标记辅助选择育种的基础。本研究依据在qLTG3-1复等位基因中广泛存在的18 bp变异设计了分子标记Gltg3-1,该分子标记存在于基因内部,可以高效准确地在杂交后代中选择到目标基因,这将在水稻低温发芽力分子育种中发挥作用。
本研究仅对在qLTG3-1中广泛存在的18 bp变异与低温发芽力表型的关系进行分析,但已有研究表明,qLTG3-1中存在丰富的序列变异[39-40],其他变异位点也可能是功能位点。特别是华南籼稻中qLTG3-1的等位基因类型并不明确,在华南籼稻中是否存在功能位点,这些问题值得进一步研究。选用遗传背景一致、供体来源多样的单片段代换系来进行qLTG3-1的等位基因效应分析将获得更客观准确的结果。
4 结论qLTG3-1是1个控制水稻低温发芽力的重要QTL,其序列变异丰富,存在多种类型的等位基因,+127位的18 bp缺失变异在水稻种质中广泛存在。分子标记Gltg3-1能特异区分这18 bp变异。基于Gltg3-1的扩增带型对品种/系分组进行低温发芽力差异性检测,发现两组的低温发芽力无显著差异,即qLTG3-1上的18 bp变异并不能标示水稻低温发芽力的强弱。因此,需要对qLTG3-1等位基因的效应进行评价,获得增强水稻低温发芽力的等位基因用于品种改良。来源于南洋占的qLTG3-1能显著提高低温发芽力,该等位基因可用于水稻低温发芽力的改良。分子标记Gltg3-1存在于基因内部,可以准确地在杂交后代中选择到目标基因。
| [1] |
杨志涛, 李媛, 张少红, 杨梯丰, 赵均良, 董景芳, 陈光辉, 刘斌. 377份多样性国际稻种低温发芽力评价[J]. 广东农业科学, 2017, 44(4): 1-6. DOI:10.16768/j.issn.1004-874X.2017.04.001 YANG Z T, LI Y, ZHANG S H, YANG T F, ZHAO J L, DONG J F, CHEN G H, LIU B. Evaluation of low temperature germinability of 377 national rice accessions[J]. Guangdong Agricultural Sciences, 2017, 44(4): 1-6. DOI:10.16768/j.issn.1004-874X.2017.04.001 |
| [2] |
杨梯丰, 张少红, 赵均良, 黄章慧, 张桂权, 刘斌. 水稻耐冷QTL定位的比较分析[J]. 分子植物育种, 2015, 13(1): 1-15. DOI:10.13271/j.mpb.013.000001 YANG T F, ZHANG S H, ZHAO J L, HUANG Z H, ZHANG G Q, LIU B. Meta-analysis of QTLs underlying cold tolerance in rice(Oryza sativa L.)[J]. Molecular Plant Breeding, 2015, 13(1): 1-15. DOI:10.13271/j.mpb.013.000001 |
| [3] |
杨梯丰, 曾瑞珍, 朱海涛, 陈岚, 张泽民, 丁效华, 李文涛, 张桂权. 水稻粒长基因GS3在聚合育种中的效应[J]. 分子植物育种, 2010, 8(1): 59-66. DOI:10.3969/mpb.008.000059 YANG T F, ZENG R Z, ZHU H T, Chen L, ZHANG Z M, DING X H, LI W T, ZHANG G Q. Effect of grain length gene GS3 in pyramiding breeding of rice[J]. Molecular Plant Breeding, 2010, 8(1): 59-66. DOI:10.3969/mpb.008.000059 |
| [4] |
秦延春. DNA分子标记及其在水稻遗传育种研究中的应用[J]. 广东农业科学, 2012, 39(7): 145-147. DOI:10.16768/j.issn.1004-874x.2012.07.058 QIN Y C. DNA molecular marker and application in genetics and breeding study of rice[J]. Guangdong Agricultural Sciences, 2012, 39(7): 145-147. DOI:10.16768/j.issn.1004-874x.2012.07.058 |
| [5] |
李玉营, 李声春, 李晓方. 分子标记辅助选择聚合水稻抗虫抗病基因育种研究进展[J]. 广东农业科学, 2016, 43(6): 119-126. DOI:10.16768/j.issn.1004-874X.2016.06.021 LI Y Y, LI S C, LI X F. Research progress of molecular marker-assisted selection for pyramiding disease and insect resistance genes in rice[J]. Guangdong Agricultural Sciences, 2016, 43(6): 119-126. DOI:10.16768/j.issn.1004-874X.2016.06.021 |
| [6] |
YANG T F, ZHANG S H, ZHAO J L, LIU Q, HUANG Z H, MAO X X, DONG J F, WANG X F, ZHANG G Q, LIU B. Identification and pyramiding of QTLs for cold tolerance at the bud bursting and the seedling stages by use of single segment substitution lines in rice(Oryza sativa L.)[J]. Molecular Breeding, 2016, 36: 96. DOI:10.1007/s11032-016-0520-9 |
| [7] |
LI L Y, CHEN H P, MAO D H. Pyramiding of rapid germination loci from Oryza Sativa cultivar'Xieqingzao B'and cold tolerance loci from Dongxiang wild rice to increase climate resilience of cultivated rice[J]. Molecular Breeding, 2019, 39: 85. DOI:10.1007/s11032-019-0985-4 |
| [8] |
刘斌. 紧跟世界科技发展前沿, 水稻分子育种初见成效——广东省农业科学院水稻分子育种进展[J]. 广东农业科学, 2020, 47(12): 12-23. DOI:10.16768/j.issn.1004-874X.2020.12.002 LIU B. Following the frontier of scientific and technological development, significant progress has been made in molecular rice breeding ─ A Brief introduction to the work in molecular rice breeding of rice research institute of Guangdong Academy of Agricultural Sciences[J]. Guangdong Agricultural Sciences, 2020, 47(12): 12-23. DOI:10.16768/j.issn.1004-874X.2020.12.002 |
| [9] |
滕胜, 曾大力, 钱前, 国广泰史, 黄大年, 朱立煌. 低温条件下水稻发芽力QTL的定位分析[J]. 科学通报, 2001, 13: 1104-1108. DOI:10.3321/j.issn:0023-074X.2001.13.012.j.issn:0023-074X.2001.13.012 TEN S, ZENG D L, QIAN Q, KUNIHIRO Y, HUANG D N, ZHU L H. Mapping analysis of QTL for germinability of rice under low temperature[J]. Scientific Bulletin, 2001, 13: 1104-1108. DOI:10.3321/j.issn:0023-074X.2001.13.012.j.issn:0023-074X.2001.13.012 |
| [10] |
MIURA K, LIN S Y, YANO M, NAGAMINE T. Mapping quantitqtive trait loci controlling low-temperature germinability in rice(Oryza sativa L.)[J]. Breeding Science, 2001, 51(4): 293-399. DOI:10.1270/jsbbs.51.293 |
| [11] |
侯名语, 王春明, 江玲, 万建民, 安井秀, 吉村醇. 水稻低温发芽力QTL定位和遗传分析[J]. 遗传学报, 2004, 31(7): 701-706. HOU M Y, WANG C M, JIANG L, WAN J M, HIDESHI Y, ATSUSHI Y. Inheritance and QTL mapping of low temperature germinability in rice (Oryza sativa L.)[J]. Acta Genetica Sinica, 2004, 31(7): 701-706. |
| [12] |
姜旋, 李辰昱, 毛婷. 水稻低温发芽性QTL的分子标记定位[J]. 武汉植物学研究, 2005, 23(3): 216-220. DOI:10.3969/j.issn.2095-0837.2005.03.003 JIANG X, LI C Y, MAO T. Mapping of QTLcontrolling low temperature germinability in rice[J]. Journal of Wuhan Botanical Research, 2005, 23(3): 216-220. DOI:10.3969/j.issn.2095-0837.2005.03.003 |
| [13] |
陈亮, 楼巧君, 孙宗修, 邢永忠, 余新桥, 罗利军. 水稻低温发芽力的QTL定位[J]. 中国水稻科学, 2006, 20(2): 159-164. DOI:10.16819/j.1001-7216.2006.02.008 CHEN L, LOU Q J, SUN Z X, XING Y Z, YU X Q, LUO L J. QTL mapping of low temperature germinability in rice[J]. Chinese Journal Rice Science, 2006, 20(2): 159-164. DOI:10.16819/j.1001-7216.2006.02.008 |
| [14] |
纪素兰, 江玲, 王益华, 刘世家, 刘喜, 翟虎渠, 吉村醇, 万建民. 水稻种子耐低温发芽力的QTL定位及上位性分析[J]. 作物学报, 2008, 34: 551-556. DOI:10.3724/SP.J.1006.2008.00551 JI S L, JIANG L, WANG Y H, LIU S J, LIU X, ZHAI H Q, YOSHIMURA A, WAN J M. QTL and epistasis for low temperature germinability in rice[J]. Acta Agronomica Sinica, 2008, 34(4): 551-556. DOI:10.3724/SP.J.1006.2008.00551 |
| [15] |
巩迎军, 阮雯君, 荀星, 董彦君, 林冬枝, 叶胜海, 张小明. 水稻芽性状耐冷性的QTL分析[J]. 分子植物育种, 2009, 7(2): 273-278. DOI:10.3969/j.issn.1672-416X.2009.02.009.j.issn.1672-416X.2009.02.009 GONG Y J, RUAN W J, XUN X, DONG Y J, LIN D Z, YE S H, ZHANG X M. QTL analysis of cold tolerance for two bud traits in rice[J]. Molecular Plant Breeding, 2009, 7(2): 273-278. DOI:10.3969/j.issn.1672-416X.2009.02.009.j.issn.1672-416X.2009.02.009 |
| [16] |
胡涛, 宋佳瑜, 吴爱婷, 刘思彤, 郭志富, 姜秀娟, 高继平, 赵明辉, 黎毛毛. 东乡野生稻低温发芽力QTL定位及超级稻耐冷改良[J]. 植物遗传资源学报, 2018, 19(4): 627-632. DOI: 10.13430/j.cnki.jpgr.20171128004. HU T, SONG J Y, WU A T, LIU S T, JIANG X J, GAO J P, ZHAO M H, GUO Z F, JIANG X J, GAO J P, ZHAO M H, LI M M. QTLs mapping for low temperature germinability in a population of Dongxiang wild rice(Oryza rufi pogou Griff. ) and super rice variety[J]. Journal of Plant Genetic Resources, 2018, 19(4): 627-632. DOI: 10.13430/j.cnki.jpgr.20171128004. |
| [17] |
王棋, 范淑秀, 郭江华, 陈兆赫, 梁银培, 刘振宇, 殷业超, 王嘉宇. 利用籼粳交RIL群体对水稻发芽期和苗期耐冷性的QTL分析[J]. 华北农学报, 2019, 34(1): 83-88. DOI:10.7668/hbnxb.201751291 WANG Q, FAN S X, GUO J H, CHEN Z H, LIANG Y P, LIU Z Y, YIN Y C, WAN J Y. QTL analysis of cold tolerance at germination and seedling stages of rice[J]. Acta Agricultural Huabei Sinica, 2019, 34(1): 83-88. DOI:10.7668/hbnxb.201751291 |
| [18] |
FUJINO K, SEKIGUCHI H, SATO T, KIUCHI H, NONOUE Y, TAKEUCHI Y, ANDO T, LIN S, YANO M. Mapping of quantitative trait loci controlling low-temperature g erminability in rice(Oryza sativa L.)[J]. Theoretical and Applied Genetics, 2004, 108: 794-799. DOI:10.1007/s00122-003-1509-4 |
| [19] |
HAN L, ZHANG Y, QIAO Y, CAO G, ZHANG S, KIM J, KOH H. Genetic and QTL analysis for low-temperature vigor of germination in rice[J]. Acta Genetica Sinica, 2006, 33(11): 998-1006. DOI:10.1016/S0379-4172(06)60135-2 |
| [20] |
JIANG L, LIU S, HOU M, TANG J, CHEN L, ZHAI H, WAN J. Analysis of QTLs for seed low temperature germinability and anoxia germinability in rice(Oryza sativa L.)[J]. Field Crops Research, 2006, 98: 68-75. DOI:10.1016/j.fcr.2005.12.015 |
| [21] |
WANG Z, WANG F, ZHOU R, WANG J, ZHANG H. Identification of quantitative trait loci for cold tolerance during the germination and seedling stages in rice(Oryza sativa L.)[J]. Euphytica, 2011, 181: 405-413. DOI:10.1007/s10681-011-0469-z |
| [22] |
LI L, LIU X, XIE K, WANG Y, LIU F, LIN Q, WANG W, YANG C, LU B, LIU S, CHEN L, JIANG L, WAN J. qLTG-9, a stable quantitative trait locus for low-temperature germination in rice(Oryza sativa L.)[J]. Theoretical and Applied Genetics, 2013, 126: 2313-2322. DOI:10.1007/s00122-013-2137-2 |
| [23] |
RANAWAKEA A, MANANGKILB O, YOSHIDAC S, ISHⅡD T, MORIE N, NAKAMURAE C. Mapping QTLs for cold tolerance at germination and the early seedling stage in rice(Oryza sativa L.)[J]. Biotechnology & Biotechnological Equipment, 2014, 28(6): 989-998. DOI:10.1080/13102818.2014.978539 |
| [24] |
BORJAS A, DE L, SUBUDHI P. Genetic analysis of germinating ability and seedling vigor under cold stress in US weedy rice[J]. Euphytica, 2016, 208: 251-264. DOI:10.1007/s10681-015-1584-z |
| [25] |
SATOH T, TEZUKA K, KAWAMOTO T, MATSUMOTO S, SATOH N, UEDA K, SAKURAI K, WATANABE A, TAKAHASHI H, AKAGI H. Identification of QTLs controlling low-temperature germination of the East European rice(Oryza sativa L.)variety Maratteli[J]. Euphytica, 2016, 207: 245-254. DOI:10.1007/s10681-015-1531-z |
| [26] |
JIANG N, SHI S, SHI H, KHANZADA H, WASSAN G, ZHU C, PENG X, YU Q, CHEN X, HE X, FU J, HU L, XU J, OUYANG L, SUN X, ZHOU D, HE H, BIAN J. Mapping QTL for seed germinability under low temperature using a new high-density genetic map of rice[J]. Frontiers in Plant Science, 2017, 8: 1223. DOI:10.3389/fpls.2017.01223 |
| [27] |
NAJEEB S, ALI J, MAHENDER A, PA NG Y, ZILHAS J, MURUGAIYAN V, VEMIREDDY L, LI Z. Identification of main-effect quantitative trait loci(QTLs)for low-temperature stress tolerance germination- and early seedling vigor-related traits in rice(Oryza sativa L.)[J]. Molecular Breeding, 2020, 40: 10. DOI:10.1007/s11032-019-1090-4 |
| [28] |
PEI R, ZHANG Z, HUANG M, HOU G, LUO J, ZHU H, LIU G, FU X, ZHANG G, WANG S. Mapping QTLs controlling low-temperature germinability in rice by using single segment substitution lines derived from 4 AA-genome species of wild rice[J]. Euphytica, 2021, 217: 58. DOI:10.1007/s10681-021-02791-2 |
| [29] |
FUJINO K, OBARA M, SHIMIZU T, KOYANAGI K, IKEGAYA T. Genome-wide association mapping focusing on a rice population derived from rice breeding programs in a region[J]. Breeding Science, 2015, 65: 403-410. DOI:10.1270/jsbbs.65.403 |
| [30] |
SALES E, VIRUEL J, DOMINGO C, MARQUE'S L. Genome wide association analysis of cold tolerance at germination in temperate japonica rice(Oryza sativa L.)varieties[J]. PLOS ONE, 2017, 12(8): e0183416. DOI:10.1371/journal.pone.0183416 |
| [31] |
SCHLAPPI M, JACKSON A, EIZENGA G, WANG A, CHU C, SHI Y, SHIMOYAMA N, BOYKIN D. Assessment of five chilling tolerance traits and GWAS mapping in rice using the USDA Mini-Core collection[J]. Frontiers in Plant Science, 2017, 8: 957. DOI:10.3389/fpls.2017.00957 |
| [32] |
SHAKIBA E, EDWARDS J, JODARI F, DUKE S, BALDO A, KORNILIEV P, MCCOUCH S, EIZENGA G. Genetic architecture of cold tolerance in rice(Oryza sativa L.)determined through high resolution genome-wide analysis[J]. PLOS ONE, 2017, 12(3): e0172133. DOI:10.1371/journal.pone.0172133 |
| [33] |
WANG H, LEE A, PARK S, JIN S, LEE J, HAM T, PARK Y, ZHAO W, KWON S. Genome-wide association study reveals candidate genes related to low temperature tolerance in rice(Oryza sativa L.)during germination[J]. 3 Biotech, 2018, 8: 235. DOI:10.1007/s13205-018-1252-9 |
| [34] |
THAPA R, TABIEN R, THOMSON M, SEPTININGSIH E. Genomewide association mapping to identify genetic loci for cold tolerance and cold recovery during germination in rice[J]. Frontiers in Genetics, 2020, 11: 22. DOI:10.3389/fgene.2020.00022 |
| [35] |
YANG J, YANG M, SU L, ZHOU D, HUANG C, WANG H, GUO T, CHEN Z. Genome-wide association study reveals novel genetic loci contributing to cold tolerance at the germination stage in indica rice[J]. Plant Science, 2020, 301: 110669. DOI:10.1016/j.plantsci.2020.110669 |
| [36] |
YANG T, ZHOU L, ZHAO J, DONG J, LIU Q, FU H, MAO X, YANG W, MA Y, CHEN L, WANG J, BAI S, ZHANG S, LIU B. The candidate genes underlying a stably expressed QTL for low temperature germinability in rice(Oryza sativa L.)[J]. Rice, 2020, 13: 74. DOI:10.1186/s12284-020-00434-z |
| [37] |
FUJINO K, SEKIGUCHI H, MATSUDA Y, SUGIMOTO K, ONO K, YANO M. Molecular identification of a major quantitative trait locus, qLTG3-1, controlling low-temperature germinability in rice[J]. Proceedings of the National Academy of Sciences, 2008, 105: 12623-12628. DOI:10.1073/pnas.0805303105 |
| [38] |
WANG X, ZOU B, SHAO Q, CUI Y, LU S, ZHANG Y, HUANG Q, HUANG J, HUA J. Natural variation reveals that OsSAP16 controls low-temperature germination in rice[J]. Journal of Experimental Botany, 2018, 69(3): 413-421. DOI:10.1093/jxb/erx413 |
| [39] |
FUJINO K, SEKIGUCHI H. Origins of functional nucleotide polymorphisms in a major quantitative trait locus, qLTG3-1, controlling low-temperature germinability in rice[J]. Plant Mol Biol, 2011, 75: 1-10. DOI:10.1007/s11103-010-9697-1 |
| [40] |
SHIM K, KIM S, LEE H, ADEVA C, JEON Y, LUONG N, KIM W, AKHTAMOV M, PARK Y, AHN S. Characterization of a new qLTG3- 1 allele for low-temperature germinability in rice from the wild species Oryza rufipogon[J]. Rice, 2020, 13: 10. DOI:10.1186/s12284-020-0370-2 |
| [41] |
杨梯丰, 张少红, 王晓飞, 董景芳, 黄章慧, 赵均良, 刘清, 毛兴学, 张桂权, 刘斌. 多样性国际稻种四个生长发育时期的耐冷性及其与籼粳性的关系[J]. 分子植物育种, 2017, 15(2): 763-773. DOI:10.13271/j.mpb.015.000763 YANG T F, ZHANG S H, WANG X F, DONG J F, HUANG Z H, ZHAO J L, LIU Q, MAO X X, ZHANG G Q, LIU B. Evaluation of cold tolerance at four growth stages and analysis of relationship between cold tolerance and indica-japonica differentiation in a diverse rice collection(Oryza sativa L.)[J]. Molecular Plant Breeding, 2017, 15(2): 763-773. DOI:10.13271/j.mpb.015.000763 |
| [42] |
张桂权. 基于SSSL文库的水稻设计育种平台[J]. 遗传, 2019, 41(8): 754-760. DOI:10.16288/j.yczz.19-105.j.yczz.19-105 ZHANG G Q. The platform of breeding by design based on the SSSL library in rice[J]. Hereditas, 2019, 41(8): 754-760. DOI:10.16288/j.yczz.19-105.j.yczz.19-105 |
| [43] |
刘冠明, 李文涛, 曾瑞珍, 张泽民, 张桂权. 水稻单片段代换系代换片段的QTL鉴定[J]. 遗传学报, 2004, 12: 1395-1400. IU G M, L I W T, ZENG R Z, ZHANG Z M, ZHANG G Q. Identification of QTLs on substituted segments in single segment substitution lines of rice[J]. Acta Genetica Sinica, 2004, 12: 1395-1400. |
| [44] |
杨梯丰. 基于SSSL的水稻耐冷QTL的定位和聚合效应[D]. 广州: 华南农业大学, 2016. DOI: 10.7666/d.D01036704. YANG T F. Identification and pyramiding of QTLs for cold tolerance by use of single segment substitution lines in rice(Oryza sativa L. )[D]. Guangzhou: South China Agricultural University, 2016. DOI: 10.7666/d.D01036704.d.D01036704. |
(责任编辑 崔建勋)
 2021, Vol. 48
2021, Vol. 48