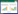文章信息
基金项目
- 广东省基础与应用基础研究基金(2021A1515012132,2020A1515011330);广东省基础与应用基础研究基金(粤桂联合基金)(2020A1515410005);贵州省自然科学基金(黔科合基础〔2018〕1184)
作者简介
- 徐晓美(1983—),女,博士,研究员,研究方向为茄果类疫病及分子育种,E-mail:xiaomeixu@gdaas.cn.
通讯作者
- 王恒明(1964—),男,硕士,研究员,研究方向为辣椒遗传育种,E-mail:1273450635@qq.com.
文章历史
- 收稿日期:2022-06-26
2. 茂名市茂蔬种业科技有限公司,广东 茂名 525000
2. Maoming Maoshu Seed Technology Co., Ltd, Maoming 525000, China
【研究意义】1年生辣椒(Capsicum annuum)是5个辣椒栽培种中最重要的栽培种,因其风味多样、营养丰富,深受消费者喜爱,同时既可鲜食又可加工,具有重要的产业价值。在广东地区,辣椒是南菜北运最重要的蔬菜,广东省年辣椒播种面积达10万hm2左右,居全省蔬菜播种面积的第2位。然而,因广东地区高温高湿,辣椒疫病病害严重,对辣椒生产造成重大损失,筛选抗疫病种质材料、培育抗疫病辣椒品种是一条经济、环保且有效的途径[1]。另外,种质材料的遗传多样性是作物育种的前提,是种质评价和利用的基础,同时也为基因资源的发掘提供必要信息。【前人研究进展】辣椒疫病由辣椒疫霉菌(Phytophthora capsici Leonian)引起,是一种发病周期短、传播速度快的土传病害[2]。目前生产上对该病害的防治主要依赖化学药剂,这不仅增加生产成本,同时也对环境造成污染。因此,选育抗病品种才是一条经济、环保且有效的途径,而实现该途径的第一步则是做好辣椒抗疫病种质资源的鉴定和筛选。何烈干等[3]利用游动孢子灌根法对208份辣椒种质材料进行疫病抗性鉴定,筛选出高抗材料18份、抗性材料23份;关天舒等[4]利用相同样方法在国内外共217份辣椒种质材料中鉴定并筛选出高抗材料84份、抗病材料57份,两者筛选结果差异较大。在种质材料遗传多样性研究中,形态学分析可以直观展示种质材料特点,也是研究种质多样性及开展作物育种工作的基础,但需要较长的种植周期去观察性状间的差异,既费时费力,影响因素也较多。而基于物种DNA序列的SSR(Simple Sequence Repeats)分子标记是一种技术成熟、应用广泛的分子标记技术[5],具有数量丰富、分布广、稳定性好、等位基因多、共显性遗传等优点[6],已全面应用于油菜[7]、水稻[8]、花生[9-10]、大豆[11]、大麦[12]等作物遗传多样性研究中。自2014年辣椒基因组测序工作完成以来,在辣椒中开发SSR分子标记变得更加便捷,SSR分子标记在辣椒种质资源遗传多样性研究中也陆续得到应用[13-15]。Christov等[16]利用21个SSR分子标记对176份1年生辣椒种质材料进行遗传多样性分析,筛选出44份材料并构建了一个迷你型核心种质库。Gu等[17]利用29个SSR标记对我国蔬菜中期基因库中的1 904份辣椒种质资源进行遗传多样性分析,最终选择248份覆盖75.6% SSR等位基因的材料作为核心种质。Zhong等[18]利用33个新开发的SSR标记对来源于25个国家的147个灌木状辣椒(C. frutescens)材料进行遗传多样性分析,并将其分为7个大类群,聚类结果与材料来源具有一致性。【本研究切入点】相比国外的辣椒核心种质,我国辣椒资源的种间材料不够丰富,但在种内存在一些特有的进化和分化情况。全方位了解和保存这些资源,可为辣椒种质资源进化驯化、特异基因挖掘和辣椒育种改良等研究奠定基础。目前,国内外对辣椒种质资源同时进行疫病抗性评价和分子标记遗传多样性分析的研究还未见报道。【拟解决的关键问题】通过辣椒全基因组筛选到20个SSR分子标记,对广东省农业科学院蔬菜研究所辣椒课题组现存的96份1年生辣椒种质材料进行SSR分子标记遗传多样性分析,同时对疫病抗性、熟性、果色、果型和果实风味5个重要性状进行调查,旨在加深对种质材料的了解并将核心种质材料加以保存,以应用于将来育种研究中。
1 材料与方法 1.1 供试辣椒材料及其重要性状调查96份待测辣椒种质材料均由广东省农业科学院蔬菜研究所辣椒课题组收集、创制的高世代纯合种质材料。抗疫病对照CM334和感疫病对照NMCA10399均受赠于美国墨西哥州立大学Paul W. Bosland教授[19]。96份材料均在广东省农业科学院钟落潭实验基地种植,进行常规化管理,并对其熟性、果色、果型和果实风味4个重要性状进行了初步调查和登记。熟性分为早熟、中熟和晚熟,其中始花节位10节以下为早熟、10~14节为中熟、15节及以上为晚熟;果色分为青熟果色和老熟果色,由肉眼观察获得;果型调查参照辣椒果实形态分类方法[20],先测量果实横径和纵径,再计算果型指数,进而判断果型;果实风味定性分为甜、微辣、中辣、辣和极辣,由3人同时生食品尝鉴定获得。
1.2 辣椒疫病抗性鉴评1.2.1 待测辣椒苗培养 所有辣椒材料均播种于50孔育苗盘中,每份材料播种25孔,每孔播种3~5粒种子,于植物培养室育苗。育苗基质为蛭石: 草炭: 土壤=1:2:1,高温灭菌30 min后备用,育苗条件为26(±2)℃的温度下光照14 h、黑暗10 h交替进行,湿度为70%,待幼苗长至6片真叶时用于接种。
1.2.2 供试菌株培养及孢子液制备 辣椒疫霉菌Byl4为本实验室分离获得[21],在固体V8汁培养基上培养。培养基配制方法如下:200 mL V8汁、3 g CaCO3和20 g琼脂,加水定容至1 000 mL,调节pH至6.3,高温灭菌30 min后倒平板备用。将保存好的辣椒疫霉菌Byl4接种于固体V8汁培养基上,在恒温箱中28 ℃黑暗条件下培养3~4 d后转移至室温(26±2 ℃)下光照、黑暗各12 h交替培养3~4 d。在培养皿中注入10 mL无菌水,4 ℃放置30 min后转移到室温(26±2 ℃)下放置30 min,诱导游动孢子释放。采用无菌毛笔刷洗、2层纱布过滤,获得游动孢子悬浮液。采用血球计数板计数,调节浓度为1×104个/mL,备用。
1.2.3 接种及抗病性鉴评 接种前12 h为待测辣椒植株充分浇水,使育苗基质水分达到饱和状态。采用注射器吸取孢子悬浮液均匀注射于辣椒植株附近约1 cm处,接种量为5 mL/ 孔。接种后于28 ℃黑暗条件下保湿培养24 h,而后在植物培养室参照育苗条件正常培育,至感病对照全部发病时(5 d左右)调查待测苗发病表型。植株发病程度分为7个等级,0、1等级为无任何病症,视为抗病植株;2、3、5、7、9等级为具有不同程度病症,视为发病植株[22],统计发病株数,计算发病率〔发病率(%)=发病株数/ 总种植株数×100〕。植株抗性等级划分参考Pecchioni等方法分为5个等级:无感病植株为免疫,0 < 发病率≤ 5% 为高抗,5% < 发病率≤ 20% 为抗病,20% < 发病率≤ 50% 为感病,发病率高于50% 为高感[23]。
1.3 分子标记检测1.3.1 DNA提取 采集6片真叶期辣椒幼苗嫩叶,采用改良CTAB法[24]提取基因组DNA,并用1% 琼脂糖凝胶电泳检测DNA纯度,采用NanoDrop2000测量DNA浓度,并统一调整DNA浓度至60 ng/μL,置于-20 ℃冰箱保存,备用。
1.3.2 分子标记筛选及分子标记检测 在茄果类基因组数据库网站(https://solgenomics.net/cview/map.pl?map_id=10)下载辣椒SSR分子标记引物序列,送生工生物工程股份有限公司合成。选取表现型差异大的10份辣椒DNA样品用于分子标记筛选,选出多态性好、条带清晰且扩增稳定的SSR标记用于后续遗传多样性分析。PCR扩增体系为10 μL:2×Taq PCR MasterMix 5 μL、灭菌超纯水2 μL、上游引物(10 μmol/L)0.5 μL、下游引物(10 μmol/L)0.5 μL、模板DNA 2 μL。PCR扩增程序为:95 ℃预变性4 min;94 ℃变性40 s,60~55 ℃(每个循环降低1 ℃)退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min,4 ℃保存。PCR产物经8.0% 非变性聚丙烯酰胺凝胶电泳分离,银染显色。
1.4 数据分析整理并记录所有条带:先统计电泳图等位基因,从上往下对等位基因数量进行统计;对于不同泳道(样品)下的条带,在相应等位基因上有条带的记为“1”、没有的记为“0”,由此获得0~1矩阵(每一行为一个泳道数据)。利用DataFormater软件[25]对数据进行格式转换后,再利用Popgene32软件[26]进行等位基因数(Na)、有效等位基因数(Ne)和Shannon多样性信息指数(I)等遗传多样性参数分析。另外,在统计软件DPS[27]的遗传聚类模块下,基于Jiccard方法将整理好的0~1矩阵数据进行材料间的遗传距离计算,得到遗传距离矩阵,利用MEGA6.0软件[28]以类平均法(UPGMA)进行聚类分析,绘制聚类图。
2 结果与分析 2.1 辣椒种质材料重要性状调查及疫病抗性鉴定对96份辣椒种质材料的熟性、果色、果型和果实风味4个性状的调查显示,在熟性方面,早熟、中熟和迟熟辣椒材料分别有19、49和28份;在果色方面,青熟果色为绿色、红熟果色为红色的辣椒材料占绝大多数,多达75份;在果型方面,有57份材料为圆锥形,牛角形、羊角形、灯笼形、螺丝形、线形和长柱形辣椒材料各有少量;在果实风味方面,中辣材料占比较多、有40份,极辣、辣、微辣和甜的材料分别有17、11、14和14份(表 1)。疫病抗性鉴定结果显示,96份材料中,免疫材料有1份,是国外引进的辣椒抗疫病资源;高抗材料有6份,其中迟熟指天椒占3份;抗病材料有24份,占材料总数的25%;感病和高感材料分别有53份和12份,分别占材料总数的55% 和13%,该调查数据与田间种植时疫病调查结果基本一致,只有编号10、28、45、54等4份材料不一致,在田间均表现为抗病(表 1)。
|
2.2 SSR分子标记筛选及分析
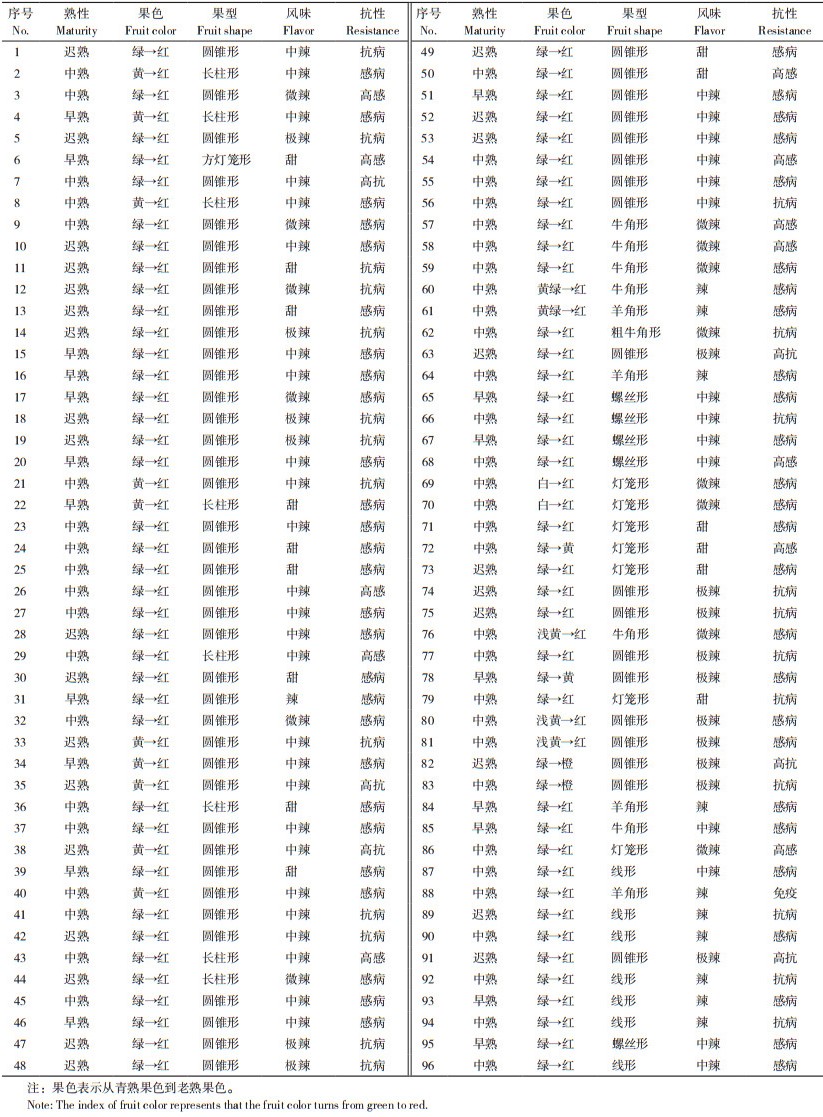
利用表现型差异较大的10份辣椒材料,从102对SSR引物中筛选出20对扩增稳定、条带清晰且多态性丰富的引物组合(图 1)。20个SSR标记较均匀地分布于辣椒12条染色体上,除第10、12染色体上没有分布外,其他染色体上均有分布(表 2)。

|
| 图 1 20个SSR分子标记扩增情况 Fig. 1 Amplification performance of 20 SSR molecular markers |
以筛选出的20对引物对96份辣椒种质资源进行遗传多样性分析,20对引物共检测出79个等位基因(Na)位点,每对引物检测到的等位基因数为2~9个、平均为3.95个,检测到的有效等位基因数(Ne)共48.0740个、平均为2.4037个;Shannon多样性信息指数(I)在0.2992 ~1.9893之间、平均值为0.9513,表明标记的遗传多态性较高;检测到的杂合度(Ho)在0~0.9792之间,平均为0.1640;期望杂合度(He)在0.1622~0.8484之间,平均为0.5287;Nei’s遗传多样性指数(H)在0.1614~0.8440之间,平均为0.5259,表明材料间遗传多样性高(表 3)。综合分析显示,筛选出的20个SSR分子标记多态性良好,能够有效分析遗传多样性。
2.3 辣椒种质材料的聚类分析
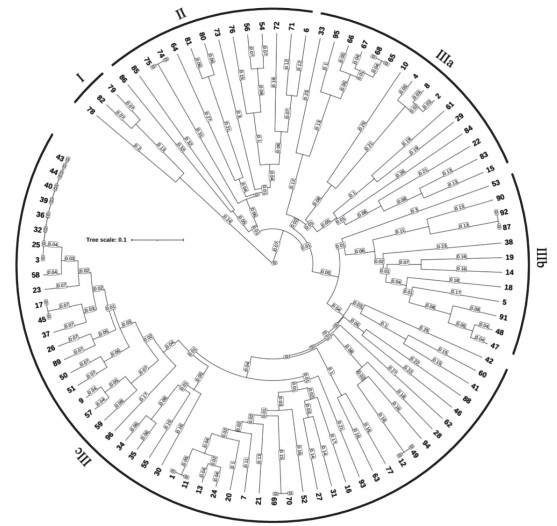
基于Jaccard方法进行遗传距离计算,利用UPGMA方法对96份辣椒种质材料进行聚类分析,结果表明在遗传距离0.37处可以将供试材料分为Ⅰ、Ⅱ、Ⅲ等3个大类群。第Ⅰ大类群所含个体数最少、仅3份,均是从美国引进的辣椒种质材料,其中编号78、79的材料分别是已育成辣椒新品种金田1号的父母本。第Ⅱ大类群包含14份材料,其中编号74、75两份材料极其相似,遗传距离仅为0.0062,两者应该是同一份材料的不同株系;在这14份材料中,各有4份材料是指天椒(编号74、75、80和81)和甜椒(编号6、71、72和73)类型,即这两类果型、果实风味等性状表现出极大差异的材料聚在一起。第Ⅲ大类群所含个体数最多、共有79份材料,在遗传距离0.32处又可将第Ⅲ大类群分为Ⅲ a、Ⅲ b和Ⅲ c等3个亚类群,3个亚类群分别包含有16、12和51份材料。在Ⅲ a亚类群中,编号65和68两份材料的遗传距离极小、仅为0.0054;Ⅲ b亚类群中,编号87和92两份材料的遗传距离也仅为0.0081;在Ⅲ c亚类群中,编号69和70,12和49,1和11,17和45,以及3、25、32、36、39、40、43和44这5组材料的遗传距离均接近于0(图 2),推测以上7组材料均来源于同一材料的不同株系。
|
| 图 2 96份辣椒种质材料的遗传多样性聚类分析 Fig. 2 Cluster analysis of genetic diversity of 96 pepper germplasm materials |
3 讨论
种质资源是遗传育种的基础,为应对当前全球气候变化带来的挑战以及人们对农产品数量需求的增加和品质要求的不断提高,收集丰富的作物种质资源并建立资源库是极为重要的一项工作。然而,数量庞大的作物种质资源在保护和管理实践中均面临着问题,因此,建立一个核心基因库,在保留作物种质基因多样性的同时尽可能去除重复或是遗传关系相近的种质资源是一条有效途径。前人或基于表型数据、或基于基因型数据或是将以上两者结合起来,构建了核心种质库[29-31]。Gu等[17]利用29对SSR标记对我国蔬菜中期基因库中的1 904份辣椒种质资源进行遗传多样性分析,最终选择248份覆盖75.6% SSR等位基因的材料作为核心种质。Christov等[13]基于21个SSR标记对176份一年生辣椒种质进行遗传多样性分析,并最终选出44份核心材料构建了一个迷你型核心基因库。本研究通过从辣椒基因组上的102个SSR分子标记中筛选出20个扩增稳定、条带清晰且多态性丰富的标记对96份辣椒资源进行分析,结果显示这20个筛选出来的分子标记遗传多态性较高,可以用于辣椒种质材料的遗传多样性分析。此外,分析结果还发现,材料74和75,65和68,87和92,69和70,12和49,1和11,17和45,以及3、25、32、36、39、40、43和44这8组材料的遗传距离均接近于0,表明它们之间亲缘关系极近,在种质保存时每组只保留其中一份即可。尽管如此,96份辣椒种质材料的平均Nei’s遗传多样性指数为0.5259,表明材料间遗传多样性高,具有应用于杂交育种的前景。
常见的辣椒疫病接种鉴定方法有离体叶片接种法、切茎接种法和游动孢子液灌根接种法,以上3种方法各有优缺点,但灌根接种法因更接近于辣椒疫病在田间的发生过程,因此更接近于辣椒材料的真实抗性水平[32-33],也最为常用。2011年开始实施的行业标准《辣椒抗疫病鉴定技术规程》(批准号:NY/T 2060.1-2011,下称“标准”)规定,接种前需用玻棒在距幼苗根茎约3 cm处扎一孔,孔深3 cm左右[34],且表型鉴定采用分级,分别为0~5级:0级为无任何症状;1级为幼苗根茎部稍有变黑,叶片不萎蔫或可恢复性萎蔫;2级为幼苗根茎部变黑达1~2 cm,叶片不可恢复性萎蔫,下部叶片偶有脱落;3级为幼苗根茎部变黑超过1~2 cm,叶片明显萎蔫或落叶明显;4级为幼苗根茎部变黑、皱缩,生长点外部叶脱落或整株萎蔫;5级为植株枯死。鉴定过程操作越繁琐,通常越容易带来误差,如《标准》规定的“在距幼苗根茎约3 cm处扎一孔”,以加快侵染速度的目的,完全可以通过增大疫霉菌游动孢子液浓度来达到。另外,由于表型分级带有“稍有”“偶有”等描述模糊的词语,同时一个级别有好几个病症共存,造成表型鉴定复杂化且主观性太强,影响表型鉴定的准确性。因此,本研究在对96份辣椒种质材料进行疫病鉴定时采用的是改良后的灌根接种法。一是加大接种液孢子总量,即由标准规定的3×103增加到5×104的量,该接种量也是通过本团队长期摸索得出并沿用至今[21, 35-37]。表型调查中植株表型分为两种,即分为无任何症状的正常植株和有不同程度病症的发病植株[22],将表型调查简化,如此尽可能减少实验误差,使疫病鉴定结果更接近于材料本身的真实水平。
4 结论通过辣椒全基因组扫描筛选到20个SSR标记,对96个辣椒种质材料进行了遗传多样性分析,20个SSR标记的Shannon信息指数平均值为0.9513,表明所选用的20个SSR分子标记遗传多态性较高,能够用于遗传多样性分析;Nei’s遗传多样性指数平均值为0.5259,表明材料间遗传多样性高,但聚类分析显示有8组材料间的遗传距离接近为0,即亲缘关系极近。因此,综合表明这96份辣椒种质材料来源丰富,但存在同质材料,在种质保存时可以适当舍弃部分材料。另外,改良后的辣椒疫病灌根鉴定方法鉴评结果与材料在田间表现基本一致,可以用于辣椒疫病抗性评价。
| [1] |
HAUSBECK M K, LAMOUR K H. Phytophthora capsici on vegetable crops: research progress and management challenges[J]. Plant Disease, 2004, 88(12): 1292-1303. DOI:10.1094/pdis.2004.88.12.1292 |
| [2] |
LEONIAN L H. Stem and fruit blight of peppers caused by Phytophthora capsici Leon.[J]. Phytopathology, 1922, 12(9): 401-408. |
| [3] |
何烈干, 周银生, 陈学军, 方荣, 马辉刚. 辣椒种质资源抗疫病性及发病速度分析[J]. 江西农业大学学报, 2017, 39(4): 691-698. DOI:10.13836/j.jjau.2017090 HE L G, ZHOU Y S, CHEN X J, FANG R, MA H G. An analysis on resistance of pepper germplasms to Phytophthora blight and disease rate[J]. Acta Agriculturae Universitatis Jiangxiensis, 2017, 39(4): 691-698. DOI:10.13836/j.jjau.2017090 |
| [4] |
关天舒, 刘长远, 王丽萍, 梁春浩, 王辉, 李柏宏. 辣椒抗疫病材料的筛选[J]. 江西农业学报, 2011, 23(11): 100-102. DOI:10.19386/j.cnki.jxnyxb.2011.11.030 GUAN T S, LIU C Y, WANG L P, LIANG C H, WANG H, LI B H. Screening of materials resistant to Phytophthora disease of pepper[J]. Acta Agriculturae Jiangxi, 2011, 23(11): 100-102. DOI:10.19386/j.cnki.jxnyxb.2011.11.030 |
| [5] |
ZIETKIEWICZ E, RAFALSKI A, LABUDA D. Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification[J]. Genomics, 1994, 20(2): 176-183. DOI:10.1006/geno.1994.1151 |
| [6] |
POWELL W, MORGANTE M, ANDRE C, HANAFEY M, VOGEL J, TINGEY S, RAFALSKI A. The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis[J]. Molecular Breeding, 1996, 2(3): 225-238. DOI:10.1007/bf00564200 |
| [7] |
HASAN M, SEYIS F, BADANI A G, PONS-KÜHNEMANN J, FRIEDT W, LÜHS W, SNOWDON R J. Analysis of genetic diversity in the Brassica napus L. gene pool using SSR markers[J]. Genetic Resources and Crop Evolution, 2006, 53(4): 793-802. DOI:10.1007/s10722-004-5541-2 |
| [8] |
SINGH N, CHOUDHURY D R, TIWARI G, SINGH A K, KUMAR S, SRINIVASAN K, TYAGI R K, SHARMA A D, SINGH N K, SINGH R. Genetic diversity trend in Indian rice varieties: an analysis using SSR markers[J]. BMC Genetics, 2016, 17(1): 1-13. DOI:10.1186/s12863-016-0437-7 |
| [9] |
TANG R H, GAO G Q, HE L Q, HAN Z Q, SHAN S H, ZHONG R C, ZHOU C Q, JIANG J, LI Y R, ZHUANG W J. Genetic diversity in cultivated groundnut based on SSR markers[J]. Journal of Genetics and Genomics, 2007, 34(5): 449-459. DOI:10.1016/s1673-8527(07)60049-6 |
| [10] |
王润风, 鲁清, 洪彦彬, 梁炫强, 陈小平, 刘国强, 洪成佳, 李少雄. 中国南方区试花生品种的遗传多样性分析[J]. 广东农业科学, 2021, 48(12): 44-53. DOI:10.16768/j.issn.1004-874X.2021.12.006 WANG R F, LU Q, HONG Y B, LIANG X Q, CHEN X P, LIU G Q, HONG C J, LI S X. Analysis on the genetic diversity of peanut varieties in regional tests of southern China[J]. Guangdong Agricultural Sciences, 2021, 48(12): 44-53. DOI:10.16768/j.issn.1004-874X.2021.12.006 |
| [11] |
KUMAR S P, SUSMITA C, SRIPATHY K V, AGARWAL D K, PAL G, SINGH A N, KUMAR S, RAI A K, SIMAL-GANDARA J. Molecular characterization and genetic diversity studies of Indian soybean 〔Glycine max (L.) Merr.〕cultivars using SSR markers[J]. Molecular Biology Reports, 2022, 49(3): 2129-2140. DOI:10.1007/s11033-021-07030-4 |
| [12] |
郭铭, 张金福, 司二静, 孙莉莎, 魏建敏, 刘海颖, 乔岩, 姚立蓉, 汪军成, 李葆春, 杨轲, 孟亚雄, 马小乐, 朱靖环, 尚勋武, 王化俊. 不同来源大麦对条纹病抗性鉴定及遗传多样性分析[J]. 植物遗传资源学报, 2022, 23(1): 72-82. DOI:10.13430/j.cnki.jpgr.20210427002 GUO M, ZHANG J F, SI E J, SUN L S, WEI J M, LIU H Y, QIAO Y, YAO L R, WANG J C, LI B C, YANG K, MENG Y X, MA X L, ZHU J H, SHANG X W, WANG H J. Resistance identification and genetic diversity analysis of barley genotypes from different sources to barley stripe disease[J]. Journal of Plant Genetic Resources, 2022, 23(1): 72-82. DOI:10.13430/j.cnki.jpgr.20210427002 |
| [13] |
傅鸿妃, 吕晓菡, 陈建瑛, 李国景. 辣椒种质表型性状与SSR分子标记的遗传多样性分析[J]. 核农学报, 2018, 32(7): 1309-1319. DOI:10.11869/j.issn.100-8551.2018.07.1309 FU H F, LYU X H, CHEN J Y, LI G J. Genetic diversity analysis based on phenotypic traits and SSR molecular markers in pepper germplasms[J]. Journal of Nuclear Agricultural Sciences, 2018, 32(7): 1309-1319. DOI:10.11869/j.issn.100-8551.2018.07.1309 |
| [14] |
贾豪, 魏小春, 姚秋菊, 原玉香, 王志勇, 姜俊, 杨双娟, 赵艳艳, 王彬, 张晓伟. 辣椒SSR标记种质资源遗传多样性的分析[J]. 分子植物育种, 2017, 15(1): 353-363. DOI:10.13271/j.mpb.015.000353 JIA H, WEI X C, YAO Q J, YUAN Y X, WANG Z Y, JIANG J, YANG S J, ZHAO Y Y, WANG B, ZHANG X W. Genetic diversity analysis of Capsicum genus germplasm resources using SSR markers[J]. Molecular Plant Breeding, 2017, 15(1): 353-363. DOI:10.13271/j.mpb.015.000353 |
| [15] |
李艳, 赵红星, 王勇, 姜俊, 孟祥锋, 魏小春, 李金玲. 169份辣椒种质资源的遗传多样性分析[J]. 河南农业科学, 2018, 47(2): 91-97. DOI:10.15933/j.cnki.1004-3268.2018.02.018 LI Y, ZHAO H X, WANG Y, JIANG J, MENG X F, WEI X C, LI J L. Genetic diversity analysis of 169 accessions of Capsicum[J]. Journal of Henan Agricultural Sciences, 2018, 47(2): 91-97. DOI:10.15933/j.cnki.1004-3268.2018.02.018 |
| [16] |
CHRISTOV N K, TSONEV S, TODOROVA V, TODOROVSKA E G. Genetic diversity and population structure analysis–a prerequisite for constructing a mini core collection of Balkan Capsicum annuum germplasm[J]. Biotechnology & Biotechnological Equipment, 2021, 35(1): 1010-1023. DOI:10.1080/13102818.2021.1946428 |
| [17] |
GU X Z, CAO Y C, ZHANG Z H, ZHANG B X, ZHAO H, ZHANG X M, WANG H P, LI X X, WANG L H. Genetic diversity and population structure analysis of Capsicum germplasm accessions[J]. Journal of Integrative Agriculture, 2019, 18(6): 1312-1320. DOI:10.1016/s2095-3119(18)62132-x |
| [18] |
ZHONG Y M, CHENG Y, RUAN M Y, YE Q J, WANG R Q, YAO Z P, ZHOU G Z, LIU J, YU J H, WAN H J. High-throughput SSR marker development and the analysis of genetic diversity in Capsicum frutescens[J]. Horticulturae, 2021, 7(7): 187. DOI:10.3390/horticulturae7070187 |
| [19] |
REEVES G, MONROY-BARBOSA A, BOSLAND P W. A novel Capsicum gene inhibits host-specific disease resistance to Phytophthora capsici[J]. Phytopathology, 2013, 103(5): 472-478. DOI:10.1094/phyto-09-12-0242-r |
| [20] |
田浩, 廖卫琴, 周安韦, 令狐昌英, 余常水. 辣椒果实形态分类[J]. 农技服务, 2014, 31(7): 62. IAN H, LIAO W Q, ZHOU A W, LING-HU C Y, YU C S. Morphological classification of pepper fruit[J]. Agricultural Technological Service, 2014, 31(7): 62. |
| [21] |
XU X M, CHAO J, CHENG X L, WANG R, SUN B J, WANG H M, LUO S B, XU X W, WU T Q, LI Y. Mapping of a novel race specific resistance gene to Phytophthora root rot of pepper (Capsicum annuum) using bulked segregant analysis combined with specific length amplified fragment sequencing strategy[J]. PLoS ONE, 2016, 11(3): e1514013. DOI:10.1371/journal.pone.0151401 |
| [22] |
MONROY-BARBOSA A, BOSLAND P W. Genetic analysis of Phytophthora root rot race-specific resistance in chile pepper[J]. Journal of the American Society for Horticultural Science, 2008, 133(6): 825-829. DOI:10.21273/jashs.133.6.825 |
| [23] |
PECCHIONI N, VALE G, TOUBIA-RAHME H, FACCIOLI P, TERZI V, DELOGU G, FISCHBECK G. Barley—Pyrenophora graminea interaction: QTL analysis and gene mapping[J]. Plant Breeding, 1999, 118(1): 29-35. DOI:10.1046/j.1439-0523.1999.118001029.x |
| [24] |
POREBSKI S, BAILEY L G, BAUM B R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J]. Plant Molecular Biology Report, 1997, 15(1): 8-15. DOI:10.1007/bf02772108 |
| [25] |
樊文强, 盖红梅, 孙鑫, 杨爱国, 张忠锋, 任民. SSR数据格式转换软件DataFormater[J]. 分子植物育种, 2016, 14(1): 265-270. DOI:10.13271/j.mpb.014.000265 FAN W Q, GAI H M, SUN X, YANG A G, ZHANG Z F, REN M. DataFormater, a software for SSR data formatting to develop population genetics analysis[J]. Molecular Plant Breeding, 2016, 14(1): 265-270. DOI:10.13271/j.mpb.014.000265 |
| [26] |
YEH F, YANG R C, BOYLE T. Popgene (v. 1.32), microsoft windowsbased freeware for population genetic analysis, 2000[Z]. 2014.
|
| [27] |
TANG Q Y, ZHANG C X. Data processing system (DPS) software with experimental design, statistical analysis and data mining developed for use in entomological research[J]. Insect Science, 2013, 20(2): 254-260. DOI:10.1111/j.1744-7917.2012.01519.x |
| [28] |
TAMURA K, STECHER G, PETERSON D, FILIPSKI A, KUMAR S. Mega6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [29] |
MONGKOLPORN O, HANYONG S, CHUNWONGSE J, WASEE S. Establishment of a core collection of chilli germplasm using microsatellite analysis[J]. Plant Genetic Resources, 2015, 13(2): 104-110. DOI:10.1017/s1479262114000768 |
| [30] |
ZEWDIE Y, TONG N, BOSLAND P. Establishing a core collection of Capsicum using a cluster analysis with enlightened selection of accessions[J]. Genetic Resources and Crop Evolution, 2004, 51(2): 147-151. DOI:10.1023/b:gres.0000020858.96226.38 |
| [31] |
LEE H Y, RO N Y, JEONG H J, KWON J K, JO J, HA Y, JUNG A, HAN J W, VENKATESH J, KANG B C. Genetic diversity and population structure analysis to construct a core collection from a large Capsicum germplasm[J]. BMC Genetics, 2016, 17(1): 142. DOI:10.1186/s12863-016-0452-8 |
| [32] |
赫卫, 张慧, 王莹. 辣椒对疫霉抗性的快速鉴定[J]. 植物保护, 2018, 44(2): 145-148. DOI:10.16688/j.zwbh.2017261 HAO W, ZHANG H, WANG Y. Rapid identification of resistance to Phytophora capsici in pepper[J]. Plant Protection, 2018, 44(2): 145-148. DOI:10.16688/j.zwbh.2017261 |
| [33] |
马辉刚, 何烈干, 陈学军, 方荣, 周坤华, 涂玉琴. 两种方法鉴定辣椒疫病抗性研究[J]. 江西农业大学学报, 2013, 35(3): 536-542. DOI:10.13836/j.jjau.2013096 MA H G, HE L G, CHEN X J, FANG R, ZHOU K H, TU Y Q. Comparative studies on the two methods for identification of Phytophthora capsici resistance[J]. Acta Agriculturae Universitatis Jiangxiensis, 2013, 35(3): 536-542. DOI:10.13836/j.jjau.2013096 |
| [34] |
易图永, 张宝玺, 谢丙炎, 高必达, 马新卫. 辣椒疫病三种接种方法的比较[J]. 中国蔬菜, 2003(2): 18-20. DOI:10.3969/j.issn.1000-6346.2003.02.006 YI T Y, ZHANG B X, XIE B Y, GAO B D, MA X W. Comparison of three inoculation methods for pepper blight[J]. China Vegetables, 2003(2): 18-20. DOI:10.3969/j.issn.1000-6346.2003.02.006 |
| [35] |
LI Y, YU T, WU T Q, WANG R, WANG H M, DU H, XU X W, XIE D S, XU X M. The dynamic transcriptome of pepper (Capsicum annuum) whole roots reveals an important role for the phenylpropanoid biosynthesis pathway in root resistance to Phytophthora capsici[J]. Gene, 2020, 728: 144288. DOI:10.1016/j.gene.2019.144288 |
| [36] |
龙誉铭, 卲建明, 赵竑博, 邹优永, 刘玉丹. 绿宝3号黄瓜霜霉病抗性鉴定及选育过程[J]. 广东农业科学, 2019, 46(7): 23-31. DOI:10.16768/j.issn.1004-874X.2019.07.004 LONG Y M, SHAO J M, ZHAO H B, ZOU Y Y, LIU Y D. Identification and breeding process of resistance to downy mildew of lvbao No.3 cucumber[J]. Guangdong Agricultural Sciences, 2019, 46(7): 23-31. DOI:10.16768/j.issn.1004-874X.2019.07.004 |
| [37] |
黄志君, 易辉玉, 李荣俏, 李林山. 广州地区桑椹菌核病病原鉴定[J]. 广东农业科学, 2017, 44(7): 91-95. DOI:10.16768/j.issn.1004-874X.2017.07.015 HUANG Z J, YI H Y, LI R Q, LI L S. Morphological and molecular identification of mulberry fruit sclerotiniose pathogen in Guangzhou[J]. Guangdong Agricultural Sciences, 2017, 44(7): 91-95. DOI:10.16768/j.issn.1004-874X.2017.07.015 |
(责任编辑 张辉玲)
 2022, Vol. 49
2022, Vol. 49